1. Moleculaire merkers in de ecologie
Moleculaire merkers zijn eigenlijk niet meer dan kleine specifieke stukjes DNA
die gerelateerd zijn aan een bepaalde eigenschap. Hiervoor coderen ze wel niet
altijd rechtstreeks voor die eigenschap.
Er zijn een groot aantal technieken beschikbaar, welke techniek gebruikt wordt
hangt af van 3 factoren :
- welk niveau van genetische polymorfie vereist is met betrekking tot de
vraagstelling (ouderschapstest kleine genetische verschillen kleine
polymorfie)
- Welke statistische testen reeds ontwikkeld zijn met betrekking tot de
beschouwde techniek. De statistische technieken lopen altijd achter op de
technieken.
- De tijd en het geld dat beschikbaar is (gelelektroferese is goedkoop)
Verder zijn er nog 2 locaties van merkers te onderscheiden. Merkers op neutral
loci. Deze merkers zijn niet rechtstreeks gerelateerd met het te onderzoeken
kenmerk. Mutatiesnelheden in neutrale loci zijn in principe constant.
En merkers op niet –neutrale loci, deze zijn per definitie betrokken in het te
onderzoeken kenmerk.
Types merkers
Allozymes :
Variante vormen van een enzyme die gecodeerd worden door verschillende
allelen van eenzelfde locus. De allozymes voeren dus eenzelfde functie uit
doorheen verschillende fylums.
Gebruikt om relaties aan te tonen tussen soorten omdat deze enzymes
aangetroffen worden in heel veel organismen (coderen voor algemene functies)
en ze doorheen de geschiedenis veranderingen zijn ondergaan waardoor ze te
scheiden zijn via gelelektroforese. Ze zijn onderhevig aan mendeljaanse
overerving.
Nucleaire merkers
Hierin heb je merkers op organel-DNA en nucleair DNA. Organel DNA
maternale overerving (Ne is de helft van biparentale overerving snellere
genetische drift.)
Nucleair DNA biparentale overerving.
VNTR : Variable number tandem repeats
Zoals de afkorting al doet vermoeden zijn er plaatsen in het genoom waar een
kleine nucleotidensequentie een variabel aantal keren voorkomt. Deze variatie
in lengte komt zeer frequent voor waardoor het mogelijk is individuen van een
soort te onderscheiden en ook ouderschapsanalyse te doen aangezien deze vntr
erfelijk is.
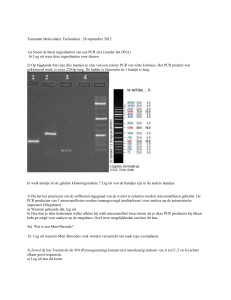
Om dus individuen te onderscheiden moet er dus gekeken kunnen worden naar
de lengte van de VNTR’s. Hiervoor dient een site met de vntr geamplificeerd te
worden (PCR nadeel is dat primers moeten ontwikkeld worden) en
vervolgens digestie ondergaan met een restrictie enzyme (die NIET knipt IN de
vntr). De VNTR’s kunnen dan door hun verschil in lengte gescheiden worden op
een gelelektroferese . De lengte kan bepaald worden door te vergelijken met de
ladder.
Binnen VNTR’s wordt er nog een onderscheid gemaakt in lengte tussen
minisattelieten en microsattelieten. Dit is een arbitrair onderscheid.
Minisatellieten zijn blokken van 10-60 bp terwijl microsatellieten blokjes zijn
van 2-13 bp.
RFLP: restriction fragment length polymorphism
Een verschil tussen 2 plaatsen op een genoom kan hier aangetoond worden
doordat er verschillend geknipt zal worden door een restrictie enzyme (nadat
eerst PCR heeft plaatsgevonden).
Vb.
ACACACAGACACAC. En ACACACACACACA
Als hier een restrictie enzyme opwerkt dat knipt na een G zullen er links 2 delen
DNA geknipt worden en rechts zal er niet geknipt worden.
Vervolgens kan met de digestieproducten een gelelektroferese gedaan worden
om de bandjes te scheiden volgens lengte. Het linkse deel zal 2 bandjes vertonen
en het rechtse deel maar 1 bandje.
ook hier kunnen ouderschapsanalyses mee uitgevoerd worden.
AFLP: amplified fragment length polymorphism
Een aangepaste methode van RFLP. Hierbij wordt EERST geknipt met een
restrictieenzyme en dan van de geknipte delen een PCR uitgevoerd.
Een nadeel bij RFLP en AFLP is het feit dat de heterozygositeit gaat onderschat
worden doordat sommige bandjes ‘geshared’ worden.
SNP’s: Single nucleotide polymorphism
Variatie in 1 nucleotide tussen leden van 1 soort. Kunnen gebruikt worden om
verschillen aan te tonen tussen verschillende groepen (bv een zieke en niet-zieke
groep.) SNP’s komen niet op alle plaatsen even vaak voor. In coils van proteïnes
bijvoorbeeld gebeurt dit vaker (extra info). Er zijn ook hotspots in het genoom
waar deze snelheid van mutatie hoger is.
Technieken
Elektroforese : eenvoudige techniek gebaseerd op de verschillende migratie in
een elektrisch veld door enerzijds verschil in grootte en anderzijds verschil in
lading.
Merkers die deze techniek gebruiken : Allozymes, RFLP, AFLP, VNTRs, SNPs.
Ook sanger sequencing gebruikt deze techniek (zie later)
ZEER goedkope techniek. Snel. Enzymes zijn echter niet zo goed bewaarbaar.
Fluorescentie hybridisatie:
Stel je hebt 2 individuen waarvan je niet weet hoe die gerelateerd zijn. Algemeen
is geweten dat zich in de populatie zich een SNP bevindt.
Nu is het mogelijk om via een micro array na te gaan wie welk genotype heeft.
Het werkt als volgt:
1) voor de specifieke sequentie wordt een complementaire probe (singlestranded) gevormd, kunstmatig gesynthetiseerd dus. Deze probe wordt
dan gebonden op een oppervlakte (roche 454 gebruikt bolletjes, zie next
generation sequencing).
2) Hierna kunnen we vanuit de samples ssDNA isoleren van het gekozen gen
met hierin de SNP. Dit ssDNA labelen we met een fluorescente groep.
3) Het gelabelde ssDNA laten we nu reageren met onze probe. Als de
sequentie volledig complementair is bindt deze sterker dan de strand met
de substitutie.
4) We wassen. Hierbij zal enkel het goede gebonden ssDNA blijven hangen
5) De graad van fluorescentie kan nu gemeten worden.
Sequencing technieken
Sanger sequencing:
1) PCR van het DNA met de promotors voor het te sequeneren gen.
2) Isolatie van single stranded DNA van het te onderzoeken gen.
3) PCR doen met dit single stranded DNA.
Hierbij worden gelabelde terminator
nucleotides willekeurig ingebouwd. Het zijn
terminator nucleotides omdat ze ddNTPs
(dideoxy) zijn ipv dNTPs (deoxy). De labels
zijn verschillend voor de 4 nucleotides
4) Je krijgt dus een PCR product met daarin
heel veel verschillende lengtes van
dubbelstrengig DNA.
5) Via een gelelektroferese het dubbelstrengig
DNA rangschikken van klein naar groot.
6) Signalen van de eindstandige nucleotides
worden afgelezen en geven de
nucleotidensequentie weer.
Next generation sequencing :
Dit zijn grootschalige methodes voor het genereren van DNA-sequenties.
Worden gebruikt voor het maken van volledig genoom. Er zijn 2 veelgebruikte
methodes : Roche 454 en Illumina sequencing.
Beiden gebruiken ssDNA dat dan sequentieel wordt blootgesteld aan een
nucleotide met detectielabel. Als dat nucleotide wordt ingebouwd dan geeft het
een signaal. Als het tweemaal wordt ingebouwd na elkaar dan verdubbelt het
signaal. Wordt het niet ingebouwd dan krijg je geen signaal.
Belangrijk punt! Next generation sequencing kan enkel uitgevoerd worden bij
organismen waarvan de genoomsequentie gekent is omdat next gen sequencing
korte reads (korte nucleotidensequenties) geeft die moeten in elkaar gepuzzeld
worden. Hiervoor dient de gekende sequentie als leidraad.
Roche 454:
1) library preparation: PCR uitvoeren van het hele genoom of het target gen
en dan alles shearen om ssDNA te bekomen.
2) 1 ssDNA streng aan een bead hangen.
3) Op elke bead apart een PCR uitvoeren waarbij de enkele gebonden ssDNA
streng gekloneerd wordt. We bekomen zo een bead met vele malen
hetzelfde ssDNA.
4) Al deze bolletje sin een picotiter plaat met welletjes laden.
5) Primer toevoegen en polymerase toevoegen
6) Nu dus telkens 1 stroom van nucleotides over de plaat laten gaan
ingebouwd geeft signaal, niet ingebouwd geeft geen signaal. Het signaal is
trouwens een lichtsignaal.
7) Assemblage van de sequenties tov een gekend genoom. De novo
assemblages (zonder gekend genoom) staat vermeld in de slides, maar
handboek moleculaire biologie zegt hier niets over.
Illumina HiSeq 2000:
Enige verschil met roche 454 is het vormen van de ssDNA library op de
surface. Hier wordt dit gedaan met een bridge PCR.
De dikkere rode en blauwe balkjes stellen primers voor. De dunne zijn de
sites waarop de primers binden.
Genomische analyse
QTL mapping: Quantitative trait locus
Een quantitative trait locus is een stuk DNA dat genen bevat die gerelateerd zijn
aan een kwantitatief kenmerk zoals grootte. QTL mapping is de studie van deze
genen om deze te lokaliseren in het genoom.
Als het genoom gekend is kan men al veel genen laten vallen die zeker geen
relatie hebben met het kwantitatief kenmerk. Als het genoom niet gekend is
kunnen de sequenties vergeleken worden met andere gekende sequenties voor
homologieën die dan duiden op eenzelfde functie. Als deze functie dan niet
gerelateerd is met het kwantitatief kenmerk kan het ook weggelaten worden.
Genome scans
Bij een genoomscan kunnen we nagaan wat de genetische differentiatie is tussen
soorten/individuen. Dit door na te gaan wat de allelfrequentie is bij
verschillende populaties. Deze allelfrequentie kan nagegaan worden door het
gebruik van merkers in het gekozen gen. Dit geeft dan volgende plot.
Zoals je ziet staat er een P waarde, wat dus betekent dat hierop statistiek
mogelijk is. Meerbepaald de statistieken van Wright. Nu wat betekent de
bekomen Fst? Fst staat voor de genetische differentiatie tussen de populaties.
Tussen 0 en 0,05 is er geringe differentiatie. 0,15 matige. 0,25 grote en meer als
0,25 is zeer grote differentiatie.
Ook belangrijk is Fst : 1/ (1+ 4.Ne.m)
Hieruit kan het aantal immigranten geschat worden per generatie.
E-DNA: environmental DNA
Hier is enkel DNA barcoding een interessant
concept. Hierbij wordt aan de hand van genetische
merkers in mitochondriaal DNA de soort
geidentificeerd.
Phylogenetic trees
Bij het tekenen van een fylogenetische boom bijvoorbeeld aan de hand van de
genetische merkers. Moet er rekening gehouden worden met parsimonie (zie
cursus diversiteit van planten) of spaarzaamheidsanalyse. Hierbij wordt er
vanuit gegaan dat een verandering in het gen/merker zo min mogelijk keren
heeft plaatsgevonden.
Voorbeeld uit de slides over de evolutie van het oog. Stel dat we aannemen dat
het octopusoog en het vertebraten homoloog zijn (en dus evolutionair verwant).
Dan tekenen we volgende boom.
Hierbij maken we maar liefst 5 verliezen van dit kenmerk.
Stel dat we er nu van uitgaan dat ze convergent zijn en dus elk apart
geevolueerd.
hier hebben we maar 2 veranderingen moeten toevoegen.
Bij het eerste model hadden we dus te maken met homoplasie: het verkeerd
veronderstellen van homologie of evolutionaire verwantschap