Tentamen Moleculaire Technieken , 28 september 2012
1a) Noem de basis ingredienten van een PCR mix (zonder het DNA)
b) Leg uit waar deze ingredienten voor dienen
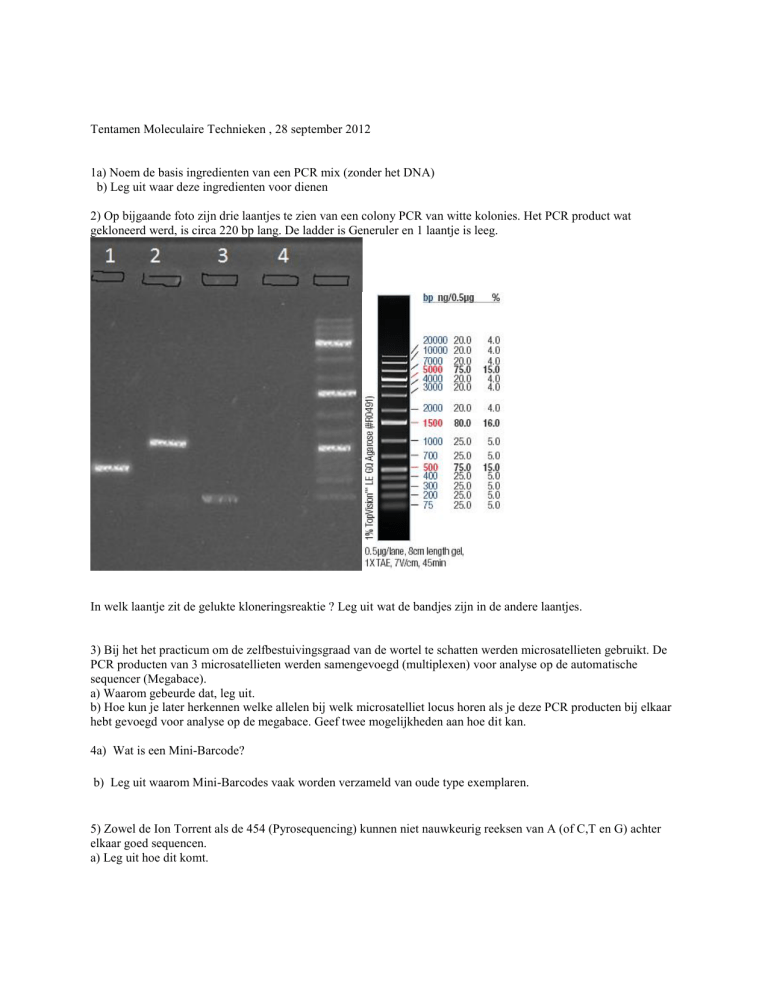
2) Op bijgaande foto zijn drie laantjes te zien van een colony PCR van witte kolonies. Het PCR product wat
gekloneerd werd, is circa 220 bp lang. De ladder is Generuler en 1 laantje is leeg.
In welk laantje zit de gelukte kloneringsreaktie ? Leg uit wat de bandjes zijn in de andere laantjes.
3) Bij het het practicum om de zelfbestuivingsgraad van de wortel te schatten werden microsatellieten gebruikt. De
PCR producten van 3 microsatellieten werden samengevoegd (multiplexen) voor analyse op de automatische
sequencer (Megabace).
a) Waarom gebeurde dat, leg uit.
b) Hoe kun je later herkennen welke allelen bij welk microsatelliet locus horen als je deze PCR producten bij elkaar
hebt gevoegd voor analyse op de megabace. Geef twee mogelijkheden aan hoe dit kan.
4a) Wat is een Mini-Barcode?
b) Leg uit waarom Mini-Barcodes vaak worden verzameld van oude type exemplaren.
5) Zowel de Ion Torrent als de 454 (Pyrosequencing) kunnen niet nauwkeurig reeksen van A (of C,T en G) achter
elkaar goed sequencen.
a) Leg uit hoe dit komt.
b) Geef het sequentie resultaat van het bovenstaande pyrogram van een 454 sequencer (A = rood, C =groen,
T=blaauw, G= zwart)
6) Op de Ion Torrent kunnen verschillende PCR producten van een zelfde individu van elkaar
worden onderscheiden door aan de primers/adaptors een staart (tag) te zetten van bv 4 baseparen.
Die 4 baseparen zijn dan de code waaraan je dat PCR producten van dat individu kunt
herkennen. Deze techniek maakt het mogelijk om PCR producten van verschillende individuen
op dezelfde chip te sequencen en later toch te kunnen herkennen.
Het mitochondriaal DNA van een bepaalde soort wordt met PCR vermeerderd, de stukken
worden gefragmenteerd tot circa 200-300 bp, van een tag voorzien en gesequenced op de 314
chip die circa 500.000 reads van 200 bp als oplevert. Aangezien men geinteresseerd is de
verschillen tussen soorten wil men meerdere soorten op de chip brengen. Het mitochondriaal
DNA is 16000 bp lang en van elke soort wil men een gemiddelde coverage bereiken van 20.
Hoeveel soorten kunnen erop 1 chip gezet worden? Geef de berekening.
7) Er wordt een ouderschaps analyse uitgevoerd met behulp van AFLPs. Van AFLPs is bekend
dat het een dominante merker is.
Hoe komt het dat zowel een heterozygoot als de ene homozygoot bij AFLP beide een bandje
produceren en de alternatieve homozygoot niet. Leg uit.
8)Bij Sanger sequencing word een PCR product vaak Forward en Reverse gesequenced. Leg uit
waarom er in de cyclesequence reaktie maar 1 primer wordt gebruikt.
9a) Wat is de beste methode (AFLP, SNP, Microsatellieten, Sanger sequencing) voor
ouderschapsanalyse van een soort waar geen enkele informatie over het genoom bekend is en je
beperkt bent in tijd (en geld). Leg uit.
b) Welke methode zou je gebruiken als het een modelsoort zoals rijst zou betreffen. Leg uit.
Antwoorden
1) Basis ingredienten van een PCR mix: Buffer (zorgt voor de juiste pH en zout
concentratiewaarbij de reaktie optimaal verloopt), dNTPs (bouwstenen voor het DNA), water
(zorgt voor de juiste verdunning), Taq polemerase (enzym dat de dNTPs aan de DNA streng
koppelt), Forward en reverse prime (startpunt voor de polymerase en stukje DNA dat bepaalt
welk stuk wordt geamplificeerd: bepalt de locatie).
MgCl2 is niet essentieel als extra component omdat de buffer dit al bevat. Dit is een component
die alleen wordt toegevoegd als de PCR niet goed loopt.
2) Laantje 2: succesvol gekloneerd DNA
Laantje 1: Vector die nagenoeg leeg is (bandje 220 bp kleiner). Vector bevat ws een kleine insertie omdat de kolonie
wit geweest moet zijn.
Laantje 3: Er is geen of zeer weinig template aanwezig in de reaktie en er is alleen een primer dimer zichtbaar.
3a) Om kosten te besparen. Door bv 6 microsatellieten in een laan op de automatische sequencer te doen worden de
kosten met 5/6 teruggebracht.
b) Door msats te leablen met een (fluorescent) label
Door msats te gebruiken waarvan de allellengte zodanig verschilt dat ze niet overlappen
4)a Answer 1: A small part (ca. 100 bp) of a regularly applied DNA barcoding marker (ca. 650
bp) providing only a third of the diagnostic characters of the full region but still useful for for
instance DNA barcoding of museum specimens
4b Answer 2: Old type specimens usually lack diagnostic characters such as colors due to
degradation over time. Two centuries ago taxonomists did not yet make very elaborate type
descriptions as they often collected the first white butterfly from a region. The description ‘white
butterfly’ was therefore sufficient. Over time many more white butterflies were collected leaving
later collectors with the puzzle which of these were already described and which were new to
science. By sequencing the type specimens (old so degraded DNA so only amplifiable with
Mini-Barcode markers) you can find out which of your putative new species are really new to
science by matching them to the sequences of type specimens. Only the ones with unique
sequences deserve formal description.
5a) In elke stap wordt 1 type base ingebouwd. Het signaal dat afgegeven wordt door het inbouwen van 1 base heeft
een kleine variatie (zie diagram). Deze variatie neemt toe als er meer van dezelfde basen achter elkaar moeten
worden ingebouwd . Het reultaat is dat op een gegeven moment dan niet meer gezien kan worden of er 10 of 11
basen zijn ingebouwd.
b )ACGGGGTAGCTGTTAGTTTGGAGTAGGCCGGAACCGCGGCGAGGAACG
6) 16000x 20 = 320000bp per soort. Beschikbare ruimte op de chip is 500.000*200=
100.000.000.
Aantal soorten op 1 chip= 100.000.000/320.000 = 10.000/32 = 312 soorten.
7) Variatie bij AFLPs ontstaat onder andere doordat er een mutatie aanwezig is in de
restrictiesite of de plek waar de selectieve basen aanhechten. Als er hier een mutatie is knipt of
het restrictieenzym niet meer of de primer kan niet aanhechten. Gevolg is dat producten van dat
allel (B) niet worden geamplificeerd en alleen het product van het andere allel (A) zichtbaar is.
Het fenotype is dat er 1 bandje zichtbaar is dat niet verschilt van het fenotype van een
homozygoot allel (A). Een homozygoot allel B produceert voor beide allelen geen product.
8) Bij Sanger sequencing word 1 primer gebruikt omdat er slechts 1 richting gesynthetiseerd mag
worden. Als er bv de forward en reverse tegelijk worden gebruikt ontstaat er een dubbel signaal.
Fragmenten met een lengte van 10 vanaf de forward primer geven dan bv een A aan terwijl de
fragmenten met lengte 10 van de reverse primer een C aangeven. Resultaat is dat er dan voor
elke fragment lengte een gemixed signaal is.
9a) Als er geen enkele informatie beschikbaar is, is AFLP de snelste methode. Voor de andere
methoden moeten eerst primers worden ontwikkeld hetgeen een tijdrovend karwei is.
b) Voor een model soort kun je het beste kiezen voor microsatellieten omdat deze zeer varaitie
vertonen tussen individuen door de hoge mutatiesnelheid in deze stukken DNA. SNPs vertonen
minder variatie en dus moeten er heel veel bekeken worden om toch een goed resultaat te
krijgen. AFLPs zijn dominante merkers en er zijn vaak problemen met de scoring van de
bandjes. Sanger sequencing levert te weinig variatie en is tijdrovend.












