UNIVERSITEIT GENT
ONZE-LIEVE-VROUWZIEKENHUIS
FACULTEIT FARMACEUTISCHE WETENSCHAPPEN
CAMPUS AALST – KLINISCH LABORATORIUM
VAKGROEP BIOANALYSE
DIENST MICROBIOLOGIE
Laboratorium voor Medische Biochemie
en Klinische Analyse
Labo Moleculaire Biologie
Academiejaar 2014-2015
ONTWIKKELING VAN EEN REAL-TIME PCR ASSAY ALS NIEUWE
DETECTIETEST IN DE DIAGNOSTIEK VAN TRICHOMONAS INFECTIES
Ian VANDENBUSSCHE
Eerste Master in de Geneesmiddelenontwikkeling
Promotor
Prof. dr. J. Van Bocxlaer
Commissarissen
dr. A. Vankeerberghen
Prof. dr. K. Boussery
UNIVERSITEIT GENT
ONZE-LIEVE-VROUWZIEKENHUIS
FACULTEIT FARMACEUTISCHE WETENSCHAPPEN
CAMPUS AALST – KLINISCH LABORATORIUM
VAKGROEP BIOANALYSE
DIENST MICROBIOLOGIE
Laboratorium voor Medische Biochemie
en Klinische Analyse
Labo Moleculaire Biologie
Academiejaar 2014-2015
ONTWIKKELING VAN EEN REAL-TIME PCR ASSAY ALS NIEUWE
DETECTIETEST IN DE DIAGNOSTIEK VAN TRICHOMONAS INFECTIES
Ian VANDENBUSSCHE
Eerste Master in de Geneesmiddelenontwikkeling
Promotor
Prof. dr. J. Van Bocxlaer
Commissarissen
dr. A. Vankeerberghen
Prof. dr. K. Boussery
SAMENVATTING
Het Onze-Lieve-Vrouwziekenhuis behoort tot de grootste niet-universitaire ziekenhuizen van
België. Het heeft, naast de campus in Aalst, ook vestigingen in Asse en Ninove. In samenwerking
met de Universiteit Gent wordt deze thesis uitgevoerd op het labo moleculaire biologie in het
klinisch laboratorium van het OLVZ, campus Aalst.
Het gebruik van PCR technieken in de diagnose van allerhande infecties is tegenwoordig niet
langer weg te denken. In de huidige screening op SOA’s wordt, door het relatief onschuldig
karakter van de parasiet, Trichomonas vaginalis naast het klassieke microscopisch onderzoek
vaak niet opgenomen in andere, gevoeligere detectieanalyses. Nochtans is een goede
diagnostiek van Trichomonas infecties essentieel ter vermijding van complicaties voor de
patiënt en een ongewenst grotere kost voor de gezondheidszorg. In dat opzicht wordt een realtime PCR (RT-PCR) methode ontwikkeld inzake de diagnose van T. vaginalis infecties.
Allereerst wordt de T. vaginalis RT-PCR geoptimaliseerd, zodat in tweede instantie een 6plex
RT-PCR methode ontwikkeld kan worden. Deze multiplex PCR screent naast T. vaginalis ook op
de aanwezigheid van de bacteriën Chlamydia trachomatis en Neisseria gonorrhoeae. Op die
manier kan een staal in één en dezelfde analyse geëvalueerd worden op het voorkomen van de
drie bovenbeschreven micro-organismen. In de optimalisatie van de 6plex RT-PCR wordt
geopteerd voor een PP mix die respectievelijk 300nM en 100nM Trichomonas primers en probe
bevat. Zodoende kan met deze condities overgegaan worden tot de validatie van de methode.
Er wordt uitgebreid gevalideerd op technisch en theoretisch vlak. De ontwikkelde test
doorstaat alle lineariteit- en specificiteitstesten, alsook de testen op houdbaarheid en precisie.
De sensitiviteit, specificiteit en accuraatheid bedragen 100%, gebaseerd op 67 geanalyseerde
stalen. Er wordt een kostenanalyse gemaakt en terugbetalingstarieven worden gerapporteerd.
Een methodevergelijking met de OSOM® Trichomonas Rapid sneltest moet een idee geven van
het verschil in performantie tussen beide methoden.
Na correcte interpretatie van alle validatiedata wordt er besloten om de ontwikkelde RT-PCR
methode op te nemen in de routine PCR analyses in het labo moleculaire biologie van het OLVZ.
Het schrijven van deze thesis zou zonder een aantal mensen niet mogelijk geweest zijn. De afgelopen
maanden waren enorm verrijkend, zowel op wetenschappelijk als op menselijk vlak.
In de eerste plaats wil ik de mensen van het labo moleculaire biologie bedanken, met in het bijzonder
mijn directe promotor dr. Anne Vankeerberghen. Bedankt Anne, voor de goede steun, uitleg en
begeleiding tijdens het uitvoeren van mijn thesis. Ook wil ik Karen, Freya, Leen, Inge, Elfi, Astrid, Magda
en Melissa bedanken voor de warme, aangename sfeer in het labo en de uitleg en begeleiding wanneer ik
die nodig had.
Naast mijn vrienden zou ik vooral mijn ouders willen bedanken voor de steun die ze mij tijdens de
afgelopen periode geboden hebben. De manier waarop jullie altijd klaar staan voor mij is onvergetelijk en
heeft mij gedurende de ganse thesisperiode gesterkt. Ook mijn oma en opa verdienen een welgemeend
dankjewel voor hun betrokkenheid en interesse.
Tenslotte wens ik klinisch bioloog Apr. An Boel en Prof. dr. Jan Van Bocxlaer te bedanken om mij de
mogelijkheid te bieden mijn thesis uit te voeren in het klinisch labo van het OLV Ziekenhuis Aalst.
Ian Vandenbussche
Aalst, mei 2015.
INHOUDSOPGAVE
1.
INLEIDING .............................................................................................................................................. 1
1.1
TRICHOMONAS VAGINALIS ........................................................................................................... 1
1.1.1
Introductie en prevalentie.................................................................................................... 1
1.1.2
Microbiologie ........................................................................................................................ 1
1.1.3
Complicaties en behandeling ............................................................................................... 2
1.1.4
Diagnose ................................................................................................................................ 3
1.2
1.1.4.1
Microscopie ....................................................................................................................... 3
1.1.4.2
Cultuur ............................................................................................................................... 3
1.1.4.3
Antigen sneltest ................................................................................................................. 3
1.1.4.4
Nucleic Acid Amplification Test (NAAT)............................................................................. 4
CHLAMYDIA TRACHOMATIS EN NEISSERIA GONORRHOEAE ........................................................ 5
1.2.1
Chlamydia trachomatis......................................................................................................... 5
1.2.2
Neisseria gonorrhoeae ......................................................................................................... 6
1.3
POLYMERASE CHAIN REACTION (PCR) .......................................................................................... 6
1.3.1
Extractie ................................................................................................................................ 6
1.3.2
PCR ........................................................................................................................................ 7
1.3.2.1
Algemeen........................................................................................................................... 7
1.3.2.2
Real-time PCR .................................................................................................................... 9
1.3.2.3
Multiplex PCR .................................................................................................................. 10
1.3.3
Rapportering ....................................................................................................................... 11
1.3.4
Targets voor PCR ................................................................................................................. 11
1.4
1.3.4.1
Trichomonas vaginalis ..................................................................................................... 11
1.3.4.2
Chlamydia trachomatis en Neisseria gonorrhoeae ......................................................... 12
SEQUENCING ............................................................................................................................... 13
2.
OBJECTIEF ............................................................................................................................................ 14
3.
MATERIAAL EN METHODEN ............................................................................................................... 15
3.1
STALEN......................................................................................................................................... 15
3.2
PRIMERS EN PROBES ................................................................................................................... 15
3.2.1
Selectie primers en probes ................................................................................................. 15
3.2.2
4.
Gebruikte primers, probes en reagentia ............................................................................ 16
3.3
EXTRACTIE ................................................................................................................................... 17
3.4
KLONEREN ................................................................................................................................... 18
3.5
REAL-TIME PCR ............................................................................................................................ 22
3.5.1
Algemeen ............................................................................................................................ 22
3.5.2
Optimalisatie ....................................................................................................................... 23
3.5.3
Validatie .............................................................................................................................. 25
3.6
GELELEKTROFORESE .................................................................................................................... 26
3.7
SEQUENCING ............................................................................................................................... 27
RESULTATEN........................................................................................................................................ 29
4.1
KLONEREN ................................................................................................................................... 29
4.2
OPTIMALISATIE ............................................................................................................................ 30
4.2.1
Optimalisatie van singleplex RT-PCR voor T. vaginalis ..................................................... 30
4.2.2
Optimalisatie van 6plex RT-PCR ......................................................................................... 32
4.3
4.2.2.1
Vergelijking met singleplex RT-PCR ................................................................................. 32
4.2.2.2
Vergelijking met 5plex RT-PCR ........................................................................................ 32
4.2.2.3
Vergelijking met externe referentie ................................................................................ 34
4.2.2.4
Checkerboard analyses.................................................................................................... 34
VALIDATIE .................................................................................................................................... 37
4.3.1
4.3.1.1
Analyse van de specificiteit ............................................................................................. 37
4.3.1.2
Bepalen van de analytische accuraatheid, sensitiviteit en specificiteit .......................... 38
4.3.1.3
Bepalen van de lineariteit................................................................................................ 39
4.3.1.4
Bepalen van de intrarun precisie..................................................................................... 42
4.3.1.5
Bepalen van de houdbaarheid en interrun precisie ........................................................ 42
4.3.2
4.4
Technische validatie ........................................................................................................... 37
Theoretische validatie: kostenanalyse en terugbetalingstarieven ................................... 43
METHODEVERGELIJKING ............................................................................................................. 43
5.
DISCUSSIE ............................................................................................................................................ 44
6.
CONCLUSIE .......................................................................................................................................... 47
7.
LITERATUURLIJST ................................................................................................................................ 48
VERKLARENDE WOORDENLIJST
5plex PCR:
PCR met 5voudige target (16S, CP, omp, porA en PhHV target)
6plex PCR:
PCR met 6voudige target (16S, CP, omp, porA, PhHV en TRIVAG
target)
A:
adenine base
acc.nr.:
accessie nummer
BHQ:
Black Hole Quencher
BLAST:
Basic Local Alignment Search Tool
bp:
baseparen
BSA:
Bovine Serum Albumine
C. trachomatis:
Chlamydia trachomatis
C:
cytosine base
CACG:
verdunningsreeks van C. trachomatis
CHTR:
Chlamydia trachomatis
CP:
cryptisch plasmide; C. trachomatis target
Ct waarde:
cycle treshold waarde
Cy5:
cyanine fluorescente stof
ddATP:
dideoxy adenosinetrifosfaat
ddCTP:
dideoxy cytidinetrifosfaat
ddGTP:
dideoxy guanosinetrifosfaat
ddNTP:
dideoxy nucleosidetrifosfaat
ddTTP:
dideoxy thymidinetrifosfaat
DMSO:
dimethylsulfoxide
DNA:
deoxyribonucleïnezuur
dsDNA:
dubbelstrengig DNA
DTCS:
dye terminator cycle sequencing
dXTP:
deoxy nucleosidetrifosfaat
E. coli:
Escherichia coli
Eff%:
efficiëntie
EtOH:
ethanol
EQC:
External Quality Control
FP:
forward primer
FS DNA:
fish sperm DNA
G:
guanine base
GAGG:
verdunningsreeks van N. gonorrhoeae
gem:
gemiddelde
HSV:
Herpes simplex virus
IC:
interne controle
LAF:
Laminar Air Flow
LOD:
Limit Of Detection
mRNA:
messenger RNA
N. gonorrhoeae:
Neisseria gonorrhoeae
NAAT:
Nucleic Acid Amplification Test
NCBI:
National Center for Biotechnology Information
O.D:
optische densiteit
omp:
outer membrane proteïne; N. gonorrhoeae target
PCR:
Polymerase Chain Reaction
PhHV:
Phocine Herpes virus
PID:
Pelvic Inflammatory Disease
porA:
porine pseudogen; N. gonorrhoeae target
PP mix:
primer-probe mix
R2:
correlatiecoëfficiënt
RIZIV:
Rijksinstituut voor ziekte- en invaliditeitsverzekering
RMO:
rechtstreeks microscopisch onderzoek
RNA:
ribonucleïnezuur
ROX:
referentie fluorescente stof voor achtergrondsignaal
RP:
reverse primer
rpm:
rotations per minute
rRNA:
ribosomaal RNA
RT-PCR:
real-time PCR
S:
Svedberg: sedimentatie eenheid
SLS:
sample loading solution
SOA:
Seksueel Overdraagbare Aandoening
SOC:
Super Optimal broth with Catabolic repression
ssDNA:
enkelstrengig DNA
stdev:
standaarddeviatie
swCT:
Zweedse variant van C. trachomatis
T. vaginalis:
Trichomonas vaginalis
T:
thymine base
TATG:
verdunningsreeks van T. vaginalis
TAE buffer:
Tris base, Acetic acid and EDTA buffer
TAT:
Turn Around Time
TE:
Tris base, EDTA buffer
TRIVAG:
Trichomonas vaginalis
TSA:
Tryptone Soy Agar
TSB:
Tryptone Soy Broth
TV :
Trichomonas vaginalis staal (gevolgd door een nummer)
V:
Volt
VZV:
Varicella zoster virus
W:
Watt
WHO:
World Health Organization
WIV:
Wetenschappelijk Instituut Volksgezondheid
Δ Ct:
delta Ct: verschil tussen Ct waarde van de 6plex en 5plex PCR
1. INLEIDING
1.1 TRICHOMONAS VAGINALIS
1.1.1 Introductie en prevalentie
Trichomoniasis, veroorzaakt door het protozoön Trichomonas vaginalis, wordt
wereldwijd aanzien als de meest frequente niet virale seksueel overdraagbare
aandoening (SOA). Bij seksueel actieve vrouwen vormt het één van de drie
hoofdoorzaken van vaginale klachten, samen met bacteriële vaginose en candida
vulvovaginitis. Infectie met T. vaginalis gaat typisch gepaard met een geelgroene
vaginale afscheiding en vaginale irritatie. Bij mannen manifesteert de infectie zich vooral
als een urethrale irritatie en een branderig gevoel na plassen of ejaculatie. Afscheiding is
hier beperkter. Het klachtenpatroon kan echter enorm variëren; vaak zijn patiënten zelfs
asymptomatisch. Hierbij dient opgemerkt te worden dat bij mannen de infectie
frequenter asymptomatisch verloopt dan bij vrouwen. Wanneer de infectie chronisch
verloopt zijn symptomen, typisch jeuk en dyspareunie, meestal milder van karakter. i,ii,iii
Prevalentie- en incidentiestudies van het WHO rapporteren op globale schaal
jaarlijks rond de 180 miljoen infecties, met een prevalentie die bij vrouwen ongeveer
tien maal hoger ligt dan bij mannen. De incidentie in beide geslachten is nagenoeg gelijk.
Deze gegevens moeten echter met de nodige voorzichtigheid bestudeerd worden. Het
zijn slechts schattingen aangezien Trichomonas infecties wegens hun relatief onschuldig
karakter niet noodzakelijk gerapporteerd dienen te worden. iv
1.1.2 Microbiologie
T. vaginalis is een facultatief anaerobe parasiet met een zeer typerende
peervormige structuur die zich kan voortbewegen door de aanwezigheid van flagellen.
Vier van deze flagellen bevinden zich anterior van de parasiet. De vijfde flagel wordt
posterior teruggevonden en wordt ook wel eens de vrije flagel genoemd.
1
Verder bezit de parasiet een prominent aanwezige nucleus en de axostyle. Deze loopt
doorheen de parasiet en biedt stevigheid aan het micro-organisme.ii
De overdracht van het protozoön gebeurt hoofdzakelijk seksueel. T. vaginalis
gedijt goed in de vrouwelijke urogenitale tractus en mannelijke urethra en prostaat, daar
waar de parasiet buiten het lichaam niet zo goed overleeft. De mens is de enige gekende
natuurlijke gastheer. Replicatie gebeurt enkel door binaire fissie. Bij lagere pH, wanneer
het micro-organisme niet meer in de trofozoïde vorm maar in de cysteuze vorm
voorkomt, blijkt T. vaginalis geen klachten te geven, maar kan het wel overleven.v
1.1.3 Complicaties en behandeling
Vroeger werd T. vaginalis beschouwd als een onschuldige commensaal van de
vagina. Tegenwoordig is deze veronderstelling echter al lang voorbijgestreefd. Zo kan
een infectie met de parasiet tot tal van complicaties leiden. Onbehandeld resulteert de
infectie vaak in een cystitis of urethritis. Eveneens rapporteerden studies een associatie
tussen T. vaginalis infectie en het verwerven van infertiliteit, cervicale neoplasie of zelfs
een verhoogd risico op een HIV infectie.vi,vii,viii Er is een verhoogde kans op vroegtijdige
geboorte bij zwangerschap en tevens kan de baby tijdens de geboorte zelf geïnfecteerd
worden door contact met de vaginale mucosa. ix Complicaties bij mannen zijn minder
frequent, al zijn er al gevallen gerapporteerd geweest van prostatitis, balanoposthitis,
epididymitis, infertiliteit en zelfs prostaatkanker.v
5-nitroimidazol geneesmiddelen zoals metronidazol zijn voorlopig de enige klasse
geneesmiddelen die een curatieve therapie bieden. Voor zowel symptomatische
personen als asymptomatische dragers wordt het best een therapie opgestart;
gewoonlijk een enkelvoudige dosis van 2g metronidazol of tinidazol. Orale toediening
krijgt de voorkeur op vaginale applicatie aangezien de therapeutische spiegels ter hoogte
van de urethra veel hoger zijn bij systemische toediening. De urethra kan immers dienen
als een soort van reservoir voor het pathogeen waardoor recidieven mogelijk zijn.x
2
1.1.4 Diagnose
1.1.4.1 Microscopie
Rechtstreeks microscopisch onderzoek (RMO) wordt aanzien als één van de oudste
technieken in de diagnostiek van trichomoniasis. Desondanks de zeer lage gevoeligheid
van de techniek, wordt deze toch nog extensief gebruikt in laboratoria. Bovendien
moeten afgenomen stalen zo snel mogelijk geanalyseerd worden opdat nog levende
kiemen waargenomen zouden kunnen worden. Overige parameters die kunnen inspelen
op de performantie van de techniek zijn de ervaring van de laborant, de parasitaire
lading en de bewaarcondities van het staal. Net omwille van bovenbeschreven lage
gevoeligheid wordt meer en meer gezocht naar nieuwere technieken met een hogere
graad van sensitiviteit.xi
1.1.4.2 Cultuur
Cultuur is een alternatief in de diagnostiek van T. vaginalis infectie bij patiënten
waarbij microscopisch onderzoek onmogelijk blijkt te zijn of wanneer er geen motiele
parasieten te zien zijn bij microscopie terwijl de patiënt wel met een verhoogde vaginale
pH en een verhoogd leukocytenaantal zit. Een groot nadeel aan diagnose met cultuur is
de lange turnaround time (TAT) van gemiddeld 48 uur. Tegenwoordig zijn zowel cultuur
als RMO op gebied van gevoeligheid voorbijgestreefd door de nucleïnezuur
amplificerende testen (NAATs).xii
1.1.4.3 Antigen sneltest
Omwille van de lage gevoeligheid van RMO zijn er een tijdje geleden nieuwe, op
immunochromatografie gebaseerde commerciële kits op de markt verschenen. De
meest frequent in gebruik genomen kit is de OSOM® Trichomonas Rapid Test (Sekisui
Diagnostics, Tokio, Japan) en deze wordt ook in deze thesis geëvalueerd.
3
De test is relatief goedkoop, makkelijk te gebruiken, vereist geen levende organismen en
kan als point of care test gebruikt worden.xiii,xiv
Het immunochromatografisch principe wordt verklaard aan de hand van
onderstaand voorbeeld met de OSOM® Trichomonas Rapid Test. Een positief staal bevat
de parasiet en dus ook parasietantigenen in oplossing. Op de absorberende kant van de
teststrook is een set gelabelde antilichamen aanwezig, gericht tegen die specifieke
antigenen. Wanneer de strook in contact wordt gebracht met de oplossing zal door
migratie het gevormde antigen-antilichaam complex ter hoogte van het afleesraam een
nieuwe set gefixeerde antilichamen ontmoeten. Deze tweede set antilichamen binden
eveneens met de parasietantigenen. Aangezien de met een kleurstof gelabelde
antilichamen nog steeds gebonden zijn op de parasietantigenen, zal ter hoogte van de
tweede bindingplaats een gekleurd lijntje ontstaan. Even verderop staat als controle nog
een derde set antilichamen, specifiek gericht tegen de gelabelde antilichamen. Dit
antigen-antilichaam complex moet
bijgevolg altijd gevormd worden,
zowel bij een positief als negatief
staal, en controleert of de migratie
correct verlopen is. (zie figuur 1.1).xv
Figuur 1.1: OSOM® Trichomonas Rapid
Test (Sekisui Diagnostics, Tokio, Japan)
1.1.4.4 Nucleic Acid Amplification Test (NAAT)
NAAT analyses zijn tegenwoordig de nieuwe gouden standaard voor wat diagnose
van trichomoniasis betreft. Ze zijn gebaseerd op het principe van PCR, waarbij genetisch
materiaal geamplificeerd wordt. Aangezien er bij PCR veel meer detectiemateriaal
geproduceerd wordt, zal bijgevolg ook de gevoeligheid veel groter zijn dan bij de
hierboven besproken technieken.xvi Een ander voordeel betreft de viabiliteit van
Trichomonas.
Levende
parasieten
zijn
hier
niet
noodzakelijk,
4
waardoor onmiddellijke analyse na afname niet nodig is. Hieruit volgt dan onmiddellijk
ook een nadeel van de NAAT techniek; het is moeilijk om te bepalen in welk stadium van
infectie de patiënt zich bevindt aangezien er geen onderscheid gemaakt wordt tussen
een levend pathogeen of een door het immuunsysteem reeds afgedood pathogeen.
1.2 CHLAMYDIA TRACHOMATIS EN NEISSERIA GONORRHOEAE
1.2.1 Chlamydia trachomatis
De meest voorkomende bacteriële oorzaak van seksueel overdraagbare genitale
aandoeningen wordt toegeschreven aan infectie met Chlamydia trachomatis. De
bacterie is strikt intracellulair en aeroob. Het merendeel van geïnfecteerde personen,
zowel mannen als vrouwen, blijkt een asymptomatisch verloop te kennen waardoor de
infectie vaak niet opgemerkt wordt en de bacterie aanwezig blijft in de genitale streek.
Nadelig is dat de bacterie op die manier onbewust overgedragen wordt. Bij
symptomatische vrouwen manifesteert de infectie zich meestal als cervicitis en
urethritis. De symptomatologie van deze laatst genoemde betreft vaak pyurie of dysurie.
De cervix blijkt de meest frequent voorkomende infectieplaats te zijn en infectie kan
onbehandeld leiden tot Pelvic Inflammatory Disease (PID) en in tweede instantie tot
infertiliteit. Bij mannen gaat infectie met C. trachomatis voornamelijk gepaard met
urethritis, epididymitis of prostatitis. Naast de genitale infectie kan C. trachomatis ook
verantwoordelijk worden geacht voor trachoma of granulaire conjunctivitis, een oculaire
infectie die bij de geboorte opgelopen kan worden en vaak in de derde wereldlanden tot
uiting komt. xvii
Diagnose gebeurt bij voorkeur met een NAAT, alhoewel ook sneltesten op de
markt zijn die snel resultaat geven en financieel makkelijker haalbaar zijn. Oudere
technieken gebaseerd op cultuur worden tegenwoordig minder uitgevoerd. Bovendien
hangen ze vaak af van de expertise van de uitvoerder van de test. Elke persoon die
seksueel actief is en symptomen vertoont die geassocieerd zouden kunnen zijn met
chlamydia, dient een screening te ondergaan, alsook de betrokken partner(s). xvii
5
1.2.2 Neisseria gonorrhoeae
Net zoals chlamydia is gonorroe een seksueel overdraagbare aandoening die in
dit geval veroorzaakt wordt door de aerobe bacterie Neisseria gonorrhoeae. Opnieuw is
een substantieel deel van de geïnfecteerde personen asymptomatisch. N. gonorrhoeae
ligt aan de basis van een vaak etterende urethritis bij mannen en cervicitis bij vrouwen.
Ook hier dient uitgekeken te worden voor ontaarding van de onbehandelde cervicitis,
wat kan leiden tot PID, chronische pijn in de onderbuik, ectopische zwangerschap of
eventueel zelfs infertiliteit. Uitzonderlijk kunnen invasieve infecties voorkomen,
resulterend in een meningitis of endocarditis. In de routine wordt meer en meer gebruik
gemaakt van een multiplex PCR (zie 1.3.2.3) waarbij zowel N. gonorrhoeae als
C. trachomatis binnen één en dezelfde PCR assay gedetecteerd kunnen worden. xviii
1.3 POLYMERASE CHAIN REACTION (PCR)
1.3.1 Extractie
Vooraleer een staal onderworpen kan worden aan PCR, dient het een extractie te
ondergaan. Een extractieprocedure zorgt ervoor dat het onbereikbare intracellulaire
DNA en RNA vrijkomt en bovendien afgezonderd wordt. Zodoende kan veel specifieker
gewerkt worden en is de hoeveelheid aan storende substanties drastisch verminderd.
Extractie wordt uitgevoerd op verschillende soorten stalen: urinestalen, vaginale swabs,
respiratoire stalen, bloedstalen… De cellen die de gewenste nucleïnezuren bevatten
worden eerst gelyseerd met een lysisbuffer. Naast de lysis zelf zorgt de aanwezigheid
van de buffer ook voor inhibitie van DNase en RNase enzymen. In de praktijk wordt
onder andere gebruik gemaakt van een silicakolom die selectief DNA bindt of van
magnetic beads die eveneens DNA en RNA adsorberen. Uiteindelijk kunnen de
nucleïnezuren in de daaropvolgende elutiestap gecollecteerd worden. Het opgezuiverde
genetisch materiaal kan vervolgens geamplificeerd worden met behulp van een NAAT. xix
6
1.3.2 PCR
1.3.2.1 Algemeen
PCR is een techniek waarbij op korte tijd nucleïnezuren geamplificeerd kunnen
worden. Door specifieke stukken van de DNA template te targetten, kan op
gecontroleerde wijze vanuit heel weinig DNA heel veel DNA gemaakt worden. Zo kan het
staal op eventuele aanwezigheid van het pathogeen gescreend worden. De PCR werd
voor het eerst ontwikkeld in 1983 door K. Mullis, en sindsdien is het een onmisbare
techniek geworden op vlak van detectie van genetisch materiaal. xx DNA bestaat uit twee
complementaire strengen die door base complementariteit aan elkaar gebonden zijn.
Een adenine base bindt via twee waterstofbindingen met een thymine base, terwijl een
cytosine en guanine base met drie waterstofbindingen verbonden zijn. Het principe van
de PCR techniek bestaat erin het gewenste DNA te denatureren tot twee afzonderlijke
strengen, zodat beiden dienst kunnen doen als template voor de vorming van een
nieuwe complementaire streng, gevormd volgens bovenstaande complementariteit (zie
figuur 1.2)
xxi,xxii
. Naast de aanwezigheid van het template DNA dienen er ook twee
primers, Taq polymerase enzym, de vier deoxynucleotiden, MgCl2 en buffer aanwezig te
zijn in de correcte hoeveelheden om de reactie optimaal te laten doorgaan. De PCR
bestaat grofweg uit 3 stappen: denaturatie, annealing en elongatie. Iedere stap heeft zijn
eigen karakteristieken en is essentieel voor de amplificatie.
In een eerste stap wordt van dubbelstrengig DNA (dsDNA) enkelstrengig DNA
(ssDNA) gemaakt door de temperatuur zodanig op te drijven dat de strengen van elkaar
loskomen. Op moleculair vlak wordt deze overgang naar enkelstrengig DNA verklaard
door de verbreking van de waterstofbindingen tussen de basen. Bijgevolg is er niets wat
de twee strengen nog kan samenhouden waardoor ze ‘uiteenvallen’. De temperatuur
voor denaturatie ligt meestal rond 95°C.
Vervolgens zakt de temperatuur tot net onder de smelttemperatuur van de
toegevoegde primers, vaak rond de 50°C tot 60°C, waardoor aan deze primers de kans
7
gegeven wordt om zich te binden met een regio op de complementaire DNA streng.
Deze binding moet specifiek zijn en de primers moeten vooral aan 3’ zijde zeer goed
aansluiten met de streng om een correcte elongatie te verkrijgen. Ze mogen ook niet te
sterk binden (i.e. te hoog GC gehalte) zodat de hierboven beschreven denaturatiestap
nog door kan gaan. Bovendien mag de primer niet met zichzelf of met een andere primer
binden. Dergelijke lus-of dimeervorming is nefast voor de efficiëntie van amplificatie.
In de elongatiefase wordt de temperatuur terug opgetrokken tot een temperatuur
waarbij het Taq polymerase een optimale enzymactiviteit bezit. De oude streng dient nu
als template voor het enzym zodat verlenging mogelijk wordt. De beschikbare
deoxynucleotides die zich in de oplossing bevinden worden nu stuk voor stuk ingebouwd
in de zich nieuw vormende streng. Het eindproduct betreft een dubbelstrengig stuk DNA
dat terug gedenatureerd wordt wanneer de eerste fase aanvat.
Het grote voordeel van de PCR reactie is de exponentiële amplificatie van enkel de
doelsequentie, gelegen tussen de twee gekozen primers. De replicatie van de nieuwe
streng start ter hoogte van de plaats waar de primer zich aan de complementaire streng
bindt en loopt verder (5’-3’ richting) tot wanneer geen complementaire streng meer
voorhanden is. Na doorlopen van 20 cycli en meer zal de aan PCR onderworpen
oplossing miljoenen amplicons bevatten. (zie figuur 1.2).
Figuur 1.2: Cycli van de PCR reactie en de basencomplementariteit
Vervolgens kan de hoeveelheid PCR product in de oplossing bepaald worden, met
gelelektroforese als een van de meest courante technieken. Tegenwoordig wordt echter
8
meer en meer geopteerd voor het principe van real-time PCR (RT-PCR). Hierbij wordt het
geamplificeerd materiaal continu gemeten en weergegeven in een amplificatieplot.
1.3.2.2 Real-time PCR
In vergelijking met de conventionele PCR kan de amplificatie bij RT-PCR onmiddellijk
gevolgd worden. Een cruciaal verschil ligt in de aanwezigheid van een TaqMan probe. Dit
oligonucleotide zal binden in het gebied tussen de twee primers en zorgt voor een
fluorescent signaal dat gedetecteerd kan worden door het RT-PCR toestel. Aan het 5’
uiteinde bevat deze probe een reporter R (zie figuur 1.3)xxiii. Na excitatie kan de reporter
bij terugval naar de grondtoestand zelf licht uitstralen van een welbepaalde golflengte.
Aan het 3’ uiteinde zit echter een quencher Q bevestigd. Deze zal het uitgestraalde licht
van de fluorofoor aan 5’ zijde
quenchen. In het RT-PCR toestel
zit een excitatiebron alsook een
detector om het uitgestraalde
licht te kunnen detecteren. De
quencher aan 5’ zijde van de
probe zorgt, naast het quenchen
op zich, er ook voor dat het Taq
enzyme niet ter hoogte van de
probe
begint
ketenverlenging.
aan
de Figuur 1.3: (a) TaqMan probe in oplossing, (b) TaqMan probe
gehybridiseerd aan complementaire streng, (c) exonuclease
activiteit van polymerase enzym breekt de probe af.
Tijdens de annealingstap van de PCR reactie zal de probe zich vasthechten aan de
DNA template. De reporter wordt geëxciteerd door het licht afkomstig van een
halogeenlamp, maar zit te dicht bij de quencher waardoor deze de emissie teniet doet.
Wanneer het polymerase enzym de nieuwe streng vormt, zal de TaqMan probe op een
gegeven moment zijn pad versperren. Dankzij de 5’-3’ exonuclease activiteit kan het
enzym de probe nucleotide per nucleotide afbreken en tegelijk toch de nieuwe streng
vormen. Dit proces creëert bijgevolg meer ruimte tussen reporter en quencher,
9
zodat quenching van de emissie niet langer mogelijk is. De sterkte van fluorescentie is
dan ook evenredig met de toename in PCR product: Hoe meer product er gevormd
wordt, hoe meer probe er afgebroken zal worden, dus hoe groter het fluorescent signaal
zal zijn. Het uitgestraalde licht gaat via lenzen en een filter naar de detector. Alle data
worden vervolgens omgezet tot een amplificatieplot.
1.3.2.3 Multiplex PCR
Binnen eenzelfde PCR run kan gebruik gemaakt worden van meerdere probes en
primers tegelijk. Hierbij zullen dus meerdere targets tegelijk geamplificeerd worden. In
de huidige screening op SOA’s wordt reeds gebruik gemaakt van een multiplex PCR die
zowel de aanwezigheid van C. trachomatis als die van N. gonorrhoeae bepaalt.xxiv
De keuzemogelijkheden inzake Taqman probes en de reporters in het bijzonder zijn
zeer divers, en net omwille van die diversiteit wordt multiplex PCR mogelijk. De
emissiegolflengtes van de verschillende reporters zijn karakteristiek, waardoor het
corresponderende geamplificeerd product eveneens specifiek geregistreerd wordt
(Figuur 1.4)xxv. Afhankelijk van de hoeveelheid beschikbare kanalen in het PCR toestel,
kan een multiplex PCR met een even groot aantal targets opgezet worden. Een rationele
keuze van reporters en quenchers is noodzakelijk, aangezien interferentie vermeden
dient te worden. Interferentie kan mogelijk leiden tot een bias op de werkelijke waarden
of veroorzaakt in ergere gevallen zelfs vals positieve waarden. Daarom wordt het best
geopteerd voor reporters die niet te dicht bij elkaar in de buurt komen qua emissie.
Figuur 1.4: Reporters met corresponderende emissiespectra
10
1.3.3 Rapportering
Een amplificatieplot geeft de toename in genormaliseerde fluorescentie weer in
functie van het aantal cycli (zie figuur 1.5).xxvi Er wordt genormaliseerd ten opzichte van
ROX, een fluorescente stof die reeds aanwezig is in de gebruikte enzymenmix en zorgt
voor een achtergrondsignaal. De fluorescentie stijgt exponentieel en snijdt grafisch met
een horizontaal gelegen treshold lijn. Deze lijn wordt getrokken op het punt in de curve
waar deze exponentieel stijgt. Vanuit het snijpunt kan op de horizontale as de cycle
treshold waarde (Ct waarde) afgelezen worden. De Ct waarde wordt gedefinieerd als het
aantal cycli nodig om een fluorescent signaal te genereren dat juist boven de
achtergrond uitkomt en is een indicatie voor hoeveel target DNA een staal bevat. Hoe
sneller de curve exponentieel zal stijgen, hoe sneller de curve ook zal snijden met de
treshold lijn en hoe lager de Ct waarde zal liggen. Een lage Ct waarde is dus
representatief voor een hoge concentratie target DNA in het geamplificeerd staal.
Figuur 1.5: Algemene amplificatieplot bij real-time PCR met treshold lijn en Ct waarde van ± 22
1.3.4 Targets voor PCR
1.3.4.1 Trichomonas vaginalis
Grondig literatuuronderzoek toonde aan dat vooral een 2-kb sequentie en een
sequentie die codeert voor het β tubuline proteïne op de voorgrond treden als meest
belovende targets. Specifieke primer/probe sets kunnen bijgevolg gebaseerd worden op
bepaalde deelsequenties, op hun beurt gelegen op de desbetreffende 2-kb sequentie.
Het lager kopiegetal van het β tubuline gen in vergelijking met de 2-kb sequentie brengt
11
een kleinere detectiekans van T. vaginalis met zich mee, wat de keuze voor het 2kbrepeat target rationaliseert. Het β tubuline target zou immers een minder aantal
positieve stalen detecteren.xxvii
1.3.4.2 Chlamydia trachomatis en Neisseria gonorrhoeae
De klassieke, reeds in de routine opgenomen 5plex PCR voor detectie van Chlamydia
trachomatis en Neisseria gonorrhoeae baseert zich op het 16S rRNA target en het
cryptisch plasmide of CP target voor C. trachomatis enerzijds en op het porA target en
het OMP target voor N. gonorrhoeae anderzijds.
Het 16S ribosomaal RNA target is een relatief kort stukje RNA van 1542 nucleotiden
dat deel uitmaakt van de 30S subeenheid van ribosomen bij prokaryoten. Het target
komt enkel voor in bacteriële cellen en vertoont in bepaalde gebieden evolutionair
verklaarbare gelijkenissen.xxviii Toch is het 16S rRNA soortspecifiek en net omwille van
dat verschil in sequentie is het mogelijk een onderscheid te maken tussen verschillende
bacteriën onderling. Naast het 16S rRNA target wordt C. trachomatis ook gedetecteerd
via het cryptisch plasmide of CP target. Deze dubbele detectie is er gekomen nadat in
2006 in Zweden een nieuwe variant van de bacterie was opgedoken (swCT). Deze
vertoont een deletie in het cryptisch plasmide gen en werd in het verleden vaak gemist
toen C. trachomatis nog gescreend werd op enkel het CP target.xxix
Ook bij N. gonorrhoeae wordt gebruik gemaakt van 2 targets. Bepaalde varianten op
de klassieke bacterie worden niet gedetecteerd aangezien deze het porine pseudogen
(porA gen) van de N. meningitidis bacterie bevatten. Het outer membrane proteïn (OMP)
gen codeert voor het beschermende buitenste membraan in Gram-negatieve cellen en
speelt een belangrijke rol als virulentiefactor. In tegenstelling tot het porA gen, dat
bovendien niet noodzakelijk tot expressie komt, is deze target wel ontegensprekelijk in
de correcte vorm aanwezig waardoor de zekerheid op detectie toch gegarandeerd kan
worden. xxx,xxxi
12
1.4 SEQUENCING
De cycle sequencing methode baseert zich in grote lijnen op dezelfde principes
als de klassieke PCR, al is er wel verschil tussen beiden. Zo wordt bij PCR bijvoorbeeld
geen gebruik gemaakt van dideoxynucleotides (ddNTP’s) en wordt hier slechts met één
primer gewerkt. Het gebruik van twee verschillende primers zou het uiteindelijke
resultaat vertroebelen doordat per lengte-eenheid twee signalen gegenereerd zouden
worden.
Een veelgebruikte DNA sequencing methode is de Sanger sequencing methode.
In eerste instantie wordt de targetsequentie geamplificeerd. De ketenverlenging is
echter niet volledig en stopt op het moment wanneer een ddNTP wordt ingebouwd.
Door een ontbrekende 3’ hydroxyl groep op de ddNTP molecules is ketenverlenging niet
meer mogelijk. Deze ddNTP’s, bestaande uit ddATP, ddGTP, ddCTP en ddTTP, hebben
alle vier hun eigen fluorescentielabel. Elk label vertoont een karakteristieke emissie
zodat na inbouw in de gerepliceerde streng een fluorescent signaal kan gegeven worden.
Door het gebruik van capillaire elektroforese zullen de kortst geamplificeerde producten
de detector eerst bereiken. Net zoals bij de klassieke gelelektroforese is de grootte van
de molecule rechtevenredig met de weerstand tijdens migratie. Bijgevolg zullen de
grootste moleculen het traagst migreren richting detector. Eenmaal de detector bereikt,
worden de labels geëxciteerd door de laser waardoor bij terugval naar grondniveau een
karakteristiek emissiepatroon opgevangen wordt door de detector. De detector herkent
op zijn beurt het ontvangen signaal als één van de vier mogelijke basen, zijde A, G, C of T
(zie figuur 1.6).xxxii De ruwe data worden via software verwerkt tot een interpreteerbaar
elektroferogram.
Figuur 1.6: Elektroferogram en bijbehorende nucleotidensequentie
13
2. OBJECTIEF
In de huidige screening op SOA’s wordt, door het relatief onschuldig karakter van de
parasiet, T. vaginalis vaak niet opgenomen in andere, gevoeligere detectieanalyses dan het
klassieke microscopisch onderzoek. Nochtans is een goede diagnostiek van Trichomonas
infecties essentieel ter vermijding van complicaties zoals cystitis of urethritis. Bovendien leidt
het ontbreken van correcte diagnoses tot een hogere transmissiefrequentie. Naast de
ongemakken voor de geïnfecteerde patiënt zelf, zorgt dit alles ook voor een ongewenst grotere
kost voor de gezondheidszorg.
Het doel van deze thesis bestaat erin een RT-PCR methode te ontwikkelen, optimaliseren
en valideren zodat T. vaginalis infecties opgespoord kunnen worden. Een T. vaginalis single RTPCR wordt geoptimaliseerd en in tweede instantie bij een reeds bestaande assay gevoegd die
screent op de aanwezigheid van de bacteriën Chlamydia trachomatis en Neisseria gonorrhoeae.
Op die manier zou binnen één en dezelfde analyse een staal geëvalueerd kunnen worden op het
voorkomen van zowel C. trachomatis, T. vaginalis als de genitale gonococ.
Genitale swabs en stalen worden allereerst onderworpen aan een extractieprocedure
waar al het aanwezige DNA en RNA geïsoleerd wordt. Op deze extracten kan vervolgens een
PCR uitgevoerd worden. In deze thesis wordt gebruik gemaakt van RT-PCR, waarbij een
specifieke TaqMan probe zorgt voor een fluorescent signaal, evenredig met de hoeveelheid
geamplificeerd product. Er wordt een 6plex PCR ontwikkeld waarmee C. trachomatis via het
16S rRNA target en CP target, N. gonorrhoeae via het OMP target en porA target en T. vaginalis
via een 2kb specifieke repeat gedetecteerd kunnen worden. Het PhHV target wordt als zesde
target aangewend als interne extractie- en inhibitiecontrole. Deze 6plex PCR wordt uitgebreid
gevalideerd en indien voldoende potent bevonden in gebruik genomen in het OLV Ziekenhuis
campus Aalst.
T. vaginalis positieve stalen worden ook geanalyseerd via RMO en een commercieel
beschikbare antigen sneltest kit: de OSOM® Trichomonas Rapid Test (Sekisui Diagnostics, Tokio,
Japan). Deze methodevergelijking levert een goed beeld van het verschil in performantie tussen
de ontwikkelde 6plex RT-PCR methode en reeds bestaande referentiemethoden.
14
3. MATERIAAL EN METHODEN
3.1 STALEN
Stalen worden gecollecteerd vanuit het OLV Ziekenhuis campus Aalst zelf, alsook
vanuit de campus in Asse en vanuit het AZ Damiaan in Oostende. Stalen positief voor
C. trachomatis, N. gonorrhoeae of T. vaginalis zijn ter beschikking voor analyse. De stalen
worden via RMO, een antigen sneltest of via PCR getest. Omwille van de patiëntenconfidentialiteit worden de staalnummers van de gebruikte stalen slechts gedeeltelijk
weergegeven of worden de staalnummers vervangen door acroniemen.
3.2 PRIMERS EN PROBES
3.2.1 Selectie primers en probes
Specifieke primers en probe worden gekozen op basis van een goed gekende en
vaak voorkomende targetsequentie in het genoom van het te detecteren microorganisme. Nu wordt gewerkt met een T. vaginalis specifieke repeat met NCBI accessie
nummer L23861. Deze sequentie staat opgelijst in de databank van NCBI. Vervolgens kan
deze sequentie geanalyseerd worden met het software programma Primer Express, dat
primer- en probe combinaties voorstelt die werken bij het temperatuurprofiel van de
gebruikte ABI Prism 7500 Fast Real-Time PCR toestellen van Life Technologies, California,
USA (serienr. 275013004 en 275013017). Met een samenvattende penalty score worden
de primers in het softwareprogramma Primer Express in volgorde van geschiktheid
geclassificeerd. De gekozen primers en probe worden bevestigend nog eens gescreend
met BLAST (Basic Local Alignment Search Tool) ten opzicht van de T. vaginalis specifieke
repeat om eventuele mismatchen op te sporen.
Primers en probes voor C. trachomatis , N. gonorrhoeae en het Phocine Herpes
Virus (PhHV: interne controle) zijn reeds ter beschikking aangezien in de routine al een
5plex RT-PCR uitgevoerd wordt die bovenstaande oligonucleotiden bevat.
15
3.2.2 Gebruikte primers, probes en reagentia
De primers (506 en 507) die nodig zijn voor het amplificeren van de T. vaginalis
targetsequentie worden aangekocht bij Life Technologies™, alsook primer 509, die bij
het sequencen in de specificiteitsvalidatie gebruikt wordt. De gebruikte TaqMan probe
(508) wordt aangekocht bij Eurogentec, Seraing, België. De sequenties en modificaties
van de oligonucleotiden worden weergegeven in Tabel 3.1.
Tabel 3.1: Overzicht van gebruikte primers en probes
Naam
Sequentie (5' - 3')
Trichomonas Forward Primer (p506)
AAG ATG GGT GTT TTA AGC TAG ATA AGG T
Trichomonas Reverse Primer (p507)
CGT CTT CAA GTA TGC CCC AGT AC
Trichomonas Probe (p508)
Cy 5®-CCG AAG TTC ATG TCC TCT CCA AGC GT- BHQ-2®
Forward primer CP CHTR (p324)
CAG CTT GTA GTC CTG CTT GAG AGA
Reverse primer CP CHTR (p325)
CTC GCT CCG GAA AAA TGG T
Probe CP CHTR (p326)
FAM®-CGT GCG GGC GAT TTG CCT TAA C- BHQ-1®
Forward primer 16S CHTR (p321)
AAG TCG AAC GGA GCA ATT GTT T
Reverse primer 16S CHTR (p322)
ACC CCA CCA ACT AGC TGA TAT CA
Probe 16S CHTR (p323)
FAM®-TCG GAT GCC CAAATA TCG CCA CA- BHQ-1®
Forward primer porA (p344)
GCA TTC AAT TTG TTC CGA GTC A
Reverse primer porA (p345)
CCG GAA CTG GTT TCA TCT GAT T
Probe porA (p346)
Cy 5®-CGT GAA AGT AGC AGG CGT ATA GGC GGA- BHQ-2®
Forward primer OMP (p347)
CAT ACC GAC TTT ATG GGT TCT GAA
Reverse primer OMP (p351)
AGC CGA CAG CAG AAA TTC TAG TAG
Probe OMP (p352)
TAMRA®-CTC TGT CCG AAC GCC GCG CAT A- BHQ-2®
Forward primer PhHV (p318)
GGG CGA ATC ACA GAT TGA ATC
Reverse primer PhHV (p319)
GCG GTT CCA AAC GTA CCA A
Probe PhHV (p320)
Yakima Yellow®-TTT TTT ATG TGT CCG CCA CCA TCT GGA TC- BHQ-1®
TOPO® Forward primer (p247)
GTA AAA CGA CGG CCA G
CAG GAA ACA GCT ATG AC
TOPO® Reverse primer (p248)
Sequencing primer (p509)
GCT GCT TGA CCA TCC GAA AT
Bovenstaande primers en probes worden vervolgens gemengd in een bepaalde
verhouding tot een primer probe mix (PP mix), die op zijn beurt gemengd kan worden
met een commercieel beschikbare TaqMan® Fast Universal PCR Mastermix van Life
Technologies™ (catnr. 4352042). Deze buffer bevat in essentie een hittestabiel gezuiverd
DNA polymerase, alle bouwstenen en stoffen voor efficiënte DNA amplificatie en
16
eveneens een fluorescente kleurstof (ROX) die dienst doet als achtergrondsignaal zodat
er genormaliseerd kan worden ten opzichte van niet-PCR gerelateerde fluorescenties.
3.3 EXTRACTIE
Er wordt gewerkt met het NucliSens easyMAG 1.0 toestel van bioMérieux, Parijs,
Frankrijk (serienr. 00108 en 1057), dat op automatische wijze DNA en RNA uit de
staalmatrices extraheert. Gecollecteerde genitale stalen worden eerst afgedraaid om
lekkage bij het openen te vermijden.
200µL homogeen staal wordt toegevoegd aan de NucliSens Lysis Buffer
(bioMérieux, part No 2000292), reeds vooraf uitverdeeld in lysis tubes. De nodige
veiligheid moet ingebouwd worden aangezien de buffer guanidine thiocyanaat bevat.
Contact met zuren kan zorgen voor een gasontwikkeling die toxisch is. Eveneens wordt
10µL 105 kopieën plasmide/mL PhHV interne controle (IC) toegevoegd. Dit is een
extractiecontrole en verzekert een goed verloop van de extractieprocedure wanneer het
plasmide DNA van de IC teruggevonden wordt na PCR. Vervolgens wordt goed
gevortexed en gedurende 10 minuten geïncubeerd bij kamertemperatuur. Het toestel
kan ook zelf de stalen lyseren, maar hier wordt dus geopteeerd voor een off board lysis
procedure. De stalen kunnen nu met behulp van pasteurse bolpipetten overgebracht
worden in de NucliSens easyMAG vessels. Eerst werd aan elk van deze vessels 50µL
NucliSens easyMAG Magnetic Silica (bioMérieux, part No 280133) toegevoegd. Deze
korrels met nucleïnezuur adsorberende eigenschappen zijn essentieel en zorgen ervoor
dat het DNA en RNA gescheiden wordt van het overig celmateriaal in de stalen. Tijdens
het wassen worden alle verontreinigingen behalve de geadsorbeerde nucleïnezuren
afgezogen, waarna in een volgende stap het gezuiverd DNA en RNA in een onderstaande
vloeistof elueert. In deze stappen maakt het toestel gebruik van NucliSens easyMAG
Extraction Buffer 1, 2 en 3 (bioMérieux, part No 280130, 280131 en 280132). Uiteindelijk
worden de silica korrels met behulp van een magnetische staaf uit de vloeistof
getrokken zodat een zo zuiver mogelijk staalextract bekomen wordt.
17
Via het bijbehorende softwareprogramma kan het toestel vanaf de pc gestuurd
worden. Eerst worden alle stalen ingegeven. Bij ‘volume’, ‘eluate’, ‘lysed or non lysed’ en
‘type’ wordt respectievelijk voor volgende parameters gekozen: 200µL, 110µL, Lysed,
Other. De geïntroduceerde vessels en reagentia moeten altijd eerst gescand worden. De
extractie start en duurt ongeveer 40 minuten, afhankelijk van het aantal te extraheren
stalen. xxxiii
3.4 KLONEREN
In eerste instantie dienen positieve plasmide controles aangemaakt te worden
vooraleer van start kan gegaan worden met de ontwikkeling van de RT-PCR methode. Er
wordt een klassieke PCR uitgevoerd op enkele stalen (*7325, *9954 en *6762) die
voldoende positief bevonden zijn voor T. vaginalis. Hiertoe worden twee verschillende
mixen aangemaakt die in eenheidsvolumes 1µL polymerase enzym (5U/µL), 2µL forward
primer 506 (10µM), 2µL reverse primer 507 (10µM), 3µL MgCl2 (50 mM), 10µL
deoxynucleotide oplossing (2mM), 10µL PCR buffer 10x en 60µL RNase/DNase vrij water
bevatten. In de tweede mix zit eveneens 1µL BSA 100x en DMSO 100x. De gebruikte
reagentia werden aangekocht bij Life Technologies™. Van elke mix wordt 90µL gemengd
met 10µL van het gewenste DNA extract en aan PCR onderworpen. Initieel wordt de
temperatuur in het GeneAmp® PCR system 2700 (Life Technologies™) toestel gedurende
5 minuten op 94°C gehouden waarna 45 cycli aangevat worden: 30 seconden op 94°C,
vervolgens 30 seconden op 60°C en 45 seconden op 72°C. De PCR producten worden
uiteindelijk nog 15 minuten op 72°C gehouden om dubbelstrengig DNA te verzekeren.
De geamplificeerde producten worden vervolgens op gel gezet om het positief karakter
van de stalen te verifiëren (Figuur 3.1). Bandjes van positieve stalen hebben een lengte
van 84bp.
Figuur 3.1: vlnr. basenpaar ladder, stalen *7325, *9954, *6762 en een negatieve controle met
DMSO en BSA mix, stalen *7325, *9954, *6762 en negatieve controle zonder DMSO en BSA mix
18
Een tweede PCR wordt uitgevoerd op de stalen die een correcte bandjeslengte
opleveren. De bandjes die het gewenste amplicon bevatten worden uit de gel gesneden
met een scalpel, waarna het DNA selectief uit de gel geëxtraheerd wordt met behulp van
de QIAquick® Gel Extraction kit van Qiagen (Hilde, Duitsland). Aan de uitgesneden
bandjes wordt 600µL QG buffer toegevoegd en geïncubeerd op 50°C gedurende 10
minuten. Er wordt na het oplossen van de gel 200µL isopropanol toegevoegd en goed
gemengd. De oplossing wordt overgebracht op QIAquick® columns en er wordt
afgedraaid gedurende 1 minuut op 13000rpm. Vervolgens wordt aan het geadsobeerde
DNA 750µL PE buffer toegevoegd en gecentrifugeerd. Er wordt droog afgedraaid om een
droge kolom te verzekeren. De kolommen worden in nieuwe 1,5mL eppendorfs
geplaatst en het gebonden DNA wordt van de kolom afgeëlueerd met 50µL EB buffer. xxxiv
Het geëxtraheerde DNA kan nu in een kloneringsvector geligeerd worden. Er
wordt gewerkt met de TOPO® TA Cloning® Kit van Life Technologies™. Deze kit bevat de
pCR™2.1-TOPO® vector waarin de beoogde sequentie geligeerd kan worden (Figuur 3.2).
2µL DNA product wordt gemengd met 1µL zoutoplossing, 1 µL TOPO® vector en dit alles
wordt aangelengd met DNase/ RNase vrij water tot een totaalvolume van 6µL. Er wordt
goed
gemengd.
Vervolgens
wordt
gedurende
30
minuten
geïncubeerd
op
kamertemperatuur zodanig dat de PCR producten voldoende de tijd krijgen om te
hybridiseren in de vector.xxxv
Figuur 3.2: de pCR™2.1-TOPO® vector klonering site en zijn omgevende sequenties
Competente E. coli bacteriën die op ijs bewaard worden ondergaan een heat
shock. Door de korttijdige temperatuursverhoging gaan de poriën in het celmembraan
van de bacteriën zich openen zodat de plasmiden makkelijker opgenomen worden.
19
De plasmiden worden in oplossing toegevoegd aan de bacteriënsuspensie en gedurende
30 seconden wordt de temperatuur op 42°C gebracht. Nadien worden de behandelde
bacteriën snel terug op ijs geplaatst en wordt er SOC (Super Optimal broth with
Catabolic repression) medium toegevoegd om de bacteriën te laten herstellen van de
hitteshock. Vervolgens wordt gedurende 1 uur geschud bij 37°C (200rpm). De bacteriën
worden uitgeplaat op ampicilline bevattende TSA platen en worden een nacht op 37°C
geïncubeerd. Omwille van het ampicilline resistentiegen van de vector zullen enkel
bacteriën die de vector effectief hebben opgenomen op de platen groeien.
20 individuele kolonies worden de dag nadien gesuspendeerd in 25µL TSB. Nu zal
een nieuwe PCR-gelelektroforese test met specifieke vectorprimers opgezet worden om
na te gaan welke kolonies de vector mét geligeerd Trichomonas amplificatieproduct
bevatten. De lengte van de PCR producten zal immers groter zijn met insert. De
gebruikte PCR mix is samengesteld uit 0,25µL TOPO® vector specifieke primers p247 en
p248 (10µM), 0,75µL MgCl2 (50mM), 2,5µL PCR buffer (10x), 2,5µL dXTP mix (2mM),
0,25µL Taq polymerase enzym (0,05 U/µL) en 16µL gepurifieerd water als
eenheidsvolumes. Er wordt 22,5µL van deze mix gemengd met 2,5µL kolonie suspensie.
De PCR bezit, op een annealingtemperatuur van 55°C na, hetzelfde temperatuurprofiel
als deze die eerder al uitgevoerd werd bij het kloneren.
Positieve kolonies, die dus de vector met het insert opgenomen hebben,
ondergaan een T. vaginalis RT-PCR. 5µL kolonie suspensie wordt gemengd met 20µL mix
die bestaat uit 12,5 µL TaqMan® Fast Universal PCR Mastermix en 7,5 µL PP mix. Deze
Trichomonas singleplex PP mix bestaat uit 0,75µL primer 506 en 507 (10mM), 1µL probe
508 (5mM) en 5µL gepurifieerd water. Na interpretatie van de data zullen de meest
geschikte kolonies, deze met de laagste Ct waardes, overnacht in tubes opgekweekt
worden. Deze TSB tubes bevatten naast de koloniesuspensie zelf ook ampicilline om
selectieve groei van de gewenste bacteriën te bekomen.
Opgekweekte kolonies worden opgezuiverd met de Wizard® Plus SV Minipreps
DNA Purification System kit van Promega (California, USA). De overnacht geïncubeerde
20
tubes worden 15 minuten afgedraaid aan 4000rpm. De bekomen pellet wordt
geresuspendeerd
met
250µL
Resuspension
Solution.
Daaropvolgend
wordt
achtereenvolgens 250µL lysis buffer, 10µL alkalisch protease oplossing en 350µL
neutralisatieoplossing toegevoegd, telkens gescheiden door een incubatieperiode van 5
minuten. Deze handelingen zorgen er respectievelijk voor dat de bacteriën gelyseerd
worden, de vrijgekomen endonucleases geïnhibeerd worden en dat de uiteindelijke
oplossing geneutraliseerd wordt. Ook zal het bacterieel genomisch DNA gefragmenteerd
worden, zonder aantasting van het plasmide DNA. Er wordt gecentrifugeerd op
14000rpm gedurende 10 minuten zodat een pellet met al het ongewenst materiaal
bekomen wordt. Het supernatans, dat het gewenste plasmide DNA bevat, wordt
overgebracht op een silicakolommetje. Nu wordt opnieuw kort gecentrifugeerd zodat
het DNA kan binden aan de kolom. Er wordt met 750 µL en 250µL Column Wash Solution
gewassen door respectievelijk 1 en 2 minuten kort af te draaien. Wanneer nu
RNase/DNase vrij water op de kolom gebracht wordt en er wordt opnieuw
gecentrifugeerd, dan zal het eluaat het gewenste plasmide DNA bevatten.xxxvi De
hoeveelheid aan gepurifieerd DNA wordt via een optic density (OD) meting met het
Nanodrop ND-1000 toestel gekwantificeerd (Isogen Life Science, Utrecht, Nederland).
Met behulp van onderstaande vergelijkingen wordt de concentratie aan plasmide DNA
weergegeven als het aantal kopieën plasmide DNA per mL.
waarin: m= g, MW= g/mol en n= mol
(3.1)
waarin: NA= 6,022 1023 moleculen/mol
(3.2)
Er wordt een verdunningsreeks opgesteld met TE 1x als verdunningsmedium. In
de eerste twee stappen wordt 1/100 verdund, vanaf 109 kopieën plasmide per mL is dat
1/10. Concreet wordt aan 5µL van de stock DNA oplossing (bij benadering 1013 kopieën
plasmide DNA per mL) 490µL TE 1x toegevoegd, alsook 5µL Fish Sperm DNA (FS DNA).
Het toevoegen van FS DNA dient om de aanwezige plasmiden te stabiliseren en om
aspecifieke bindingen van de oligonucleotiden met oppervlakten te vermijden. Deze
nieuwe oplossing van 1011 kopieën plasmide per mL wordt op dezelfde wijze nogmaals
21
verdund tot een oplossing die 109 kopieën plasmide per mL bevat. Aan 50µL van deze
nieuw bekomen oplossing wordt opnieuw 5µL FS DNA toegevoegd en aangelengd tot
500µL met TE buffer 1x. Deze stap wordt herhaald tot een concentratie van 10 3 kopieën
plasmide per mL bereikt wordt. Met deze aangelegde verdunningsreeks zal verder
gewerkt worden tijdens de optimalisatie van de RT-PCR methode.
3.5 REAL-TIME PCR
3.5.1 Algemeen
Allereerst wordt de PCR mix aangemaakt die de gewenste primers, probes en
enzym bevat voor de RT-PCR. Op basis van het aantal stalen wordt volgens in een 5:3
verhouding per staal 12,5µL TaqMan® FAST universal Mastermix gemengd met 7,5 µL PP
mix. Vervolgens kan dan per welletje 20µL van bovenstaande homogene mix uitverdeeld
worden in de 96 well FAST platen (MicroAmp Fast Optical 96-well reaction plates, Part
No 4346906). 5µL van het gewenste DNA extract wordt toegevoegd aan de 20µL mix die
reeds in het welletje aanwezig is. Naast de analyse van de stalen wordt altijd een
positieve controle meegenomen die de amplificatie van het target in de RT-PCR
controleert. Ook een negatieve controle, om eventuele contaminatie uit te sluiten en de
specificiteit van de RT-PCR te bevestigen, wordt meegenomen op de plaat. Als positieve
controle wordt 5µL plasmide extract van het target toegevoegd, terwijl een negatieve
controle net geen target DNA bevat. De gevulde platen worden afgesloten met een
doorzichtige ‘adhesive cover’ om verdamping en contaminatie te vermijden.
De gevulde en afgedekte plaat wordt gedurende een paar seconden op de
trilplaat gezet en ondergaat een korte spin om te verzekeren dat alle luchtbelletjes zich
bovenaan het vloeistofoppervlak bevinden zodat de PCR niet gestoord wordt. De plaat
wordt in een ABI Prism 7500 Fast Real-Time PCR toestel geplaatst en er wordt een
‘advanced setup’ gecreëerd. Er wordt gekozen voor een ‘quantitation run’ en in het
venster van ‘plate setup’ worden de gewenste targets toegevoegd. Deze targets, meer
22
bepaald de 2kb specfieke repeat voor T. vaginalis, het 16S- en CP target voor
C. trachomatis, het omp- en porA target voor N. gonorrhoeae en het PhHV target voor
de interne controle, kunnen dan toegewezen worden aan welletjes in het venster van
‘plate layout’. De correcte ‘run method’ wordt gekozen waarna de run gestart kan
worden. Initieel wordt de plaat 5 minuten op 50°C gehouden, waarna de temperatuur
wordt opgetrokken en voor 20 seconden op 95°C gehouden wordt. Vanaf dan worden 45
cycli gestart: 3 seconden op 95°C en vervolgens 30 seconden op 65°C. Het resultaat
wordt onmiddellijk via ‘real-time imaging’ weergeven in een amplificatieplot.xxxvii
3.5.2 Optimalisatie
Zoals beschreven in ‘3.4 Kloneren’, wordt een plasmide verdunningsreeks
aangemaakt ter optimalisatie van de T. vaginalis RT-PCR. Vervolgens wordt via trial and
error naar de meest geschikte primer -en probeconcentraties gezocht. Verschillende
samenstellingen worden uitgeprobeerd op de T2 verdunningsreeks. Een overzicht van de
samenstelling van de PP mixen wordt weergeven in onderstaande tabel (Tabel 3.2).
Tabel 3.2: Buffer en PP mix samenstellingen in de optimalisatie van de 6plex PCR (20µL totaalvolume)
Reagens
[stock]
Taqman Fast
FP p506
RP p507
Probe p508
water
2x
10µM
10µM
5µM
Reagens
[stock]
Taqman Fast
FP p506
RP p507
Probe p508
water
2x
10µM
10µM
5µM
150nM
primer
200nM
probe
12,50µL
0,375µL
0,375µL
1,00µL
5,75µL
100nM
probe
12,50µL
0,75µL
0,75µL
0,50µL
5,50µL
100nM
probe
12,50µL
1,50µL
1,50µL
0,50µL
4,00µL
600nM
primer
150nM
probe
12,50µL
1,50µL
1,50µL
0,75µL
3,75µL
300nM
primer
150nM
probe
12,50µL
0,75µL
0,75µL
0,75µL
5,25µL
200nM
probe
12,50µL
0,75µL
0,75µL
1,00µL
5,00µL
200nM
probe
12,50µL
1,50µL
1,50µL
1,00µL
3,50µL
900nM
primer
200nM
probe
12,50µL
2,25µL
2,25µL
1,00µL
2,00µL
23
Er wordt, na het bereiken van een optimum in samenstelling aan primers en
probe, een grote hoeveelheid aan 6plex PCR PP mix aangemaakt (Tabel 3.3). Deze
samenstelling kan bij succesvol afronden van de optimalisatie en validatie van de
methode als definitief beschouwd worden.
Tabel 3.3:Definitieve 6plex PCR PP mix samenstelling
primer
target
Conc.
volume
p 347
OMP
100 µM
3,75µL
p 351
OMP
100 µM
p352
OMP
p344
primer
target
Conc.
volume
p322
CHTR16S
10 µM
37,50µL
3,75µL
p323
CHTR16S
5 µM
25,00µL
5 µM
50,00µL
p324
CHTR CP
10 µM
37,50µL
PorA
100 µM
3,75µL
p325
CHTR CP
10 µM
37,50µL
p345
PorA
100 µM
3,75µL
p326
CHTR CP
5 µM
25,00µL
p346
PorA
5 µM
50,00µL
p506
TRIVAG
100 µM
3,75µL
p318
PhHV
10 µM
12,50µL
p507
TRIVAG
100 µM
3,75µL
p319
PhHV
10 µM
12,50µL
p508
TRIVAG
50 µM
2,50µL
p320
PhHV
5 µM
12,50µL
water
12,50µL
p321
CHTR16S
10 µM
37,50µL
totaal
375,00µL
Aangezien het T. vaginalis target niet het enige target is dat opgenomen wordt in
de 6plex PCR methode, wordt ook van de andere targets een positieve controle
verdunningsreeks opgesteld. Startmateriaal van de 16S en CP target voor C. trachomatis
en de omp en porA target voor N. gonorrhoeae is reeds ter beschikking zodat de
kloneringsprocedure voor deze positieve controles niet herhaald dient te worden. Net
zoals de verdunningsreeks van T.vaginalis wordt ook nu 50µL oplossing aangelengd tot
500µL met TE buffer 1x, inclusief de 5µL FS DNA. De bekomen verdunningsreeksen zullen
vervolgens gebruikt worden om vergelijkende testen uit te voeren tussen de nieuw
ontwikkelde PCR en de huidige routine PCR’s.
Ook worden er op analoge wijze verdunningsreeksen gemaakt van DNA
extracten. Dit om de performantie van de PCR methode op reële stalen, in tegenstelling
tot positieve controles, te kunnen beoordelen. De verdunningsreeksen van extracten
worden onder andere gebruikt om de lineariteit van de 6plex RT-PCR te valideren, alsook
om checkerboard analyses uit te voeren. Checkerboard analyses geven de gevoeligheid
24
van een RT-PCR assay weer in aanwezigheid van een bepaalde concentratie van een
ander micro-organisme. Elk welletje op de 96 well plaat wordt zoals normaal gevuld met
20 µL TaqMan® FAST universal Mastermix – PP mix mengsel, waarbij nu niet langer 5µL
DNA template toegevoegd wordt, maar 2,5µL van een bepaalde verdunning van het ene
micro-organisme en 2,5µL van een bepaalde verdunning van het tweede (Tabel 3.4). De
gehanteerde lettercode is gebaseerd op het principe dat ‘A’ de onverdunde oplossing
voorstelt, zodanig dat bij aflopen van het alfabet er telkens tiendelig verdund wordt.
Tabel 3.4: Voorstelling van een checkerboard setup tussen T. vaginalis (T reeks) en C. trachomatis (C reeks)
TA
TB
TC
CA
CB
CC
2,5 µL 1x T. vag
2,5 µL 1x T. vag
2,5 µL 1x T. vag
2,5µL 1x C. trach
2,5µL 0,1x C. trach
2,5µL 0,01x C. trach
2,5µL 0,1x T. vag
2,5µL 0,1x T. vag
2,5µL 0,1x T. vag
2,5µL 1x C. trach
2,5µL 0,1x C. trach
2,5µL 0,01x C. trach
2,5µL 0,01x T. vag
2,5µL 0,01x T. vag
2,5µL 0,01x T. vag
2,5µL 1x C. trach
2,5µL 0,1x C. trach
2,5µL 0,01x C. trach
3.5.3 Validatie
De specificiteit van de methode wordt in drievoud nagegaan. Op vijf
verschillende positieve stalen wordt een sequentie-analyse uitgevoerd, beschreven
onder 3.7 Sequencing. Ten tweede worden een aantal positieve en negatieve stalen
onderworpen aan gelelektroforese, aangehaald onder hoofdstuk 3.6, waarbij enkel de
bandjes van het gewenste target zichtbaar mogen zijn. Er wordt ten derde ook nog een
RT-PCR uitgevoerd op organismen die frequent voorkomen in genitale stalen, afkomstig
van het labo microbiologie, om de specificiteit van de PCR voor T. vaginalis,
N. gonorrhoeae en C. trachomatis te bevestigen.
Om een idee te verwerven over de analytische accuraatheid, sensitiviteit en
specificiteit van de ontwikkelde RT-PCR methode, worden de resultaten van een aantal
geteste positieve stalen vergeleken met de resultaten afkomstig van gevalideerde
25
referentiemethoden. Er wordt gebruik gemaakt van 22 opgestuurde stalen vanuit
Oostende, alsook van de resultaten die bekomen werden in de test met de verwante
organismen. Ten derde worden alle routinestalen gedurende een week parallel
geëvalueerd met de 6plex PCR. Ten slotte wordt ook de 2015 EQC (External Quality
Control) reeks van C. trachomatis en N. gonorrhoeae onderworpen aan de ontwikkelde
RT-PCR.
Bij het bepalen van de lineariteit wordt teruggegrepen naar de eerder
aangelegde verdunningsreeksen van DNA extracten. Echter, om een eventueel verlies
aan gevoeligheid door de extractiestap op te merken wordt besloten om de
verdunningsreeks van T. vaginalis aan te maken vanaf een positief staal zelf. Er wordt
verdund in eSwab® medium. Van twee positieve stalen, TV 13 en TV 15 – Oostende,
wordt zo’n verdunningsreeks opgesteld en in drievoud geëxtraheerd. Op die manier
wordt vanuit een correct en volledig perspectief de lineariteit geëvalueerd. Zowel de
verdunningsreeks van C. trachomatis, N. gonorrhoeae als die van T. vaginalis wordt in
drievoud getest via RT-PCR, met zowel de 6plex PP mix als de referentie 5plex PP mix.
Met als doel de intrarun precisie van de 6plex PCR te bepalen wordt gebruik
gemaakt van staal TV13 – Oostende voor T. vaginalis detectie, van staal *7703 en *7708
voor respectievelijk C. trachomatis en N. gonorrhoeae. De interrun precisie wordt
gekoppeld aan de test op houdbaarheid, waar positieve stalen (*1349, *9233, *7997,
*9212 en *2214) via een strikt protocol geanalyseerd worden. Op gefixeerde data wordt
een positief staal, bewaard op kamertemperatuur of in de koelkast, geëxtraheerd en
onderworpen aan PCR. De stalen worden getest op de dag van afname, 4 dagen na
afname, 8 dagen na afname en 15 dagen na afname.
3.6 GELELEKTROFORESE
Het Mupid®-One elektroforesetoestel van Eurogentec (serienr. 072566) scheidt
PCR producten onder invloed van een spanningsveld doorheen een agarosegel. De gel
bevindt zich in 0,5x TAE buffer; Tris Acetaat EDTA buffer (10x) twintig maal verdund
26
(Sigma-Aldrich, catnr. T8280-1L). Tijdens de run wordt ook altijd een 100bp ladder
meegenomen van Life Technologies (catn° 10488-058). Het te detecteren DNA wordt,
vooraleer het geladen wordt op de gel, gemengd met een ladingsbuffer van Life
Technologies (catn° 10482-028). Visualisatie nadien gebeurt met een VWR GenoView
UV-lamp en fototoestel.
1,8g afgewogen agarose (Life Technologies, lotnr. 4041901) wordt opgelost in
100mL 0,5x TAE buffer. Hiertoe wordt de suspensie in de microgolfoven geplaatst op
360W, tot wanneer het kookpunt bereikt wordt. Aan de oplossing wordt vervolgens
10µL GelRed™ Red Stain 10000x (catnr. 41003) toegevoegd en uitgegoten in een mal om
te stollen. De gestolde gel kan vervolgens in het toestel gebracht worden, waarbij het
vloeistofniveau van TAE buffer de bovengrens van de gel moet overschrijden. Aan 10µL
PCR product wordt 2µL ladingsbuffer toegevoegd en in de welletjes van de gel
gepipetteerd. Eén welletje wordt ook gevuld met de 100bp ladder. De run wordt gestart
en loopt gedurende 20minuten op 100V. Na de run worden de bandjes met behulp van
UV licht zichtbaar gemaakt en wordt het beeld fotografisch vastgelegd. xxxviii
3.7 SEQUENCING
Er wordt een klassieke PCR ingezet met p506 en p509 voor de stalen die
gesequenced dienen te worden. Het temperatuurprofiel van de PCR is identiek aan deze
die eerder al gebruikt werd bij het kloneren. De geamplificeerde producten worden
allereerst op gel gezet om te verifiëren of de amplificatie goed verlopen is. De
geamplificeerde DNA producten worden vervolgens opgezuiverd. DNA selectieve filters
(Amicon R Ultra-0.5 Centrifugal Filter Devices) worden gevuld met 360µL TE buffer 1x
(1/100 TE 100x catnr. 117K8612), waaraan 40µL amplificatieproduct toegevoegd wordt.
Na centrifugatie bevindt het gewenste DNA zich in de filter. Het filtraat wordt
weggegooid. Deze filters worden nu omgekeerd op nieuwe eppendorfs geplaatst die
15µL TE buffer 1x bevatten. Er wordt opnieuw gecentrifugeerd zodat het gefilterde DNA
terechtkomt in de TE buffer 1x en beschikbaar is voor verdere handelingen.
27
3µL hiervan wordt aangelengd tot 15µL met water. 3µL van deze 1/5 DNA oplossing
wordt samengevoegd met 1µL gewenste primer (3,33µM primer 509), 8µL water en 8µL
DTCS (GenomeLab™ Dye Terminator Cycle Sequencing Quick Start Kit), wat resulteert in
een totaalvolume van 20µL. Deze oplossing ondergaat een cycle sequencing programma
van 30 cycli. Het GeneAmp PCR system 2400 toestel van Life Technologies (serienr.
803P5100308) zorgt voor een initiële verwarming tot een temperatuur van 96°C, zodat
vanaf nu de cycli aangevat kunnen worden: 20 seconden op 96°C, vervolgens 20
seconden op 50°C en 4 minuten op 60°C. Nadien worden de geamplificeerde producten
op 4°C gehouden.
Na cycle sequencing wordt een alcoholprecipitatie uitgevoerd ter opzuivering van
de bekomen fragmenten. Lege 1,5mL eppendorfs worden gevuld met 5µL
neerslagmedium. Dit bevat 1µL glycogeen (Genome Lab™ DTCS Quick Start Kit), 2µL 3M
natriumacetaat pH 5.2 en 2µL 100 mM EDTA (Sigma-Aldrich). De cycle sequencing
producten worden vervolgens toegevoegd aan dit medium. Er wordt 60µL EtOH 100%
toegevoegd en er wordt gecentrifugeerd gedurende 20 minuten bij 4°C op 14000 rpm.
De bekomen pellets worden gewassen met 200µL 70% EtOH, waarna kort afgedraaid
wordt en deze stap herhaald wordt. Nadat de pellets minimaal 20 minuten aan de lucht
gedroogd hebben, wordt aan elke pellet 40µL Sample Loading Solution (SLS) toegevoegd.
De volledige inhoud wordt vervolgens overgepipetteerd naar de overeenkomstige
welletjes van de plaat van de capillaire sequencer. Op alle vloeistofoppervlaktes wordt
een druppel minerale olie aangebracht tegen verdamping.xxxix
Naast de plaat die het DNA bevat, wordt een tweede 96-well plaat gevuld. In de
overeenkomstige welletjes wordt een scheidingsbuffer (Beckman Coulter, California,
USA) gepipetteerd. Beide worden in het toestel gebracht. De fluorescent gemerkte
geamplificeerde DNA fragmenten worden gescheiden met behulp van het CEQ 8000
Genetic Analytic System toestel van Beckman Coulter (serienr. 3067187) via capillaire
elektroforese. In het toestel bevinden zich acht capillairen waardoor in achtvoud
gewerkt kan worden.
28
4. RESULTATEN
4.1 KLONEREN
Volgens de procedure beschreven in ‘3.4 Kloneren’, zou bij een klassieke PCR van
het T. vaginalis target een bandjeslengte van 84bp bekomen moeten worden. De
bekomen resultaten bewezen deze veronderstelling (Figuur 3.1), waardoor beslist werd
om verder te werken met staal *6762 zonder gebruik van DMSO en BSA in de mix.
Na hybridisatie van de geamplificeerde producten in de kloneringsvector,
heatshock in E. coli bacteriën en overnacht groeien op de selectieve TSA – ampicilline
bodem worden de bacteriekolonies op de platen geëvalueerd. 20 zuivere kolonies
worden opgepikt en gesuspendeerd in TSB medium, waarop een nieuwe PCRgelelektroforese test uitgevoerd wordt met specifieke vector primers zoals vroeger al
aangehaald (Figuur 4.1). Bandjes met een lengte van 328bp komen overeen met de
lengte van het beoogde product, i.e. het geamplificeerd stuk vector (244bp) met de
insert (84bp). Bandjes die verder gemigreerd zijn bevatten hoogstwaarschijnlijk het
insert niet, zodat deze ook niet in aanmerking komen voor de daaropvolgende RT-PCR.
Onder andere kolonie 1 behoort tot deze laatste groep.
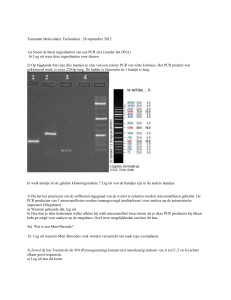
Figuur 4.1: vlnr. basenpaar ladder, positieve controle, negatieve controle en alle kolonies (1-14)
Kolonies 2 en 11 bleken na single RT-PCR analyse de twee meest belovende
kolonies met respectievelijke Ct-waarden van 15,29 en 15,44. Na het overnacht
opgroeien van deze kolonies en opzuivering van de plasmiden wordt de optische
densiteit van het bekomen plasmide DNA gemeten (Tabel 4.1).
Tabel 4.1: Gemeten absorbantie en concentratie van de opgezuiverde plasmiden, waarbij een 260/280nm ratio
van 1,8 tot 2 zich in een voldoende zuivere DNA oplossing vertaalt.
Kolonie 2
Kolonie 11
Absorbantie (260nm)
2,053
1,760
260nm/280nm
1,88
1,89
conc. (ng/µL)
102,7
88,0
29
De opzuivering van kolonie 2 leverde 102,7 ng plasmide DNA op. Volgens
vergelijking (3.1) en wetende dat het moleculair gewicht van één mol plasmide met
T. vaginalis insert gelijk is aan 2,7
106 g, wordt een opbrengst van 3,81 10-14 mol
plasmide bekomen. Rekening houdend met het getal van Avogadro wordt via
vergelijking (3.2) een totale opbrengst van 2,29 1010 kopieën per µL of 2,29 1013
kopieën per mL berekend. (kolonie 11: 1,96 1013 kopieën per mL).
Om na te gaan of de positieve controles wel effectief het gewenste insert
bevatten, wordt de nucleotidenvolgorde van het PCR product opgehelderd door
sequencing. Zowel de T2 als de T11 positieve controle bevat het gewenste insert
eenmaal. Het resultaat kan vervolgens nog eens via BLASTing bevestigd worden door de
gevonden nucleotidensequentie te screenen ten opzichte van de databank van NCBI. Het
resultaat wordt weergegeven in Figuur 4.2. De primer -en probe bindingsplaatsen
vertonen bovendien geen enkele mismatch (100% match), wat de identiteit van het
insert bevestigd als zijnde het T. vaginalis target.
Figuur 4.2: Gescreende sequentie van het insert (T2) ten opzichte van de T. vaginalis sequentie (acc.nr. L23861)
4.2 OPTIMALISATIE
4.2.1 Optimalisatie van singleplex RT-PCR voor T. vaginalis
In een eerste optimalisatiestap wordt de concentratie aan primers gevarieerd in
het PP mengsel, terwijl de probeconcentratie op 200nM constant gehouden wordt. Op
de T2 T. vaginalis verdunningsreeks wordt met verschillende concentraties primer een
RT-PCR uitgevoerd. Concentraties van 300nM en 600nM primer blijken de beste
resultaten te geven. Binnen deze bekomen primerconcentraties wordt vervolgens de
30
concentratie aan probe gevarieerd van 100nM tot 200nM. De resultaten staan vermeld
in Tabel 4.2. Naast de Ct waarden worden ook de R2, de procentuele efficiëntie en de
maximale fluorescentie gerapporteerd. De R2 en efficiëntie spelen een grote rol in de
mate van de lineariteit van de RT-PCR. De maximale fluorescentie is best niet te laag,
wat een verminderde detectiekracht met zich meebrengt, of te hoog, wat de potentiële
kans op interferentie met de andere signalen van de multiplex PCR vergroot.
Tabel 4.2: Analyse verdunningsreeks T2 in optimalisatie van [primers en probe] (‘ng’ = niet gedetecteerd)
conc. primers
150nM
conc. probe
verdunningsreeks
1,00E+09
1,00E+08
1,00E+07
1,00E+06
1,00E+05
1,00E+04
1,00E+03
1,00E+02
R2
Eff%
max
fluorescentie
200nM
Ct
20,78
24,58
27,93
31,64
34,96
38,71
ng
ng
1,00
91,05
1,40
300nM
100nM
Ct
20,86
24,46
27,75
31,20
33,99
37,43
ng
ng
1,00
101,64
1,10
150nM
Ct
20,79
24,35
27,75
31,42
33,77
38,39
ng
ng
1,00
95,82
1,55
600nM
200nM
Ct
20,77
24,29
27,67
31,26
35,27
36,95
43,90
ng
0,99
98,63
1,85
100nM
Ct
21,06
24,54
28,01
31,40
34,12
ng
ng
ng
1,00
100,99
1,10
900nM
150nM 200nM 200nM
Ct
Ct
Ct
20,75
20,68
20,54
24,35
24,36
24,18
27,83
27,71
27,55
31,02
31,16
31,06
34,60
33,68
33,98
ng
ng
ng
ng
ng
ng
ng
ng
ng
1,00
1,00
1,00
95,39 101,75 97,92
1,85
2,30
2,35
De conditie waarbij gebruik gemaakt wordt van 300nM primers en 100nM probe
blijkt de meest interessante te zijn. Deze veronderstelling is gebaseerd op het feit dat de
Ct waarde idealiter stijgt met 3,3 eenheden per 1/10 verdunning. Dit omdat de
tiendelige verdunning bij iets meer dan de 3e cyclus weggewerkt wordt door
amplificatie. Het target wordt geamplificeerd tot respectievelijk een verdunning van 105
en 104 kopieën per mL bij 600nM en 300nM primers. Tevens is de fluorescentie bij een
probeconcentratie van 100nM reeds voldoende hoog, wat ervoor zorgt dat een hogere
probeconcentratie niet nodig is. Ook de R2 en de efficiëntie geven bij deze condities
mooie cijfers weer, zodat met deze ‘300-100’ conditie verder gewerkt zal worden. Er
wordt nu geprobeerd om bovenstaande ontwikkelde single RT-PCR bij een reeds
bestaande 5plex RT-PCR voor C. trachomatis en N. gonorrhoeae te voegen. Deze 6plex
31
RT-PCR methode zal zowel ten opzichte van de zonet geoptimaliseerde singleplex RTPCR als ten opzichte van de reeds gevalideerde routine 5plex RT-PCR vergeleken worden.
4.2.2 Optimalisatie van 6plex RT-PCR
4.2.2.1 Vergelijking met singleplex RT-PCR
Zowel de 6plex RT-PCR als de geoptimaliseerde referentie singleplex RT-PCR worden
getest met de eerder aangemaakte T2 T. vaginalis plasmide verdunningsreeks.
Resultaten staan in onderstaande Tabel 4.3. De Ct waarden (Ct waarde van de 6plex
PCR verminderd met de Ct waarde van de referentie PCR) geven negatieve resultaten
weer, wat impliceert dat de 6plex RT-PCR op vlak van gevoeligheid zelfs iets beter scoort
dan de singleplex RT-PCR. In absolute waarde verschillen de ΔCt’s maximaal 0,46 zodat
de twee PCR methodes zo goed als vergelijkbaar geacht kunnen worden.
2
Tabel 4.3: Vergelijking tussen een singleplex PCR (R =0,998 en Eff%=100,2) als referentie en de
2
ontwikkelde 6plex PCR (R =1 en Eff%=95,2)(‘ng’ = niet gedetecteerd)
Singleplex RT-PCR (Ct)
6plex RT-PCR (Ct)
ΔCt
20,54
24,11
27,50
30,94
33,70
ng
20,38
23,93
27,31
30,79
34,16
ng
-0,16
-0,18
-0,19
-0,15
0,46
nvt
1,00E+09
1,00E+08
1,00E+07
1,00E+06
1,00E+05
1,00E+04
4.2.2.2 Vergelijking met 5plex RT-PCR
Net zoals bij de vergelijking met de singleplex RT-PCR wordt er gebruik gemaakt van
de aangemaakte plasmide verdunningsreeksen van alle aanwezige targets om de
vergelijkende test uit te voeren. In Tabel 4.4 staan de bekomen resultaten.
2
Tabel 4.4: Vergelijking tussen een referentie 5plex PCR (R >0,99 en Eff%>80% voor alle targets) en de
2
ontwikkelde 6plex PCR met 16S target (R >0,99 en Eff%>80% voor alle targets)
5plex RT-PCR (Ct)
verdunningsreeks
1,00E+09
16S
20,02
CP
16,86
omp
17,11
porA
19,80
32
Tabel 4.4: Vervolg
1,00E+08
1,00E+07
1,00E+06
1,00E+05
1,00E+04
1,00E+03
23,56
26,97
30,71
34,10
36,22
ng
verdunningsreeks
1,00E+09
1,00E+08
1,00E+07
1,00E+06
1,00E+05
1,00E+04
1,00E+03
16S
20,19
23,71
27,26
30,81
34,05
ng
ng
verdunningsreeks
1,00E+09
1,00E+08
1,00E+07
1,00E+06
1,00E+05
1,00E+04
1,00E+03
16S
0,18
0,15
0,29
0,10
-0,05
nvt
nvt
20,39
20,56
23,97
23,87
27,68
27,19
31,02
30,69
34,74
33,98
37,77
38,12
6plex RT-PCR (Ct)
CP
omp
16,28
16,86
19,73
20,15
23,15
23,63
26,76
27,00
30,14
30,32
33,45
34,42
38,01
38,07
ΔCt
CP
omp
-0,58
-0,25
-0,66
-0,41
-0,81
-0,24
-0,92
-0,19
-0,88
-0,37
-1,29
0,44
0,24
-0,05
23,43
26,74
30,18
33,59
ng
ng
porA
18,45
22,06
25,34
28,82
31,61
35,94
ng
porA
-1,35
-1,37
-1,40
-1,37
-1,99
nvt
nvt
Wanneer de verschillen tussen 5plex en 6plex RT-PCR geanalyseerd worden, valt op
dat ook hier het verschil niet erg groot is; maximaal 1,99. Bij targets 16S en porA wordt
bij een verdunning van 104 kopieën per mL afwisselend wel en geen signaal
gedetecteerd door de 5plex- en 6plex RT-PCR. Vanaf 104 kopieën per mL leunt de
methode dus dicht tegen de detectielimiet aan, wat zowel voor de 5plex als 6plex RTPCR gezegd kan worden. Ook dient opgemerkt te worden dat met de 6plex RT-PCR vaak
een gevoeligere en dus lagere Ct waarde bekomen wordt dan met de vergelijkende
5plex RT-PCR. Deze resultaten impliceren bijgevolg dat de nieuwe methode onvoldoende
afwijkingen bevat ten opzichte van de huidige diagnostiek om de methode te verwerpen.
33
4.2.2.3 Vergelijking met externe referentie
In samenwerking met het klinisch labo van het AZ Damiaan in Oostende worden
een aantal verdachte stalen door beide laboratoria geanalyseerd. Op die manier wordt
een extra referentie ingebouwd inzake de kwaliteit van de ontwikkelde methode.
Extractie van 22 stalen waarop vervolgens RT-PCR op uitgevoerd wordt levert volgende
resultaten (Tabel 4.5). De resultaten tonen een accuraatheid (% correct gescoorde stalen
ten opzichte van de referentiemethode) van 100%, zodat deze analyse de
detectiekwaliteit van de geoptimaliseerde 6plex RT-PCR bevestigt. Slechts 1 staal werd in
AZ Damiaan ook getest op C. trachomatis en N. gonorrhoeae. Ook dit resultaat sluit aan
bij de resultaten die bekomen worden met de ontwikkelde 6plex RT-PCR.
Tabel 4.5: Vergelijking tussen de Ct waarden van de PCR analyse Aalst en PCR analyse Oostende. (‘ng’ = niet
gedetecteerd, ‘nu’ = niet uitgevoerd)
T.vaginalis
staal
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16 -> 22
Aalst (Ct)
24,89
28,67
ng
24,91
24,43
22,32
ng
24,54
23,48
ng
22,84
22,34
19,36
21,55
20,92
ng
Oostende (Ct)
23,66
25,58
ng
20,12
22,90
19,00
ng
20,05
18,94
ng
19,87
18,72
15,76
17,68
18,00
ng
C. trachomatis
Aalst (Ct)
ng
ng
ng
ng
ng
ng
ng
23,49
35,98
20,24
ng
ng
ng
ng
ng
ng
Oostende (Ct)
nu
nu
nu
nu
nu
nu
nu
22,26
nu
nu
nu
nu
nu
nu
nu
nu
N. gonorrhoeae
Aalst (Ct)
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
Oostende (Ct)
nu
nu
nu
nu
nu
nu
nu
ng
nu
nu
nu
nu
nu
nu
nu
nu
4.2.2.4 Checkerboard analyses
Het uitvoeren van checkerboard analyses is essentieel in het bepalen van
eventueel gevoeligheidsverlies van de 6plex PCR wanneer alle primers en probes worden
34
samengevoegd in een gezamenlijke PP mix. Het geeft namelijk aan in welke mate de
detectie van een bepaald target beïnvloed wordt door de aanwezigheid van andere
targets en dus ook van andere amplificatieproducten. Eventuele inhibitie van de PCR kan
op die manier uitgesloten worden. Er wordt een checkerboard analyse uitgevoerd voor
T.vaginalis in combinatie met C. trachomatis (Tabel 4.6) en een tweede voor T.vaginalis
in combinatie met N. gonorrhoeae (Tabel 4.7). De lettercodes worden verklaard in ‘3.5.2
Optimalisatie’.
Tabel 4.6: Checkerboard analyse 1 (‘ng’ = niet gedetecteerd, ‘nu ‘= niet uitgevoerd)
Ct waarden van T. vaginalis bij 1/10 verdunningsstappen van C. trachomatis
TA
TB
TC
TD
TE->TG
blanco
CA
CB
CC
CD
CE
CF
CG->CH
blanco
CA
CB
CC
CD
CE
CF
CG
CH
24,23
24,74
25,16
25,06
25,11
25,32
25,14
25,27
28,42
28,48
28,62
28,86
28,63
28,92
28,73
29,14
33,95
32,99
32,77
31,86
32,19
31,85
31,97
32,22
ng
ng
ng
ng
ng
35,99
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
Ct waarden van C. trachomatis bij 1/10 verdunningsstappen van T. vaginalis
TA
TB
TC
TD
TE
TF
TG
TH
19,13
19,13
19,39
19,29
19,27
19,31
19,33
nu
22,60
22,80
22,82
22,85
22,86
23,10
22,78
nu
26,04
26,27
26,34
26,34
26,37
27,08
26,27
nu
29,35
29,82
29,79
29,79
29,72
29,84
29,73
nu
33,54
33,47
33,09
33,33
32,88
33,39
33,24
nu
41,31
39,73
39,00
38,62
41,50
ng
37,59
nu
ng
ng
ng
ng
ng
ng
ng
nu
ng
ng
ng
ng
ng
ng
ng
nu
blanco
nu
28,54
31,56
38,70
ng
ng
blanco
18,99
22,57
26,11
29,47
32,89
36,85
ng
ng
Concluderend tonen de resultaten weinig tot geen verlies in gevoeligheid voor
beide analyses. Bij elke concentratie aan C. trachomatis wordt per T. vaginalis
verdunningsstap ongeveer eenzelfde Ct waarde voor T. vaginalis waargenomen. Deze
conclusie is gerechtvaardigd tot verdunning TC; vanaf dan wordt het Trichomonas target
niet langer gedetecteerd. Hetzelfde kan gezegd worden bij de Ct waarden voor
C. trachomatis, tot verdunning CE. De laatste kolom geeft de waarden onvertekend weer
aangezien hier geen invloed is van een aanwezig tweede DNA target.
35
Tabel 4.7: Checkerboard analyse 2 (‘ng’ = niet gedetecteerd, ‘nu‘= niet uitgevoerd)
Ct waarden van T. vaginalis bij 1/10 verdunningsstappen van N. gonorrhoeae
GA
GB
GC
GD
GE
GF
GG
GH
blanco
TA
17,98
20,16
20,85
20,92
20,87
20,89
20,96
21,01 20,86
TB
18,16
21,49
23,71
24,50
24,67
24,65
24,68
24,86 24,54
TC
18,19
21,62
24,79
27,16
28,14
28,21
28,26
28,58 28,14
TD
18,34
21,76
24,90
28,29
30,49
31,66
31,98
31,94 31,52
TE
18,26
21,82
24,99
28,44
31,40
34,57
35,52
36,12 34,97
TF
18,38
21,74
25,01
28,45
31,51
34,16
nu
ng
ng
TG
18,28
21,70
24,94
28,50
31,52
34,87
40,39
ng
ng
blanco 18,30
21,75
24,95
28,40
31,58
35,24
38,77
ng
ng
Ct waarden van N. gonorrhoeae (omp) bij 1/10 verdunningsstappen van T. vaginalis
TA
TB
TC
TD
TE
TF
TG
TH
blanco
GA
17,95
17,98
17,96
18,06
18,07
18,09
18,10
nu
17,96
GB
21,21
21,57
21,62
21,69
21,75
21,68
21,62
nu
21,52
GC
24,66
24,67
24,89
24,85
24,92
24,93
24,85
nu
24,64
GD
29,20
27,95
28,18
28,50
28,29
28,48
28,36
nu
28,01
GE
34,92
32,98
31,97
31,56
31,65
31,90
31,67
nu
31,13
GF
44,06
42,42
41,11
36,68
35,27
36,31
36,14
nu
35,81
GG
ng
ng
ng
ng
ng
ng
ng
nu
ng
GH
ng
ng
ng
ng
ng
ng
ng
nu
ng
blanco
ng
ng
ng
ng
ng
ng
ng
nu
ng
In tegenstelling tot Checkerboard 1 is deze tabel iets moeilijker te interpreteren.
Er moet namelijk rekening gehouden worden met het dubbel karakter van de
‘Trichomonas’ detectietarget. Aangezien de probe van de porA target van
N. gonorrhoeae ook een Cy5 reportermolecule bevat, zullen de uiteindelijke Ct waarden
2 reële waarden representeren; deze van T. vaginalis en die van N. gonorrhoeae. Toch
zijn waardevolle inzichten te verwerven uit bovenstaande tabel. Met behulp van de
blanco kolom kan bepaald worden van wanneer de 2e invloed precies begint te spelen.
Het complex karakter van deze dubbele detectie wordt verder behandeld in ‘5.
Discussie’, verderop in deze thesis. Eveneens is er slechts een minimaal effect van T.
vaginalis op de detectie van het N. gonorrhoeae omp target. Hiermee rekening houdend
kan besluitend gezegd worden dat er net zoals bij de resultaten in Tabel 4.6 geen verlies
in detectiegevoeligheid waargenomen wordt wanneer T. vaginalis samen met
N. gonorrhoeae in een staal voorkomt.
36
4.3 VALIDATIE
4.3.1 Technische validatie
4.3.1.1 Analyse van de specificiteit
De specificiteit van de PCR wordt op drie verschillende manieren geëvalueerd. Er
wordt in eerste instantie op 5 verschillende positieve stalen een sequentieanalyse
uitgevoerd om de identiteit van het micro-organisme te bevestigen. De bekomen
sequenties worden via BLAST gescreend ten opzichte van de gekende T. vaginalis repeat.
Resultaten worden weergegeven in Tabel 4.8. De sequenties stemmen in grote mate
overeen met de gekende T. vaginalis repeat, waardoor de specificiteit van de PCR
bevestigd wordt.
Tabel 4.8: Gelijkenis tussen cycle sequencing producten van positieve stalen met de T. vaginalis repeat
staal
TV 6
TV 11
TV 12
TV 14
TV 15
primer
p509
p509
p509
p509
p509
% match met T. vaginalis target
95%
94%
93%
95%
96%
Ten tweede worden een aantal verschillende positieve stalen onderworpen aan
een agarosegelelektroforese (Figuur 4.3). De stalen die gebruikt worden zijn deze vanuit
de TV 1- 22 reeks; de 22 stalen afkomstig van Oostende. De lengte van de bandjes op de
gel dienen in overeenstemming te zijn met de specifieke vooropgestelde lengtes, wat
een amplificatie van het correcte target impliceert. Geen enkele andere lengtes van
bandjes mogen teruggevonden worden aangezien dat kan wijzen op aspecifieke
amplificatie van nucleïnezuren. Vergelijking met de negatieve controle leert dat de
bandjes effectief van gewenste lengte zijn, zijnde 84bp voor T. vaginalis en 212bp voor
het 16S target van C. trachomatis. De interne controle, eveneens rond de 100 bp lang,
geeft hier weliswaar een lichte vertroebeling van de resultaten, aangezien ook de
negatieve controle een intens bandje oplevert.
37
Figuur 4.3: Gelelektroforese voor specificiteitsvalidatie: vlnr: ladder, positieve stalen TV13, TV14, TV15,
TV9, TV12, TV10, negatieve stalen TV16, TV17, TV18, TV19 en controles. (T.vag= Trichomonas positief,
CHTR= Chlamydia positief)
Er wordt als derde test ook nog een RT-PCR uitgevoerd op organismen die
voorkomen in genitale stalen (Tabel 4.9). Geen enkel staal buiten de positieve controles
gaf positieve resultaten, waardoor met voldoende zekerheid gezegd kan worden dat de
ontwikkelde PCR methode specifiek is voor de beoogde targets.
Tabel 4.9: Overzicht van geanalyseerde verwante micro-organismen
Staphylococcus epidermidis
Pseudomonas aeruginosa
Enterococcus faecalis
Candida glabrata
Gardnerella vaginalis
Escherichia coli
Streptococcus agalactiae
Herpes simplex virus (HSV)
Lactobacillus rhamnosus
Herpes simplex virus (HSV)
Candida albicans
Varicella zoster virus (VZV)
4.3.1.2 Bepalen van de analytische accuraatheid, sensitiviteit en specificiteit
Op verschillende positieve en negatieve referentiestalen wordt de nieuw
ontwikkelde PCR, de 6plex PCR, uitgevoerd. Hierbij wordt gebruik gemaakt van de 22
opgestuurde stalen vanuit Oostende, van de test op genitale organismen, van verdachte
C. trachomatis en N. gonorrhoeae routinestalen en van 2015 EQC stalen (WIV), eveneens
positief aan C. trachomatis of N. gonorrhoeae. Op basis van deze resultaten wordt
bijgevolg een berekening gemaakt van de sensitiviteit (% positieve stalen ten opzichte
van alle positieve stalen met referentiemethode), de specificiteit (% negatieve stalen ten
opzichte van alle negatieve stalen met referentiemethode) en de accuraatheid (%
correct gescoorde stalen ten opzichte van de referentiemethode), waarbij voor allen
38
90% als de absolute ondergrens beschouwd wordt. De resultaten van bovenstaande
testen worden gebundeld in onderstaande tabellen (Tabel 4.10 en Tabel 4.11).
Tabel 4.10: Overzicht van testen voor bepaling van de accuraatheid, sensitiviteit en specificiteit
Test
aantal stalen
positief
positief gedetecteerd
aanwezig organisme
Stalen Oostende
Genitale organismen
Routine stalen
EQC stalen
TOTAAL
22
12
23
10
67
12
0
2
8
22
12
0
2
8
22
TRIVAG
DIVERS
CHTR en GONO
CHTR en GONO
Tabel 4.11: Resultaten EQC stalen met referentie (5plex RT-PCR) en Trichomonas single
bevestingingsPCR (‘ng’ = niet gedetecteerd, ‘*’ = 1 op 10 verdund staal)
1
2
3
4
5
6
7
8
9
10
5plex RT-PCR (Ct)
CHTR
omp
porA
ng
ng
ng
ng
29,02
27,22
19,80
25,34
23,79
17,65
ng
ng
21,45
30,70
28,51
15,76
ng
ng
16,75
17,99
17,66
19,26
21,27
20,91
42,87
17,32
17,00
ng
ng
ng
6plex RT-PCR (Ct)
CHTR
omp
TRIVAG
ng
ng
ng
ng
29,16
25,53
19,99
25,53
22,20
21,78 *
ng
ng
21,54
30,62
26,43
16,22
ng
ng
17,11
18,43
16,00
19,64
21,65
19,32
ng
17,65
15,56
ng
ng
ng
Single RT-PCR (Ct)
TRIVAG
ng
ng
ng
ng
ng
ng
ng
ng
ng
ng
Zowel de sensitiviteit (22/22), specificiteit (45/45) als accuraatheid geven een
waarde van 100%, waarbij één zeer zwak CHTR positief staal (EQC 9) buiten beschouwing
gelaten wordt aangezien voor klinische stalen meestal een hogere titer geregistreerd
wordt. Op de EQC stalen, waarvan sommige N. gonorrhoeae positief, wordt ook nog een
Trichomonas single RT-PCR uitgevoerd (Tabel 4.11 – laatste kolom). Deze analyse
bevestigt dat het bekomen ‘TRIVAG’ signaal van de 6plex RT-PCR afkomstig is van de
porA target van N. gonorrhoeae en niet van T. vaginalis zelf.
4.3.1.3 Bepalen van de lineariteit
De lineariteit van de PCR wordt bepaald door analyse van amplificatieplots
afkomstig van aangemaakte verdunningsreeksen. Voor detectie van C. trachomatis en
39
N. gonorrhoeae wordt vertrokken van verdunningsreeksen van positieve staalextracten,
terwijl voor T. vaginalis een verdunningsreeks gebruikt wordt vanuit een positief staal
(TV 13) om ook de extractiestap te betrekken in de lineariteitsvalidatie. Een efficiëntie
van 80-120%, een R2 van minstens 0,99 en een weinig afwijkende Δ Ct waarde waarbij
vergeleken wordt met een referentiemethode zijn essentieel voor een voldoende sterke
lineariteit van de RT-PCR. Alle resultaten die buiten bovengenoemde intervallen liggen,
worden als te afwijkend beschouwd waardoor de methode niet in gebruik kan genomen
worden. Zowel de 5plex als 6plex PCR worden telkens in triplo geanalyseerd (Tabel 4.12).
Tabel 4.12: Overzicht van de lineariteitsanalyse: vergelijking met referentie 5plex PCR (‘/’ = geen waarde)
5plex
6plex
ΔCt
gem Ct
stdev
gem Ct
stdev
1x
21,85
0,05
21,88
0,06
0,03
0,1x
25,5
0,08
25,64
0,08
0,14
0,01x
29,04
0,12
29,22
0,08
0,18
0,001x
32,46
0,04
32,62
0,13
0,16
0,0001x
36,11
0,09
37,23
0,94
1,12
1x
20,65
0,18
20,8
0,02
0,15
0,1x
24,17
0,09
24,23
0,06
0,06
0,01x
27,65
0,2
27,7
0,12
0,05
0,001x
31,12
0,15
31,16
0,14
0,04
0,0001x
34,66
0,38
34,7
0,99
0,04
1x
20,66
0,08
20,18
0,03
-0,48
0,1x
24,03
0,04
23,52
0,01
-0,51
0,01x
27,33
0,09
26,95
0,07
-0,38
0,001x
30,78
0,24
30,17
0,18
-0,61
0,0001x
34,06
0,21
34
0,06
-0,06
1x
/
/
18,72
0,10
/
0,1x
/
/
22,29
0,22
/
0,01x
/
/
26,16
0,10
/
0,001x
/
/
29,97
0,35
/
0,0001x
/
/
33,93
0,96
/
CHTR
OMP
porA
TRIVAG
40
Wanneer de gemiddelden en de standaarddeviatie van de 5plex PCR methode
vergeleken worden met de gemiddelden en standaarddeviaties van de 6plex PCR, valt op
dat de verschillen minimaal zijn. Er
is
bijgevolg
geen
extensieve
afwijking van de Δ Ct waarde ten
opzichte van het nulpunt, waardoor
aan één van de drie criteria, zijnde
een weinig afwijkende Δ Ct, reeds
voldaan is. Grafisch omzetten van
deze Ct waarden in functie van een
bepaalde
concentratie
verdunningsreeks
in
de
resulteert
in
(aantal kopieën per mL)
volgende figuur (Figuur 4.4). De
(kopieën/mL)
grafieken worden met behulp van
de
analysesoftware
van
Life Figuur 4.4: Voorbeeld van lineaire regressie van de T.
vaginalis verdunningsreeks met ABI software
Technologies opgesteld.
De resultaten van deze lineaire regressiemodellen, meer bepaald de R2 en de
efficiëntie, worden in een samenvattende tabel (Tabel 4.13) geplaatst om een correcte
beoordeling van de lineariteit van de PCR methode te kunnen maken. Ook deze
resultaten vallen net als de Δ Ct waarden binnen de vooropgestelde limieten waardoor
besloten kan worden dat er in voldoende mate aan de lineariteit van de ontwikkelde
6plex PCR methode wordt voldaan.
Tabel 4.13: Samenvattende resultaten van de lineaire regressiemodellen
R2 en Eff% over een gebied van 1x tot 0,0001x
6plex
R
2
5plex
Eff%
R
2
Limiet
Eff%
CHTR
0,997
84,25
0,999
91,33
omp
1
94,1
1
93,43
porA
0,993
95,72
1
98,64
TRIVAG
0,999
83,03
/
/
R
2
> 0,990
Eff%
>80%
41
4.3.1.4 Bepalen van de intrarun precisie
Een sterk (10x verdund) en een zwak (10 000x verdund) positief staal dienen voor
de intrarun precisie in drievoud getest te worden binnen eenzelfde run. De verdunning
van de sterk positieve stalen tot zwak positieven gebeurt voor C. trachomatis en
N. gonorrhoeae na de extractieprocedure aangezien niet voldoende verse positieve
stalen voor handen waren. In het verleden toonde het validatiedossier van de 5plex RTPCR echter aan dat de extractieprocedure voldoende stabiel is, zodat deze handeling
gerechtvaardigd is. Voor T. vaginalis wordt wel gewerkt met verdunningen vanaf het
staal zelf (TV 15). Alle verdunningen werden vervolgens in drievoud geëxtraheerd. Er
wordt een intrarun precisie van maximaal 2 Ct getolereerd, die hier nooit overschreden
wordt (Tabel 4.14).
Tabel 4.14: De Ct waarden en het verschil op de waarden van staal TV15, *7703 en *7708 (Δ = grootste
waarde – kleinste waarde) voor de intrarun precisie.
T. vaginalis
1
2
3
Δ
0,1x
20,94
21,10
21,03
0,16
C. trachomatis
0,0001x
33,25
33,41
33,60
0,35
0,1x
21,92
21,92
21,80
0,12
0,0001x
32,77
32,63
32,45
0,31
N. gonorrhoeae omp
0,1x
20,80
20,82
20,80
0,04
0,0001x
31,07
31,36
31,06
0,30
4.3.1.5 Bepalen van de houdbaarheid en interrun precisie
In functie van de tijd worden een aantal stalen geanalyseerd op hun
houdbaarheid. Op verschillende gefixeerde meetpunten wordt dan RT-PCR uitgevoerd.
Eveneens wordt uit deze resultaten de interrun precisie bepaald (Tabel 4.15).
Tabel 4.15: Analyse van de houdbaarheid van positieve stalen. (‘KT’ = kamertemperatuur, ‘2-8°C’ = koelkast)
Staal
Eerste analyse (dag 0)
Tweede analyse (dag 4)
Derde analyse (dag 8)
Vierde analyse (dag 15)
Δ Ct
*1349
(CHTR)
KT (24,57)
KT (23,98)
KT (24,02)
KT (22,00)
2,57
*9233
(CHTR)
KT (24,51)
KT (24,14)
2-8°C (24,22)
2-8°C (23,97)
0,54
*7997
(TRIVAG)
KT (22,34)
2-8°C (/)
2-8°C (21,40)
2-8°C (21,45)
0,92
*9212
(TRIVAG)
KT (23,30)
KT (/)
KT (30,54)
KT (34,62)
11,32
*2214
(GONO omp)
KT (27,48)
2-8°C (/)
2-8°C (27,34)
2-8°C (23,35)
4,13
42
Voor staal *1349, *9233 en *7997 wordt de vooropgestelde limiet (Δ Ct van 3)
niet overschreden, zodanig dat aan de houdbaarheidsvoorschriften en de interrun
precisie voldaan wordt. Staal *9212 en *2214 voldoen duidelijk niet. De variatie op de
waarden is ook beduidend kleiner wanneer het staal in de koelkast bewaard wordt.
4.3.2 Theoretische validatie: kostenanalyse en terugbetalingstarieven
Het toevoegen van de primers en de probe voor T. vaginalis aan de 5plex RT-PCR
mix resulteert in slechts een minimale meerkost (€0,05). De grootste kost bij RT-PCR
blijkt, naast de extractieprocedure van €4,75 per staal, de 96 well plaat te zijn met €4,67
per plaat. Het uitvoeren van een multiplex PCR methode is daarom een financieel
interessante optie aangezien de plaat optimaal benut wordt doordat op meerdere
targets binnen dezelfde assay getest wordt. De totaalkost per staal wordt bekomen door
bij deze reagentiakost de personeelskost te voegen: €21,00 + €7,45 = €28,45.
Het RIZIV voorziet volgens artikel 24 een terugbetaling van respectievelijk €25,00;
€10,00 en €5,00 voor volgende bepalingen: ‘Chlamydia trachomatis opzoeken door
moleculaire amplificatie (550255, 550266)’,’Opsporen van Neisseria gonorrhoeae door
een techniek van moleculaire amplificatie (5509115, 550922)’ en ‘Parasieten, opzoeken
in andere dan faecesmonsters en dan bloed (550815, 550826)’.xl
4.4 METHODEVERGELIJKING
Een commercieel beschikbare antigen sneltest, de OSOM® Trichomonas Rapid Test,
wordt in de praktijk gebruikt om snel en eenvoudig een T. vaginalis infectie te kunnen
diagnosticeren. Het gebruik van een NAAT, meer bepaald de 6plex PCR, geeft echter veel
betere resultaten op vlak van gevoeligheid en specificiteit. Drie verdachte T. vaginalis
stalen (*5333, *7997 en *9212) werden microscopisch bevestigd, waarop zowel met de
6plex PCR als met de sneltest een analyse uitgevoerd wordt. Na RT-PCR worden Ct
waarden bekomen van respectievelijk 18,69 , 22,34 en 23,30. Alle drie de stalen gaven
eveneens een positief resultaat met de sneltest. Op basis van deze resultaten kan dus
geen onderscheid gemaakt worden inzake gevoeligheid tussen de twee methoden.
43
5. DISCUSSIE
De aanmaak van de positieve plasmide controles en de keuze van welke kolonies er
opgezuiverd dienen te worden steunen in eerste instantie vooral op de bekomen resultaten
van de uitgevoerde klassieke PCR-gelelektroforese testen. Interpretatie van deze resultaten
zijn niet altijd voor de hand liggend door het ontbreken van een numerieke waarde.
Eveneens is het zo dat de hoeveelheid geladen DNA ervoor kan zorgen dat de migratie
wordt beïnvloedt; meer bepaald het verder doormigreren van het bandje bij een grote
hoeveelheid aan DNA. In dat opzicht verloopt de interpretatie van de data bekomen met RTPCR een stuk vlotter.
Tijdens de optimalisatie wordt de T. vaginalis RT-PCR op punt gebracht en bij een reeds
bestaande 5plex PCR gevoegd, waarbij het belangrijk is de data output van deze finale 6plex
RT-PCR op een correcte manier te interpreteren. Doordat zowel het signaal van het porA
target van N. gonorrhoeae als het signaal van het T. vaginalis 2kb target in hetzelfde kanaal
terechtkomen, is het onmogelijk om te beslissen of het staal nu Trichomonas of
N. gonorrhoeae positief is. Daarom zal altijd een bevestigende T. vaginalis single RT-PCR
uitgevoerd moeten worden om te kunnen differentiëren. Een nieuw positief signaal
bevestigt dan onvermijdelijk een T. vaginalis positief staal, zoniet is er sprake van een
N. gonorrhoeae positief staal.
Deze dualiteit in detectie wordt mooi geïllustreerd aan de hand van de uitgevoerde
checkerboard analyse tussen T. vaginalis en N. gonorrhoeae (Tabel 4.7 ). Bij verdunningsstap
TC van T. vaginalis, gepaard met een aanwezigheid verdunningsstap GA van N. gonorrhoeae,
wordt een vertekende waarde van 18,19 gevonden. Deze waarde wordt verklaard aan de
hand van het feit dat ook de N. gonorrhoeae verdunning een T. vaginalis positief signaal
levert. De blanco kolom leert dat GA, omwille van het porA target, in afwezigheid van het
T. vaginalis target zelf verantwoordelijk is voor een T. vaginalis positief signaal met een Ct
van 18,30. Telkens wanneer de N. gonorrhoeae verdunningen een sterker T. vaginalis
signaal geven dan de T. vaginalis verdunningsreeks zelf, zal het uiteindelijke signaal
44
overstemd worden. Pas wanneer de Ct waarde van T. vaginalis onder de blanco waarde van
de N. gonorrhoeae verdunning ligt, zal deze reële waarde ook effectief te zien zijn.
In de specificiteitsvalidatie wordt onder andere een sequentieanalyse uitgevoerd op de
geamplificeerde producten van verschillende positieve stalen. De sequenties die initieel
bekomen worden met p506 zijn niet gerapporteerd omdat deze te veel gestoord zijn.
Waarschijnlijk worden door de aanwezigheid van repeated sequences in het genetisch
materiaal van het micro-organisme meerdere cycle sequencing producten gevormd. Op die
manier kunnen geen uniforme sequentie van de target oligonucleotides met bijbehorende
fluorescentie meer afgelezen worden, zodat chaotische sequenties op de data ouput te zien
zijn. Gebruik makende van p509 wordt deze storing enorm gereduceerd. Het percentage
overeenkomst met de T. vaginalis sequentie in de NCBI databank bedraagt nu meer dan
92%, wat de identificatie van de geamplificeerde producten als T. vaginalis PCR product in
grote mate bevestigt.
De lineariteit van de ontwikkelde RT-PCR kan gegarandeerd worden tot een met een 10
000x verdunning van het oorspronkelijk staal. Vanaf dan blijkt het T. vaginalis target niet
langer opgenomen te worden. Numeriek gezien betekent dit dat de bovengrens van detectie
rond een Ct waarde van 34 ligt. In een poging deze bovengrens naar boven te verplaatsen,
werd de samenstelling van de gebruikte PP mix opnieuw herbekeken. De in de optimalisatie
bekomen ‘300-100’ conditie werd even opzij geschoven en er werd opnieuw geëvalueerd
met 200nM T. vaginalis probe in plaats van 100nM. Deze nieuwe condities gaven echter
geen betere resultaten, waarop besloten werd om de oude condities te behouden.
Aangezien alle geteste T. vaginalis positieve stalen lage Ct waardes niet groter dan 25
opleveren, lijkt het dan ook slechts een kleine beperking van de ontwikkelde RT-PCR
methode te zijn.
Wanneer positieve stalen gedurende een gegeven tijdsperiode meerdere malen
geëvalueerd worden, bestaat de mogelijkheid dat er een variatie op de bekomen waarden
zit. Deze variatie wordt mooi geïllustreerd in de test op houdbaarheid voor C. trachomatis
en N. gonorrhoeae. Vooraleer de stalen geëxtraheerd werden (*1349, *9233 en *2214)
45
ondergingen ze een vortexstap. Nu is het mogelijk dat veel van het gewenste materiaal zich
in de wisser zelf bevindt, dat tijdens het vortexen in variabele mate vrijkomt. Op die manier
zou de grote variatie in detectie, namelijk een ΔCt van 2,57 en 4,13, verklaard kunnen
worden voor C. trachomatis en N. gonorrhoeae stalen. T. vaginalis stalen blijken, wanneer
bewaard op kamertemperatuur, erg onderhevig te zijn aan degradatie. Deze degradatie,
duidelijk merkbaar in de test op houdbaarheid, dient in de toekomst zeker nog verder
geanalyseerd te worden. Het verhogen van de testfrequentie van een staal kan een voor de
hand liggende oplossing zijn. Besluitend kan wel geconcludeerd worden dat wanneer de
stalen bewaard worden op kamertemperatuur, de degradatie significant kleiner is.
Een aantal routinestalen worden beschikbaar gesteld waarop de gevalideerde 6plex RTPCR nog getest kan worden. Met behulp van deze testen kunnen extra inzichten verworven
worden over de performantie en karakteristieken van de ontwikkelde RT-PCR methode. Een
cervicale en vaginale swab, beiden positieve T. vaginalis routinestalen afkomstig van
dezelfde patiënt, worden ook met de 6plex RT-PCR geëvalueerd om de rol van het soort
staal op de detectie te bepalen. Resultaten tonen dat de manier van staalname geen grote
invloed heeft op de detectie van T. vaginalis, aangezien de Ct waarden van de twee
bepalingen met respectievelijk 21,23 en 23,46 niet ver uiteen liggen. Een N. gonorrhoeae
positief routinestaal wordt ook positief bevonden met de 6plex RT-PCR (Ct omp 27,48 en Ct
TRIVAG 25,41). Omwille van de dubbele detectie in het ‘TRIVAG’ kanaal, dient de
Trichomonas single RT-PCR aangewend te worden zoals vroeger beschreven. In deze RT-PCR
wordt geen significante amplificatie van een target teruggevonden, wat erop duid dat het
bekomen ‘TRIVAG’ signaal afkomstig is van het porA target van N. gonorrhoeae.
De OSOM® Trichomonas sneltest en de ontwikkelde 6plex RT-PCR blijken even goed te
scoren op vlak van sensitiviteit. Drie positieve T. vaginalis stalen gaven drie positieve
signalen bij beide methodes. De hoeveelheid aan beschikbare stalen, met name 3
gedurende een aantal maanden, is echter zeer gering zodat dit resultaat hoogst
waarschijnlijk vertekend is. Analyse van meerdere positieve stalen zou een oplossing kunnen
zijn om de verwachte hogere gevoeligheid van de RT-PCR methode op de voorgrond te doen
treden.
46
6. CONCLUSIE
Om T. vaginalis infecties zo accuraat mogelijk op te kunnen sporen beschrijft deze thesis de
ontwikkeling van een nieuwe multiplex RT-PCR methode. Deze NAAT zorgt ervoor dat een
verdacht staal naast detectie van T. vaginalis, ook geëvalueerd kan worden op de aanwezigheid
van C. trachomatis en N. gonorrhoeae. Op die manier wordt een grote stap vooruit gezet in de
diagnostiek van de T. vaginalis parasiet en SOA’s in het algemeen.
Met behulp van verdunningsreeksen van aangemaakte positieve plasmidecontroles wordt
een T. vaginalis RT-PCR geoptimaliseerd. Condities waarbij in de aangewende PP mix 300nM
T. vaginalis primers en 100nM T. vaginalis probe aanwezig zijn blijken de meest belovende,
waardoor met deze concentraties ook verder gewerkt wordt. De T. vaginalis RT-PCR wordt bij
een reeds bestaande RT-PCR gevoegd die screent op de aanwezigheid van C. trachomatis en
N. gonorrhoeae. Vergelijkingsstudies tussen deze nieuwe 6plex RT-PCR en reeds in gebruik
genomen referentiemethoden kunnen geen opmerkbare verschillen aantonen, zodat de
ontwikkelde methode verder getest en aan een grondige validatie onderworpen wordt.
De 6plex RT-PCR ondergaat een specificiteitsvalidatie in drievoud, waarbij sequencing
aantoonde dat de geamplificeerde producten wel degelijk het gewenste Trichomonas target
betreffen. Een test op andere genitale organismen en een PCR-gelelektroforese van positieve
T. vaginalis stalen leerde dat de methode effectief specifiek is voor het bewuste organisme. De
lineariteit van de methode wordt bevestigd over een gebied van 1x tot 0,0001x verdunning, met
een gerapporteerde R2 en efficiëntie die groter is dan de beoogde 0,99 en 80%. De precisie en
de houdbaarheid voldoen, met uitzondering van twee stalen, aan de vooropgestelde limiet die
een afwijking van maximaal 3 Ct waarden als grens stelt. De analytische accuraatheid van de
ontwikkelde RT-PCR wordt bepaald aan de hand van 67 geanalyseerde stalen en bedraagt 100%.
De methodevergelijking met de antigen sneltest levert geen merkbare verschillen op tussen
de twee methoden. Drie positieve T. vaginalis stalen, bevestigd via RMO, zorgen zowel met de
RT-PCR als met de sneltest voor een positief signaal. Na succesvol afronden van de validatie van
de ontwikkelde 6plex RT-PCR wordt de methode opgenomen in de routine PCR analyses.
47
7. LITERATUURLIJST
i
Anderson MR, Klink K, Cohrssen A. Evaluation of vaginal complaints. JAMA 2004; 291:1368.
ii
Jane R. Schwebke, Donald Burgess. Trichomoniasis. Clin Microbiol Rev. 2004 Oct; 17(4): 794–
803.
iii
Jack D Sobel, MD. Trichomoniasis. Up To Date. Sep 2014.
iv
World Health Organization (WHO). Department of Reproductive Health and Research.
WHO, 2012. Global incidence and prevalence of selected curable sexually transmitted infections
– 2008.
v
Centers
for
Disease
Control
and
http://www.cdc.gov/dpdx/trichomoniasis/lifecycle. (13/02/2015).
Prevention:
CDC.
vi
Grodstein F, Goldman MB, Cramer DW. Relation of tubal infertility to history of sexually
transmitted diseases. Am J Epdemiol 1993; 137:577.
vii
Zhang ZF, Begg CB. Is Trichomonas vaginalis a cause of cervical neoplasia? Results from a
combined analysis of 24 studies. Int J Epidemiol 1994; 23:682.
viii
McClelland RS, Sangare L, Hassan WM, et al. Infection with Trichomonas vaginalis increases
the risk of HIV-1 acquisition. J Infect Dis 2007; 195:698.
ix
Silver BJ, Guy RJ, Kaldor JM, et al. Trichomonas vaginalis as a cause of perinatal morbidity: a
systematic review and meta-analysis. Sex Transm Dis 2014; 41:369.
x
Workowski KA, Berman S. Centers for Disease Control and Prevention: CDC. Sexually
transmitted diseases treatment guidelines, 2010. MMWR Recomm Rep 2010; 59:1.
xi
Hobbs, M.M. and A.C. Seña. Modern diagnosis of Trichomonas vaginalis infection. Sex Transm
Infect, 2013; 89:434-8
xii
T Crucitti, E Van Dyck, A Tehe, S Abdellati, B Vuylsteke, A Buve, and M Laga. Comparison of
culture and different PCR assays for detection of Trichomonas vaginalis in self collected vaginal
swab specimens. Sex Transm Infect. 2003 Oct; 79(5): 393–398.
xiii
Huppert, J.S., et al. Use of an immunochromatographic assay for rapid detection of
Trichomonas vaginalis in vaginal specimens. J Clin Microbiol, 2005. 43(2): p. 684-7.
xiv
L. Campbell, V. Woods, T. Lloyd, S. Elsayed and D. L. Church. Evaluation of the
OSOM Trichomonas Rapid Test versus Wet Preparation Examination for Detection
48
of Trichomonas vaginalis. Vaginitis in Specimens from Women with a Low Prevalence of
Infection. J Clin Microbiol. 2008 Oct; 46(10): 3467–3469.
xv
Bijsluiter OSOM® Trichomonas Rapid Test (Sekisui Diagnostics, Tokio, Japan).
xvi
Coleman, J.S., C.A. Gaydos, and F. Witter. Trichomonas vaginalis vaginitis in obstetrics and
gynecology practice: new concepts and controversies. Obstet Gynecol Surv, 2013; 68(1) p. 4350.
xvii
Jeanne Marrazzo, MD, MPH. Clinical manifestations and diagnosis of Chlamydia trachomatis
infections. Up To Date. Jul 2014.
xviii
Peter A Leone, MD. Epidemiology and pathogenesis of Neisseria gonorrhoeae infection. Up
To Date. Jun 2014.
xix
Boom R, Sol CJ, Salimans MM, Jansen CL, Wertheim-van Dillen PM, van der Noordaa J. Rapid
and simple method for purification of nucleic acids. J Clin Microbiol. 1990;28(3):495-503.
xx
History of PCR. http://www.scienceisart.com/A_PCR/PCRhistory_2.html (8/04/2015)
xxi
PCR Method. Environmental Information.
http://enfo.agt.bme.hu/drupal/en/node/8912 (8/04/2015)
xxii
DNA structure. Bio-Physics wiki.
http://www.bio-physics.at/wiki/index.php?title=DNA_Structure (8/04/2015)
xxiii
Real-time PCR: The TaqMan method (Davidson college)
http://www.bio.davidson.edu/Courses/Molbio/MolStudents/spring2003/Pierce/realtimepcr.ht
m (23/02/2015)
xxiv
Mahony JB, Luinstra KE, Tyndall M, Sellors JW, Krepel J, Chernesky M. Multiplex PCR for
detection of Chlamydia trachomatis and Neisseria gonorrhoeae in Genitourinary specimens. J
Clin Microbiol. 1995 Nov;33(11):3049-53.
xxv
7500 dyes. 7500 and 7500 Fast RT-PCR Systems support, Life Technologies.
http://www.lifetechnologies.com/content/dam/LifeTech/global/technicalsupport/images/7500-Dye.jpg (08/04/2015)
xxvi
Source Molecular. Real-time PCR quantification.
http://www.sourcemolecular.com/microbial-source-tracking/quantification.html.(24/02/2015).
49
xxvii
Schirm J, Bos PA, Roozeboom-Roelfsema IK, Luijt DS, Möller LV. Trichomonas vaginalis
detection using real-time TaqMan PCR. J Microbiol Methods. 2007 Feb;68(2):243-7. Epub 2006
Sep 26.
xxviii
W G Weisburg, S M Barns, D A Pelletier, and D J Lane. 16S ribosomal DNA amplification for
phylogenetic study. J Bacteriol. 1991 Jan; 173(2): 697–703.
xxix
Hermann B. Update on the new variant of Chlamydia trachomatis: prevalence and
diagnostics. Eurosurveillance 2008; Volume 13, issue 26.
xxx
Koebnik R, Locher KP, Van Gelder P. Structure and function of bacterial outer membrane
proteins: barrels in a nutshell. Mol Microbiol. Jul 2000;37(2):239-53.
xxxi
Genetic Home References.
http://ghr.nlm.nih.gov/gene/POR. (13/03/2015)
How do we sequence DNA? The University of Michigan DNA Sequencing Core.
http://seqcore.brcf.med.umich.edu/doc/educ/dnapr/sequencing.html. (13/03/2015)
xxxii
xxxiii
Vankeerberghen Anne. Werkinstructies OLV Ziekenhuis Aalst: NucliSens easyMAG
(WI.MB.5). Sep 2014.
xxxiv
QIAquick® Gel Extraction kit Manual: Gelextraction protocol.
xxxv
TOPO® TA Cloning® Kit for Sequencing: User Guide
xxxvi
Wizard® Plus SV Minipreps DNA Purification System Technical Bulletin: Complete Protocol.
xxxvii
Vankeerberghen Anne. Werkinstructies OLV Ziekenhuis Aalst:ABI 7500 Fast Real-Time PCR
System (WI.MB.12). Jan 2015
xxxviii
Vaeyens
Freya.
Werkinstructies
elektroforesetoestel.(WI.MB.15). Apr 2013
OLV
Ziekenhuis
Aalst:
Mupid-One
xxxix
Vankeerberghen Anne. Werkinstructies OLV Ziekenhuis Aalst: CEQ 8000 Genetic Analytic
System (WI.MB.1). Aug 2011
xl
NomenSoft RIZIV: Nomenclatuur van de geneeskundige verstrekkingen: Hoofdstuk V. Speciale
technische geneeskundige verstrekkingen - Afdeling 11. Klinische biologie - Artikel. 24: § 1.
Worden beschouwd als verstrekkingen waarvoor de bekwaming van geneesheer, specialist voor
klinische biologie (P), vereist is - 5/ MICROBIOLOGIE (2010)
50
LECTURES UGENT
1. Research on the causes of human cancer and scientific strategies for cancer prevention and
control (Dr. Inge Huybrechts)
In general terms, cancer can’t be referred to as just one type of disease. In fact, a lot of
different cancers have been identified, all having their specific characteristics. Cancer can be
defined as an uncontrollable cell proliferation, caused by external events such as radiation or
chemical exposure or genetic errors such as natural mutations. Eventually, the growing tumor
can invade surrounding tissues or even worse, spread throughout the body using the
bloodstream or lymphatic system. It is the leading cause of death worldwide, even
outnumbering the death counts caused by malaria, TBV and HIV/AIDS combined. Lung, prostate
and colorectal cancers are listed as the most common cancers in men, whereas breast,
colorectum, cervix uterus and lung cancer are most common in women.
When the major cancer risk factors are analysed, it is important to make a difference
between genetic pre-disposition and environmental exposure. It is known that risk factors as
‘tobacco use’ and ‘obesity ‘are responsible for over 50% of the deaths from cancer. Data have
also shown that there is a higher risk of getting cancer in less developed countries. In order to
obtain and maintain awareness of the problem that is cancer, several organizations, even from
governmental point of view, have been founded.
The International Agency for Research on Cancer (IARC) is as research affiliate part of the
World Health Organization (WHO) and it investigates mainly the causes of cancer. They also
administer monographs, the IARC Monographs, in which is stated how strong the scientific
evidence is for a cancer association. It is commonly referred as the encyclopaedia of carcinogens.
All studies and summaries on that specific agent or topic are critically reviewed by working group
members, specialists, representatives of (inter)national health agencies and observers. In order
to reduce the lifetime risk of developing cancer significantly, scientific strategies for cancer
prevention and control (e.g. NME cohorts) should be considered as very helpful. Maintaining a
healthy weight, implementing a healthy diet and having enough exercise therefore are the
primary goals in order to acquire that aforementioned objective.
2. The evolution of microbiology in cystic fibrosis (Prof. John LiPuma)
Cystic fibrosis (CF) is caused by an autosomal mutation in both copies of the gene that is
responsible for the production of a protein that acts as a regulator in transmembrane transport.
The lungs are typically most affected. Common symptoms are difficulty in breathing and an
excessive sputum production, besides poor growth, male infertility and clubbing of the fingers.
Currently, there is no cure for CF. The often established lung infections can be treated with
antibiotics, however the microbiology in cystic fibrosis is more complex than a normal acquired
infection. The evolution of microbiology in CF can be summarized in three major eras.
1
In the pre-Pseudomonas era, studies showed an association between CF and
Staphylococcus aureus. In addition, a lot of antibiotics were discovered and developed but also
overused in order to treat the CF patients with lung infections. However, since the life
expectancy of patients with CF was very poor in history, science managed to create a ‘new
species’: adolescents and adults with CF. Later, a more damaging bacterium was found
correlated to CF, starting a new era: the Pseudomonas aeruginosa era. As a matter of fact, P.
aeruginosa as such is not that good in invading the lung. However the damaged lungs of a CF
patient can easily be invaded. Microbiologists identified a mucoid variant, which is responsible
for a much shorter life expectancy in comparison with other pathogens. Also an inter-spread of
virulent epidemical clones is discovered, so susceptibility can be questioned.
In the Pseudomonas 2.0 era, scientists try to take a closer look at bacterial communities
in the lung. Because of the interbacterial quorum sensing, the virulence sometimes is
unpredictable. Also, within a species there is a possibility of difference in phenotype, resulting in
even more diversity. Today, research focusses not only on the structure of the bacterial
communities, also the activity of the communities are evaluated during the several stages of
sickness of the patient.
3. Zijn managementvaardigheden in farmaceutische zorg belangrijk voor de toekomst? Een kijk
vanuit NederBelgisch perspectief (Apr. Hendrik de Rocker)
De laatste jaren is het verstrekken van farmaceutische zorg een belangrijke aanvulling
geworden op het takenpakket van de huidige officina-apotheker. Deze evolutie uit zich op
meerdere vlakken. Naast een shift in focus van geneesmiddel naar patiënt, moet de apotheker
ook rekening houden met de financiële draagkracht van de maatschappij voor de
gezondheidszorg. In essentie betekent dit preventie bieden waar mogelijk, zodat behandelen
vermeden kan worden. Vanuit Nederland blijkt dat samenwerking tussen collega-apothekers en
een geïntegreerde visie eveneens een gunstige invloed teweeg kan brengen. Deze zogenaamde
geïntegreerde farmaceutische zorg (GFZ) zou verkeerd medicijngebruik moeten voorkomen, de
effectiviteit moeten verhogen en de medicijnkosten moeten verlagen.
Managing van de zorg, en meer in detail managing van de medicatie, maakt het mogelijk
een standaard werkwijze toe te passen waarin de medicatie voor elke patiënt als geschikt,
effectief en veilig beschouwd kan worden. Van medicatiemanagement is het slechts een kleine
stap naar farmaceutische patiëntenzorg (FPZ). Hier wordt gestreefd naar het optimaal
geneesmiddelengebruik van de individuele patiënt, met als doel de kwaliteit van het leven te
verbeteren. De bovenvermelde GFZ is hier een praktisch voorbeeld van. Besluitend kan gezegd
worden dat de apotheker, naast zijn verantwoordelijkheid als verstrekker van farmaceutische
zorg, zich ook een functie moet aanmeten in het managen en beperken van de ontwikkeling van
chronische ziekten op de populatie. De apotheker is de enige, geschikte persoon die dit veelvoud
aan taken op een optimale manier kan uitvoeren.
2
3
4
5
6