Een verhaaltje over GIST en evolutie
Van de gist Saccharomyces cerevisae, ofwel bakkersgist, zijn de genen zo te verschuiven dat
een nieuwe variant ontstaat.Die lijkt precies op een nauwverwante gist, genaamd
Saccharomyces mikatae.
Gist
Saccharomyces cerevisiae (wijngist, bakkersgist, biergist)
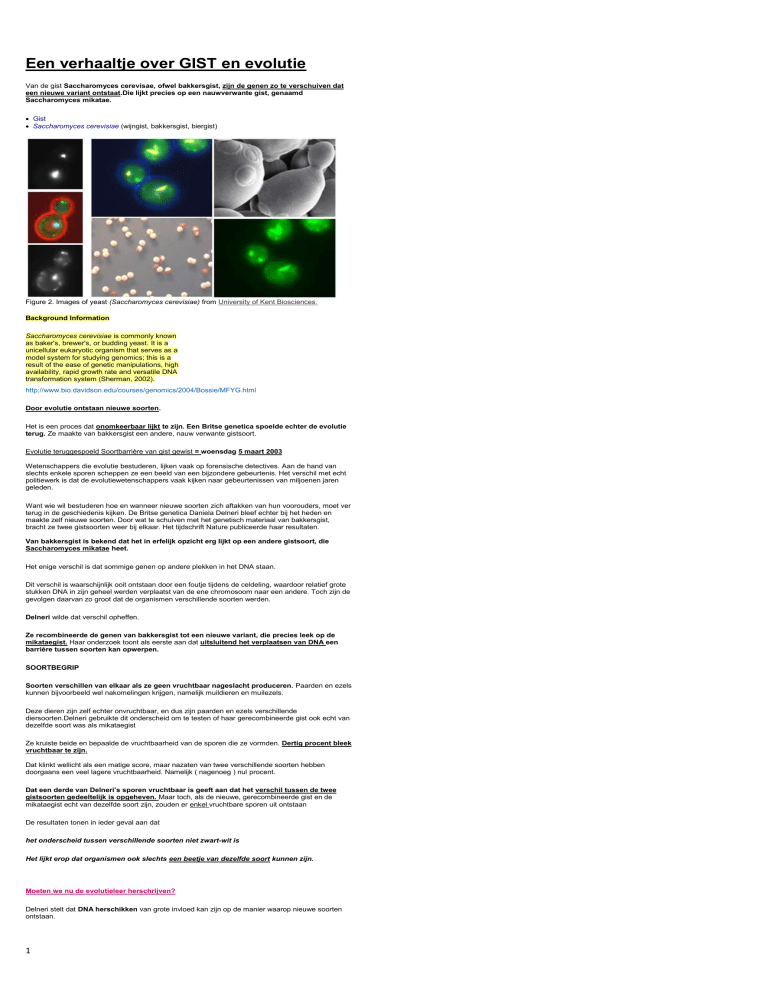
Figure 2. Images of yeast (Saccharomyces cerevisiae) from University of Kent Biosciences.
Background Information
Saccharomyces cerevisiae is commonly known
as baker's, brewer's, or budding yeast. It is a
unicellular eukaryotic organism that serves as a
model system for studying genomics; this is a
result of the ease of genetic manipulations, high
availability, rapid growth rate and versatile DNA
transformation system (Sherman, 2002).
http://www.bio.davidson.edu/courses/genomics/2004/Bossie/MFYG.html
Door evolutie ontstaan nieuwe soorten.
Het is een proces dat onomkeerbaar lijkt te zijn. Een Britse genetica spoelde echter de evolutie
terug. Ze maakte van bakkersgist een andere, nauw verwante gistsoort.
Evolutie teruggespoeld Soortbarrière van gist gewist = woensdag 5 maart 2003
Wetenschappers die evolutie bestuderen, lijken vaak op forensische detectives. Aan de hand van
slechts enkele sporen scheppen ze een beeld van een bijzondere gebeurtenis. Het verschil met echt
politiewerk is dat de evolutiewetenschappers vaak kijken naar gebeurtenissen van miljoenen jaren
geleden.
Want wie wil bestuderen hoe en wanneer nieuwe soorten zich aftakken van hun voorouders, moet ver
terug in de geschiedenis kijken. De Britse genetica Daniela Delneri bleef echter bij het heden en
maakte zelf nieuwe soorten. Door wat te schuiven met het genetisch materiaal van bakkersgist,
bracht ze twee gistsoorten weer bij elkaar. Het tijdschrift Nature publiceerde haar resultaten.
Van bakkersgist is bekend dat het in erfelijk opzicht erg lijkt op een andere gistsoort, die
Saccharomyces mikatae heet.
Het enige verschil is dat sommige genen op andere plekken in het DNA staan.
Dit verschil is waarschijnlijk ooit ontstaan door een foutje tijdens de celdeling, waardoor relatief grote
stukken DNA in zijn geheel werden verplaatst van de ene chromosoom naar een andere. Toch zijn de
gevolgen daarvan zo groot dat de organismen verschillende soorten werden.
Delneri wilde dat verschil opheffen.
Ze recombineerde de genen van bakkersgist tot een nieuwe variant, die precies leek op de
mikataegist. Haar onderzoek toont als eerste aan dat uitsluitend het verplaatsen van DNA een
barrière tussen soorten kan opwerpen.
SOORTBEGRIP
Soorten verschillen van elkaar als ze geen vruchtbaar nageslacht produceren. Paarden en ezels
kunnen bijvoorbeeld wel nakomelingen krijgen, namelijk muildieren en muilezels.
Deze dieren zijn zelf echter onvruchtbaar, en dus zijn paarden en ezels verschillende
diersoorten.Delneri gebruikte dit onderscheid om te testen of haar gerecombineerde gist ook echt van
dezelfde soort was als mikataegist
Ze kruiste beide en bepaalde de vruchtbaarheid van de sporen die ze vormden. Dertig procent bleek
vruchtbaar te zijn.
Dat klinkt wellicht als een matige score, maar nazaten van twee verschillende soorten hebben
doorgaans een veel lagere vruchtbaarheid. Namelijk ( nagenoeg ) nul procent.
Dat een derde van Delneri’s sporen vruchtbaar is geeft aan dat het verschil tussen de twee
gistsoorten gedeeltelijk is opgeheven. Maar toch, als de nieuwe, gerecombineerde gist en de
mikataegist echt van dezelfde soort zijn, zouden er enkel vruchtbare sporen uit ontstaan
De resultaten tonen in ieder geval aan dat
het onderscheid tussen verschillende soorten niet zwart-wit is
Het lijkt erop dat organismen ook slechts een beetje van dezelfde soort kunnen zijn.
Moeten we nu de evolutieleer herschrijven?
Delneri stelt dat DNA herschikken van grote invloed kan zijn op de manier waarop nieuwe soorten
ontstaan.
1
Haar collega Ken Wolfe waarschuwt echter dat we niet al teveel waarde moeten hechten aan de
recombinatie. Het werpt slechts een barrière op tussen verschillende soorten, zo stelt hij in een
commentaar in dezelfde editie van Nature. Herschikken alleen is niet genoeg om nieuwe soorten
te vormen.
Als voorbeeld geeft Wolfe aan dat sommige soorten slechts één gen van elkaar verschillen,
zonder dat daar ooit DNA is herschikt.
Niettemin geven de resultaten van Delneri een nieuw raamwerk voor andere onderzoekers die
soortvorming bestuderen, zo schrijft ze zelf. En wellicht ontstaat er dan ook meer duidelijkheid over de
evolutie van de mens.
Veel van de zesduizend genen in gist hebben overeenkomsten met menselijk DNA.
Aschwin Tenfelde
D. Delneri et al.: Engineering evolution to study speciation in yeasts. In: Nature, vol.422, p.68,
(6 maart 2003)
-àK. Wolfe: Speciation reversal. In: Nature, vol. 422, p.25, (6 maart 2003)
"genome duplication "
VERDUBBELEN IS DE SLEUTEL
De genetische samenstelling van vele gistsoorten wordt gedetailleerd onder de loep genomen. Dat
levert nuttige inzichten op in de evolutie van het leven.
André Goffeau van de Université Catholique de Louvain stelt in Nature dat het er steeds meer naar
uitziet dat
de verdubbeling van genen of langere stukken van de chromosomen - of zelfs van volledige
genensets - een sleutelmechanisme in de evolutie is.
http://www.sciencedaily.com/releases/2004/03/040308071448.htm
http://www.biomedcentral.com/news/20040701/01
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?cmd=Retrieve&db=PubMed&list_uids=9192896&dopt=
Abstract&holding=f1000
http://www.nature.com/nature/links/040701/040701-2.html
Achtergrond wilde gist soorten
http://www.cara-online.com/wildyeast.htm
BIERGIST
GENOOM VERDUBBELING
(gist historie II )
How yeasts evolve
Author: Cathy Holding
[email protected]
[2 July 2004]
http://genomebiology.com/researchnews/default.asp?arx_id=gb-spotlight-20040702-01.
De publikatie van vier nieuw ontrafelde genoom sequences van vier vershillende gistsoorten
ondersteunt verder de " genoom verdubbeling " theorie ....
De publikatie van vier nieuwe genome sequences van gist lijkt het controversieele idee ____( in 1997
gelanceerd ) dat volledige genoom verdubbeling ergens heeft plaatsgevonden in de evolutionaire lijn
van Saccharomyces cerevisiae (Nature 2004, 430:35-44)____ te bevestigen
De erg goede genoom- representatieve " steekproef " sequencies ___ vrij van belangrijke
hiaten ___ van de vier gist- species
illustreren de modellen voor respektievelijk
--->tandem gen-duplikatie
--->segmentele duplikatie ( co-duplicatie van tientallen tot en met honderden genen )
--->volledige genome duplikatie
zei co-auteur Giles Fischer, die samen met Bernard Dujon et al , het onderzoek deed aan het CNRS (
Centre Nationale de la Recherche Scientifique ( France.) )
" Een van de belangrijkste motoren / mechanismen die de evolutie aandrijven zijn genetische
verdubbeling , gevolgd door genetisch verlies ...
en .... We vonden niet slechts één enkel zulk mechanisme , we vonden er drie "
"Gedurende de laatste 2 of 3 jaar , zijn ongeveer 10 nieuwe dergelijke gist sequences afgewerkt
en gedecodeerd ,en de huidige onderlinge vergelijkingen ervan wijst duidelijk op de huidige
connclusies " zei André Goffeau, professor van het " Institut des Sciences Vie, Universié
Catholique de Louvain, Belgie , die het begeleidende " News and Views article." schreef in datzelfde
nummer van nature
" Het kompleet genoom duplicatie , mechanisme ___ werd voor het eerst voorsgesteld door
Ken Wolfe, professor "the department of Genetics, Trinity College Dublin - "is nu buiten elke
redelijke twijfel ondubbelzinnig aangetoond " zei Goffeau nog .
"Dujon was één van de grote tegenstanders van mijn hypothesis ",zei Wolfe, die wel geen deel
nam aan de studie
(Ken Wolfe http://www.tcd.ie/Genetics/staff/Ken_Wolfe.html)
De franse onderzoekers deden in 2000 een beperkte "sequensing " van 13 gistsoorten en
trokken daaruit een aantal voorbarige konklusies die geen
ondersteuning voor mijn genoom-duplikatie idee , betekenden : Het was zelfs geen " kladje " =
het betrof slechts een steekproef die voor 0.2 van elk
genoom statistisch geldig was ... Ze zijn nu kompleet van gedachten veranderd __ dat is
natuurlijk erg bevredigend voor mij ... "
"Ik dentk dat al deze verschillende mechanismen die we aan het werk zien in die groep van
species waarschijnlijk behouden zijn gebleven in de meeste biota "
indien al niet overal als universeel gelden ... "
"Noteer ook dat gist-soorten onderling meer met elkaar verschillen dan de mens en zee invertebrata , vissen , vogels en zoogdieren
De vier gist genomen in de studie , zijn afkomstig van
Candida glabrata, een menselijk pathogeen;
http://www.doctorfungus.org/thefungi/Candida_glabrata.htm
2
http://www.ukneqasmicro.org.uk/cglab.htm
Kluyveromyces lactis, een veel gebruikt genetisch laboratorium proefkonijn
http://supfam.mrc-lmb.cam.ac.uk/SUPERFAMILY/cgi-bin/gen_list.cgi?genome=kl
A mutant Kluyveromyces lactis strain
that arrests late in the cell cycle. K.
lactis is a budding yeast, meaning that it
makes new cells by building a small
"bud" on one side of the cell, which
grows until it is nearly the same size as
the original cell and then cleaves off.
Normally, a population of cells would
contain cells with buds of many different
sizes. However, since this mutant is
blocked before cell division, the cells
get to the large budded
http://newton.uor.edu/FacultyFolder/Silveira/Silveira.html
Debaryomyces hansenii, een zout-intolerante gist
http://cbi.labri.fr/Genolevures/elt/DEHA
Yarrowia lipolytica, een methaan-gebruikende gistsoort .
http://cbi.labri.fr/Genolevures/elt/YALI
http://imb.usal.es/ingles/main/research.htm
"de moleculaire vershillen , zoals ze kunnen worden gemeten door de percentages te bekijken
die de identiteit van de homologe eiwitten uitmaken , tussen deze verschillende gisten zijn erg
groot " zei Fischer nog
" Men kan de fijnere details van de evolutionaire trajekten ( op het genoom-organisatieniveau )
aflezen aan de verschillende genomen binnen hetzelfde
phylum " zei Anthony Poole, ( Department of Molecular Biology and Functional Genomics /Stockholm
University. )
"we hebben allen de neiging datgene wat we opperclakkig zien als onze standaard-waarden te
gebruiken ,als we een mens en een vis oppervlakkig bekijken
denken we meteen dat ze erg verschillen , terwijl gisten er voor ons allemaal gelijk uitzien
...Dat is ook de reden ook waarom er nog meer gedetailleerde
studies nodig zijn ... "
Poole, eveneens niet betrokken bij de studie , zei dat vroegere analyses die gebruik maakten van
kleinere data-sets steeds weer eindigden met meer en meer
generalisaties
" Als je onderlinge vergelijkingen kunt maken met deze nieuwere gegevens , dan zullen er
onvermijdelijk meer interessante
vraagstukken/vraagstellingen opduiken "
LINKS
http://nitro.biosci.arizona.edu/courses/EEB600A-2003/lectures/lecture25/lecture25.html
Over genen verdubbelingen
http://www.vib.be/nl/nieuws/Pages/DNA-verdubbelingen-blijken-essentieel-geweest-voor-evolutievissen-en-mensen.aspx
3
Verdubbeling van DNA als drijvende kracht achter de evolutie van planten
http://www.vib.be/nl/biotech-basics/Pages/Genomics-bij-planten.aspx
http://www.vib.be/nl/Pages/default.aspx
Biergist
http://www.biw.kuleuven.be/LFoRCe/Press%20releases/Verstrepen%20evolutie%20bier.pdf
http://www.consumed.nl/artikelen/112/Evolutie_mogelijk_door_kopieren_van_genen
Voeding en genen
donderdag 13 september 2007 /De standaard
Wie vlot zetmeel verteert, haalt meer calorieën uit z'n brood.
Photo News
(c) Layla Aerts
Het erfgoed van mensen is aangepast aan het plaatselijke voedingspatroon.
Miljoenen subtiele variaties in het erfelijk materiaal (DNA) van mensen bepalen waarom we er
allemaal anders uitzien (behalve eeneiige tweelingen)
en waarom sommige mensen vatbaarder zijn voor ziekten dan andere personen.
Maar er zijn ook genetische variaties tussen bevolkingsgroepen die de leden optimaal aangepast
maken aan het beschikbare voedsel of het traditionele
voedingspatroon van de groep, hebben wetenschappers ontdekt.
Met name het zetmeelgehalte van het dieet blijkt uitzonderlijk sterk weerspiegeld in de genen, melden
George Perry van de Arizona State University en
zijn collega's in een vervroegde publicatie van het vakblad Nature Genetics. ( zie hierbo ven (1) )
Daardoor verraden die genen misschien iets meer over de levenswijze van de eerste mensen.
Volkeren die vooral leven van de landbouw of van jacht en gevonden voedsel uit de natuur zoals
wortels en knollen, eten relatief veel zetmeel.
Bewoners van regenwouden en polaire gebieden hebben meestal veel minder zetmeel in hun dieet
en verhoudingsgewijs meer eiwit (van vlees) en suiker
(van fruit, honing en melk).
Dat verschil in voedingsgewoonte blijkt weerspiegeld in de productie van het speekseleiwit amylase,
dat het zetmeel helpt verteren in de mond, de maag en de darmen. Het amylase is namelijk niet bij
alle mensen even overvloedig aanwezig.
Uit de recente genetische analyses blijkt dat sommige mensen in hun erfgoed meer dan tien
exemplaren hebben van het gen (de erfelijke instructie in het DNA)
dat verantwoordelijk is voor de aanmaak van het amylase.
Andere hebben maar twee of meer exemplaren van het gen, en daardoor ook een lagere
amylaseproductie in het speeksel.
Het amylase-gen blijkt veelvuldiger aanwezig in een bevolkingsgroep met een zetmeelrijk dieet
(Japanners) dan in een volk met een zetmeelarm dieet
(de Yakut, een visetend volk uit het oosten van Rusland), stelden de wetenschappers vast.
De variatie blijkt ongewoon sterk vergeleken met andere genetische variaties tussen mensen, en lijkt
een interessant gevolg van natuurlijke selectie.
Wie effici챘nt zetmeel kan verteren, haalt immers meer calorie챘n uit zijn voedsel.
Dat kan een levensreddend voordeel zijn, bijvoorbeeld bij een zware epidemie van diarree.
In volkeren die weinig zetmeel eten, hebben mensen met een verhoogd aantal amylase-genen weinig
voordeel, en is er dus geen selectiedruk die het
genen-aantal in de populatie opdrijft.
Het verhoogde aantal amylase-genen is (nog) niet vastgesteld bij chimpansees, nauwe
verwanten van de mens.
De structuur van de menselijke amylase-genen onderling blijkt bovendien relatief weinig diversiteit te
vertonen, wat erop kan wijzen dat het
verhoogde aantal amylase-genen een kenmerk is dat pas in de jongste 200.000 jaar verworven is.
Die evolutie was mogelijk een gevolg van een veranderend eetpatroon van de vroege mensachtigen,
bijvoorbeeld omdat ze van leefgebied veranderden.
Verder onderzoek zou meer details kunnen blootleggen over het precieze tijdstip en de
omstandigheden van die overgang.
Wat hebben het speeksel van mensen , bakkersgist en parende fruitvliegjes gemeenschappelijk ?
(vertaling van een blogartikel van Carl Zimmer
bron :
http://scienceblogs.com/loom/2007/09/10/todays_odd_bedfellows_spit_yea.php#more )
Mensenspeeksel, bakkersgist en sex bij fruitvliegjes , illustreren allemaal een manier waarop nieuwe
soorten van genen evolueren.
( toename van genkapitaal door verdubbelingen )
Wetenschappers publiceerden in Nature Genetics (9 september 2007 on line) een document (1)
waarin zij een enzym (amylase (2) bestudeerden die in
menselijk speeksel wordt geproduceerd en zetmeel(3) opsplitst.
De menselijke amylase genen delen een gemeenschappelijk voorgeslacht met het amylase gen dat
in onze nauwste verwant, de chimpansee wordt gevonden.
Maar ze verschillen onderling ( bij mens en chimp ) op sommige belangrijke punten .
In plaats van één amylase gen, hebben mensen verschillende amylase-genen .
De menselijke amylase genen varieeren van 2 tot en met 15 exemplaren (drie keer zoweel als het
gemiddelde aantal van 2 tot en met 5 amylase genen varianten
bij ( de reeds onderzochte )chimpansees. )
Maar onze extra exemplaren zijn niet willekeurig over de wereldbevolking verdeeld .
Europese Amerikanen en knol-etende Afrikaanse jager-verzamelaars (bijvoorbeeld de Hadza
)bezitten zoals bekend , heel wat exemplaren.
Yakuts, die in Siberië leven en meestal vis eten, en de centraal-afrikaanse Mbuti pygmeeen die
meestal jachtwild eten hebben er minder.
Dat geeft het volgende patroon ;
Mensen met weinig amylase genen zijn jagers die niet veel zetmeel in hun dieet hebben zitten .
De mensen met heel wat genen eten ook heel wat zetmeel.
Hoe kon dit patroon zich ontwikkelen ?
4
Gameten (eieren en sperma ) , bezitten nogal regelmatig brokken van het DNA dat soms toevallig
werd gekopieerd tijdens de gametogenese .
In plaats van dat één exemplaar van een gen aan de volgende generatie wordt doorgegeven , kan
een ouder toevallig er twee inbrengen in de genetische
erfenis van zijn/haar nageslacht .
Wetenschappers bekijken menselijke genomen steeds gedetailleerden , en ze vinden meer en meer"
variaties in het aantal (en de functies )van gekopieerde exemplaren van genen ".
De verdubbeling van het gen is iets dat af en toe voorkomt.
In sommige gevallen, kan het extra gen zeer schadelijk zijn.(4)
In andere gevallen kan het op een of andere manier geen effect hebben( neutraal
/redundant/overbodig / back up ? ) .
Maar er zijn ook gevallen waar het wat goeds schijnt te doen.
De extra amylase genen zijn daarvan een goed voorbeeld .
Meer dezelfde varianten van genen betekent uiteindelijk meer gen -product ---> ( in dit geval )
amylase.
Meer amylase betekent meer krachtig speeksel voor het opsplitsen van zetmeel, wat meer voeding
voor overleving betekent(= verhoogde fitness ).
Door het aantal amylase exemplaren met andere verdubbelde genen te vergelijken, kwamen de
wetenschappers tot de (voorlopige) conclusie dat grotere
aantallen van genvarianten door natuurlijke selectie worden gedreven.
De eerste verhoging in amylase kan er gekomen zijn toen de hominiden begonnen knollen te rooien ,
een miljoen jaar geleden of meer.
Een veel recentere verhoging van amylsasegenen werd aangezwengeld door de opkomst van de
landbouw in sommige delen van de wereld.
Deze soorten voordelen kunnen de vermenigvuldiging van genen, niet alleen in mensen maar ook in
andere organismen/soorten /species drijven.
In Juli, publiceerden Ierse wetenschappers een studie over de bakkersgist . (5)
De "voorouders" van de huidige bakkersgist ondergingen een verdubbeling van hun volledig
genoom.
De extra genen stonden het gist toe om meer energie te onttrekken uit veranderen( = vergisten ) van
suiker in alcohol.
Een opeenhoping van genexemplaren (met dezelfde functies ) in een genoom, geeft ook de
mogelijkheid (bij de redundante gen-exemplaren ) nieuwe functies tot ontwikkeling te laten
evolueren : hier komt het fruitvlieg onderzoek aan bod .
Bij sommige soorten van Fruitvliegje, is tussen de mannetjes en het wijfjes een biochemische oorlog
tussen de geslachten , aan de gang ( inclusief een wapenwedloop ) .
De mannetjes kunnen hun reproductief succes opvoeren door hun vrouwelijke partners chemisch te
manipuleren.
Zij doen dit met een cocktail van chemische producten die in de rudimentaire sperma-vloeistof worden
gemengd.
Enkele chemicalieen bevorderen de productie van eieren, sommige andere chemische producten
veroorzaken een "dichting " in de vrouwelijke
geslachts-apparatuur die andere mannetjes de toegang beletten door een "stop" te veroorzaken
De wijfjes ontwikkelen chemische producten om de mannelijke chemische producten tegen te gaan(
door ze te versnijden : af te breken of aan te wenden voor andere doelen ) .
Veranderingen in de genen die het mannetje of het wijfje een tijdelijk genetisch en reproductief
overgewicht in deze bewapeningswedloop gaven ,
stonden dus onder sterke selectiedruk ...
In Genetica PLOS,(6) publiceerden de wetenschappers een lijst van vrouwelijke reproductieve
prote챦nen die sterke tekens van natuurlijke selectie vertonen.
Een dozijn van deze proteïnen zijn proteasen(7) -- die zijn goed bij het op maat snijden van andere
proteïnen.
De vliegen maken slechts deze proteasen aan in hun reproductieve systemen , en toch zijn deze
proteïnen niet dicht verwant aan andere proteïnen die
ook in diezelfde reproductieve apparatuur worden aangemaakt.
In plaats daarvan, zijn hun dichtste verwanten, families van proteasen die de vliegen in hun
ingewanden aanmaken om voedsel te verteren.
De beste evolutionaire verklaring voor deze resultaten, besluiten de wetenschappers , is als volgt:
de spijsverteringsproteasegenen werden toevallig gekopieerd in de voorvaderen van de
vliegen, en werden geleidelijk aan geaccumuleerd in grote families.
Sommigen van hen veranderden(= muteerden ) zodat zij slechts bepaalde proteïnen in de
reproductieve opstelling produceerden .
Zij bleken te kunnen helpen bij het afbreken van de "smerige "molecules in de spermavloestof
, en als dusdanig werden ze eruit geselekteerd
door het NS proces . Andere veranderingen deden zich later voor en werden eveneens door
natuurlijke selectie uitgezift en wel omdat ze de wijfjes
die ze bezaten in staat stelden eiweitten te produceren die de eiwitten van de mannetjes
kunnen versnijden en aan te passen
De inhoud van voormelde drie papers (1)(5)(6)( die ongeveer rond de zelfde tijd verschenen )zijn
eigenlijk ook ietwat ironisch :
Wij neigen ertoe om gist en fruitvliegen als modelorganismen voor onszelf , te denken :
wetenchappers bestuderen ze om aanwijzingen over onze eigen biologie (en onze eigen evolutie) te
verkrijgen.
Maar in het geval van genverdubbeling en de functie-verandering van nieuwe genen , zijn wij mensen
het model.
De variaties van zetmeel-verterende enzymen in menselijk speeksel illustreren hoe organismen
de grondstof voor natuurlijke selectie kunnen produceren
om nieuwe soorten genen tot stand te brengen.
Noten
(1)
http://www.nature.com/ng/journal/vaop/ncurrent/abs/ng2123.html;jsessionid=681A0D535576C0AE77
A810704E158C67
Diet and the evolution of human amylase gene copy number variation
George H Perry1,2, Nathaniel J Dominy3, Katrina G Claw1,4, Arthur S Lee2, Heike Fiegler5, Richard
Redon5, John Werner4, Fernando A Villanea3,
Joanna L Mountain6, Rajeev Misra4, Nigel P Carter5, Charles Lee2,7,8 & Anne C Stone1,8
Starch consumption is a prominent characteristic of agricultural societies and hunter-gatherers in arid
environments.
In contrast, rainforest and circum-arctic hunter-gatherers and some pastoralists consume much less
starch (1, 2, 3. )
This behavioral variation raises the possibility that different selective pressures have acted on
amylase, the enzyme responsible for starch hydrolysis( 4.)
We found that copy number of the salivary amylase gene (AMY1) is correlated positively with salivary
amylase protein level and that individuals
from populations with high-starch diets have, on average, more AMY1 copies than those with
traditionally low-starch diets.
Comparisons with other loci in a subset of these populations suggest that the extent of AMY1 copy
number differentiation is highly unusual.
This example of positive selection on a copy number–variable gene is, to our knowledge, one of the
first discovered in the human genome.
Higher AMY1 copy numbers and protein levels probably improve the digestion of starchy foods and
may buffer against the fitness-reducing effects of
intestinal disease.
School of Human Evolution and Social Change, Arizona State University, Tempe, Arizona 85287,
USA.
Department of Pathology, Brigham and Women's Hospital, Boston, Massachusetts 02115, USA.
Department of Anthropology, University of California, Santa Cruz, California 95064, USA.
School of Life Sciences, Arizona State University, Tempe, Arizona 85287, USA.
The Wellcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge
CB10 1SA, UK.
Department of Anthropological Sciences, Stanford University, Stanford, California 94305, USA.
5
Harvard Medical School, Boston, Massachusetts 02115, USA.
These authors contributed equally to this work.
Correspondence to:
Nathaniel J Dominy3 e-mail: [email protected]
(1,2,3,4 : http://www.nature.com/ng/journal/vaop/ncurrent/full/ng2123.html#B4 )
(2)
http://nl.wikipedia.org/wiki/Amylase
http://en.wikipedia.org/wiki/Amylase
(3)
http://nl.wikipedia.org/wiki/Zetmeel
http://en.wikipedia.org/wiki/Starch
http://www.lsbu.ac.uk/water/hysta.html
(4)
vergelijk met verdubbeling en vertrippeling ( trisonimie chromosoom 21 ) van chromosoon 21 bij het
syndroom van down
http://nl.wikipedia.org/wiki/Syndroom_van_Down
Trisonomie 21
(5)
http://www.nature.com/msb/journal/v3/n1/full/msb4100170.html
Increased glycolytic flux as an outcome of whole-genome duplication in yeast
Gavin C Conant1 & Kenneth H Wolfe1
Smurfit Institute of Genetics, Trinity College, University of Dublin, Dublin, Ireland
Correspondence to: Gavin C Conant1 Smurfit Institute of Genetics, University of Dublin, Trinity
College, Dublin 2, Ireland. Tel.: +353 1 896 1288;
Fax: +353 1 679 8558;
Email:
[email protected]
Received 21 March 2007; Accepted 27 June 2007; Published online 31 July 2007
This is an open-access article distributed under the terms of the Creative Commons Attribution
License, which permits distribution, and reproduction
in any medium, provided the original author and source are credited. This license does not permit
commercial exploitation or the creation of derivative
works without specific permission.
Abstract
After whole-genome duplication (WGD), deletions return most loci to single copy. However, duplicate
loci may survive through selection for increased dosage. Here, we show how the WGD increased
copy number of some glycolytic genes could have conferred an almost immediate selective
advantage to an ancestor of Saccharomyces cerevisiae, providing a rationale for the success of the
WGD. We propose that the loss of other redundant genes throughout the genome resulted in
incremental dosage increases for the surviving duplicated glycolytic genes. This increase gave postWGD yeasts a growth advantage through rapid glucose fermentation; one of this lineage's many
adaptations to glucose-rich environments. Our hypothesis is supported by data from enzyme kinetics
and comparative genomics. Because changes in gene dosage follow directly from post-WGD
deletions, dosage selection can confer an almost instantaneous benefit after WGD, unlike
neofunctionalization or subfunctionalization, which require specific mutations. We also show
theoretically that increased fermentative capacity is of greatest advantage when glucose resources
are both large and dense, an observation potentially related to the appearance of angiosperms
around the time of WGD.
(6)
http://genetics.plosjournals.org/perlserv/?request=get-document&doi=10.1371/journal.pgen.0030148
Gene Duplication and Adaptive Evolution of Digestive Proteases in Drosophila arizonae
Female Reproductive Tracts
Erin S. Kelleher1*, Willie J. Swanson2, Therese A. Markow1
1 Department of Ecology and Evolutionary Biology, University of Arizona, Tucson, Arizona, United
States of America, 2 Department of Genome Sciences, University of Washington, Seattle,
Washington, United States of America
It frequently has been postulated that intersexual coevolution between the male ejaculate and the
female reproductive tract is a driving force in the rapid evolution of reproductive proteins. The dearth
of research on female tracts, however, presents a major obstacle to empirical tests of this hypothesis.
Here, we employ a comparative EST approach to identify 241 candidate female reproductive proteins
in Drosophila arizonae, a repleta group species in which physiological ejaculate–female coevolution
has been documented. Thirty-one of these proteins exhibit elevated amino acid substitution rates,
making them candidates for molecular coevolution with the male ejaculate. Strikingly, we also
discovered 12 unique digestive proteases whose expression is specific to the D. arizonae lower
female reproductive tract. These enzymes belong to classes most commonly found in the
gastrointestinal tracts of a diverse array of organisms. We show that these proteases are associated
with recent, lineage-specific gene duplications in the Drosophila repleta species group, and exhibit
strong signatures of positive selection. Observation of adaptive evolution in several female
reproductive tract proteins indicates they are active players in the evolution of reproductive tract
interactions. Additionally, pervasive gene duplication, adaptive evolution, and rapid acquisition of a
novel digestive function by the female reproductive tract points to a novel coevolutionary mechanism
of ejaculate–female interaction.
Funding. This research was funded by the University of Arizona, and the NSF-IGERT program in
Evolutionary, Functional and Computational Genomics at the University of Arizona. ESK was
supported by a National Science Foundation Integrative Graduate Education and Research
Traineeship in evolutionary, functional, and computational genomics at the University of Arizona. WJS
was supported by NIH grant HD42563.
Competing interests. The authors have declared that no competing interests exist.
Editor: Harmit S. Malik, Fred Hutchinson Cancer Research Center, United States of America
Citation: Kelleher ES, Swanson WJ, Markow TA (2007) Gene Duplication and Adaptive Evolution of
Digestive Proteases in Drosophila arizonae Female Reproductive Tracts. PLoS Genet 3(8): e148
doi:10.1371/journal.pgen.0030148
Received: May 23, 2007; Accepted: July 13, 2007; Published: August 31, 2007
6
Copyright: 짤 2007 Kelleher et al. This is an open-access article distributed under the terms of the
Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction
in any medium, provided the original author and source are credited.
Abbreviations: EST, expressed sequence tag; GO, gene ontology; MYA, million years ago; RT-PCR,
reverse transcriptase PCR
* To whom correspondence should be addressed. E-mail: [email protected]
A previous version of this article appeared as an Early Online Release on July 18, 2007
(doi:10.1371/journal.pgen.0030148.eor).
Author Summary
In a broad range of organisms, including humans, molecular interactions between the male ejaculate
and the female reproductive tract play integral roles in sexual reproduction. Although these
interactions are essential, the biochemical composition of the male ejaculate can change rapidly over
short evolutionary time periods. It is often hypothesized that this rapid evolution reflects a
coevolutionary relationship with the female reproductive tract. The paucity of research on females,
however, presents a formidable challenge to empirical tests of this hypothesis. In this study, we
sought to identify proteins in the female reproductive tracts of D. arizonae that may be interacting or
coevolving with the male ejaculate. Unexpectedly, we discovered that D. arizonae females produce an
array of “digestive” enzymes in their reproductive tracts. These classes of enzymes are normally
found in the gut, where they degrade ingested food for nutritional uptake. In D. arizonae, these
enzymes have resulted from recent gene duplications, and natural selection has caused rapid and
radical changes in their amino acid sequences. We propose that this pattern of duplication and
diversification reflects the “female side” of a coevolutionary relationship with the male ejaculate.
Exploring the “male side” of this relationship is an important avenue for future research
(7)
http://nl.wikipedia.org/wiki/Protease
Proteasen zijn enzymen die andere eiwitten afbreken.
Een andere naam voor protease is peptidase.
"Prote" komt van proteïne en een enzym op -ase breekt iets af.
Een voorbeeld van een protease is pepsine.
Pepsinogeen, geproduceerd in cellen van de maagwand, wordt onder invloed van zoutzuur omgezet
in pepsine.
Pepsine breekt eiwitten uit de voeding af tot aminozuren.
De dikke slijmlaag in de maag voorkomt dat het pepsine de eigen maagwand afbreekt.
In cellen bevinden zich grote complexen van proteasen.
Deze worden proteasomen genoemd. Een andere groep proteasen zijn de caspasen.
Definitief afscheid van de "degeneratie "kwakkel ?
GEN , GENEN en GENOOM duplikaties ;
zijn natuurlijk passende antwoorden op de creationistische claim ; dat er
geen (genetische ) informatie-winst of aangroei van genenkapitaal mogelijk is
alleen maar genverlies en degeneratie ....
Deze gist historie is in elk geval een van de definitieve nagels in de doodkist van Sheele's EO pseudo
-wetenschappelijke " rethoriek "
Voorbeelden verschillende duplikaties
http://www.ingenta.com/isis/searching/Expand/ingenta?pub=infobike://bsc/kid/2004/00000065/000000
06/art00004
http://www.biomedcentral.com/1471-2229/4/10
http://www.google.be/search?q=cache:hrdCEBgqeDIJ:www.catalase.com/evogenedup.htm+tandem+
gene+duplication&hl=nl
Gene Duplication Adapts To Changing Environment
http://www.sciencedaily.com/releases/2002/03/020304081153.htm
LINKS genetische duplicaties /verdubbelingen
http://www.sciencedaily.com/releases/2002/03/020304081153.htm
http://www.medicalnewstoday.com/medicalnews.php?newsid=20010
http://www.don-lindsay-archive.org/creation/dup_favorable.html
http://genome.osu.edu/ibgp730/readings/lecture_02-Op-01.pdf
http://www.genetics.org/cgi/content/full/151/4/1531
De aangroei van het gen-kapitaal ....
PZ Myers
..... many kinds of mutations very commonly produce additional DNA structures ....
http://scienceblogs.com/pharyngula/2007/03/a_straightforward_example_of_c.php
Een antwoord aan de miskleun van
creationist Rob McEwen
Dit kan eveneens worden gezien als een mogelijke falsificatie van sommige claims van P. Scheele en
P. Borger
Gene duplication versus ID
http://www.pandasthumb.org/archives/2004/05/gene_duplication_versus_id.html
Positive Darwinian selection after gene duplication in primate ribonuclease genes - J Zhang, HF
Rosenberg, M Nei - 1998 - National Acad Sciences
Evolutionary mechanisms of origins of new gene function have been a subject of
long-standing debate. Here we report a convincing case in which positive ...
Geciteerd door 465 - Verwante artikelen - Het Internet
Evolution by gene duplication: an update
J Zhang - Trends Ecol. Evol, 2003 - nslij-genetics.org
The importance of gene duplication in supplying raw genetic material to
biological evolution has been recognized since the 1930s. Recent genomic ...
Gen duplicatie in schimmels / 2007
Ilan Wapinski
http://www.sciencedaily.com/releases/2007/09/070911155206.htm
http://www.broad.mit.edu/cgi-bin/news/display_news.cgi?id=3781
http://lib.bioinfo.pl/pmid:17805289
http://www.nature.com/nature/journal/v449/n7158/abs/nature06107.html
http://www.nature.com/nature/journal/v449/n7158/suppinfo/nature06107.html
http://web.archive.org/web/20070226194902/www.nadarwin.nl/Gtica/rip.html
Repeat-induced point mutation (RIP) en evolutie door genduplicaties
Een artikel van Marnix Medema.
Om tot een goed begrip te komen van het proces van macro-evolutie is het essentieel om de
achterliggende genetische mechanismen te ontrafelen. Eén van de meest essentiële
mechanismen die noodzakelijk zijn voor macro-evolutie is het ontstaan van nieuwe genen.
Immers, zonder nieuwe genen kan een organisme zijn genetisch potentieel niet vergroten.
Bovendien is genengroei noodzakelijk om de kwantitatieve genetische verschillen tussen alle
organismen te verklaren. Al tientallen jaren wordt als verklaring hiervoor de hypothese
7
aangehouden dat nieuwe genen ontstaan door genduplicatie. Soms wordt tijdens het
overschrijven van het DNA een gedeelte van een chromosoom (één of meerdere genen) 'per
ongeluk' twee keer overgeschreven, waardoor de dochtercel twee versies van dit gedeelte
overhoudt. Als dit in de kiemlijn van een organisme gebeurt, zullen de nakomelingen van het
organisme twee versies van dit gedeelte hebben. Als er twee versies van een gen in het genoom
voorkomen, kan een van de twee versies muteren, zonder dat dit schadelijke gevolgen heeft,
omdat de andere versie van het gen de oude functie in stand houdt. Na mutaties in de
genetische code van één of beide genen zullen de genen functioneel kunnen gaan verschillen,
en zijn er uit een enkel gen twee nieuwe genen ontstaan.
De hypothese dat nieuwe genen kunnen ontstaan door genduplicatie en -divergentie wordt
ondersteund door het feit dat in alle organismen waarvan het genoom bekend is veel genen
bestaan die zeer sterk op elkaar lijken. De eiwitten waarvoor deze genen coderen hebben vaak
vrijwel dezelfde aminozuurcode, terwijl door kleine, doch belangrijke veranderingen de genen
verschillende functies hebben gekregen. De argumentatie die creationisten doorgaans inbrengen
tegen dit bewijsmateriaal is dat het feit dat genen op elkaar lijken geen bewijs is dat ze ook
dezelfde afstamming hebben. Een Schepper (of Ontwerper) zou immers logischerwijs ook
dezelfde bouwplannen aanhouden in al zijn ontwerpen, net zoals mechanici gebruik maken van
dezelfde onderdelen (electriciteitsdraden, weerstanden, etc.) voor het ontwerpen van
verschillende machines, die ook verschillende functies hebben.
Hoe komen we nu te weten of genen op elkaar lijken doordat ze een gemeenschappelijke
oorsprong in de evolutie hebben, of doordat ze ontstaan zijn uit de gelijkvormige bouwplannen
van een Schepper? Wat we nodig hebben is een organisme waarin geen genduplicaties
optreden. Als de verklaring voor de homologie van genen ligt in het feit dat ze ontstaan zijn door
genduplicatie en -divergentie, zullen in een dergelijk organisme vrijwel geen homologe genen
voorkomen. Maar als de homologie van genen verklaard moet worden door de bouwplannen van
een Schepper, zal ook een organisme zonder genduplicaties veel homologe genen bevatten.
Gelukkig bestaat er een organisme, waarin geen genduplicaties optreden. De schimmel
Neurospora crassa bevat een uitzonderlijk mechanisme, RIP (Repeat-Induced Point-mutation)
geheten, dat ervoor zorgt dat gedupliceerde delen van zijn genoom onmiddelijk van alle
functionaliteit ontdaan worden. Dit mechanisme detecteert gedupliceerde sequenties en muteert
hierin zo'n 30% van de C:G baseparen naar A:T baseparen. Een dergelijke hoeveelheid
ongerichte mutaties die tegelijk op een sequentie aanvat betekent onherroepelijk het einde van
de functionaliteit van de sequentie. Hoewel dit mechanisme vooral werkt in de verdediging tegen
genetische 'parasieten' zoals transposons, is het in dit organisme onmogelijk dat nieuwe genen
zouden ontstaan door genduplicatie en -divergentie. Als we vervolgens kijken naar het aantal op
elkaar lijkende genen dat Neurospora crassa bevat, blijken dat er zeer weinig te zijn. Slechts
0,1% van de exonen (functionele sequenties die samen genen vormen) van Neurospora crassa
deelt >80% van zijn nucleotide-sequentie met een ander gen, vergeleken met 16% bij
Sacharomyces cerevisiae. Onderstaande figuur geeft een overzicht van de homologie in de
aminozuursequentie van de verschillende genen in Neurospora crassa, naast de homologie
tussen genen in twee andere schimmels (S. cerevisiae en S. pombe), de worm C. elegans, het
fruitvliegje D. melanogaster en de plant A. thaliana.
De conclusie uit deze gegevens is overduidelijk: nieuwe genen ontstaan door genduplicatie. Als
er geen genduplicatie mogelijk is, is het aantal homologe genen dat nog gevonden wordt
miniem. De kleine hoeveelheden homologe genen die nog in Neurospora crassa gevonden
worden, zijn bovendien allen zeer kleine genen, die bij hun ontstaan te klein waren geweest om
door het RIP-mechanisme gedetecteerd en uitgeschakeld te worden. De enige juiste verklaring
voor de homologie tussen verschillende genen binnen een organisme is dus het feit dat ze zijn
ontstaan door genduplicaties, en een gemeenschappelijke oorsprong hebben. Als we kijken naar
de evolutie op het niveau van genen, valt dus niet te ontkennen dat deze plaatsvindt. Geen
twijfel over mogelijk.
Bron:
- RIP: the evolutionary cost of genome defense. JE Galagan, EU Selker
(2004). TRENDS in Genetics 20(9): blz. 417-423
Aangroei van coderend genkapitaal ?
Het wordt steeds duidelijker hoe evolutionaire noviteiten ontstaan.
En creationisten maar blijven roepen "Informatie kan nooit toenemen".....
Genetici hebben 5 genen in de fruitvlieg Drosophila ontdekt die uit het niets ontstaan lijken te
zijn.
De meest gangbare manier waarop nieuwe genen ontstaan is het veranderen van oude
genen.
Meestal het wijzigen van kopieën ( duplicaties ) van andere genen.
Maar als je geen voorlopers( voor"ouders" ) kunt ontdekken, dan is de vraag: waar komt een
heel nieuw gen vandaan?
Het moet ergens vandaan komen.
Genetici hebben nu voor het eerst aannemelijk kunnen maken dat genen ontstaan uit
zogenaamd junk DNA.
Dat is DNA dat niet voor de productie van eiwitten dient.
Genetici noemen een deel daarvan 'noncoding DNA'( er is ook verongelukt DNA ) : DNA dat
niet codeert voor eiwitten.
En daar is genoeg van.
Door een foutje ( in het doorgeven van ( ook dit ) erfelijke materiaal aan de volgende
generatie --> mutatie ? ) kan het gebeuren dat dat noncoding DNA toch afgelezen wordt
alsof het een echt gen was.
En dan krijg je toch een geheel nieuw eiwit.
Tenslotte kan dat nieuwe eiwit ( soms ) ook nog iets nuttigs doen.
Daarna doet natuurlijke selectie de rest
Manyuan Long (2007) 'An evolutionary geneticist is surprised by genes of unknown
origin', Nature 449, 511 (4 October 2007) Journal Club.
http://pondside.uchicago.edu/ceb/faculty/Long.html
Mia T. Levine (2006) 'Novel genes derived from noncoding DNA in Drosophila
melanogaster are frequently X-linked and exhibit testis-biased expression', PNAS June
8
27, 2006 vol. 103 no. 26 9935-9939
http://www.pnas.org/cgi/reprint/0509809103v1
(Korthof )
Noncoding DNA codeert niet voor eiwitten.
Als er dan op een gegeven moment wel eiwit geproduceerd wordt op basis van zo'n stuk DNA,
dan is er een nieuw eiwit.
Dat nieuw eiwit heeft geen voorloper eiwitten; is niet ontstaan uit een wijziging van bestaande
eiwitten.
Zo ontstaat er eiwitcoderende informatie uit niet eiwitcoderende informatie.
In die zin ontstaat er informatie uit het niets.
Zo moeilijk is dat niet te begrijpen
Er is een nieuwe mutatie geweest die een stuk betekenisloos ( non coding) DNA heeft
veranderd in een stuk DNA dat een eiwit tot expressie brengt.
: een 'experimenteel' eiwit waarvan niet van tevoren te zeggen is wat het doet.
De conclusie dat
'Blijkbaar codeert Noncoding DNA wél voor eiwitten' is dus fout.
Genetici die het genoom onderzoeken kunnen gewoon zien of een stuk DNA wordt afgelezen en
of er een eiwit geproduceerd wordt.
Daar bestaan tests voor.
Als dat eiwit niet gevonden wordt, dan is het noncodingDNA.
Het is dus niet zo dat wetenschappers niet weten wat er met DNA gebeurt, ze weten door het
doen van tests positief dat er geen eiwit geproduceerd wordt van een stuk DNA.
Tenslotte:
is ook deze bewering overbodig (en misleidend ) ;
Junk-DNA heeft blijkbaar méér functie dan tot voor kort werd aangenomen.
met moet immers het woord 'junk-DNA' niet gebruiken in deze context
.Non-coding is trouwens niet hetzelfde als het zogenaamde junk ... ( zie boven )
(Martin)
* De uitdrukking dat "DNA codeert" betekent, dat er een uitleessysteem is, dat het DNA
interpreteert om vervolgens eiwitten te produceren.
Dus "coderen" heeft alleen betekenis in samenhang met de interpretatieregels. Veel mensen
begrijpen dit niet .
bovenvermeld voorbeeld toont aan, dat "informatie" niet noodzakelijkerwijze door een intelligent
wezen in dat DNA gestopt moet zijn.
Ex nihilo, dus.
In het voorbeeld wordt blijkbaar een eiwit geproduceerd buiten de geldende regels om: het was
een foutje.
Net zoals wanneer een mens "atoombom" leest terwijl er "autobom" staat.
Waar komt die informatie "atoombom" vandaan?
#19 #20
http://evolutie.blog.com/2147411/?page=2#cmts
Dubbel DNA vergroot overlevingskans
Planten die een ramp willen overleven, kunnen maar beter wat extra erfelijk materiaal aan boord
hebben.
Niet alleen de dinosaurussen stierven 65 miljoen jaar geleden uit door catastrofale gebeurtenissen,
ook 60 procent van alle plantensoorten verdween van de aardbodem door een meteorietinslag of
vulkaanuitbarstingen.
http://noorderlicht.vpro.nl/artikelen/23672997/
Waarom overleefde die andere 40 procent wel?
Wat was hun evolutionaire voordeel?
Dubbel DNA, schrijven drie Belgische bio-informatici in PNAS. Yves van de Peer en zijn collega’s zijn
gespecialiseerd in het ontcijferen van planten-DNA, van gras en tomaten tot rijst en bomen.
Daarbij bleef het ze opvallen dat deze planten in de loop van hun leven op aarde hun DNA één of
meerdere malen hebben verdubbeld.
De wetenschappers wisten deze verdubbelingen te dateren en stelden vast dat de meest recente
duplicaties zo'n 65 miljoen jaar geleden
plaatsvonden, dus samen met de "Krijt-Paleogeen-massa-extinctie". .
Een of meerdere catastrofale gebeurtenissen (zoals komeetinslag, toegenomen vulkanische activiteit,
...) veroorzaakten toen branden en stofwolken, die een lange tijd het zonlicht tegenhielden.
Zestig procent van de plantensoorten en heel wat diersoorten, waaronder de dinosaurussen,
overleefden de gewijzigde klimaatomstandigheden niet.
Rond de tijd van de grote catastrofe dus.
De soorten die wel overleefden, waren beter aangepast aan de nieuwe omstandigheden.
Dat hadden ze wellicht te danken aan DNA-duplicatie,( in het gevalvan planten ook veelal een een
komplete genoomduplicatie ) die hen een evolutionair voordeel gaf.
In de loop van de geschiedenis hebben planten een of meerdere keren hun DNA verdubbeld.
De catastrofe was een mazzeltje voor deze planten.
Onder constante omstandigheden is verdubbeld DNA juist nadelig voor organismen, omdat het allerlei
nieuwe eigenschappen veroorzaakt.(1)
Maar die nieuwe eigenschappen komen juist goed van pas als de omgeving ingrijpend verandert.
Bijvoorbeeld als de lava en de meteorieten je om de oren vliegen.
De wetn schappers concluderen dat planten met dubbel DNA het best waren aangepast aan de
veranderde omstandigheden.
Afwijking
Duplicaties van het DNA zijn een eerder uitzonderlijke "afwijking".
Ze veroorzaken zeer uitgesproken eigenschappen en zijn in ongewijzigde omstandigheden eerder
nadelig voor de soort.
Eerder onderzoek van Yves Van de Peer had al aangetoond dat DNA-verdubbelingen bij vroege
voorouders van gewervelde dieren en vissen cruciaal waren voor de ontwikkeling van
gewervelde dieren, en dus ook van de mens
http://www.sciencedaily.com/releases/2002/03/020304081153.htm
Gene Duplication Adapts To Changing Environment
http://www.sciencedaily.com/releases/2007/09/070911155206.htm
New Method Can Reveal Ancestry Of All Genes Across Many Different Genomes
http://www.medicalnewstoday.com/articles/143261.php
http://www.ugent.be.
http://noorderlicht.vpro.nl/noorderlog/bericht/41720344/
Bouwe van Straten
(1)
(Hierdoor zijn dier- en plantensoorten ook veel complexer kunnen worden
(belga/edp)
24/03/09
9












