Ecomorfologie
Oefeningen 'comparative method'
1. Testen van een trade-off tussen twee prestatiematen adhv de methode van de
gestandaardiseerde onafhankelijke contrasten.
Spierfysiologische en biomechanische principes voorspellen een negatief verband tussen
snelheid en uithoudingsvermogen. Soorten met een relatief grote hoeveelheid witte
spiervezels (anaëroob, snelle contracties, maar snel vermoeid) zouden een hoge
sprintsnelheid hebben, maar een klein uithoudingsvermogen. Soorten met een grote
proportie rode spiervezels (aëroob, minder snelle contractie, fatigue-resistent) zouden
trager zijn, maar een betere uithouding hebben.
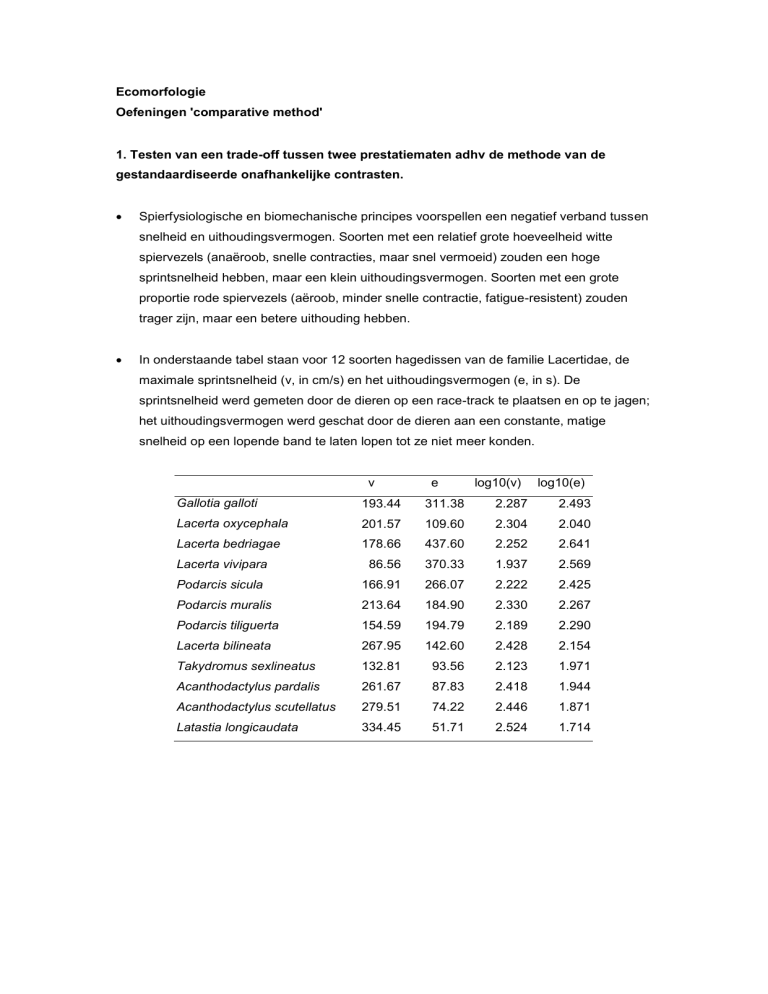
In onderstaande tabel staan voor 12 soorten hagedissen van de familie Lacertidae, de
maximale sprintsnelheid (v, in cm/s) en het uithoudingsvermogen (e, in s). De
sprintsnelheid werd gemeten door de dieren op een race-track te plaatsen en op te jagen;
het uithoudingsvermogen werd geschat door de dieren aan een constante, matige
snelheid op een lopende band te laten lopen tot ze niet meer konden.
v
e
log10(v)
log10(e)
Gallotia galloti
193.44
311.38
2.287
2.493
Lacerta oxycephala
201.57
109.60
2.304
2.040
Lacerta bedriagae
178.66
437.60
2.252
2.641
Lacerta vivipara
86.56
370.33
1.937
2.569
Podarcis sicula
166.91
266.07
2.222
2.425
Podarcis muralis
213.64
184.90
2.330
2.267
Podarcis tiliguerta
154.59
194.79
2.189
2.290
Lacerta bilineata
267.95
142.60
2.428
2.154
Takydromus sexlineatus
132.81
93.56
2.123
1.971
Acanthodactylus pardalis
261.67
87.83
2.418
1.944
Acanthodactylus scutellatus
279.51
74.22
2.446
1.871
Latastia longicaudata
334.45
51.71
2.524
1.714
De verwantschap tussen de verschillende soorten wordt gegeven door de onderstaande
boom. De getallen langs de takken geven de divergentietijden aan in miljoenen jaren.
50
Gallotia galloti
26
26
2
36
Lacerta vivipara
13
2
Podarcis sicula
10
13
10
5
Podarcis muralis
23
5
Podarcis tiliguerta
33
Lacerta bilineata
40
5
Takydromus sexlineatus
25
Acanthodactylus pardalis
5
25
15
Acanthodactylus scutellatus
Latastia longicaudata
30
50
40
30
20
10
0
miljoen jaar
Kijk eerst met 'klassieke' (i.e. niet-fylogenetische) statistiek na of er een verband bestaat
tussen uithouding en snelheid.
Voor de fylogenetische verwerking maken we gebruik van het programma-pakket PDAP
(Phenotypic Diversity Analysis Programs), geschreven door Garland en medewerkers.
De eerste stap bestaat erin de fylogenetische boom in te brengen. Dit kan via het
programma PDTREE.EXE.
Wanneer je dit programma opstart, kom je terecht in de hoofdmenu, met keuze tussen
******************************************************************************************************
(I)nput a tree
(als je er al één gemaakt hebt)
(C)reate a tree from scratch
(dat ga je nu doen)
(E)dit the tree currently in memory
(voor het aanbrengen van veranderingen)
(A)nalyze tip data with Felsenstein's contrasts
(de echte analyse, zie verder)
(S)ave the tree currently in memory to disk
(niet vergeten te doen)
(Q)uit this program and exit to DOS
(dat zal nog even duren)
******************************************************************************************************
Creëer nu je boom (druk op C). Het programma vraagt hoe 'hoog' de boom moet zijn,
dus de afstand van de wortel tot de tippen. In dit geval : 50 miljoen jaar (zie figuur).
Je komt direct terug in de hoofdmenu. Om de boom interactief op te bouwen, druk E. Je
krijgt nu een nieuw menu, met de volgende opties.
******************************************************************************************************
(E)dit the tree graphically. You will not see this menu.
(B)ranch length manipulations.
(T)ip data manipulations.
(F)ull screen print. For a prettier screen dump.
(R)eturn to the Main Menu.
******************************************************************************************************
Druk opnieuw op E om de boom aan te passen.
Het volgende scherm toont je enkel de (vooralsnog embryonale) boom. Je kan doorheen
de boom bewegen door gebruik te maken van de pijltjes op je toetsenbord. Het linkse
pijltje brengt je naar het linkse kind, het rechtse naar het rechtse, en het neerwaartse
pijltje brengt je naar de voorouder. Door op één van volgende toetsen te drukken, kan je
de boom aanpassen.
******************************************************************************************************
(A)dd a branch to the branch you are on.
(C)hange the name and/or values of a node.
(D)raw a horizontal line through current node.
(E)dit just the subtree descended from the current node.
(G)o to tip or node name.
(L)ength change - alter the length of a branch.
r(O)tate node - exchange left and right subtrees.
(P)op = undo the E command and work with the whole tree again.
(R)eturn will get you back here.
(S)calar extension of all branches at current height.
(T)rim off the branch you are on and all of its children.
(V) - Reroot the tree at the current node or branch.
*******************************************************************************************************
Je zal vooral gebruik maken van A, C, R en misschien L.
Een vork bijmaken kan door op de bewuste tak te gaan staan en op A te drukken. Je moet
dan beslissen of je eerst op de linker (L) of op de rechter tak (R) terecht wil komen. Daarna
moet je ingeven hoe ver de nieuwe tweesprong van de vorige ligt. Is dit bijvoorbeeld 5 miljoen
jaar, voer dan '1e6' in. Dit is kort voor 1 x 106 jaren. Geef daarna het nieuwe kruispunt ('node')
een naam. Die moet bestaan uit twee karakters (vb. aa, AA, aA, n1, ...), maar gebruik geen
combinatie die je daarvoor al eens gebruikt hebt. Tenslotte vraagt het programma naar de
eigenschappen van de nieuwe tip-soort. Voer de waarden in voor de twee variabelen.
Herhaal dit nu tot de ganse boom afgewerkt is.
Om de gestandaardiseerde onafhankelijke contrasten te berekenen, keer je terug naar de
hoofdmenu (druk op R). Daar kies je (A)nalyze tip data with Felsenstein’s (1985)
contrasts.
Een nieuw menu verschijnt, met volgende opties :
**********************************************************************************************************
(V)iew diagnostic graphs and statistics (screen 1-8)
(W) view regression graphs and statistics (screen 9-11)
(S)ave .STA file (screen display statistics and contrasts)
(J)ust save .FIC file (just contrasts)
**********************************************************************************************************
De eerste optie (V) gaat na of een aantal voorwaarden i.v.m. de taklengten voldaan zijn. Dit is
allemaal nogal technisch en hier van minder belang. We verwijzen de geïnteresseerden naar
de handleiding van PDAP.
Gebruik de tweede optie om de resultaten van de fylogenetische analyse te bekijken. Je moet
eerst kiezen welke van de twee variabelen x is, en welke y. In het voorbeeld hier speelt dat
geen rol.
Het volgende scherm geeft een grafiek van de contrasten in variabele x t.o.v. de contrasten in
variabele y. Links krijg je ook een aantal statistieken. In dit geval zijn we vooral
geïnteresseerd in de correlatiecoëfficiënt (corr) en zijn significantie (zie t en df onderaan).
Er zijn nog twee bijzonderheden op te merken aangaande de grafiek. Eén : de waarden voor
de X-variabelen zijn ‘gepositiveerd’, dit wil zeggen dat bij het berekenen van de contrasten in
variabele x steeds de kleinere waarde is afgetrokken van de grotere waarde. Twee : de
regressielijn is door de oorsprong geforceerd.
Tenslotte wijzen we erop dat het programma nu rekening houdt met de (variabele) lengte van
de takken in de boom. (In feite wordt het contrast gedeeld door de vierkantswortel van de
som van de twee taklengten.) Het programma veronderstelt dus een graduele evolutie, met
meer veranderingen langs langere takken in de boom. Als je niet akkoord gaat met dit model
van evolutie, dan moet je de taklengten veranderen.
Dat kan in de Edit-module, onder de optie (B)ranch length manipulations. Denk je
bijvoorbeeld dat veranderingen vooral plaatsgrijpen tijdens speciaties (‘speciational
evolution’), zet dan alle taklengten gelijk aan één (optie (C)onstant). Twee ander veel
gebruikte manipulaties zijn die van Grafen en van Pagel. Zie de PDAP handleiding voor meer
uitleg.
2. De home-range van herbivore en carnivore zoogdieren : een fylogenetische
(co-)variantie-analyse via computersimulatie
Ecologen beweren vaak dat de home-range van dieren varieert naargelang het trofische
niveau waarop ze zich bevinden. Secundaire consumenten (carnivoren) zouden een
grotere home-range nodig hebben dan primaire consumenten (herbivoren), omdat het
voedsel van carnivoren meer verspreid voorkomt en minder joules per oppervlakteeenheid oplevert dan het voedsel van herbivoren.
We zullen dit idee toetsen bij twee clades van zoogdieren : de Carnivora (voornamelijk
vleeseters, maar ook enkele omnivoren) van de ungulaten (in deze gegevensset allen
herbivoor).
Omdat de home-range van een dier sterk gecorreleerd is met zijn lichaamsgrootte, zullen
we in de analyse massa meenemen als een covariabele : we willen dus testen of een
herbivoor een kleinere home-range heeft dan een carnivoor van vergelijkbare grootte.
De massa’s en home-range-groottes van 49 zoogdieren staan vermeld in de tabel op de
volgende bladzijde.
Het cladogram op de bladzijde daarna geeft een idee van de fylogenetische
verwantschappen van de 49 zoogdieren in de gegevensset.
De boom en de gegevens over home-range en massa zitten reeds in het bestand
49lbr.inp. Je kan dit bestand oproepen in het programma PDTREE.EXE om de boom
eens te bekijken en te controleren. Merk op dat massa en home-range-grootten eerst
logaritmisch (log10) getransformeerd werden.
Een conventionele an(c)ova kan je uitvoeren met het programma PDSINGLE.EXE, of met
eender welk ander statistisch pakket.
PDSINGLE.EXE vereist een bestand met de extensie .PDI. Je kan dit bestand aanmaken
door 49lbr.inp in te lezen in het programma PDTREE.EXE en het te bewaren als
49lbr.pdi. Gebruik daartoe de (S)ave optie in de hoofdmenu.
PDSINGLE.EXE vraagt eerst hoeveel sets je wil vergelijken, en vervolgens welke soorten
in welke set thuishoren. Het invoeren van deze gegevens is helaas wat archaïsch. Volg
de instructies op het scherm. Nadat je ook nog hebt opgegeven welk van de twee
variabelen moet beschouwd worden als co-variabele (in dit geval : massa, dus variabele
1), worden de resultaten neergeschreven in een bestand met extensie .sgl. Bekijk de F-
waarden voor het ‘main’ effect (verschillen tussen groepen) en voor de co-variabele (het
belang van massa). Zoek op of deze F-waarden significant zouden zijn als we gewone Ftabellen zouden gebruiken.
Voor de fylogenetische an(c)ova gebruiken we de programma’s PDSIMUL.EXE en
PDANOVA.EXE. Het eerste programma gaat de simulaties uitvoeren en de 1000 (2000,
3000, ...) nieuw gecreëerde sets virtuele beesten wegschrijven in een .SIM bestand. Het
tweede programma voert op deze nieuwe sets 1000 (2000, 3000, ...) variantie-analysen
uit, en bewaart alle F-waarden in het bestand *.FIC.
Na het opstarten van PDSIMUL.EXE krijg je een (vrij onoverzichtelijk) menu, waarin je
dingen kan veranderen door HOOFDLETTERS in te drukken.
Zeker aan te passen is (N), het aantal simulaties dat moet uitgevoerd worden. Zet dit
minstens op 1000.
Eventueel kan je ook het evolutiemodel aanpassen. Je kan kiezen tussen een graduele
vorm van evolutie, een speciationele vorm, en een evolutie van onderbroken
evenwichten.
‘Gradual Brownian motion’ is het meest eenvoudige evolutiemodel. Het veronderstelt
dat elke verandering aan een kenmerk onafhankelijk is van de vorige verandering,
onafhankelijk van de startwaarde van het kenmerk, en even goed positief als
negatief. Kenmerken zullen meer veranderen langs lange takken dan langs korte.
‘Speciational Brownian’ verschilt van het vorige model in het feit dat alle taklengten
als even groot beschouwd worden. Je kan dit ook interpreteren alsof er maar één
verandering optreedt, aan beide dochters, tijdens de speciatie.
‘Punctuated Equilibrium’ doet iets gelijkaardigs als het vorige model, maar gaat ervan
uit dat slechts in één dochter veranderingen optreden, en dat de andere ‘dochter’ er
hetzelfde uitziet als de voorouder.
Het programma voorziet ook twee varianten van het Uhlenbeck & Ornstein evolutiemodel,
dat volgens sommige auteurs een adequate beschrijving van veranderingen onder
natuurlijke selectie zou geven. We gaan hier niet dieper op in, zie handleiding en de
verwijzingen daarin.
Er is ook de mogelijkheid om grenzen te stellen aan de mate van veranderingen in de
kenmerken. Je zou bijvoorbeeld in dit geval een bovengrens kunnen opleggen aan de
variabele massa hier. Het grootste bekende terrestrische zoogdier is Baluchitherium (een
fossiel ook bekend als Indricotherium of Paraceratherium), en het woog maximaal 15000
kg. Aan de andere kant weegt de kleinste zoogdier ongeveer 1-2 g.
Als je grenzen oplegt, kan je ook nog beslissen wat er moet gebeuren als zo’n grens
bereikt wordt. Bij ‘Throw out’ begint de simulatie gewoon van voor af aan. Bij ‘Replace’
gaat het programma na of een gekozen verandering zou resulteren in een waarde die
buiten de grenzen ligt. Zo ja, wordt een andere verandering gekozen. ‘Flip’ doet hetzelfde
als ‘Replace’, maar door het teken van de verandering om te draaien. Voor de andere
mogelijkheden, raadpleeg de handleiding.
Als alles naar je zin is, druk je op (J)ust do it, en de simulaties zijn onderweg. De nieuwe
sets gegevens worden neergeschreven in een bestand *.SIM.
PDANOVA.EXE werkt zoals PDSINGLE.EXE maar voert zoveel ANOVA’s uit als er
gesimuleerde sets zijn. Het programma genereert een ganse familie bestanden, maar het
belangrijkste is het bestand *.F. Hierin worden de 1000 (2000, 3000, ...) F-waarden
bewaard. Het is een lang tekstbestand, dat je eventueel kan invoeren in een spreadsheet
(vb. EXCEL).
*.F bevat volgende kolommen :
1) F voor een ANOVA op variabele 1
2) F voor een ANOVA op variabele 2
3) F voor de betekenis van de co-variabele
4) F voor het verschil in ‘adjusted means’ tussen de geduide groepen (het ‘main’
effect)
5) F voor de variatie verklaard door het totale model (main + covariate)
6) F voor de verschillen tussen de hellingen
Je kan deze reeksen F-waarden gebruiken als nul-distributie, waartegen je de Fwaarden bekomen met de traditionele an(c)ova, kan testen. Als de traditionele F-waarde
groter is dan 95 % van de F-waarden bekomen via de simulaties, dan mag je stellen dat
je een significant effect gevonden hebt.
3. Testen van een correlatie tussen ‘lekking’ gedrag en seksueel dimorfisme in grootte bij
vogels : het tellen van evolutionaire overgangen
Evolutionaire biologen beweren dat seksuele selectie leidt tot de evolutie van
seksueel dimorfisme. Seksueel dimorfosme wordt verondersteld sterk tot uiting te
komen bij polygame soorten. Soorten die ‘lekken’ worden als voorbeeld gebruikt
omdat het lekking systeem een extreme vorm van polygamie is. We zullen dit idee
testen binnen 1 familie vogels (Tetraonidae).
In onderstaande table staan voor 18 soorten vermeld of de sort lekking gedrag
vertoont en of ze seksueel dimorph is in lichaamsgrootte.
(0 = kenmerk afwezig; 1 = kenmerk aanwezig; ? = gegevens ontbreken)
species
Lekking gedrag
Seksueel dimorfisme
Centrocercus urophasianus
1
1
Dendragapus falcipennis
0
1
Dendragapus canadensis
0
1
Dendragapus obscurus
0
?
Lagopus leucurus
0
0
Lagopus mutus
0
0
Lagopus lagopus
0
0
Tetrao urogallus
1
1
Tetrao parvirostris
1
1
Tetrao textrix
1
1
Tetrao mlokosiewiczi
1
1
Bonasa sewerzowi
0
1
Bonasa bonasia
0
0
Bonasa umbellus
0
0
Tympanuchus cupido
1
1
Tympanuchus pallidicincus
1
1
Tympanuchus phasianellus
1
1
Phasianinae
0
1
De verwantschappen tussen de verschillende soorten wordt gegeven in volgende
boom (de nummers komen overeen met de verschillende soorten in de table):
Kijk eerst met klassieke statistische methoden na of er een verband is tussen het
voorkomen van lekking gedrag en seksueel dimorfisme.
Voor de fylogenetische verwerking maken we gebruik van het programma MacClade
(v. 4.0)
In een eerste stap moeten de gegevens en de fylogenetische boom ingegeven
worden in het programma. Dit gebeurt rechtstreeks in MacClade. Bij het aanmaken
van een nieuwe file kan je de gegevens intypen per soort. Na het ingeven van de
gegevens maakt het programma automatische een willekeurige boom van de 18
soorten (ga naar ‘Tree Window’ onder ‘Windows’ menu).
De boom wordt aangepast adhv de toolbox linksonder. Om taken van plaats te
veranderen gebruik je het pijltje. Vervolgens ga je op de tak die je wil verplaatsen
staan (kleurt grijzig) en verbindt je deze met de rest van de boom op de juiste plaats.
Vervolgens kan je dan je gegevens langsheen de takken opsporen. Hiervoor gebruik
je het ‘Trace’ menu – Trace character. Nu krijg je per kenmerk (in dit geval lekking
gedrag en seksueel dimorfisme) een boom met daarop de ‘evolutie’ van het kenmerk.
Het paneel rechtsonder geeft aan over welk kenmerk het gaat, wat de kleurencode
voorstelt en hiermee kan je ook van het ene naar het andere kenmerk verspringen.
De methode die we in dit voorbeeld gaan gebruiken bestaat eruit na te gaan waar er
transities (overgangen van ene staat naar de andere – bv. Van niet-lekking naar
lekking) gebeuren en welke de overeenkomstige staat is van de twee kenmerken.
Merk op dat er een tak niet kan opgelsot worden voor het kenmerk seksueel
dimorfisme. Beide oplossingen zijn even parsimonieus en er kan hier geen uitsluitsel
gegeven worden over de ancestrale vorm. In onze analyse gaan we beide
mogelijkheden bekijken. Je kan de staat van de tak aanpassen via ‘Resolving
options’ onder het ‘Trace’ menu.
Om gemakkelijk te zien waar, welke karakterstaten optreden ga je naar ‘Trace
labeling’ onder het ‘Display’ menu. Duid ‘use labels to indicate states reconstructed’
aan. Deze verschijnen nu op de boom (elk kenmerk apart).
De volgende stap is om na te gaan aan welke node er overgangen gebeuren. Als er
inderdaad een verandering heeft plaats gehad noteren we de UITEINDELIJKE staat
voor beide kenmerken. Dit wordt gedaan voor heel de boom. Let op: de taken
waarlangs geen veranderingen plaatsvinden tellen niet mee!
Of inderdaad de combinatie ‘lekking – seksueel dimorfisme’ meer voorkomt dan de
anderen, kan getest worden adhv een chi kwadraat test.











