Workshop: Van DNA tot evolutionaire boom
Deze workshop heeft als doel om aan de hand van enkele praktische voorbeelden en
bijbehorende oefeningen duidelijk te maken hoe men van DNA gegevens kan komen
tot een cladogram. Op de bijgevoegde CD zijn een begeleidende tekst, een
interactieve presentatie en de gebruikte software programma's terug te vinden.
Interpretatie van een fylogenetische boom
Op een fylogenetische boom zijn vier zaken af te lezen: 1) het tijdsaspect, 2) het
vertakkingpatroon of de cladistische verwantschappen, 3) het patristisch aspect of
met andere woorden hoe sterk soorten in een bepaalde periode zijn veranderd ten
opzichte van de oudersoort en 4) het fenetisch aspect of met andere woorden hoe
sterk soorten op een bepaald tijdstip op elkaar gelijken.
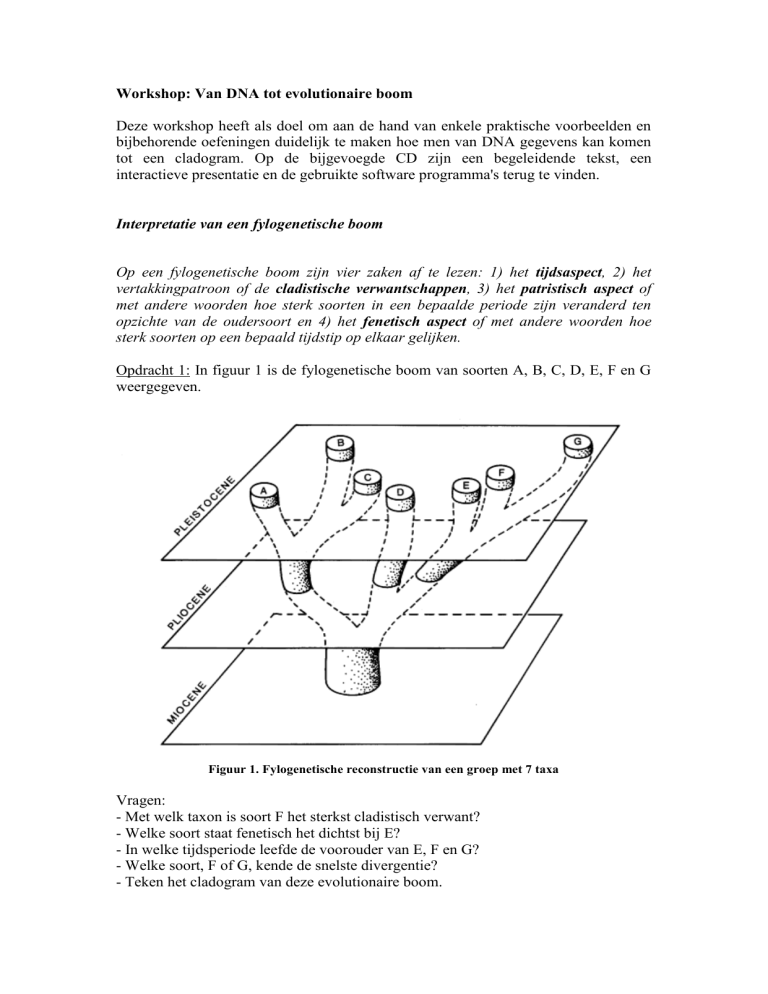
Opdracht 1: In figuur 1 is de fylogenetische boom van soorten A, B, C, D, E, F en G
weergegeven.
Figuur 1. Fylogenetische reconstructie van een groep met 7 taxa
Vragen:
- Met welk taxon is soort F het sterkst cladistisch verwant?
- Welke soort staat fenetisch het dichtst bij E?
- In welke tijdsperiode leefde de voorouder van E, F en G?
- Welke soort, F of G, kende de snelste divergentie?
- Teken het cladogram van deze evolutionaire boom.
Lengte van kenmerken en lengte van een cladogram
De lengte van een kenmerk op een bepaald cladogram is het minimaal aantal
overgangen tussen kenmerktoestanden dat volgens het cladogram vereist is om de
verspreiding van de kenmerktoestanden over de taxa te verklaren.
De lengte van een cladogram behorende bij een bepaalde gegevensmatrix is de som
van de lengte van alle kenmerken uit die matrix op dat cladogram.
Opdracht 2: Figuur 2 toont een cladogram van 4 taxa gebaseerd op de matrix
weergegeven in tabel 1. Zet de kenmerken uit op het cladogram en bepaal de lengte
van elk kenmerk. Bepaal eveneens de lengte van het gehele cladogram.
1
2
3
4
1
A
G
G
G
2
G
G
G
G
3
A
C
C
A
4
C
C
C
G
5
T
G
C
A
Tabel 1. Matrix met 4 soorten en 5 kenmerken
Figuur 2. Cladogram gebaseerd op de matrix van Tabel 1
Van DNA gegevens tot cladogram
Veranderingen in het DNA kunnen gebruikt worden om een cladogram te
reconstrueren. Indien twee taxa een bepaalde mutatie met elkaar delen, dan is de kans
groot dat deze mutatie ontstaan is in de voorouder van die twee taxa en niet twee keer
onafhankelijk van elkaar is ontstaan. Om manueel een boom op te stellen dienen
volgende stappen uitgevoerd te worden:
1) opsporen van de (fylogenetisch) informatieve kenmerken
2) opstellen van de evolutie van elk kenmerk
3) zoeken naar overlap (congruentie) tussen de evolutie van de verschillende
kenmerken
4) bepalen van de meest parsimone boom.
We zullen deze stappen illustreren aan de hand van een voorbeeld. Onderstaande
matrix omvat 11 kenmerken en 8 taxa. Elk kenmerk heeft vier kenmerktoestanden: A,
C, G, T. Taxon X en Y vormen de buitengroep.
1
2
3
4
5
6
7
8
9
10
11
X
A
C
G
T
A
T
G
G
A
G
T
Y
A
C
G
T
A
T
G
G
A
G
T
A
G
C
G
T
A
T
G
G
A
G
A
B
G
A
C
T
A
T
G
G
A
G
T
C
G
A
C
A
A
T
G
G
A
G
A
D
G
A
C
A
G
T
G
G
A
G
T
E
G
A
C
A
G
A
C
G
A
G
T
F
G
A
C
A
G
A
C
G
A
A
T
G
G
A
C
A
G
A
G
C
C
G
T
H
G
A
C
A
G
A
G
C
C
G
T
Stap 1: opsporen van informatieve kenmerken
Alle kenmerken blijken informatief behalve kenmerk 10.
Stap 2: opstellen van de evolutie van elk kenmerk
Kenmerk 1 groepeert de taxa A tot H.
De evolutie van kenmerk 2 is identiek aan de evolutie van kenmerk 3; beide
groeperen taxon B tot H.
Kenmerk 4 groepeert C tot H.
Kenmerk 5 groepeert D tot H.
Kenmerk 6 groepeert E tot H.
Kenmerk 7 groepeert E en F.
Kenmerk 8 en 9 groeperen G en H.
Kenmerk 11 groepeert A en C.
Stap 3: zoeken naar congruentie tussen de evolutie van de verschillende kenmerken
Geen enkele van de kenmerken geeft de evolutie van de taxa, deze kan enkel
opgespoord worden door de congruentie tussen de verschillende kenmerkevoluties op
te sporen.
De samenvoeging van de kenmerkevolutie van kenmerk 1, 2 en 3 laat ons toe om het
volgende cladogram te construeren: (A(B,C,D,E,F,G,H)). Houden we ook rekening
met de informatie vervat in het vierde kenmerk, dan bekomen we het volgende
cladogram (A(B(C,D,E,F,G,H))) enz.
Stap 4: Het bepalen van de meest parsimone boom
De fylogenie van kenmerk 11 is niet congruent met het cladogram gebaseerd op de
kenmerken 1 tot 10. Er zijn twee mogelijk oplossingen. Bij afwezigheid van
congruentie zal men de meest spaarzame (= parsimone) oplossing verkiezen.
Opdracht 3: In Tabel 2 is een matrix weergegeven met vier taxa en 10 kenmerken met
'out' als buitengroep en taxon 1 tot 3 als binnengroep.
Vragen:
- Welke kenmerken zijn informatief?
- Wat is de evolutie voor elk kenmerk afzonderlijk?
- Wat is het meest spaarzame cladogram?
- Wat is de lengte van de verschillende kenmerken op het bekomen cladogram en wat
is de lengte van het cladogram?
Out
1
2
3
1
C
A
A
A
2
T
T
T
T
3
A
A
C
C
4
C
C
C
C
5
C
C
T
T
6
G
T
T
T
7
C
C
A
A
8
T
T
T
T
9
A
A
A
A
10
C
A
C
A
Tabel 2. Matrix met 4 soorten en 10 kenmerken
Opdracht 4 (optioneel): In tabel 3 is een matrix weergegeven met 8 taxa en 10
kenmerken. Het eerste taxon vormt de buitengroep.
O
1
2
3
4
5
6
7
1
A
A
A
A
C
C
C
C
2
G
G
G
G
T
T
T
G
3
T
T
T
T
A
A
T
T
4
G
C
C
G
G
G
G
G
5
T
C
T
T
T
T
T
T
6
C
G
G
C
C
C
C
C
7
T
A
A
T
A
A
A
A
Tabel 3. Matrix met 8 soorten en 10 kenmerken
8
C
C
C
C
T
C
C
C
9
T
A
A
A
A
A
A
A
10
C
C
C
C
C
G
G
G
Vragen:
- Welke kenmerken zijn informatief?
- Wat is de evolutie voor elk kenmerk afzonderlijk?
- Wat is het meest spaarzame cladogram?
- Wat is de lengte van de verschillende kenmerken op het bekomen cladogram en wat
is de lengte van het cladogram?
Het gebruik van computerprogramma's voor het zoeken naar de meest parsimone
boom
Bovenstaande opdrachten hebben hopelijk duidelijk gemaakt hoe men met behulp van
cladistiek de verwantschappen tussen taxa kan achterhalen. De voorbeelden waren
echter misleidend in het feit dat ze heel weinig taxa en kenmerken omvatten. In de
praktijk willen we vaak de evolutie van enkele tientallen of zelfs honderden soorten
ophelderen en maken we gebruik van enkele honderden tot duizenden kenmerken. Het
manueel bepalen van de meest spaarzame boom is dan ook uitgesloten. Maar ook
computers hebben het er moeilijk mee. Het aantal berekeningen nodig om tot de meest
spaarzame boom te komen neemt immers exponentieel toe met het aantal taxa dat
onderzocht wordt. Er zijn echter specifieke computeralgoritmes uitgewerkt om ook in
die gevallen nog steeds een goede kans te hebben om de meest spaarzame boom te
vinden.
Tot slot van deze workshop zullen we kort PHYLIP introduceren. PHYLIP is een
software pakket dat op http://evolution.genetics.washington.edu/phylip.html
afgehaald kan worden. Het bestaat uit verschillende programma's waarvan we er hier
drie zullen bespreken. Deze zijn ook terug te vinden op de bijgevoegde CD en kunnen
vrij voor educatieve doelstellingen gebruikt worden.
Opdracht 5: Opstellen van een file die door de PHYLIP programma's gelezen kunnen
worden
Om het programma uit te testen en onze eigen resultaten te controleren, gebruiken we
de matrix met taxa A tot G en 20 DNA kenmerken die we eerder in deze workshop
hebben gebruikt. Om door het programma gelezen te kunnen worden moet het
bestand er als volgt uitzien:
8 20
Out
Taxon_A
Taxon_B
Taxon_C
Taxon_D
Taxon_E
Taxon_F
Taxon_G
ATTACTTAGTAAACCATAGT
ACTCCTTGGTAAACCATAGT
ACTCCTCGGTAAACCATAGT
ACTCCTCGGTAAACCATAGT
ACTAATTAGTAAACCATAGC
ACTAATTAGCAAACCATAGT
TCTAATTAGCAAACCATAGT
TCTAATTAGCAAACCATAGT
De eerste regel vermeldt het aantal taxa en het aantal kenmerken. Vervolgens volgt de
matrix met de taxonnaam die 10 tekens moet omvatten (kortere namen kunnen
eventueel met spaties worden aangevuld zoals in bovenstaand voorbeeld het geval is)
gevolgd door de kenmerktoestanden voor elk kenmerk. Ontbrekende gegevens worden
aangeduid met een streepje (-), onzekere waarden met een vraagteken (?). Het
bestand moet als een txt file bewaard worden en in dezelfde folder geplaatst worden
als de PHYLIP programma's waarmee gewerkt zal worden.
Opdracht 6: Uitvoeren van een cladistische analyse met behulp van het PHYLIP
programma dnapenny
Plaats het bestand die je hierboven hebt aangemaakt in de PHYLIP folder. Dubbel
klik op het dnapenny icoon. Het programma probeert automatisch een file met de
naam ‘infile’ te openen. Indien jouw file een andere naam heeft, dan kan je die naam
intypen.
Het programma geeft nu een lijst met opties. Je hoeft deze niet te wijzigen voor deze
opdracht en je kan dus ‘Y’ typen.
Het programma zal nu de kortste boom berekenen en schrijft deze weg in de
bestanden ‘outtree’ en ‘outfile’ die in de PHYLIP folder bewaard zullen worden.
Indien je het bestand ‘outtree’ opent, krijg je de berekende boom te zien in
haakjesnotatie. Het bestand ‘outfile’ geeft de bomen visueel weer.
Om de berekende boom beter te visualiseren moeten we een tweede PHYLIP
programma opstarten, namelijk drawgram.
Dubbel klik op het icoontje van drawgram. Het programma probeert een bestand te
vinden met de naam ‘intree’, zoniet wordt gevraagd de naam van het bestand in te
typen, in ons geval ‘outtree’.
Je krijg opnieuw een aantal keuzemogelijkheden, die vooral te maken hebben met hoe
de boom er grafisch uit zal zien. Druk ‘Y’ en bekijk de boom. Is hij gelijk aan de
boom die wij manueel hebben opgesteld?
Opdracht 7: Evolutie van de primaten
Het bestand ‘primaten’ bevat een matrix van 12 taxa en 898 kenmerken. De volgende
genera zijn vertegenwoordigd in de matrix: Lemur (lemuren), Homo (waartoe de
mens behoort), Pan (waartoe de bonobo’s en de chimpansees behoren), Gorilla
(gorilla’s), Pongo (orang-oetans), Hylobates (gibbons), Macaca (makaken; in de
matrix vertegenwoordigd door 4 soorten), Saimiri (doodshoofdaapjes) en Tarsius
(spookdiertjes). Open deze matrix in het programma Notepad om je nog eens
vertrouwd te maken met het bestandsformaat van PHYLIP.
Vragen:
Wat is de fylogenie van de primaten volgens deze DNA gegevens? Voer hiertoe een
cladistische analyse uit met het programma dnapenny en open het bestand ‘outfile’.
Hoeveel cladogrammen bekomen we? Hoe kan je dat verklaren?
Welke geslacht is de zustersoort van de mens volgens de bekomen cladogrammen?
Van welke groep is de orang-oetan de zustersoort?
Vormen de makaken een natuurlijke (= monofyletische) groep?










