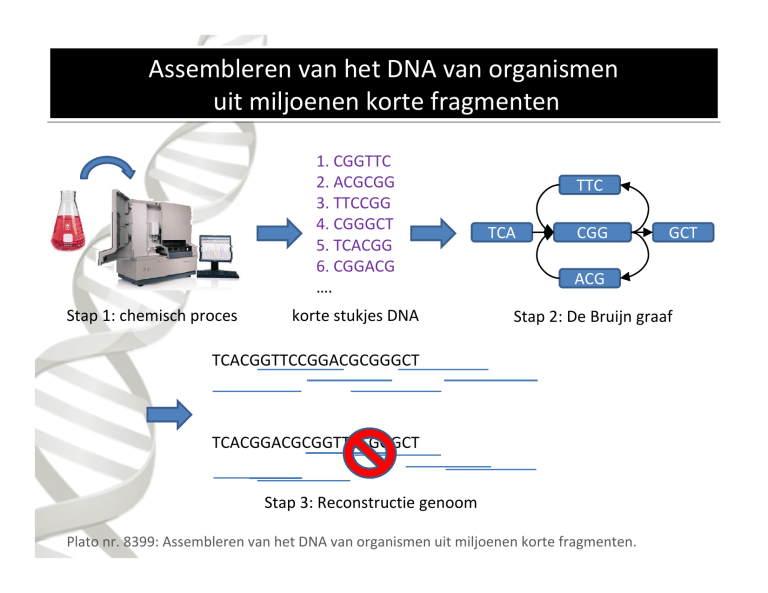
Assembleren van het DNA van organismen
uit miljoenen korte fragmenten
1. CGGTTC
2. ACGCGG
3. TTCCGG
4. CGGGCT
5. TCACGG
6. CGGACG
….
Stap 1: chemisch proces
korte stukjes DNA
TTC
TCA
CGG
GCT
ACG
Stap 2: De Bruijn graaf
TCACGGTTCCGGACGCGGGCT
TCACGGACGCGGTTCCGGGCT
Stap 3: Reconstructie genoom
Plato nr. 8399: Assembleren van het DNA van organismen uit miljoenen korte fragmenten.
Uitdagingen
Doelstellingen
Zeer grote datasets
• miljoenen tot miljarden reads
• enkele tot honderden GBytes
aan input data
• Parallelisatie (MPI, MapReduce)
• DNA data compressie
Data bevat fouten
• sample contamination
• sequencing fouten
• insert length fouten
• Algoritmes voor de detectie en correctie van fouten.
Genoom bevat herhalingen
• Verschillende reconstructies
zijn mogelijk.
• Gebruik maken van paired‐end reads om correct genoom te
assembleren.
samenwerking met
Plato nr. 8399: Assembleren van het DNA van organismen uit miljoenen korte fragmenten.
Enhanced suffix arrays: Geheugenefficiënte indexstructuren in de bioinformatica
Contact: [email protected]
DNA bevat de informatie voor eiwitsynthese
Het uitlezen van die informatie
gebeurt in een tweestapsproces:
Het coderend DNA (1% van genoom) is het bouwplan voor een bepaald eiwit.
Op het niet‐coderend DNA binden eiwitten die:
‐ De transcriptiemolecules helpen positioneren
‐ De transcriptiesnelheid beïnvloeden
Het zoeken van de locaties in het DNA waar deze
regulerende eiwitten binden is een uitdagend
computationeel probleem in de bioinformatica.
Plato nr. 8394: Enhanced suffix arrays: geheugenefficiënte indexstructuren in de bioinformatica.
Indexstructuren lenen zich ertoe om op efficiënte manier DNA te doorzoeken.
Het nadeel van indexstructuren is dat ze veel
geheugen vereisen:
40 – 70 Byte 120 a 200 GByte voor
per karakter
volledig genoom!!
Enhanced suffix arrays (ESA) zijn veel
compacter maar de bijhorende algoritmes een
stuk uitdagender!
Ontwerpt een duurzaam software‐
pakket gebaseerd op ESA.
Werkt mee aan geavanceerde
detectie‐algoritmen.
Mogelijkheid tot een doctoraat.
Plato nr. 8394: Enhanced suffix arrays: geheugenefficiënte indexstructuren in de bioinformatica.
Graafgebaseerde similariteitsmaten voor de detectie van functioneel gelijkaardige genen in een gen‐interactienetwerk
• Genen zijn stukken functioneel DNA
– Genactiviteit bepaalt de functie van een cel in een organisme
– Produceren proteïnen of bouwstenen voor DNA verwerking
– Werken niet zelfstandig: • genen participeren in complexe biologische processen door te interageren met omgevingsfactoren en andere genen => pathways (ketens) van genen die bijvoorbeeld verantwoordelijk zijn voor voortplanting, ziekteontwikkeling, celwandconstructie, …
– Aflijnen van pathways is cruciaal in het begrijpen van celmechanismen, het ontwikkelen van geneesmiddelen, het gericht kruisen van planten om de droogteresitentie te verhogen, …
Plato nr. 8401: Graafgebaseerde similariteitsmaten in een gen‐interactienetwerk. Doel
•
Genen kunnen voorgesteld worden in een interactiegraaf
– Gen‐gen interacties kunnen gedownload worden uit publieke databases, voor verschillende organismen (b.v. gist, muis of mens)
•
Stelling: “nabijheid in een interactiegraaf is een maat voor de functionele similariteit van genen”
•
We onderzoeken of bepaalde nabijheidsmaten gebruikt kunnen worden om genen te identificeren die gelijkaardige biologische functies hebben
– Kortste paden
– Maximum flow
– Random walks
– Kernels op grafen
=> Implementatie (c++, matlab) en testing met nadruk op performantie en schaalbaarheid
•
Evaluatie aan de hand van publieke databases met gekende genfuncties
Plato nr. 8401: Graafgebaseerde similariteitsmaten in een gen‐interactienetwerk. Zoeken naar graafmotieven met behulp van
parallelle programmeertechnieken
• Genen interageren en beinvloeden mekaar
• Clusters van functioneel gerelateerde genen werken als “biologische eenheden”.
• Netwerken zijn groot (6.000 knopen en 80.000 interacties)
Plato nr. 8402: Zoeken naar graafmotieven met behulp van parallelle programmeertechnieken.
Doel
• Stap 1: het identificeren van elementaire graafmotieven in een biologisch
interactie netwerk.
• Stap 2: clusteren van graafmotieven in betekenisvolle biologische units.
• Optimaliseren van een score‐functie.
• Stap 3: het ontwikkelen van een parallel algoritme dat gebruik kan maken
van een computer cluster om stap 1 en 2 aanzienlijk te versnellen.
Plato nr. 8402: Zoeken naar graafmotieven met behulp van parallelle programmeertechnieken.












