Single nucleotide polymorfismen in genen betrokken in
miRNA biogenese en HPV status als biomerkers voor
predictie van radiotherapie outcome in patiënten
behandeld voor oropharynx kanker
Laura De Backer
Verhandeling ingediend tot
het verkrijgen van de graad van
Master in de Biomedische Wetenschappen
Promotor: Prof. Dr. H. Thierens
Vakgroep Medische Basiswetenschappen
Academiejaar 2012-2013
1
2
Single nucleotide polymorfismen in genen betrokken in
miRNA biogenese en HPV status als biomerkers voor
predictie van radiotherapie outcome in patiënten
behandeld voor oropharynx kanker
Laura De Backer
Verhandeling ingediend tot
het verkrijgen van de graad van
Master in de Biomedische Wetenschappen
Promotor: Prof. Dr. H. Thierens
Vakgroep Medische Basiswetenschappen
Academiejaar 2012-2013
3
“De auteur en de promotor geven de toelating deze masterproef voor consultatie beschikbaar
te stellen en delen ervan te kopiëren voor persoonlijk gebruik. Elk ander gebruik valt onder
de beperkingen van het auteursrecht, in het bijzonder met betrekking tot de verplichting
uitdrukkelijk de bron te vermelden bij het aanhalen van resultaten uit deze masterproef.”
16 mei 2013
HANDTEKENING STUDENT
Laura De Backer
HANDTEKENING PROMOTOR
Prof. Dr. H. Thierens
4
Voorwoord
Deze thesis is tot stand gekomen dankzij de hulp en steun van verschillende mensen die ik
hier graag zou bedanken.
Mijn eerste woord van dank gaat uit naar mijn promotor Prof. Dr. H. Thierens en mijn
begeleidster Kim De Ruyck om mij de mogelijkheid te geven dit onderzoek uit te voeren.
Dankzij hun heb ik ook de kans gekregen om een deel van dit onderzoek in Maastricht uit te
voeren. Dit was nooit mogelijk geweest zonder hun steun en vertrouwen. Bedankt!
Een speciaal woord van dank gaat hierbij uit naar mijn begeleidster Kim De Ruyck. Zij heeft
met veel geduld alle technieken aangeleerd en was steeds bereid om vragen te beantwoorden
en te helpen. Ook Sofie De Langhe zou ik graag bedanken voor de hulp wanneer nodig.
Daarnaast ook een dikke ‘merci’ aan iedereen van de dienst Medische Fysica (INW). Door de
aangename sfeer die steeds aanwezig was, was het zeer leuk werken op het labo.
Bedankt aan de dienst Pathologie van het UZ Gent, om mee te willen werken aan dit project.
Ook de Universiteit Maastricht, het Maastro Research Lab en de dienst Pathologie van het AZ
Maastricht, bedankt om mij te verwelkomen in jullie labo en mij de technieken aan te leren.
De meisjes van de Medische Stralingswetenschappen zijn de volgende die ik graag zou
bedanken. We zaten elke dag samen in het labo te werken en te schrijven. Een leuke babbel,
etentjes, muziek en het vele lachen tussendoor maakte het werken aan die thesis veel
aangenamer.
Mijn vrienden zou ik ook graag bedanken, omdat ze er steeds voor mij waren. Mijn
kotgenoten verdienen hierbij een speciaal woord van dank. Dankzij hun had ik steeds een
leuke ‘thuis’ om naar terug te keren na een lange dag in het labo.
Mijn ouders. Bedankt om mij de kans te geven om Biomedische Wetenschappen te studeren.
Zonder jullie steun was dit mij nooit gelukt.
En als laatste Stijn. Om naar mij te luisteren, mij te steunen en in mij te geloven. Maar vooral
bedankt om mij steeds weer aan het lachen te brengen.
Zonder jullie allemaal was deze thesis niet mogelijk geweest. - BEDANKT!
5
Inhoud
Afkortingen ............................................................................................................................9
Samenvatting ..........................................................................................................................1
Inleiding .................................................................................................................................2
1. Hoofd- en halskanker .....................................................................................................2
1.1 Anatomie .................................................................................................................2
1.2 Risicofactoren..........................................................................................................3
1.2.1 Alcohol en tabak ..............................................................................................3
1.2.2 Virussen ...........................................................................................................3
1.2.3 Andere risicofactoren .......................................................................................3
1.3 Orofaryngeale kanker ..............................................................................................4
2. Het Humaan Papillomavirus ...........................................................................................5
2.1 Mechanisme van HPV-geïnduceerde carcinogenese .................................................5
2.1.1 Proteïne E5.......................................................................................................6
2.1.2 Proteïnen E6 en E7 ...........................................................................................6
2.1.2.1 Proteïne E6...........................................................................................6
2.1.2.2 Proteïne E7...........................................................................................7
2.1.2.3 Verdere interacties van E6 en E7 ..........................................................7
2.2 HPV gerelateerde tumoren en betere prognose .........................................................8
2.3 Detectie van HPV ....................................................................................................9
3. MicroRNA’s ................................................................................................................ 10
3.1 MicroRNA’s: functie ............................................................................................. 10
3.2 Biosynthese ........................................................................................................... 11
3.3 MicroRNA’s, HPV en orofaryngeale kanker .......................................................... 12
4. Single Nucleotide Polymorfismen ................................................................................ 12
4.1 SNPs in biosynthese genen .................................................................................... 12
4.2 SNPs in pre-microRNA genen ............................................................................... 13
6
4.2.1 pre-microRNA146a rs2910164 G>C .............................................................. 14
4.2.2 pre-microRNA196a2 rs11614913 C>T ........................................................... 14
4.3 SNPs in target genen.............................................................................................. 14
4.3.1 KRAS rs61764370 T>G ................................................................................. 14
4.3.2 SMC1B rs3747238 A>G ................................................................................ 15
5. Probleem- en doelstelling ............................................................................................. 15
Materialen en methoden ....................................................................................................... 17
1. Introductie .................................................................................................................... 17
2. Studiepopulatie ............................................................................................................ 17
3. DNA purificatie (Puregene kit)..................................................................................... 17
4. DNA isolatie uit buffy coats ......................................................................................... 18
5. Concentratie- en kwaliteitsbepaling van het DNA ........................................................ 19
6. Polymerase Chain Reaction (PCR) ............................................................................... 20
6.1 Principe ................................................................................................................. 20
6.2 PCR mix ................................................................................................................ 20
6.3 PCR primers .......................................................................................................... 21
6.4 PCR controle ......................................................................................................... 22
7. SNP bepaling ............................................................................................................... 22
7.1 Restriction Fragment Length Polymorphism (RFLP) .................................................. 22
7.2 High Resolution Melting (HRM) ........................................................................... 23
7.3 Geoptimaliseerde protocols.................................................................................... 25
8. Linkage Analyse .......................................................................................................... 25
9. Kwaliteitscontroles....................................................................................................... 25
10. HPV status bepaling ................................................................................................... 26
10.2 Polymerase Chain Reaction (PCR)....................................................................... 26
10.2.1 DNA isolatie uit tumormateriaal ................................................................... 26
10.2.2 PCR mix ...................................................................................................... 26
7
10.2.3 PCR controle ................................................................................................ 27
10.2.4 ELISA .......................................................................................................... 27
10.3 Chromogenic In Sity Hybridisatie ........................................................................ 28
10.4 p16-Immunohistochemie ..................................................................................... 28
11. Statistische analyse..................................................................................................... 29
Resultaten............................................................................................................................. 29
1. Optimalisatie protocols ................................................................................................ 30
1.1 XPO5 rs2227301 G>A........................................................................................... 30
1.2 XPO5 rs699937 C>T ............................................................................................. 31
1.3 SMC1B rs3747238 A>G........................................................................................ 31
1.4 Drosha rs17410035 C>A ....................................................................................... 32
1.5 Pre-microRNA146a rs2910164 G>C ..................................................................... 33
1.6 Pre-microRNA196a2 rs11614913 C>T .................................................................. 33
2. Patiënt karakteristieken ................................................................................................ 33
3. Associatie met overleving (OS, DFS, DSS) .................................................................. 34
3.1 Univariate Kaplan-Meier analyse van patiënt-gerelateerde en klinische factoren met
overleving. .................................................................................................................. 34
3.2 Univariate Kaplan-Meier analyse van associatie tussen miRNA genotypes en
overleving ................................................................................................................... 37
4. Associatie met tumor HPV status ................................................................................. 40
4.1 Associatie van tumor HPV status met patiënt-gerelateerde en klinische factoren.... 40
3.4 Associatie van tumor HPV status met microRNA genotypes.................................. 42
5. Bepaling van de tumor HPV status met behulp van CISH en p16-IHC ......................... 42
Discussie .............................................................................................................................. 43
Besluit .................................................................................................................................. 46
Referentielijst ....................................................................................................................... 47
8
Afkortingen
°C
Celsius
µl
Microliter
2j-OS
2jaar-Overall Survival
3’UTR
3’-Untranslated Regio
3DRT
3D Radiotherapie
3j-OS
3jaar-Overall Survival
A
Absorbantie
A
Adenine
AGO
Argonaut
AL
Antilichaam
AZ
Academisch Ziekenhuis
BC
Buffy Coat
Bp
Basepaar
BSA
Bovine Serum Albumine
C
Cytosine
Cdk-4
Cyclin-Dependent Kinase
CISH
Chromogenic In Situ Hybridisation
CSF1-R
Colony Stimulating Factor 1-Receptor
DFS
Disease Free Survival
DNA
DeoxyRiboNucleicAcid
DSS
Disease Specific Survival
E6AP
E6 Associated Protein
9
EBV
Epstein-Barr Virus
EGFR
Epidermal Growth Factor Receptor
ELISA
Enzyme-Linked ImmunoSorbent Assay
EtBr
Ethidiumbromide
FWD
Forward
G
Guanine
GP5+/6+
General Primers 5+/6+
H2O
Water
HPV
Humane Papillomavirus
HPV-
Humane Papillomavirus negatief
HPV+
Humane Papillomavirus positief
HR
High Risk
HRM
High Resolution Melting
hTERT
Human Telomerase Reverse Transcriptase
IHC
Immunohistochemistry
IMRT
Intensity-Modulated Radiation Therapy
ISH
In Situ Hybridisation
K-RAS
Kirsten Rat Sarcoma
KT
Kamertemperatuur
LD
Linkage Disequilibrium
MAF
Minor Allel Frequency
MgCl2
Magnesiumchloride
Min.
Minuut
10
miRNA
Micro Ribonucleic Acid
Ml
Milliliter
mRNA
Messenger Ribonucleic Acid
NFX1
Nuclear Transcription Factor, X-box binding 1
N-stage
Nodal-stage
Nt
Nucleotide
OPC
Oropharyngeal Cancer
OS
Overall Survival
PCR
Polymerase Chain Reaction
PDGFβ-R
Platelet-Derived Growth Factor β-Receptor
PFS
Progression Free Survival
Pre-miRNA
Precursor Micro Ribonucleic Acid
Pri-miRNA
Primair Micro Ribonucleic Acid
PY
Pack-Years
RAS
Rat Sarcoma
Rb
Retinoblastoma
RBC
Rode bloedcel
REV
Reverse
RFLP
Reverse Fragment Lenght Polymorphism
RISC
RNA Inducing Silencing Complex
RNA
Ribonucleic Acid
SMC1B
Structural Maintenance of Chromosomes 1B
SNP
Single Nucleotide Polymorphism
11
T
Thymine
Ta
Annealing Temperatuur
TE-buffer
Tris-EDTA-buffer
Tm
Melting Temperatuur
TNF
Tumor Necrosis Factor
T-stage
Tumor-stage
U
Unit
UV
Ultraviolet
UZ
Universitair Ziekenhuis
V
Volt
XPO5/Ran-GTP complex
Exportin5/Ran-Guanosinetrifosfaat complex
12
Samenvatting
Orofaryngeale kankerpatiënten kunnen ingedeeld worden in twee klinisch en biologisch
verschillende groepen naargelang hun prognose na behandeling. Hierbij wordt een
onderscheid gemaakt tussen HPV+ en HPV- orofaryngeale kankerpatiënten, waarbij de HPV+
patiënten een betere overleving vertonen in vergelijking met de HPV- patiënten. Om deze
patiënten nog beter in te delen in verschillende groepen naar prognose na behandeling, wordt
er in deze studie gezocht naar additionele, niet-tumorale biomerkers. Hiervoor worden 10
SNPs in microRNA genen bepaald in 72 orofaryngeale kankerpatiënten en worden de
genotypes, samen met de tumor HPV status, gelinkt aan overleving (Overall Survival (OS),
Disease Free Survival (DFS), Disease Specific Survival (DSS) ). De associatie tussen de
miRNA SNPs, patiënt-gerelateerde factoren, klinische factoren en de tumor HPV status wordt
eveneens bestudeerd. Er worden 6 SNPs in 2 biosynthese genen onderzocht, waarvan 4 SNPs
in het Drosha gen (rs639174 , rs3805500, rs7719666 en rs17410035) en 2 SNPs in het XPO5
gen (rs2227301 en rs699937). Daarnaast worden 2 SNPs worden in 2 pre-microRNA genen
onderzocht, pre-microRNA146a (rs2910164) en pre-microRNA196a2 (rs11614913), en 2
SNPs in microRNA targetgenen: KRAS (rs61764370) en SMC1B (rs3747238). De tumor
HPV status werd bepaald met behulp van 2 technieken: p16-Immunohistochemische kleuring
(p16-IHC) en In Situ Hybridisatie (ISH), die eveneens onderling met elkaar vergeleken
worden. Enkele statistisch significante associaties werden terug gevonden. Allereerst werd
een associatie gevonden tussen een SNP in het Drosha gen (rs3805500 T>C) en overleving
(OS, DFS). Patiënten die drager zijn van het CC genotype vertonen een slechtere OS (3-jaar
OS = 22,2 %) en DFS in vergelijking met patiënten drager van het TT/TC genotype (3-jaar
OS = 69,6%). Patiënten met een T1/2/3 tumor vertonen een betere OS en DFS in vergelijking
met patiënten met een T4 tumor. Het aantal drinks/week vertoont eveneens een significante
associatie met overleving: patiënten die minder dan 10 drinks/week consumeerden, vertonen
een betere OS, DFS en DSS in vergelijking met patiënten die 10 of meer drinks/week
nuttigden. Sterk significante associaties werden gevonden tussen HPV status en rookgedrag,
alcoholgebruik en de 2-jaar OS. HPV+ patiënten worden gekenmerkt door een betere 2j-OS
en blijken eerder niet zware rokers en drinkers te zijn. Deze studie kon bevestigen dat HPV+
en HPV- patiënten als 2 aparte entiteiten met verschillende karakteristieken (overleving, rooken drinkgedrag) beschouwd kunnen worden. Daarnaast is een additionele biomerker
gevonden, die toelaat patiënten nog beter in te delen naar overleving.
1
Inleiding
1. Hoofd- en halskanker
Hoofd- en halskanker, waartoe orofaryngeale kanker behoort [1], is wereldwijd de zesde
meest voorkomende kanker met meer dan 500 000 nieuwe gevallen die per jaar worden
vastgesteld [2]. In 2008 kregen in België 2515 patiënten (1935 mannen en 580 vrouwen) de
diagnose van hoofd- en halskanker. Dit maakte hoofd- en halskanker in België voor mannen
de 4e en voor vrouwen de 9e meest frequente vorm van kanker [3]. In Nederland ging dit in
2008 om 2863 gevallen (1961 mannen en 902 vrouwen) [4]. De 5-jaaroverleving is 50% [2]
en diegenen die overleven, hebben een levenslang verhoogd risico om te sterven aan cardiale
en respiratorische ziekten en op het ontstaan van secundaire tumoren. Hierbij is de long de
meest voorkomende plaats van metastase, gevolgd door de mediastinale lymfeknopen, lever
en bot [5]. Hoofd- en halskanker wordt 2-4x meer bij mannen dan bij vrouwen vastgesteld [6]
en de gemiddelde leeftijd van diagnose is 60 jaar [5].
1.1 Anatomie
Hoofd- en halskanker is een verzamelnaam voor kankers die ontstaan in verschillende
anatomische regio’s: de mondholte, de orofarynx, de nasofarynx, de hypofarynx en de larynx
[7]. Zo goed als al deze kankers zijn squameuze cel carcinoma’s [5].
Figuur 1. Hoofd- en halskanker: de anatomische regio’s [12]
2
1.2 Risicofactoren
1.2.1 Alcohol en tabak
Naast besmetting met het Humane Papillomavirus zijn de voornaamste risicofactoren voor
het ontwikkelen van hoofd- en halskanker het gebruik van tabak en consumptie van alcohol
[5]. Dit door de directe effecten van nicotine en polycyclische aromatische hydrocarbonaten
bij het gebruik van tabak [8] en acetaldehyde, een metaboliet in de afbraak van ethanol, bij
alcoholconsumptie [9]. Hevige rokers hebben 5-25x meer kans op het ontwikkelen van hoofden halskanker en in combinatie met het gebruik van alcohol stijgt het risico verder [8].
Tabakgebruik en alcoholconsumptie verhogen dus afzonderlijk het risico, maar samen
vertonen ze een multiplicatief gecombineerd effect [5]. Eveneens is aanzienlijke blootstelling
aan zowel alcohol als tabak geassocieerd met mutaties in het p53 tumor suppressorgen [8].
1.2.2 Virussen
Er zijn twee types virussen die gelinkt worden aan het ontstaan van kanker in de hoofd- en
halsregio: het Epstein-Barr virus (EBV) en het Humane Papillomavirus (HPV). Besmetting
met het Epstein-Barr virus wordt geassocieerd met kanker ter hoogte van de nasofarynx,
terwijl besmetting met het Humane Papillomavirus geassocieerd is met het ontstaan van
orofaryngeale kanker [10]. Hoewel het gebruik van tabak en consumptie van alcohol de
voornaamste risicofactoren zijn voor het ontwikkelen van hoofd- en halskanker, ziet men de
laatste jaren een daling in incidentie van hoofd- en halskanker geassocieerd met het gebruik
van tabak en alcohol [7,11], en een sterke toename van HPV+ orofaryngeale kanker
(geïllustreerd in figuur 2) [7]. Deze verschuivingen in incidentie zijn vooral te verklaren door
de bewustmaking van de volksgezondheid en de antitabak campagnes enerzijds en door de
verandering in levensstijl en seksueel gedrag anderzijds [6]. Zo zijn HPV+ tumoren sterk
geassocieerd met seksueel gedrag [10], waaronder het aantal vaginale en orale seksuele
partners [7]. De prevalentie van HPV+ hoofd- en halskanker varieert van 20% tot 60% tussen
de verschillende publicaties [11].
1.2.3 Andere risicofactoren
Andere risicofactoren die geassocieerd zijn met hoofd- en halskanker zijn vitamine A
deficiëntie, ijzerdeficiëntie bij het Plummer-Vinson syndroom en blootstelling aan bepaalde
stoffen zoals asbest, nikkel en radium [8].
3
Figuur 2. Incidentie HPV+ en HPV- orofaryngeale kanker [7]
1.3 Orofaryngeale kanker
De orofarynx is het gedeelte van de farynx achter de mond en omvat het achterste 1/3e van de
tong, het zachte verhemelte, de zij- en achterwanden van de keel en de keelamandelen [12].
Op basis van de risicofactoren kunnen er twee biologisch en klinisch verschillende groepen
van elkaar onderscheiden worden: HPV+ orofaryngeale tumoren en HPV- orofaryngeale
tumoren [7]. HPV+ orofaryngeale kankerpatiënten zijn doorgaans jonger bij aanvang van de
eerste symptomen [1], blanke mannen, infrequente of niet-rokers en matige tot nietalcoholische drinkers. HPV- orofaryngeale kankerpatiënten daarentegen worden gekenmerkt
door een oudere leeftijd en een grote associatie met het gebruik van tabak en consumptie van
alcohol [7]. Een ander belangrijk verschil tussen deze groepen is de overleving na
behandeling [13]. Zo vertonen HPV+ patiënten een betere Progression Free (PFS) en Overall
Survival (OS) in vergelijking met de HPV- patiënten. In de studie van Ang et al. bedroeg de 3
jaar OS na chemoradiotherapie (cisplatine (100 mg/m2) in combinatie met radiotherapie) voor
HPV+ patiënten 82,4%, terwijl deze voor de HPV- patiënten nog slechts 57,1% bedroeg.
Eenzelfde tendens kon worden opgemerkt voor de 3 jaar PFS (73,3% vs. 43,4%
respectievelijk) [6]. Bij HPV+ orofaryngeale kanker patiënten wordt de OS eveneens
gedetermineerd door het aantal pakjaren tabak gebruik ( ≤ 10 vs. > 10) en de nodal stage (Nstage: N0-N2a vs. N2b-N3). De tumor stage (T-stage: T2-T3 vs. T4) en het aantal pakjaren
tabakgebruik bepaalt de OS bij de HPV- orofaryngeale kankerpatiënten [14]. Op basis van
4
deze factoren konden beide studies de patiënten indelen in drie groepen, naargelang de kans
op sterfte: laag, gemiddeld en hoog risico [6,14]. p16 wordt hier gebruikt als biomerker voor
HPV infectie [14].
Figuur 3. Indeling van de patiënten in 3 categorieën naargelang HPV status, aantal pakjaren en N- of T-stage
[14]
2. Het Humaan Papillomavirus
Het Humaan Papillomavirus (HPV) is een circulair dubbelstrengig DNA virus [10] dat
voornamelijk de basale squameuze epitheliale cellen van de orofarynx infecteert [15] met een
voorkeur voor de cellen van de verhemelte- en tongamandelen [7]. Er zijn meer dan 100
unieke HPV types gekend, die onderverdeeld worden in de types die een voorkeur hebben om
de huid te infecteren, en de types die een voorkeur hebben om de mucosa te infecteren. In
deze laatste categorie kunnen de invasieve carcinoma’s ingedeeld worden. Verder worden de
HPV types onderverdeeld in hoog-risico en laag-risico types. Goedaardige proliferaties zijn
geassocieerd met de laag-risico types, terwijl maligniteiten geassocieerd worden met de hoogrisico types. Op basis van associatie met halskanker, zijn er zo’n 15 hoog-risico of oncogene
HPV types geïdentificeerd [10], waaronder HPV 16, 18, 31, 33 en 35 [15]. HPV-16 is het
meest voorkomende type en wordt in 87-90% van de HPV+ orofaryngeale kankers terug
gevonden [10]. Deze hoog-risico types zijn in staat orale epitheliale cellen te transformeren
via E6 en E7, de virale oncoproteïnen [15].
2.1 Mechanisme van HPV-geïnduceerde carcinogenese
HPV behoort tot de familie van de Papovaviridae: kleine icosahedrale virussen [16] die
bestaan uit een viraal genoom dat ongeveer 8000 basenparen groot is [10]. Dit genoom is
samengesteld uit 3 grote regio’s: de vroege genen (E1-8), de late genen (L1-2) en een lange
coderende regio [15]. De vroege genen coderen voor 2 regulerende proteïnen (E1 en E2) en 3
oncoproteïnen (E5, E6 en E7), terwijl de late genen coderen voor 2 structurele capside
proteïnen (L1 en L2) [10]. De capside proteïnen zijn verantwoordelijk voor de initiële binding
5
tussen het virus en target cel, door hun affiniteit voor proteoglycaan heparine sulfaat groepen
op de basale membraan [7]. De oncoproteïnen zijn verantwoordelijk voor carcinogenese [10].
2.1.1 Proteïne E5
Oncoproteïne E5 is een hydrofoob proteïne dat preferentieel terug gevonden wordt in het
Golgi
apparaat
en
in
het
plasmamembraan.
E5
bindt
met
verschillende
transmembraanproteïnen, waaronder EGFR, PDGFβ-R en CSF1-R [17], en speelt een rol in
de vroege infectie door het initiëren van de celproliferatie. Tijdens de integratie van het viraal
genoom naar het gastheergenoom, wordt de coderende sequentie voor E5 vaak verwijderd,
waaruit blijkt dat E5 enkel betrokken is bij de vroege infectie en niet bij de latere stadia van
carcinogenese. Deze rol wordt vervuld door andere proteïnen, namelijk E6 en E7 [10].
2.1.2 Proteïnen E6 en E7
Oncoproteïnen E6 en E7 zijn verantwoordelijk voor carcinogenese en behoud van een
kwaadaardig fenotype [10] door degradatie van belangrijke signaalelementen. Dit doen ze
door interactie met hun PDZ-domein, een structureel domein dat terug gevonden wordt in
meer dan 400 proteïnen [7]. De belangrijkste targets van E6 en E7 zijn respectievelijk p53 en
pRb, beide tumor suppressor proteïnen [10]. Deze proteïnen zijn verantwoordelijk voor het
regelen van de celcyclus. Ze kunnen de celcyclus stoppen tijdens de G1 en G2 fase zodat de
DNA schade gedetecteerd en hersteld kan worden. Wanneer dit proces verloren gaat, wordt de
celcyclus niet gestopt en gaat deze gewoon door zonder herstel van schade. Dit kan dus
bijgevolg leiden tot een accumulatie van DNA schade en een grotere kans op ontstaan van
mutaties [18].
2.1.2.1 Proteïne E6
E6 is een oncoproteïne van 151 aminozuren lang [19] dat verschillende cellulaire targets
heeft, waarvan de belangrijkste de tumor suppressor p53 is. E6, gebonden aan E6AP (E6
Associated Proteins), bindt met p53 [20] wat een ubiquitine-gemedieerde degradatie of directe
inactivatie van p53 (via complexvorming) tot gevolg heeft [21]. Aangezien p53 een
belangrijke rol heeft in het stoppen van de celcyclus en behoud van de DNA integriteit tijdens
de celcyclus checkpoints [18], leidt degradatie van p53 tot disregulatie van deze celcyclus
checkpoints (G1/S en G2/M). Bijgevolg leidt dit door een accumulatie van mutaties [21] tot
genomische instabiliteit [10]. Dit proces wordt afgebeeld in onderstaande figuur (figuur 4).
6
2.1.2.2 Proteïne E7
E7 is een oncoproteine van 98 aminozuren lang, dat onder andere bindt met proteïnen van de
Retinoblastoma familie (Rb, p107, p130) [19], ook gekend als de pocket proteïnen [20]. Deze
proteïnen zijn verantwoordelijk voor de regulatie van de celcyclus op verschillende
checkpoints [19]. In normale, niet geïnfecteerde cellen bindt pRb aan E2F [22], een
transcriptiefactor die betrokken is in de progressie van de celcyclus naar de S-fase [21] alsook
in differentiatie, ontwikkeling, proliferatie en apoptose [19]. Wanneer E2F niet gebonden is
aan pRb, worden S-fase genen overgeschreven en vindt celproliferatie plaats [10]. Wanneer
pRb E2F bindt, worden deze genen niet overgeschreven en blokkeert de celcyclus progressie.
pRb kan geïnactiveerd worden op 2 manieren: via fosforylatie door cdk-4 [22] of door
binding aan E7. In beide gevallen gaat de binding tussen pRb en E2F verloren en vindt
progressie van de cel naar de S-fase plaats [21]. p16, een cdk-inhibitor, blokkeert de
fosforylatie van pRb door cdk-4 en blokkeert dus op deze manier de G1-S fase progressie.
Wanneer cellen geïnfecteerd zijn met HPV, gaan deze cellen p16 overexpresseren ter
compensatie van het verlies van pRb door E7. Omwille van deze overepressie, kan p16
gebruikt worden als surrogaat marker voor HPV infectie [22]. Dit proces kan terug gevonden
worden in figuur 4.
2.1.2.3 Verdere interacties van E6 en E7
Naast binding op p53 en pRb, binden E6 en E7 nog op tal van andere targets met oncogene
effecten die verder bijdragen tot genomische instabiliteit. E7 bindt zo op verschillende andere
leden van de Retinoblastoma familie, waaronder p107 en p130. Daarnaast is het eveneens in
staat te binden op p21CIP1 en p27KIP1 die, net als p16, cdk-inhibitors zijn. Binding van E7 met
de cdk-inhibitors en de overige leden van de Retinoblastoma familie leidt tot inactivatie van
deze targets met ongecontroleerde celproliferatie en carcinogenese tot gevolg. Daarnaast zorgt
E6, gebonden aan E6AP, voor degradatie van NFX1, een transcriptiefactor. Dit leidt tot
activatie van hTERT en induceert telomerase activiteit. E6 bindt eveneens met Bak, waardoor
Bak zijn apoptotische werking verliest [10]. Expressie van E6 en E7 zijn noodzakelijk voor
kwaadaardige transformatie [23] maar dit is echter niet voldoende. Er vinden bijkomende
processen plaats, maar deze zijn tot hiertoe ongekend [10].
7
Figuur 4. Mechanisme van HPV-geïnduceerde carcinogenese [10]
2.2 HPV gerelateerde tumoren en betere prognose
Zoals reeds beschreven in 1.3 is er een belangrijk verschil tussen de OS en PFS voor patiënten
met HPV+ orofaryngeale kanker en patiënten met HPV- orofaryngeale kanker. Er bestaan
verschillende hypothesen voor het verschil in prognose na behandeling. Afwezigheid van
‘field cancarization’, bewaking van het immuunsysteem voor virus-specifieke antigenen en
een intacte apoptotische respons op straling [24] kunnen als oorzaken voor een betere
overleving bij HPV+ tumoren beschouwd worden. Ook zijn HPV+ kankers minder
geassocieerd met het gebruik van tabak en alcohol, vertonen zij minder p53 mutaties, hebben
ze de neiging om focaal te blijven en is er minder kans op het ontwikkelen van tumoren in de
longen, slokdarm of elders in de hoofd- en halsregio [25]. De behandeling van orofaryngeale
kanker gebeurt voornamelijk door radiotherapie en chemoradiotherapie [26] waarbij gebruik
gemaakt wordt van stralingsgeïnduceerde celdood. Hierbij vertonen HPV+ tumoren een
grotere gevoeligheid in vergelijking met HPV- tumoren [27]. Verschillende hypothesen
worden hierrond gesteld. Vu et al. veronderstellen een verhoogde immuunrespons na
radiotherapie via verschillende mechanismen, zoals opregulatie van TNF receptors [15]
terwijl Pang et al. de verantwoordelijkheid van de stralingsgeïnduceerde gevoeligheid bij een
isovorm van E6, E6*I leggen. E6*I zorgt er namelijk voor dat de cellen gevoeliger zijn aan
stralingsgeïnduceerde apoptose door hun de toegang tot de G1 fase te verhinderen en hen in
apoptose te laten gaan [27]. Lassen et al. stellen de hypothese dat tumorcellen die p16
expresseren minder hypoxisch zijn minder snel herbevolken na bestraling [28].
8
2.3 Detectie van HPV
Aantonen van een infectie met HPV gebeurt doorgaans via 3 technieken: Polymerase Chain
Reaction (PCR), In Situ Hybridisatie (ISH) en Immunohistochemie (IHC) [7]. De voor- en
nadelen van deze technieken zijn opgesomd in tabel 1. PCR detecteert geamplificeerd viraal
mRNA of viraal DNA [11,29] en wordt door zijn hoge gevoeligheid [11] gezien als de
gouden standaard in research laboratoriums [30]. Ondanks dit grote voordeel, is deze techniek
moeilijk praktisch uitvoerbaar in de meeste klinische laboratoriums [31] gezien het complexe
weefselverwerking en expertise vereist [32]. Bovendien kan met de PCR techniek geen
onderscheid gemaakt worden tussen HPV aanwezig in neoplastische cellen en HPV aanwezig
in niet neoplastisch epitheel en stroma [29]. Zowel IHC als ISH zijn technisch minder
veeleisende methodes [7]. Bij ISH wordt het HPV DNA gevisualiseerd met behulp van
signaal amplificatie [31]. ISH overkomt bepaalde limitaties van PCR doordat ISH in staat is
om aan te tonen dat het virus zich in de tumorcellen bevindt [11]. ISH toont op deze manier
dus enkel de klinisch relevante infecties aan [29]. Hoewel ISH een hoge specificiteit kent,
heeft het een lage sensitiviteit en is het duurder in vergelijking met IHC [11]. Steeds meer
onderzoekers ijveren voor het gebruik van p16 als biomerker voor de bepaling van de HPVstatus omwille van de vele voordelen die hiermee verbonden zijn. De p16-IHC techniek is
eenvoudig uitvoerbaar, kent een lage kost en heeft een hoge sensitiviteit [33]. Daarnaast zijn
monoclonale antilichamen tegen p16 commercieel verkrijgbaar [29] en is deze techniek
beschikbaar in de meeste routine pathologische laboratoria [11].
Tabel 1. Overzicht van de voor- en nadelen van de technieken voor HPV detectie
Techniek
Voordelen
Nadelen
PCR
Hoge sensitiviteit
Moeilijk uitvoerbaar in klinische
laboratoria
Vals positieven wegens te sensitief
[11]
Lage specificiteit [29]
Contaminatie errors
Geen zekerheid over de
aanwezigheid in tumorcellen
Verknipt DNA kan primerbinding
verhinderen
9
Vervolg tabel 1. Overzicht van de voor- en nadelen van de technieken voor HPV detectie
Techniek
Voordelen
Nadelen
ISH
Technisch minder veeleisend
Lage sensitiviteit
Toont enkel klinisch relevante
Duur
infecties aan
Hoge specificiteit
p16-IHC
Technisch minder veeleisend
Lage specificiteit [34]: subset
Goedkoop
HPV-tumoren overexpresseren ook
Hoge sensitiviteit
p16
Monoclonale lichamen tegen p16
zijn commercieel verkrijgbaar
3. MicroRNA’s
Op basis van de tumor HPV status, worden patiënten met orofaryngeale kanker ingedeeld in
twee biologisch en klinisch verschillende groepen, zoals reeds vermeld in 1.3. Het verband
tussen polymorfismen in microRNA genen en kankerrisico is reeds onderzocht in
verschillende studies. Hoe deze polymorfismen bijdragen tot prognose is tot hiertoe echter
weinig bestudeerd. Deze polymorfismen zouden gebruikt kunnen worden om orofaryngeale
kanker patiënten nog beter in te delen naar prognose na behandeling.
3.1 MicroRNA’s: functie
Mature microRNA’s zijn korte, niet-coderende [35], RNA moleculen van zo’n 19-22
nucleotiden lang [36] die betrokken zijn in belangrijke biologische processen zoals apoptose,
proliferatie, differentiatie, angiogenese en immuunrespons [37]. Sequentiespecifieke binding
van microRNA’s op hun target mRNA leidt tot inhibitie van translatie en/of degradatie van dit
mRNA [36]. Op deze manier kan één enkele microRNA de expressie van 100den target
mRNA’s controleren en wordt ongeveer 1/3e van de proteïne coderende genen in het
menselijk genoom gecontroleerd door microRNA’s. Het spreekt voor zich dat disregulatie van
de normale microRNA functie, en dus disregulatie van apoptose, proliferatie, differentiatie,
angiogenese en immuunrespons, een cruciale rol speelt in het ontstaan van kanker. Zo kunnen
microRNA’s zich gedragen als tumorsuppressors of als oncogenen. Gedereguleerde
microRNA’s
zijn
op
deze
manier
geassocieerd
met
initiatie,
progressie
en
behandelingsoutcome van verschillende kankers [37], waaronder hoofd- en halskanker [38].
10
3.2 Biosynthese
Een mature miRNA molecule wordt gevormd in 3 stappen. In een eerste stap wordt het
primaire miRNA (pri-miRNA) gevormd door transcriptie van het miRNA gen met behulp van
RNA polymerase II [39]. Dit pri-miRNA is 500-3000 nucleotiden lang [36] en wordt door een
nucleair RNase, DROSHA, omgevormd tot een precursor miRNA (pre-miRNA) [39, 40] van
ongeveer 70-100 nucleotiden lang [40]. Deze dubbelstrengige haarspeldstructuur [36] wordt
getransporteerd naar het cytoplasma door het exportin 5 (XPO5)/Ran-GTP complex [40] waar
de finale stap, de maturatie, plaats vindt [39]. Hier wordt het pre-miRNA verder gesplitst door
een RNase III endonuclease, DICER, met vrijlating van 2 complementaire korte RNA
sequenties tot gevolg [40]: de mature miRNA sequentie en zijn kortlevend complementaire
sequentie aangeduid als miR* [36]. Argonaut proteïnen (AGO1-4) vormen een complex met
GEMIN3 en GEMIN4 en binden selectief op 1 van de 2 sequenties wat leidt tot vorming van
het RISC [40] (RNA-Inducing Silencing Complex [36]). De streng met het minst stabiele 5’
eind wordt preferentieel geselecteerd en geladen in het RISC [41]. Het RISC vergemakkelijkt
de binding van het miRNA op zijn target mRNA doordat het de bindingsplaats in de 3’UTR
van het target mRNA herkent en vervolgens het miRNA hierop richt [40]. Deze binding leidt
tot inhibitie van translatie en/of degradatie van het target mRNA [36]. Dit proces kan terug
gevonden worden in onderstaande figuur.
Figuur 5. MicroRNA biosynthese [36]
11
3.3 MicroRNA’s, HPV en orofaryngeale kanker
Gezien de regulerende rol van microRNA’s in apoptose en immuunrespons, kunnen
polymorfismen in microRNA’s zorgen voor een veranderde apoptotische en immuunrespons.
Deze veranderende respons kan ervoor zorgen dat HPV enerzijds ontsnapt aan apoptose en
anderzijds ontsnapt aan het immuunsysteem en op deze manier niet geklaard wordt. Op deze
manier spelen microRNA polymorfismen een rol in de ontwikkeling van orofaryngeale
kanker en verschillende outcomes zoals HPV status en overleving [42].
4. Single Nucleotide Polymorfismen
Een Single Nucleotide Polymorfisme (SNP) is een niet-repetitieve vorm van sequentie
variatie dat grotendeels verantwoordelijk is voor de variatie tussen verschillende individuen
[36]. SNPs worden geassocieerd met diversiteit binnen een populatie alsook met gevoeligheid
voor bepaalde ziekten en individuele respons op medicatie [37]. Naargelang de
basenverandering die plaats vindt, worden SNPs ingedeeld in verschillende klassen (klasse 14). Dit wordt weergegeven in tabel 2. SNPs met betrekking tot microRNA’s kunnen
voorkomen in de microRNA genen zelf, in de bindingsplaats voor het microRNA en in de
microRNA biosynthese genen [36].
Tabel 2. De SNP klassen. Bron: HRM Guide, Applied Biosystems
SNP klasse
Basenverandering
Voorkomen in het
menselijk genoom
1
C/T & G/A
64%
2
C/A & G/T
20%
3
C/G
9%
4
A/T
7%
4.1 SNPs in biosynthese genen
SNPs in biosynthese genen zijn gelinkt met kankerrisico, prognose en respons op medicatie
[36]. Verschillende studies deden reeds onderzoek naar het verband tussen SNPs in genen
betrokken in de biosynthese van microRNA’s, waaronder AGO1, GEMIN3 en Dicer, en het
ontstaan van kanker. De selectie van de biosynthese genen en de SNPs gebeurde op basis van
de studie van Zhang et al., waarin het verband werd onderzocht tussen SNPs in biosynthese
genen en het risico op secundaire tumoren en/of herval bij patiënten met hoofd- en halskanker
12
in een vroeg stadium. Er werd gekozen voor het 4 SNPs in het Drosha gen en 2 SNPs in het
XPO5 gen. Zoals reeds vermeld in 3.1 is Drosha een nucleair RNase dat instaat voor de
vorming van het pre-microRNA uit het pri-microRNA. Het gen dat codeert voor Drosha
bevindt zich op chromosoom 5. Hierin worden 4 SNPs onderzocht (rs639174 G>A,
rs3805500 T>C, rs7719666 C>T en rs17410035 C>A). Zhang et al. konden in hun studie een
verband aantonen tussen de 4 SNPs en het ontstaan van een 2e primaire tumor en/of terugkeer
in patiënten met hoofd- en halskanker in een vroeg stadium. Daarnaast konden zij ook
vaststellen dat hoe meer risicovolle genotypes een persoon bezat, hoe groter het risico op
herval werd. XPO5 is verantwoordelijk voor het transport van het pre-microRNA naar het
cytoplasma. Het gen voor XPO5 is gelokaliseerd op chromosoom 6 waarin 2 SNPs
(rs2227301 G>A en rs699937 C>T) onderzocht worden [43]. Een overzicht van de SNPs die
in dit eindwerk onderzocht worden, wordt weergegeven in onderstaande tabel.
Tabel 3. Overzicht van de SNPs in de biosynthese genen
Gen
SNP
Locatie
Basenverandering
MAF *
Drosha
rs639174
Intron
G>A
24,6 %
Drosha
rs3805500
Intron
C>T
32,5 %
Drosha
rs7719666
Intron
C>T
49,1 %
Drosha
rs17410035
3’-UTR
C>A
33,2 %
XPO5
rs2227301
3’-UTR
G>A
26,5 %
XPO5
rs699937
Intron
C>T
34,8 %
LD **
rs10520985
MAF * = Minor Allele Frequency, in Kaukasische populatie / LD ** = Linkage Disequilibrium
4.2 SNPs in pre-microRNA genen
Reeds vele studies toonden aan dat afwijkende expressie van microRNA’s geassocieerd is met
etiologie, diagnose en prognose van verschillende kankers, waaronder orofaryngeale kanker
[42]. Zo werd in de studie van Liu et al. het verband tussen 4 SNPs in 4 pre-microRNA genen
(pre-microRNA146a, -149, -196a2 en -499) en het risico op het ontwikkelen van squameuze
cell carcinoma’s in de hoofd- en halsregio onderzocht. Hoewel voor slechts 1 SNP (premicroRNA499 rs3746444 T>C) een significante associatie kon aangetoond worden, werd een
gecombineerd risico gevonden voor de 4 SNPs. Wanneer een individu de 4 risicovolle
13
genotypes bezat, had deze 1,4x meer kans op het ontstaan van hoofd- en halskanker. Ook
werd opgemerkt dat dit risico groter was voor patiënten met orofarynx kanker, wat kan wijzen
op een mogelijke interactie tussen deze pre-microRNA genen en de HPV oncogene proteïnen
[38]. Hierop ging de studie van Guan et al. verder. Zij onderzochten of bovenvernoemde
SNPs de HPV-16 status en overleving kon voorspellen in patiënten met squameuze cel
carcinoma ter hoogte van de orofarynx. Dit is de eerste studie die op zoek gaat naar
biomerkers voor prognose van patiënten met orofarynx kanker. Zij konden een significante
associatie aantonen tussen de SNPs in 2 pre-microRNA genen (pre-microRNA 146a
rs2910164 G>C en pre-microRNA 196a2 rs11614913 C>T) en HPV-16 positieve
orofaryngeale kanker alsook met overleving na behandeling [42]. Deze 2 SNPs worden in de
volgende paragrafen verder besproken.
4.2.1 pre-microRNA146a rs2910164 G>C
Deze SNP (G>C) komt voor in het pre-microRNA146a en heeft een MAF van 23,5%. Zoals
eerder vermeld konden Liu et al. geen verband aantonen tussen deze SNP en het risico op het
ontstaan van hoofd- en halskanker [38] terwijl dit door Orsos et al. wel aangetoond kon
worden [44]. Een associatie met het ontstaan van gliomen [45] en borstkanker [46] kon door
andere onderzoekers eveneens aangetoond worden. Zoals eerder vermeld, werd in studie van
Guan et al. een positieve associatie gevonden tussen deze SNP en verschillende klinische
eindpunten, waaronder OS, DFS en DSS. Een associatie kon ook aangetoond worden tussen
deze SNP en een grotere kans op aanwezigheid van een HPV-16 positieve tumor [42].
4.2.2 pre-microRNA196a2 rs11614913 C>T
Deze SNP (C>T) komt voor in het pre-microRNA196a2 en heeft een MAF van 44,2%. Ook
hier konden Liu et al. [38] geen rechtstreeks verband aantonen tussen de SNP en het risico op
squameuze cel carcinoma’s in de hoofd- en halsregio. Twee andere studies deden onderzoek
naar deze SNP in relatie met het ontstaan van kanker, waarbij zij een significante associatie
konden aantonen met het ontstaan van hoofd- en halskanker [35] en borstkanker [45]. Guan
et al. konden voor deze SNP eveneens een associatie aantonen met OS, DFS en DSS alsook
met een grotere kans op aanwezigheid van een HPV-16 positieve tumor [42].
4.3 SNPs in target genen
4.3.1 KRAS rs61764370 T>G
Deze SNP (T>G) komt voor in het 3’UTR van het KRAS gen met een MAF van 12,5%. RAS
proteïnen zijn G-proteïnen die betrokken zijn in verschillende intracellulaire pathways,
14
betrokken in proliferatie, apoptose, differentiatie, celadhesie, cytoskeletale integriteit en
celmigratie [47]. De expressie van deze proteïnen wordt op negatieve wijze gecontroleerd
door let-7 [48], een lid van de microRNA familie [49]. Mutaties in RAS worden frequent
terug gevonden in bepaalde tumoren. Wanneer deze signalisatie geïnhibeerd wordt, neemt de
gevoeligheid voor stralingsgeïnduceerde celdood toe [48]. Verschillende studies konden een
associatie aantonen tussen dragers van het variante allel en een toegenomen risico op kanker,
waaronder colorectale kanker [50] niet-kleincellig longcarcinoom [51] en ovariumkanker
[52]. Eén studie kon een associatie aantonen tussen deze SNP en overleving bij orale kanker
patiënten. Zo toonden Christensen et al. aan dat dragers van het variante allel een
verminderde overleving vertoonden in vergelijking met dragers van het wild type allel [53].
4.3.2 SMC1B rs3747238 A>G
Deze SNP (A>G) komt voor in de 3’UTR van het SMC1B gen, gelegen op chromosoom 22,
met een MAF van 46%. SMC1B, Structural Maintenance of Chromosomes proteïnen [54],
zijn proteïnen die een belangrijke rol spelen in de dynamiek van chromosoom cohesie en
condensatie [55] tijdens de meiose en de mitose. Slechts één studie deed onderzoek naar het
verband tussen SNPs in het SMC1B gen en het risico op het ontwikkelen van kanker. Zhang
et al. konden hierbij een significante associatie aantonen tussen het homozygoot variante
genotype en het risico op secundaire tumoren en/of herval bij patiënten met hoofd- en
halskanker. Personen die homozygoot variant waren voor dit genotype, hadden 1,74x meer
risico op het ontwikkelen van hoofd- en halskanker. Het variante genotype creëert miRNA
bindingsplaatsen
voor
transcripten
die
getarget
worden
door
microRNA609
en
microRNA124a. Deze nieuwe bindingsplaatsen resulteren in een verminderde expressie van
SMC1B wat kan leiden tot genomische instabiliteit en een groter kankerprogressie risico [43].
5. Probleem- en doelstelling
Gezien de toenemende incidentie van HPV+ orofaryngeale kanker en de betere prognose na
chemoradiotherapie voor deze patiënten, wordt de bepaling van de HPV status voor
orofaryngeale kanker patiënten steeds belangrijker. Er is echter nog geen duidelijkheid over
welke techniek het best aangewezen is voor HPV status bepaling. Additionele biomerkers die
toelaten patiënten verder in te delen naar hun respons op therapie zijn cruciaal. Voornamelijk
voor HPV- patiënten, die een minder goede prognose hebben na chemoradiotherapie, zou
verdere stratificatie op basis van deze biomerkers zinvol zijn. SNPs in microRNA biosynthese
genen, pre-microRNA genen en target genen zijn mogelijke nieuwe biomerkers. In dit
eindwerk zullen SNPs in microRNA genen, microRNA biosynthese genen, microRNA target
15
genen en HPV status geassocieerd worden met de outcome van orofaryngeale kanker
patiënten, behandeld met radiotherapie.
16
Materialen en methoden
1. Introductie
In deze thesis wordt de HPV status van orofaryngeale kankerpatiënten vastgesteld, in
combinatie met de bepaling van 10 SNPs. Er worden 6 SNPs bepaald in genen die betrokken
zijn in de biosynthese van microRNA’s (4 SNPs in het Drosha gen, 2 SNPs in het XPO5 gen),
2 SNPs in microRNA target genen (1 SNP in het KRAS gen, 1 SNP in het SMC1B gen) en 2
SNPs in 2 pre-microRNA genen (pre-microRNA 146a en 196a2). Voor de SNP bepaling
wordt het DNA geïsoleerd uit bloedstalen. De plaats van interesse in het gen wordt vervolgens
geamplificeerd met behulp van specifieke primers (de Polymerase Chain Reaction (PCR)). De
SNP bepaling zelf gebeurt vervolgens met behulp van Restriction Fragment Length
Polymorphism (RFLP) en High Resolution Melting (HRM). De HPV status wordt bepaald
aan de hand van twee technieken: In Situ Hybridisatie (ISH) en Immunohistochemie (IHC).
2. Studiepopulatie
De populatie die gebruikt wordt voor deze studie bevat in totaal 136 orofaryngeale
kankerpatiënten (72 patiënten uit Gent, 64 patiënten uit Maastricht). Deze patiënten werden
geselecteerd uit een groep hoofd- en halskanker patiënten die aan de volgende eisen voldoen:
kanker ter hoogte van de orofarynx, curatieve (chemo)radiotherapie voor de primaire tumor,
aanwezigheid van DNA en tumor materiaal en beschikbaarheid van follow-up data. Voor de
patiënten die in Gent behandeld werden, werden bloedstalen verzameld in samenwerking met
de dienst Radiotherapie van het UZ Gent. Tumormateriaal werd beschikbaar gesteld via de
dienst Pathologie van het UZ Gent. Voor de patiënten die in Maastricht behandeld werden,
werden buffy coats verzonden naar de dienst Medische fysica van de Universiteit Gent. Het
tumormateriaal werd ter beschikking gesteld op de dienst Pathologie van het AZ Maastricht.
Wegens het te laat doorkrijgen van de follow-up data van de patiënten uit Maastricht, werd
beslist om enkel de data te gebruiken van de patiënten uit Gent. Er worden dus uiteindelijk 72
orofaryngeale kankerpatiënten beschouwd in deze studie.
3. DNA purificatie (Puregene kit)
Om de cellen te lyseren wordt 9 ml RBC (Rode bloedcel) lyse toegevoegd aan 3 ml vol bloed.
Hierna worden de buisjes enkele malen omgedraaid en vervolgens 5 minuten geïncubeerd op
kamertemperatuur. Gedurende de incubatie worden de buisjes opnieuw enkele malen
17
omgekeerd. Vervolgens worden de buisjes gecentrifugeerd gedurende 5 minuten aan 2000xg
en het supernatans verwijderd, zodat ongeveer 200 µl vloeistof over blijft. Om de cellen
opnieuw op te lossen in de overgebleven vloeistof, worden de buisjes gevortext. Nadien wordt
3 ml cellyse oplossing toegevoegd en opnieuw gevortext. Na deze stap kunnen de stalen
gedurende 2 jaar bewaard worden op kamertemperatuur. De volgende stap is de proteïne
precipitatie. Er wordt 1 ml proteïne precipitatie buffer toegevoegd aan het cellysaat en dit
geheel wordt gevortext aan hoge snelheid gedurende 20 seconden. De stalen worden 10
minuten op ijs geïncubeerd en vervolgens gecentrifugeerd gedurende 5 minuten aan 2000xg.
Na deze stap zijn de eiwitten geprecipiteerd en vormen ze een donkerbruin pellet. Als dat niet
zo is, worden de laatste stappen van de proteïne precipitatie (vortexen, incuberen op ijs en
centrifugeren) herhaald. Het supernatans wordt overgegoten in een 15 ml buisje dat 3 ml
100% isopropanol bevat. De buisjes worden ongeveer 50x omgedraaid tot het DNA, onder de
vorm van witte draden, zichtbaar wordt. Het geheel wordt gedurende 3 minuten
gecentrifugeerd aan 2000xg waarna het supernatans wordt afgegoten. De buisjes laat men kort
afdruipen op absorberend papier. Vervolgens wordt 3 ml van 70% ethanol toegevoegd en
worden de buisjes enkele malen omgedraaid om het DNA pellet te wassen. De buisjes worden
gecentrifugeerd aan 2000xg gedurende 1 minuut en het supernatans wordt opnieuw afgegoten,
zodat enkel het pellet overblijft. De overgebleven vloeistof wordt afgepipetteerd en de buisjes
worden 10 minuten gedroogd aan de lucht. In de laatste stap wordt het DNA gehydrateerd
door toevoegen van 500 µl DNA hydratie oplossing en zacht te vortexen gedurende 5
seconden. Nadien worden de buisjes 1 uur geïncubeerd op 65°C en daarna overnacht op
kamertemperatuur. De dag nadien worden de buisjes kort gecentrifugeerd en wordt de DNA
oplossing overgebracht in een 1,5 ml buisje en bewaard op -20°C.
4. DNA isolatie uit buffy coats
Buffy coats (BC) van ongeveer 500 µl worden bereid uit 9 ml bloed. DNA wordt geïsoleerd
uit 40 µl van deze BC’s. Voor de cellyse wordt 40 µl BC toegevoegd aan 120 µl RBC lyse in
1,5 ml epjes. De buisjes worden gemengd door deze enkele malen om te keren of kort te
vortexen. Vervolgens worden de epjes geïncubeerd gedurende 5 minuten op kamertempertuur
(KT) en enkele malen omgekeerd tijdens de incubatie. Nadien worden de epjes
gecentrifugeerd gedurende 20 seconden aan 16.000xg en het supernatans verwijderd tot er 1020 µl vloeistof overblijft. De buisjes worden hevig gevortext om de cellen opnieuw op te
lossen in de overblijvende vloeistof. 600 µl cellyse oplossing wordt toegevoegd en op en neer
gepipetteerd om de cellen te lyseren. Indien celklompjes aanwezig zijn, worden de epjes
18
geïncubeerd op 37°C of op KT tot de oplossing homogeen is. De epjes worden afgekoeld tot
KT vooraleer verder gegaan wordt. Na deze stap kan het protocol onderbroken worden
gedurende 2 jaar. Voor de proteïne precipitatie wordt 200 µl proteïne precipitatie buffer
toegevoegd aan het cellysaat. De epjes worden gevortext aan hoge snelheid gedurende 20
seconden. Vervolgens wordt het geheel gecentrifugeerd gedurende 3 minuten aan 16.000xg.
Na deze stap vormen de neergeslagen eiwitten een donkerbruin pellet, indien dit niet het geval
is, worden de epjes opnieuw gevortext aan hoge snelheid gedurende 20 seconden, 5 minuten
geïncubeerd op ijs en vervolgens opnieuw gecentrifugeerd gedurende 3 minuten aan
16.000xg. Voor de DNA precipitatie wordt het supernatans overgegoten in een 1,5 µl epje dat
600 µl 100% isopropanol (KT) en 1 µl glycogen (20 mg/ml) bevat. De epjes worden 50x
omgedraaid zodat het DNA pellet zichtbaar wordt en worden gecentrifugeerd gedurende 1
minuut aan 16.000xg. Na deze stap is het DNA zichtbaar als een klein wit pellet. Het
supernatans wordt afgegoten en op absorberend papier wordt het epje kort afgedropen zonder
dat het pellet mee wordt weggegoten. Om het DNA pellet te wassen, wordt 800 µl 70%
ethanol (KT) toegevoegd en de buisjes enkele malen omgekeerd. De epjes worden
gecentrifugeerd gedurende 1 minuut aan 16.000xg en het supernatans wordt afgepipetteerd.
De buisjes worden gedurende 8-10 minuten gedroogd aan de lucht. Voor de DNA hydratie
wordt 40 µl DNA hydratie oplossing toegevoegd en zacht gevortext gedurende 5 seconden.
De epjes worden gedurende 1 uur geïncubeerd op 65°C en daarna overnacht op KT. De epjes
worden zacht gevortext op verschillende tijdstippen (na 30 minuten en na 1 uur tijdens de
incubatie op 65°C, en ongeveer om het uur tijdens de incubatie op KT). De volgende dag
worden de epjes kort gecentrifugeerd en de DNA oplossing overgebracht in een 1,5 ml buisje
met schroefdop. De epjes worden bewaard op -4°C.
5. Concentratie- en kwaliteitsbepaling van het DNA
De DNA kwantiteit en kwaliteit wordt bepaald met fotospectrometrie. De kwaliteit van het
DNA wordt bepaald door de ratio van de absorbantie bij 260 nm en bij 280 nm (A260/A280).
Deze waarde moet tussen 1,8 en 2,0 liggen. Uitgaande van de stockoplossing wordt een
werkoplossing van 25 ng/µl gemaakt door toevoegen van een bepaalde hoeveelheid TE (TrisEDTA) buffer.
19
6. Polymerase Chain Reaction (PCR)
6.1 Principe
Polymerase Chain Reaction (PCR) wordt gebruikt om een bepaalde DNA sequentie te
amplificeren met behulp van een specifiek primerpaar. PCR berust op een thermische cyclus,
waarbij er verschillende keren in temperatuur gestegen en gedaald wordt, zodat het DNA
wordt gedenatureerd en enzymatische replicatie van het DNA plaats kan vinden. Deze
thermische cyclus bestaat uit 3 stappen: denaturatie, annealing en elongatie. In de
denaturatiestap wordt de temperatuur verhoogd tot 95°C, waardoor de dubbele DNA helix uit
elkaar valt en het DNA gesplitst wordt in 2 enkele strengen. Tijdens de annealingsstap wordt
de temperatuur verlaagd tot de Ta, een primer specifieke temperatuur. De Ta kan berekend
worden aan de hand van de formule Ta = Tm-2, waarbij Tm = [(#T/A x2) + (#G/C x4)]. Door
deze verlaging in temperatuur, binden de primers aan hun complementaire sequentie op het
enkelstrengig DNA. In de laatste stap, de elongatiestap, wordt de temperatuur opnieuw
verhoogd tot 72°C zodat het Taq polymerase een nieuwe DNA streng, complementair aan de
template streng, kan synthetiseren. Er zijn 2 PCR programma’s die gebruikt kunnen worden:
een algemeen programma en een touchdown programma. Een vergelijking tussen de beide
programma’s kan terug gevonden worden in onderstaande tabel.
Tabel 4. PCR programma’s
Algemeen
Touchdown
95°C (5min)
94°C (2min)
95°C (30sec), Ta (30sec), 72°C (30sec) : 35x
94°C (20sec), Ta (15sec) - 1°C per cyclus, 72°C
(1min) : 12x
72°C (10min)
94°C (40sec), Ta-12 (40sec), 72°C (30sec) : 24x
15°C (10min)
72°C (10min)
15°C (10min)
6.2 PCR mix
Er wordt gebruik gemaakt van een standaard PCR mix, weergegeven in onderstaande tabel.
De mix bevat dNTP’s, Buffer Kappa, MgCl2 Kappa, Forward Primer (FWD), Reverse Primer
(REV), Kappa Taq, DNA en H2O in een eindvolume van 15 µl.
20
Tabel 5. PCR Standaardmix
Reagens
Concentratie
Hoeveelheid (µl)
Eindconcentratie
dNTPs
1 mM
3
200 µM
Buffer Kappa
5X
3
1X
MgCl2 Kappa
25 mM
0,9
1,5 mM
Forward Primer
50 µM
0,3
1 µM
Reverse Primer
50 µM
0,3
1 µM
Kappa Taq
5 U/µl
0,0072
0,36 U
DNA
25 ng/µl
2
50 ng
H2O
-
5,43
-
Er wordt steeds een negatieve controle mee geanalyseerd. Deze negatieve controle kent
dezelfde samenstelling als de te analyseren stalen, maar er wordt 2 µl H2O toegevoegd in de
plaats van het DNA.
6.3 PCR primers
Voor bepaalde SNPs waren de protocols, en de primers, reeds geoptimaliseerd en beschikbaar
op de dienst Medische Fysica van de Universiteit Gent. Voor de overige SNPs, moesten de
protocols nog geoptimaliseerd worden en dus de primers ontworpen worden. Deze primers
moeten aan enkele eisen voldoen. Zo is de ideale primerlengte ongeveer 20 nucleotiden lang
en bevatten de primers best niet veel herhalingen van eenzelfde nucleotide om lusvormingen
te vermijden. Een DNA fragment van ongeveer 600 nucleotiden is ideaal voor een PCR
gevolgd door RFLP. Voor HRM is de lengte van het geamplificeerd fragment best tussen 80
en 200 nucleotiden. De forward primer begint en de reverse primer eindigt preferentieel met
een G of C. De Tm van de forward en reverse primer ligt best zo dicht mogelijk bij elkaar. De
sequentie van het gen waarin de SNP zich bevindt, kan worden opgezocht via Ensembl [56]
of NCBI [57]. De locatie van de SNP in deze sequentie kan teruggevonden worden via zijn
specifieke rs code. Wanneer de primers gekozen zijn, kunnen deze getest worden via In Silico
PCR. Dit programma kan online terug gevonden worden (UCSC Genome Bioinformatics
[58]).
Voor PCR gevolgd door RFLP worden de geleverde primers gebracht op een concentratie van
250 µM door een bepaalde hoeveelheid TE buffer toe te voegen. Vervolgens wordt een
21
werkoplossing gemaakt van 50 µM door de stockoplossing 5x te verdunnen. Voor HRM
wordt een stockoplossing gemaakt van 100 µM en wordt deze 20x verdund naar een
werkoplossing van 5 µM.
6.4 PCR controle
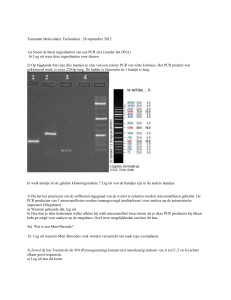
Om na te gaan of de PCR reactie juist is doorgegaan, wordt deze gecontroleerd op een agarose
gel. Deze gel wordt gemaakt door 1,5 g agarose toe te voegen aan 100 ml buffer en
vervolgens te laten opkoken in de microgolf tot de oplossing volledig doorzichtig is. De
oplossing wordt uitgegoten in een houder met kammetjes, die de lanen zullen vormen. Van
zodra de gel is afgekoeld en opgesteven, worden de stalen geladen op de gel.
In de eerste laan wordt 1 µl DNA ladder geladen, in de overige lanen worden de stalen en de
negatieve controle geladen door een mix van 2 µl staal, 1 µl ladingsbuffer en 8 µl H2O in de
laantjes op de gel te brengen. Het elektroforesetoestel wordt ingesteld op 135V voor 25
minuten en na afloop wordt de gel gekleurd met Ethidiumbromide (EtBr) gedurende 30
minuten. Om het resultaat te bekijken wordt de gel onder een UV-lamp geplaatst. Aan de
hand van de ladder kan bepaald worden of de PCR reactie juist doorgegaan is. Aan de hand
van de negatieve controle kan bepaald worden of er al dan niet contaminatie opgetreden is.
7. SNP bepaling
De SNP bepaling gebeurt via PCR-RFLP, via HRM of via spike-in HRM.
7.1 Restriction Fragment Length Polymorphism (RFLP)
RFLP is gebaseerd op het fragmenteren van de DNA streng doordat een restrictie enzym een
specifieke DNA sequentie herkent en deze knipt. Het restrictie enzym wordt geselecteerd
zodat afhankelijk van het genotype, al dan niet meer of minder geknipt wordt. Selectie van het
restrictie enzym gebeurt in silico via RestrictionMapper [59]. Voor RFLP wordt er standaard
gewerkt in een eindvolume van 30 µl. De hoeveelheid restrictie enzym en PCR product dat
moet worden toegevoegd, wordt bepaald in de optimalisatie. De buffer die wordt toegevoegd
is afhankelijk van het enzym en bedraagt steeds 10% van het eindvolume. Eveneens
afhankelijk van het enzym, is of al dan niet Bovine Serum Albumine (BSA) moet worden
toegevoegd. Indien dit het geval is, is de hoeveelheid BSA steeds 10% van de hoeveelheid
buffer dat toegevoegd is. Dit alles wordt aangelengd met H2O tot een eindvolume van 30 µl.
Hierna worden de epjes gevortext en gecentrifugeerd en gedurende een bepaalde periode, dat
vastgelegd is tijdens de optimalisatie, geïncubeerd in een warmwaterbad op een specifieke
22
temperatuur, afhankelijk van het gebruikte enzym. Na deze incubatie is het DNA geknipt in
verschillende fragmenten door het restrictie enzym en kan dit geanalyseerd worden met
behulp van gelelektroforese. Hiervoor wordt een 2% agarose gel gemaakt door 2 g agarose in
100 ml buffer op te lossen. Opnieuw wordt eerst 1 µl ladder geladen en om te kijken of het
PCR product effectief geknipt werd, wordt ongeknipt product op de gel geladen (1 µl
ladingsbuffer + 2 µl ongeknipt PCR product + 8 µl H2O). In de overige lanen wordt 20 µl
digest product + ladingsbuffer (10% van het eindvolume) op de gel geladen. Het
elektroforesetoestel wordt ingesteld op 135V gedurende 30 min. Opnieuw wordt de gel
gekleurd gedurende 30 minuten met EtBr en het resultaat bekeken onder een UV-lamp.
7.2 High Resolution Melting (HRM)
HRM is een high-troughput methode gebaseerd op PCR smeltcurves. De PCR reactie zorgt
ervoor dat de regio van interesse geamplificeerd wordt in aanwezigheid van een kleurstof die
bindt op het dubbelstrengige DNA. Op dit moment is de kleurstof sterk fluorescerend.
Vervolgens wordt de temperatuur opgedreven zodat het dubbelstrengige DNA denatureert.
Door dit proces wordt de kleurstof losgelaten en daalt de fluorescentie. Dit hele verloop wordt
in real time geregistreerd en kent als resultaat een karakteristieke smeltcurve, dat afhankelijk
is van verschillende factoren zoals de GC-inhoud, de lengte, de sequentie en de
heterozygositeit. Er wordt standaard gewerkt in een eindvolume van 20 µl. Een standaard
PCR-HRM mix wordt weergegeven in onderstaande tabel.
Tabel 6. Standaard PCR-HRM mix
Reagens
Concentratie
Hoeveelheid (µl)
dNTP mix
10 mM
0,4
buffer
10X
2
MgCl2
25 mM
1,2
Forward primer
5 µM
1,2
23
Vervolg tabel 6. Standaard PCR-HRM mix
Reagens
Concentratie
Hoeveelheid (µl)
Reverse primer
5 µM
1,2
AmpliTaq
5 U/µl
0,05
HRM Dye
20X
1
DNA
25 ng/µl
1
H2O
-
11,95
Bepaalde moeilijkheden zoals een hoge GC inhoud of een moeilijke SNP (klasse 3 of 4)
kunnen ervoor zorgen dat de genotypes moeilijker onderscheiden kunnen worden. Wanneer
een sequentie een hoge GC inhoud heeft, kan dit probleem verholpen worden door de MgCl2
concentratie op te drijven en al dan niet GC-enhancer toe te voegen. In het geval van een
klasse 3 of 4 SNP, waarbij de homozygoten niet goed van elkaar onderscheiden kunnen
worden op de smeltcurve, kan er gewerkt worden via vorming van heteroduplexen om ze op
deze manier wel te kunnen onderscheiden. Hiervoor worden twee HRM’s uitgevoerd. Op
basis van de eerste HRM kan een onderscheid gemaakt worden tussen de heterozygoten en de
homozygoten. In een tweede HRM, waarbij de heterozygoten buiten beschouwing gelaten
worden, wordt aan de PCR-HRM mix 1 µl spike-in DNA meer en 1 µl H2O minder
toegevoegd. Het spike-in DNA is DNA waarvan men weet dat deze homozygoot normaal is
voor de SNP. Dit DNA vormt heteroduplexen met het DNA van de homozygoot varianten,
waardoor deze een afwijkende vorm vertonen op de smeltcurve. Op deze manier zijn de
homozygoten van elkaar te onderscheiden op de smeltcurve. De HRM smeltcurve wordt
bekomen door het uizetten van de fluorescentie (%) in functie van de temperatuur (°C).
Typisch worden er 3 soorten smeltcurven gevormd: 2 die eenzelfde verloop en vorm hebben
maar door een verschil in smelttemperatuur een verschuiving vertonen ten opzichte van elkaar
(de homozygoten) en 1 die een andere vorm en verloop kent in vergelijking met de
homozygoten (de heterozygoten). Een voorbeeld van zo’n HRM-grafiek wordt weergegeven
in figuur 6.
24
Figuur 6. SNP bepaling SMC1B
7.3 Geoptimaliseerde protocols
Een overzicht van de protocols die reeds geoptimaliseerd waren binnen de onderzoeksgroep
wordt weergegeven in onderstaande tabel. De protocols kunnen terug gevonden worden in
bijlage. De SNP in KRAS werd bepaald aan de hand van 2 methoden: HRM en PCR-RFLP.
De stalen die niet duidelijk waren via HRM, werden gegenotypeerd aan de hand van PCRRFLP.
Tabel 7. Geoptimaliseerde SNPs
SNP
Gebruikte methode
Drosha rs639174 (G>A)
Drosha rs3805500 (T>C)
PCR-RFLP
PCR-RFLP
Drosha rs10520985 (C>T)
KRAS rs61764370 (T>G)
HRM
PCR-RFLP & HRM
8. Linkage Analyse
Om na te gaan of en hoe sterk bepaalde SNPs met elkaar gelinkt zijn werd een analyse
uitgevoerd met Haploview waarbij sequentie-informatie vanuit HapMap wordt opgeladen.
Indien twee SNPs een r² hebben hoger dan 0.8 dan wordt slechts één van beide SNPs
onderzocht.
9. Kwaliteitscontroles
Vijftien procent van alle bepalingen wordt opnieuw gedaan om te controleren of de genotypes
juist bepaald zijn. Indien dit niet het geval is, wordt de reeks opnieuw uitgevoerd.
25
10. HPV status bepaling
Drie verschillende technieken om HPV infectie aan te tonen zijn beschikbaar: Chromogenic
In Situ Hybridisatie, Immunohistochemie en Polymerase Chain Reaction.
10.2 Polymerase Chain Reaction (PCR)
Om na te gaan of er een infectie met HPV aanwezig is, kan gebruik gemaakt worden van de
PCR. Deze PCR wordt gerund met GP5+ en GP6+ primers, die een breed spectrum HPV
types amplificeren. De GP6+ primer is gekoppeld aan biotine, dat nodig is wanneer men een
typering wenst uit te voeren via ELISA. Indien een infectie met HPV aanwezig is, wordt een
fragment van ongeveer 150 bp geamplificeerd. De HPV status bepaling via PCR werd
uitgevoerd op de dienst Pathologie van het AZ Maastricht. Details van de primers en het
gebruikte PCR programma worden in tabel 8 en tabel 9 weergegeven.
Tabel 8. Details PCR primers
GP5+
5’-TTTGTTACTGTGGTAGATACTAC-3’
GP6+-biotine
5’-GAAAAATAAACTGTAAATCATATTC-3’
Tabel 9. PCR programma
PCR-programma GP5+/GP6+
94°C : 4min
94°C (4min) – 40°C (2min) – 72°C (1,30 min) : 40x
72°C (4min)
4°C (∞)
10.2.1 DNA isolatie uit tumormateriaal
De DNA isolatie uit tumormateriaal (paraffine coupes) is uitgevoerd op de dienst Pathologie
van het AZ Maastricht. Dit gebeurt op volledige automatische wijze met het Maxwell 16
toestel. De gebruikte isolatiekit voor deze DNA isolatie is de Maxwell® 16 FFPE Plus LEV
DNA Purification Kit (Promega).
10.2.2 PCR mix
De PCR mix die wordt gemaakt voor de PCR wordt beschreven in onderstaande tabel. Er
wordt gewerkt in een eindvolume van 100 µl. Het aantal µl DNA en water (MilliQ) dat moet
worden toegevoegd, wordt berekend aan de hand van de concentratie van het DNA, gemeten
na fotospectrometrie. De mix wordt steeds in duplo gemaakt en er worden steeds twee
26
positieve controles (een high risk en een low risk), een negatieve controle (DNA geïsoleerd
uit een tonsil, zonder HPV infectie) en een blanco controle (water, bevat geen DNA) mee
geanalyseerd. Details van de PCR mix worden weergegeven in tabel 10.
Tabel 10. PCR mix
Concentratie stockoplossing
Absolute hoeveelheid
Aantal µl in 100 µl eindvolume
per monster
10x PCR reaction buffer
1x
10,0
MgCl2 (25nmol/µl)
2,0 nmol/µl (200nmol)
8,0
dNTPs (25 nmol/µl)
0,4 nmol/µl (40 nmol)
1,6
GP5+ (100 pmol/µl)
1,0 pmol/µl (100 pmol)
1,0
GP6+-biotine (100 pmol/µl)
1,0 pmol/µl (100 pmol)
1,0
JumpStart Red Taq (1U/µl)
0,02 U/µl (2 U)
2,0
Totaalvolume
23,6
10.2.3 PCR controle
Om na te gaan of het fragment geamplificeerd is, wordt een agarose gelelectroforese
uitgevoerd op een 2% agarose gel. Van zodra de gel opgekookt is in de microgolfoven, wordt
SYBR Safe toegevoegd aan de gel, alvorens deze wordt uitgegoten in een houder met
kammetjes. Van zodra de gel afgekoeld en opgesteven is, worden de stalen op de gel geladen:
10 µl van elk PCR product, gevolgd door de positieve en negatieve controles. De ladder wordt
steeds in het midden van de gel geladen. Het elektroforese toestel wordt ingesteld op 130V
gedurende 18min. Na deze stap zijn de stalen aangekleurd en kan het resultaat direct via de
computer afgebeeld worden. Aan de hand van de ladder kan bepaald worden of een bandje
gevormd is rond 150 bp. Indien dit zo is, wordt de staal als HPV positief beschouwd. HPV
positieve stalen worden getypeerd via ELISA (zie 10.2.4).
10.2.4 ELISA
Wanneer na PCR een staal als positief beschouwd wordt, kan een typering via ELISA
uitgevoerd worden, om het juiste HPV type te bepalen. Hiervoor wordt gewerkt met 3
verschillende probe(mixen): een probe voor HPV type 16, een high risk probemix (HPV type
18, 31, 33, 35, 39, 45, 51, 52, 56, 58, 59, 66 en 68) en een low risk probemix (HPV type 6, 11,
40 en 42).
27
10.3 Chromogenic In Sity Hybridisatie
Bij Chromogenic In Situ Hybridisatie (CISH) wordt gebruik gemaakt van een gelabelde probe
(een complementaire DNA sequentie) om een specifieke DNA sequentie in een weefsel aan te
tonen. Hierbij maakt het gebruik van peroxidase of alkaline fosfatase reacties, die gezien
kunnen worden onder een standaard microscoop. De CISH techniek wordt toegepast op
paraffine coupes [60]. De HPV status bepaling aan de hand van deze techniek werd
uitgevoerd op de dienst Pathologie van het UZ Gent en de data werden ter beschikking gesteld
aan de dienst Medische Fysica van de Universiteit Gent.
10.4 p16-Immunohistochemie
Immunohistochemie (IHC) maakt gebruik van antilichamen (AL) die specifieke antigenen
herkennen en binden. In dit geval is het AL gericht tegen p16 INK4a, dat betrokken is in de
celcyclus. In de klinische praktijk correleert p16 met HPV infectie. De p16-IHC kleuring
gebeurt op volledig automatische wijze op de dienst Pathologie van het AZ Maastricht. De
aankleuring is nucleair en cytoplasmatisch. De techniek wordt eveneens toegepast op
paraffine coupes. De gegevens van het AL en de condities van de kleuring worden
weergegeven in onderstaande tabellen.
Tabel 11. Gegevens antilichaam p16-IHC
Naam
Fabrikant
Art. Nr.
p16
Santa Cruz
sc-56330
Isotype
Clone
Clonaliteit
IgG22a
JC8
Monoclonaal
Tabel 12. Condities p16-IHC kleuring
Endogeen peroxidase
Antigen retrieval
Blocking
Verdunning
Incubatie primair AL
Secundaire detectie
Substraat
Tegenkleuring
Controle weefsel
Envision FLEX peroxidase blocking reagent (Dako) (5 min.)
FLEX TRS High (Dako)
Nvt
1:400
Kamertemperatuur (20 min.)
Envision FLEX (20 min.) + Mouse Linker (Dako) (15 min.)
Liquid DAB+ (Dako)
Gill II hematoxiline
Positieve tumor
De bepaling van de HPV status aan de hand van de p16-IHC kleuring voor de stalen uit Gent
werd uitgevoerd op de dienst Pathologie van het UZ Gent, waarna de gegevens ter
beschikking gesteld werden aan de dienst Medische Fysica van de Universiteit Gent.
28
11. Statistische analyse
De verschillende eindpunten die in dit eindwerk onderzocht worden zijn Overall Survival
(OS), Disease Free Survival (DFS) en Disease Specific Survival (DSS). OS en DSS worden
beiden gedefinieerd als het verschil in tijd tussen de diagnose en sterfte of laatste follow-up.
Bij OS wordt geen rekening gehouden met de reden van de doodsoorzaak. Dit wordt wel
gedaan bij de DSS, waarbij enkel sterfte ten gevolge van kanker progressie of ziekte
gerelateerde oorzaak wordt beschouwd. De DFS wordt gedefinieerd als de kans om vrij te
blijven van ziekte na een bepaalde behandeling voor een groep van individuen die aan kanker
lijden. De DFS geeft op deze manier aan hoe effectief een bepaalde behandeling is. Tijd tot
herval wordt berekend uitgaande van het einde van radiotherapie tot de laatste follow-up of
optreden van herval (lokaal, regionaal, op afstand). Vervolgens worden deze eindpunten
geassocieerd met verschillende factoren, zoals geslacht, rookgedrag en alcoholgebruik maar
ook met de SNPs in de microRNA genen en de tumor HPV status. Er wordt gewerkt met het
programma SPSS versie 20. Voor associatie van verschillende factoren (patiënt-gerelateerde,
klinische en microRNA SNPs) met overleving (OS, DFS en DSS) wordt gebruik gemaakt van
de univariate Kaplan-Meier analyse. Associatie van diezelfde factoren met de tumor HPV
status, wordt uitgevoerd met behulp van de Chi-square analyse. Associatie van de leeftijd
wordt geanalyseerd aan de hand van de Mann-Whitney test. De associatie tussen tumor HPV
status en microRNA SNPs wordt geanalyseerd aan de hand van logistieke regressie. Een
resultaat wordt als statistisch significant beschouwd wanneer de p-waarde kleiner of gelijk is
aan 0,05.
29
Resultaten
Aanvankelijk werd deze studie opgestart met een groep orofaryngeale kankerpatiënten
behandeld met (chemo)radiotherapie in het UZ Gent of in het AZ Maastricht. Uiteindelijk
werd uit deze grote groep patiënten slechts een kleine groep patiënten overgehouden. Doordat
de follow-up data van de patiënten behandeld in het AZ Maastricht te laat ter beschikking
gesteld werd, werd besloten om enkel verder te werken met de patiënten behandeld in het UZ
Gent. Enkel patiënten die een curatieve (chemo)radiotherapie behandeling ondergingen voor
een primaire orofarynx tumor werden in beschouwing genomen.
1. Optimalisatie protocols
De protocols voor de SNP bepalingen in Drosha rs639174 G>A, rs10520985 C>T, rs3805500
T>C en in KRAS rs61764370 T>G werden reeds geoptimaliseerd binnen de onderzoeksgroep
en zijn terug te vinden in bijlage. Voor de overige 5 SNPs werd de genotypering
geoptimaliseerd binnen deze studie. Voor één SNP (XPO5 rs2227301 G>A) gebeurde deze
genotypering aan de hand van de PCR-RFLP reactie. Voor de overige SNPs (XPO5 rs699937
C>T, SMC1B rs3747238 A>G, Drosha rs17410035 C>A, pre-microRNA146a rs2910164
G>C en pre-microRNA196a2 rs11614913 C>T) werd het genotype bepaald met behulp van
HRM.
1.1 XPO5 rs2227301 G>A
De PCR reactie werd geoptimaliseerd door het zoeken naar geschikte primers. Deze primers
worden beschreven in onderstaande tabel (tabel 13). Er werd gebruik gemaakt van een
standaard PCR mix die getest werd met het algemeen PCR programma (56°C) en het
touchdown PCR programma (58°C). Na beide programma’s werd als resultaat een bandje
terug gevonden op 542bp, wat verwacht wordt na amplificatie met deze primers. Beide
programma’s kunnen gebruikt worden voor de PCR reactie.
Tabel 13. Details optimalisatie primers XPO5 rs2227301 G>A
Primernummer
Primersequentie (5’-3’)
Primerlengte
Tm
F. primer 446
TATATCCTGAACTTCACTGAC
21 nt
58 °C
R. primer 447
CACTGTCCCTTCGTGGAC
18 nt
58 °C
Fragmentlengte
542 nt
Vervolgens werd gezocht naar een geschikt restrictie enzym via RestrictionMapper. Er werd
30
gekozen voor het BsrI enzym. Dit enzym knipt 1x wanneer het homozygoot normale
genotype (GG) aanwezig is zodat 2 fragmenten gevormd worden met een grootte van 265 en
277 bp. In aanwezigheid van het variante genotype (AA) blijft het fragment ongeknipt met
een grootte van 542 bp. Wanneer een individu drager is van het heterozygote type (GA)
worden er fragmenten gevormd van 265, 277 en 542 bp. De digestmix wordt gemaakt met 2U
van het restrictie enzym en gedurende 3 uur geïncubeerd op de optimale incubatietemperatuur
van BsrI (65°C). Het aantal units nodig van het restrictie enzym en het aantal uur incubatie
werd reeds geoptimaliseerd voor een ander protocol met hetzelfde restrictie enzym binnen de
onderzoeksgroep. Na afloop wordt
het
resultaat
gecontroleerd met
behulp
van
gelelektroforese. De drie verschillende genotypes kunnen duidelijk op de gel onderscheiden
worden. Het protocol voor deze SNP kan in bijlage B terug gevonden worden.
1.2 XPO5 rs699937 C>T
De genotypering van deze SNP werd geoptimaliseerd aan de hand van HRM. Voor de
optimalisatie werd gebruik gemaakt van 2 standaard HRM mixen, met elk een andere forward
primer maar eenzelfde reverse primer. Er wordt steeds gewerkt met een standaard HRM mix.
De verschillende mixen met hun primers staan beschreven in onderstaande tabel.
Tabel 14. Details primers optimalisatie XPO5 rs699937 C>T
Mix
1
2
Primernr.
F.primer 448
R.primer 450
F.primer 449
R.primer 450
Primersequentie (5’-3’)
TTCTTCAGTTTCTTCATCCATG
ACAGTGCCTAATCAGAGATTC
CCATTTAGCCAATACTTACTGA
ACAGTGCCTAATCAGAGATTC
Primerlengte
22 nt
21 nt
22 nt
21 nt
Tm
60 °C
60 °C
60 °C
60 °C
Fragmentlengte
170 nt
105 nt
Het standaard HRM programma werd ingesteld (Ta = 60°C). Voor de optimalisatie worden 6
stalen per mix met gekend genotype geanalyseerd met de HRM. Na afloop van de HRM
reactie kon gezien worden dat met behulp van de eerste mix de verschillende genotypes het
best van elkaar gescheiden kunnen worden. Het protocol voor deze SNP kan in bijlage terug
gevonden worden.
1.3 SMC1B rs3747238 A>G
Voor de optimalisatie van deze SNP werd eveneens gekozen voor een HRM reactie in plaats
van een PCR-RFLP reactie. Dit omdat geen geschikt restrictie enzym gevonden kon worden.
Ook hier werd gebruik gemaakt van 2 standaard HRM mixen, beschreven in onderstaande
tabel.
31
Tabel 15. Details primers optimalisatie SMC1B rs3747238 A>G
Mix
1
2
Primernr.
F.primer 451
R.primer 452
F. primer 451
R.primer 454
Primersequentie (5’-3’)
ATCTTCACTCTGATCTTGCC
CAATACTAACATAGTTTCTCTG
ATCTTCACTCTGATCTTGCC
GTTCAACAGTTTATTAACAATC
Primerlengte
20 nt
22 nt
20 nt
22 nt
Tm Fragmentlengte
58°C
142 nt
58°C
58°C
100 nt
56°C
Voor de optimalisatie van deze SNP werd eerst gestart met een standaard HRM programma
op een Ta = 58°C. Gezien op basis van deze HRM reactie de verschillende genotypes niet van
elkaar onderscheiden konden worden, werd de HRM reactie herhaald met een Ta = 60°C. Dit
resultaat was echter nog slechter dan de eerste HRM, waardoor gekozen werd om de HRM
nog eens te herhalen maar om nu te dalen in temperatuur (Ta = 56°C). Ook dit gaf een slecht
resultaat. Een nieuw optimalisatie experiment werd opgesteld waarbij gewerkt met 3 mixen
met een stijgende hoeveelheid MgCl2. Mix 1 bevatte 1,4 µl MgCl2 per staal, mix 2 1,6 µl
MgCl2 en mix 3 1,8 µl MgCl2. Op basis van de resultaten van de vorige HRM’s, werd
besloten om verder te werken met mix 2 (forward primer 451 en reverse primer 454) en met
een Ta van 58°C. Uit het resultaat van deze optimalisatie kon afgeleid worden dat de mix met
het hoogste gehalte aan MgCl2 (1,8 µl) het beste resultaat gaf. Dit protocol kan terug
gevonden worden in bijlage C.
1.4 Drosha rs17410035 C>A
Ook voor deze SNP werd gekozen voor een genotypering aan de hand van HRM. Voor de
optimalisatie werd gewerkt met 2 standaard HRM mixen met eenzelfde forward primer, maar
een verschillende reverse primer. Er werd eveneens gebruik gemaakt van een standaard HRM
programma (Ta = 60°C). Na afloop van de HRM reactie kon gezien worden dat met behulp
van de 2e mix een betere scheiding tussen de verschillende genotypes bekomen werd. Het
protocol kan terug gevonden worden in bijlage C.
Tabel 16. Details primers optimalisatie Drosha rs17410035 C>A
Mix
1
2
Primernr.
F. primer 420
R. primer 421
F. primer 420
R. primer 422
Primersequentie (5’-3’)
ATCACTGTGGTAATTTGCAAG
AAATCTTACAAGAGCTGTACC
ATCACTGTGGTAATTTGCAAG
TGGCACCAACCTAACAGAG
Primerlengte
21 nt
21 nt
21 nt
19 nt
Tm Fragmentlengte
58°C
130nt
58°C
58°C
96 nt
58°C
32
1.5 Pre-microRNA146a rs2910164 G>C
De HRM voor deze SNP werd geoptimaliseerd door een medestudent. Na optimalisatie bleek
dat deze HRM echter geen onderscheid kon maken tussen de homozygoot varianten en de
wildtypes. Op basis hiervan, werd gekozen om deze SNP te genotyperen met behulp van
spike-in HRM. Allereerst wordt een standaard HRM uitgevoerd waaruit 2 groepen afgeleid
kunnen worden: de heterozygoten en de homozygoten (varianten en wildtypes). Na
afzonderen van de heterozygoten, wordt een tweede HRM uitgevoerd met 1 µl spike-in DNA,
zodat heteroduplexen gevormd worden. Na deze HRM kunnen de homozygoot varianten en
wildtypes van elkaar onderscheiden worden. Het protocol voor deze HRM kan in bijlage C
terug gevonden worden.
1.6 Pre-microRNA196a2 rs11614913 C>T
Voor de optimalisatie van deze SNP werd gebruik gemaakt van twee mixen zoals beschreven
in onderstaande tabel. Met behulp van de standaard HRM mix (Ta = 60°C) werd deze HRM
geoptimaliseerd. Hierbij vertoonde de HRM met mix 2 een beter resultaat. Het protocol kan in
bijlage C terug gevonden worden.
Tabel 17. Details primers optimalisatie pre-microRNA196a2 rs11614913 C>T
Mix
1
2
Primernr.
F. primer 410
R. primer 412
F. primer 411
R. primer 412
Primersequentie (5’-3’)
CAGCTGATCTGTGGCTTAG
AACCGACTGATGTAACTCAG
AGATGCAAAGCTGAATCTCC
AACCGACTGATGTAACTCAG
Primerlengte
19 nt
20 nt
20 nt
20 nt
Tm Fragmentlengte
58°C
90 nt
58°C
58°C
123 nt
58°C
2. Patiënt karakteristieken
De gemiddelde leeftijd van de 72 orofaryngeale kanker patiënten opgenomen in deze studie is
58,7 jaar (± 9,3 jaar). 77% van deze groep patiënten is mannelijk, 90% is of was gebruiker
van tabak en 93% consumeert of consumeerde alcohol. Het aantal pack-years wordt berekend
door de volgende formule: (aantal sigaretten gerookt per dag x het aantal jaren gerookt)/20.
De 2-jaar OS, DFS en DSS bedragen respectievelijk 73,6%, 63,9% en 85,9%. De 3-jaar OS,
DFS en DSS bedragen 68,1%, 59,7% en 82,8% respectievelijk. De overige karakteristieken
worden weergegeven in onderstaande tabel.
33
Tabel 18. Patiënt karakteristieken
Variabele
Geslacht
Man
Vrouw
Aantal
%
56
16
77,8%
22,2%
Type roken
Current/Former
Never
65
7
90,3%
9,7%
Pack-years
≤ 10 PY
> 10 PY
Niet gekend
15
56
1
20,8%
77,8%
1,4%
Type alcoholgebruik
Current/Former
Never
67
5
93,0%
7,0%
Drinks/week
< 10 dranken/week
≥ 10 dranken/week
25
47
34,7%
65,3%
Subsite
Tonsil
Andere
Niet gekend
33
36
3
46,0%
50,0%
4,0 %
T-stage
1-2-3
4
56
16
77,8%
22,2%
N-stage
0
1-2-3
13
59
18,0%
82,0%
3. Associatie met overleving (OS, DFS, DSS)
Tot survival behoort OS (overall survival, dood door eender welke oorzaak), DSS (disease
specific survival, dood door kanker progressie of ziekte gerelateerde dood) en DFS (disease
free survival, dood of herval of metastase).
3.1 Univariate Kaplan-Meier analyse van patiënt-gerelateerde en klinische factoren met
overleving.
Verschillende klinische factoren worden geanalyseerd en geassocieerd met OS, DSS en DFS.
De resultaten van deze analyse kunnen terug gevonden worden in onderstaande tabel.
34
Het aantal drinks/week en T-stage van de tumor vertonen een significante associatie met
overleving. Het aantal drinks/week (< 10 drinks/week vs. ≥ 10 drinks/week) vertoonde een
significante associatie met OS, DSS en DFS (p-waarden 0,051, 0,019 en 0,010
respectievelijk). Orofaryngeale kankerpatiënten die minder dan 10 drinks/week nuttigden,
vertoonden een betere OS, DSS en DFS in vergelijking met patiënten die 10 of meer drinks/
week nuttigden. Voor de T-stage kon een significante associatie aangetoond worden met OS
en DFS (p-waarden 0,019 en 0,047 respectievelijk). Patiënten met een T4 tumor vertoonden
een slechtere OS en DFS in vergelijking met patiënten met een T1/2/3 tumor. Deze associaties
kunnen terug gevonden worden in figuur 7 en figuur 8. Enkele klinische factoren vertonen een
‘borderline’ significante associatie met survival. Dit geldt voor associatie van N-stage (N0 vs.
N1/2/3) met DSS (p-waarde = 0,094), chemotherapie met OS (p-waarde = 0,074), type
radiotherapie (3DRT vs. IMRT) met OS en DFS (p-waarden = 0,097 en 0,092 respectievelijk)
en HPV p16-IHC status met DFS (p-waarde = 0,086).
Tabel 19. Associatie van patiënt-gerelateerde en klinische factoren met overleving
Variabele
Aantal
Sterfte
(eender
welke
oorzaak)
p
Sterfte
(kanker
progressie of
ziekte
gerelateerd)*
p
Sterfte,
herval of
metastase
p
Geslacht
m
v
56
16
26
6
0,610
11
1
0,362
30
7
0,829
Type roken
current/former
never
65
7
30
2
0,212
11
1
0,525
35
2
0,169
Packyears
≤ 10 PY
> 10 PY
15
56
5
27
0,209
2
10
0,440
5
32
0,103
Type alcoholgebruik
current/former
never
67
5
30
2
0,893
12
0
0,370
35
2
0,896
35
Vervolg tabel 19. Associatie van patiënt-gerelateerde en klinische factoren met overleving
Variabele
Aantal
Sterfte
(eender
welke
oorzaak)
Drinks/week
< 10 drinks/week
≥ 10 drinks/week
25
47
8
24
0,051
1
11
0,019
8
29
0,010
T-stage
T4
T1-2-3
16
56
9
23
0,019
3
9
0,244
6
27
0,047
N-stage
N0
N1-2-3
13
59
5
27
0,235
0
12
0,094
5
32
0,155
Chemotherapie
Ja
Nee
42
30
21
11
0,074
9
3
0,225
23
14
0,272
Heelkunde
Ja
Nee
21
51
11
21
0,395
4
8
0,944
12
25
0,479
Halsevidement
Ja
Nee
47
25
22
10
0,420
10
2
0,294
24
13
0,240
Type radiotherapie
3DRT
IMRT
15
57
8
24
0,097
1
11
0,218
8
29
0,092
14
55
19
4
27
6
0,235
2
10
3
0,370
4
32
13
0,110
48
24
HPV status - CISH
Positief
Negatief
HPV status - p16IHC
Postitief
Negatief
p
0,250
Sterfte
(kanker
progressie of
ziekte
gerelateerd)*
2
p
0,472
Sterfte,
herval of
metastase
p
0,086
29
*Totaal aantal patiënten voor analyse naar DSS toe bedroeg in totaal 64 patiënten.
De resultaten in bovenstaande tabel werden bekomen aan de hand van de Kaplan-Meier analyse.
36
A
B
C
Figuur 7. Associatie tussen aantal drinks/week (< 10 drinks/week vs. ≥ 10 drinks/week) en OS (A), DSS (B)
en DFS (C)
A
B
Figuur 8. Associatie tussen T-stage (T4 vs. T1/2/3) en OS (A) en DFS (B)
3.2 Univariate Kaplan-Meier analyse van associatie tussen miRNA genotypes en overleving
De resultaten na univariate analyse met Kaplan-Meier worden weergegeven in tabel 20.
Wegens het lage aantal patiënten worden enkel de resultaten van het dominante model
weergegeven. Er werden geen statistisch significante associaties gevonden tussen een SNP in
37
een microRNA gen en overleving. Voor de SNP in het SMC1B gen en associatie met DSS
kon geen statistische analyse uitgevoerd worden, aangezien in de homozygoot normale groep,
geen enkele patiënt gestorven is ten gevolge van kanker progressie of de ziekte. Volgens het
recessieve model werd een statistisch significante associatie gevonden tussen een SNP in
Drosha (rs3805500 (T>C)) en survival (OS en DFS). Orofaryngeale kanker patiënten die
drager zijn van het TT/TC genotype vertonen een betere OS en DFS in vergelijking met
dragers van het homozygoot variante genotype (CC). Zo bedraagt de 3j-OS voor patiënten die
drager zijn van het TT/TC genotype 69,6%, terwijl dit voor dragers van het CC genotype nog
slechts 22,2% bedraagt. Na correctie voor multiple testing met de Bonferroni correctie, bleef
dit resultaat statistisch significant. Alle SNPs zijn in Hardy-Weinberg equilibrium. De
resultaten worden weergegeven in tabel 21 en figuur 9. De tabel met de overige resultaten van
de univariate Kaplan-Meier analyse volgens het recessieve model kunnen terug gevonden
worden in bijlage E.
Tabel 20. Associatie miRNA genotypes en overleving volgens het dominant model
miRNA
genotypes
Totaal
aantal
patiënten
Sterfte
(eender
welke
oorzaak)
p
Sterfte
(kanker
progressie of
ziekte
gerelateerd)**
p
Sterfte,
herval of
metastase
p
GG
48
19
1*
8
1
22
1
GA+AA
22
12
0,311
3
0,972
14
0,284
CC
23
10
1
3
1
12
1
CT+TT
45
21
0,831
8
0,928
23
0,909
TT
31
13
1
3
1
16
1
TC+CC
39
18
0,180
8
0,303
20
0,350
CC
31
15
1
7
1
15
1
CA+AA
31
15
0,917
3
0,217
18
0,518
Drosha
rs639174
rs10520985
rs3805500
rs17410035
38
Vervolg tabel 20. Associatie miRNA genotypes en overleving volgens het dominant model
miRNA
genotypes
Totaal
aantal
patiënten
Sterfte
(eender
welke
oorzaak)
p
Sterfte
(kanker
progressie of
ziekte
gerelateerd)**
p
Sterfte,
herval of
metastase
p
41
29
21
10
1
0,293
5
6
1
0,491
24
12
1
0,450
29
41
14
17
1
0,373
3
8
1
0,441
16
20
1
0,609
GG
GC+CC
196a2
rs11614913
36
33
15
15
1
0,444
6
5
1
0,998
17
18
1
0,463
CC
CT+TT
26
44
13
18
1
0,756
5
6
1
0,476
15
21
1
0,977
17
55
7
25
1
0,546
0
12
/
/
9
28
1
0,369
56
14
25
6
1
0,088
10
1
1
0,350
29
7
1
0,129
XPO5
rs2227301
GG
GA+AA
rs699937
CC
CT+TT
pre-microRNA
146a
rs2910164
SMC1B
rs3747238
AA
AG+GG
KRAS
rs61764370
TT
TG+GG
* 1 wordt gebruikt als referentie. De resultaten uit bovenstaande tabel werden gevonden via Kaplan-Meier
analyse. / ** Totaal aantal patiënten voor analyse naar DSS toe bedroeg in totaal 64 patiënten.
39
Tabel 21. Associatie tussen Drosha rs3805500 (T>C) met overleving volgens recessief model
Sterfte
(eender
welke
oorzaak)
Sterfte
(kanker
progressie of
ziekte
gerelateerd)*
Totaal
miRNA
aantal
Sterfte, herval of
genotypes patiënten
p
p
metastase
p
Drosha
rs3805500
TT+TC
64
27
0,003
10
0,435
32
0,050
CC
6
4
1
4
Deze resultaten werden gevonden na analyse via Kaplan-Meier. / *Totaal aantal patiënten voor analyse naar DSS
toe bedroeg in totaal 64 patiënten.
Figuur 9. Associatie Drosha rs3805500 (T>C) met OS
4. Associatie met tumor HPV status
Hierbij worden de patiënten ingedeeld in twee groepen naargelang de tumor HPV status
(HPV+ vs. HPV-). De tumor HPV status wordt hierbij geassocieerd met de patiëntgerelateerde en klinische factoren enerzijds en met de microRNA SNPs anderzijds.
4.1 Associatie van tumor HPV status met patiënt-gerelateerde en klinische factoren
Wanneer de patiënten ingedeeld worden in twee verschillende groepen naargelang de tumor
HPV status (HPV+ vs. HPV-) en geassocieerd worden met verschillende klinische factoren,
komen hier enkele factoren uit als statistisch significant. Rookgedrag (aantal pakjaren, p <
0,001), type drinken (p = 0,001), alcohol gebruik (aantal dranken/week, p < 0,001) en de 2jaar OS (p = 0,018) kunnen geassocieerd worden met de HPV status (bepaald volgens CISH).
Dit wordt weergegeven in onderstaande tabel. Voor de tumor HPV status bepaald met p16IHC kleuring, wordt een significatie associatie gevonden met type roken (p = 0,036), aantal
packyears (p < 0,001), type drinken (p = 0,010) en het aantal drinks/week (p < 0,001). HPV+
hebben een verschillend rookgedrag (≤10 PY) en drinkgedrag (<10 drinks/week) in
40
vergelijking met HPV- patiënten. Na 2 jaar zijn alle HPV+ patiënten nog in leven, terwijl dit
aantal bij de HPV- patiënten nog slechts 67% bedraagt. Dit wordt weergegeven in tabel 22.
Tabel 22. Associatie van patiënt-gerelateerde en klinische factoren met HPV status (CISH, p16-IHC)
HPV+
(CISH)
HPV(CISH)
p
HPV+
(p16)
HPV(p16)
0,332
p
Leeftijd
Geslacht
m
v
0,856
12
2
44
11
0,238
18
2
37
11
0,217
Type roken
Current/former
Never
11
3
52
3
0,058
16
4
46
2
0,036
Roken (PY)
≤10 PY
>10 PY
8
6
6
48
< 0,001
10
10
4
43
< 0,001
Type drinken
Current/former
Never
10
4
54
1
0,001
16
4
47
1
0,010
Drinks/week
<10
≥10
11
3
13
42
< 0,001
14
6
9
39
< 0,001
Subsite
tonsil
other
9
5
24
28
0,228
12
8
21
24
0,321
T-stage
T4
T1-2-3
2
12
14
41
0,377
4
16
12
36
0,658
N-stage
N0
N1-2-3
2
12
9
2
0,850
2
18
9
39
0,372
Chemo
ja
nee
11
3
31
24
0,128
15
5
27
21
0,147
2j-OS
100%
67%
0,018
84,2%
66,7%
0,129
3j-OS
84,6%
60,1%
0,084
73,3%
60,7%
0,248
2j-DSS
100%
78,1%
0,071
89,2%
79;9%
0,363
3j-DSS
91,7%
74,8%
0,169
83,2%
76,1%
0,481
De resultaten in deze grafiek werden bekomen na Chi-Square analyse. Met uitzondering van de OS en DSS, die
bekomen werd na Kaplan-Meier analyse.
41
3.4 Associatie van tumor HPV status met microRNA genotypes
De associatie van de verschillende microRNA genotypes en tumor HPV status (bepaald door
CISH of p16-IHC) werd uitgevoerd aan de hand van logistieke regressie. Deze analyse
leverde echter geen statistisch significante resultaten op wegens te lage aantallen in de
verschillende groepen. De tabel met resultaten kan terug gevonden worden in bijlage.
5. Bepaling van de tumor HPV status met behulp van CISH en p16-IHC
In deze studie werd de tumor HPV status bepaald aan de hand van 2 technieken: CISH en
p16-IHC. De resultaten hiervan worden weergegeven in tabel 23. Na bepaling van de tumor
HPV status met behulp van CISH wordt in deze studiepopulatie 20% van de orofaryngeale
kankerpatiënten als HPV positief bevonden. Na bepaling van de tumor HPV status met behulp
van p16-IHC worden er 30% van de patiënten als HPV positief bevonden. 11% van de HPV
negatieven, bepaald volgens CISH, wordt als positief beschouwd na bepaling met p16-IHC.
Een derde techniek, PCR, werd eveneens toegepast op de stalen uit Maastricht. De techniek
kon niet worden toegepast op de stalen van de patiënten uit Gent wegens gebrek aan tijd.
Tabel 23. Resultaten tumor HPV status bepaling volgens CISH en p16-IHC kleuring
Techniek
HPV+
HPV-
CISH
14 (20%)
55 (80%)
p16-IHC
20 (30%)
48 (70%)
42
Discussie
Vooraleer gestart kon worden met het genotyperen van de patiëntenstalen moesten enkele
SNPs geoptimaliseerd worden. Voor 1 SNP (XPO5 rs2227301 G>A) werd gekozen voor een
optimalisatie met PCR-RFLP. De PCR optimalisatie verliep vlot. Hoewel er bij controle van
de PCR een fractie smeer op de gel waargenomen kon worden, werden er toch duidelijke
bandjes, van de verwachtte grootte, gevormd bij de twee verschillende programma’s. De
optimalisatie voor RFLP verliep eveneens vlot. Het gebruikte restrictie enzym werd reeds
gebruikt voor een andere SNP in een ander protocol. Hetzelfde aantal units en dezelfde
incubatietijd werden uitgeprobeerd na de PCR reactie. Bij controle van de RFLP konden de
verschillende genotypes duidelijk op gel bepaald worden. Voor de overige 5 SNPs werd
gekozen voor een optimalisatie met HRM. De optimalisaties voor XPO5 (rs699937 C>T),
Drosha (rs17410035 C>A) en pre-microRNA196a2 (rs11614913 C>T) verliepen vlot. Er
werd telkens gebruik gemaakt van 2 standaard HRM mixen, met verschillende
primercombinatie, waarbij steeds 1 van de 2 mixen een goed resultaat gaf en gebruikt kon
worden. Een mogelijke reden voor de vlotte optimalisaties zou kunnen zijn dat de SNPs (C>T
en C>A) respectievelijk behoren tot klasse 1 en klasse 2 SNPs. Deze basenveranderingen
leiden tot een grotere verschuiving in smelttemperaturen, waardoor de verschillende
genotypes op de smeltcurve makkelijker te onderscheiden zijn, in vergelijking met klasse 3 of
4 SNPs. Voor de optimalisatie van de SMC1B SNP (rs3747238 A>G) moest verschillende
keren in temperatuur veranderd worden en de MgCl2 concentratie aangepast worden vooraleer
een goede scheiding van de verschillende genotypes bekomen kon worden. De SNP in premicroRNA146a (rs2910164 G>C) werd initieel geoptimaliseerd door een medestudent voor
een standaard HRM reactie. Deze standaard HRM reactie kon de verschillende genotypes
echter niet van elkaar onderscheiden, waardoor besloten werd om over te gaan op de spike-in
HRM techniek. Een groot voordeel verbonden aan deze techniek is dat de homozygoot
normalen en varianten goed van elkaar gescheiden worden zodat geen twijfel mogelijk is. Een
nadeel verbonden met deze techniek is dat, omwille van het gebruik van spike-in DNA, veel
materiaal en consumables vereist zijn.
Een tweede aspect van dit eindwerk is de associatiestudie met overleving. In deze studie werd
een 2-j OS en 3j-OS gevonden van 73,6% en 68,1% respectievelijk. Deze percentages
verschillen licht met wat in de literatuur terug gevonden kan worden. In de studie van
Perisanidis et al. werd een 2j-OS terug gevonden van 66% bij patiënten met orofaryngeale
kanker die behandeld zijn met preoperatieve chemoradiotherapie [62]. Een andere studie vond
43
een 3j-OS bij orofaryngeale kanker patiënten behandeld met chemoradiotherapie terug van
79,7% [63]. Verschillende patiënt-gerelateerde en klinische factoren werden vervolgens
geassocieerd met overleving. Twee statistisch significante associaties werden hierbij gemaakt:
het aantal drinks/week en de T-stage. De kleine studiepopulatie groep is een zeer beperkende
factor in de analyse van deze factoren met overleving. Associatie van de microRNA SNPs
met overleving leverde een statistisch significant resultaat op. Zo vertoonde Drosha
rs3805500 T>C een statistisch significante associatie met overleving (OS en DFS). Door de
kleine studiepopulatie is het aantal homozygoot varianten dat binnen één SNP teruggevonden
wordt steeds zeer laag. Hierdoor verkleint de kans dat een statistisch significant resultaat
gevonden kan worden. Het verband tussen SNPs in microRNA biosynthese genen, target
genen en pre-microRNA genen en overleving werd tot hiertoe weinig bestudeerd, waardoor
voor selectie van de SNPs vooral gebaseerd werd op studies die de relatie tussen deze SNPs
onderzochten met het risico op het ontwikkelen van hoofd- en halskanker. Voor de
beschouwde SNPs in de pre-microRNA genen (146a en 196a2) en in KRAS zijn er wel
studies uitgevoerd naar overleving. Zo werd in de studie van Guan et al. een statistisch
significant verband gevonden tussen de SNP in het pre-microRNA146a en premicroRNA196a2 en OS, DFS en DSS [42]. Deze studie kon dit resultaat echter niet
bevestigen, waarschijnlijk door de te kleine studiepopulatie. In de studie van Christensen et
al. kon een statistisch significante associatie aangetoond worden tussen de SNP in het gen
voor KRAS en overleving bij orale kanker patiënten, waarbij dragers van het variante
genotype een verminderde overleving vertoonden [53]. Ook dit resultaat kon niet bevestigd
worden in deze studie.
Voor de associatie van deze SNPs, patiënt-gerelateerde factoren en klinische factoren met de
tumor HPV status werd de HPV status bepaald aan de hand van 2 technieken (ISH en p16IHC). Hieruit blijkt dat 20% van de orofaryngeale kanker patiënten als HPV positief
beschouwd kunnen worden na bepaling met CISH terwijl dit 30% is na bepaling met p16IHC. In de literatuur wordt eveneens een verschillend aantal HPV positieven gevonden na
analyse met de twee verschillende technieken. Zo wordt in de studie van Singhi en Westra een
vergelijking gemaakt tussen de resultaten na CISH en p16-IHC. Zij vonden een infectie met
HPV terug in 69% van alle hoofd- en halskanker patiënten, bepaald aan de hand van CISH,
terwijl dit 76% werd na bepaling met p16-IHC. 24% van alle HPV- (bepaald met CISH) werd
positief bevonden na bepaling met p16-IHC [61]. In deze studie worden 11% van de HPV
negatieven (bepaald met CISH) positief bevonden na kleuring met p16-IHC. Het aantal HPV
44
positieve patiënten in deze studie ligt lager dan de cijfers die worden gerapporteerd in de
literatuur. Een oorzaak hiervoor zou kunnen zijn dat er in deze studie gewerkt is met
patiëntenstalen die dateren van voor 2010. Het is immers pas sinds de laatste jaren dat een
stijging van het aantal HPV+ patiënten waargenomen wordt, onder meer door een
veranderende levensstijl. Een reden voor het verschil in aantal HPV positieven na de twee
verschillende technieken, zou kunnen zijn dat CISH gebruik maakt van probes die enkel
gericht zijn tegen High Risk (HR) types. Besmetting met een ander HPV type, wordt aan de
hand van CISH niet gedetecteerd op deze manier [61]. Een andere verklaring zou kunnen zijn
dat IHC p16 detecteert, en dat p16 overexpressie aanwezig kan zijn zonder de aanwezigheid
van een HPV infectie [31]. De associaties die gevonden werden met de tumor HPV status,
verschilt naargelang de gebruikte techniek. HPV status bepaling aan de hand van de CISH
techniek, wordt geassocieerd met de 2j-OS, rookgedrag en drinkgedrag. Patiënten die HPV+
zijn, vertonen in deze studie een betere 2j-OS in vergelijking met HPV- patiënten. Dit is een
bevestiging van wat reeds veel onderzocht en bewezen werd in andere studies. In de studie
van Guan et al. werd eveneens een significante associatie aangetoond met overleving en
rookgedrag. Daarnaast werd in de studie van Guan et al., net zoals in andere studies, een
associatie aangetoond met geslacht en leeftijd, wat in deze studie niet bevestigd kon worden.
HPV+ patiënten zijn immers doorgaans jonger en mannelijk. Guan et al. konden echter geen
significante associatie aantonen met alcoholgebruik, wat in deze studie wel gevonden werd.
Associatie van de SNPs met de tumor HPV status leverde geen significante resultaten op. Dit
werd wel aangetoond in de studie van Guan et al. voor de SNPs in het pre-microRNA146a en
pre-microRNA196a2. Ook hier ligt een mogelijke verklaring in het feit dat deze studie slecht
72 patiënten telde, terwijl de studie van Guan et al. 309 patiënten telde. Voor de overige SNPs
is geen vergelijkende literatuur beschikbaar.
45
Besluit
Deze studie kon enkele statistische significante associaties aantonen. Zo is een statistisch
significante associatie tussen een SNP in het gen voor Drosha (rs385500 T>C) en overleving
(OS en DFS) aangetoond. Patiënten met het homozygoot normaal (TT) of het heterozygoot
(TC) genotype vertonen hierbij een betere OS en DFS in vergelijking met patiënten met het
homozygoot variante genotype (CC). Daarnaast werd een associatie gevonden tussen T-stage
en OS en DFS: patiënten met een T4 tumor vertoonden een significant slechtere overleving in
vergelijking met patiënten met een T1/2/3 tumor. Ook het aantal drinks/week kon
geassocieerd worden met de overleving (OS, DFS en DSS). Hierbij vertoonden matige
alcoholdrinkers (< 10 drinks/week) een betere overleving in vergelijking met patiënten die 10
of meer drinks/week nuttigden. Verschillende andere factoren, waaronder de N-stage en type
radiotherapie, vertoonden een borderline significantie met overleving. Associatie met de
tumor HPV status leverde eveneens enkele statistisch significante associaties op. Zo kon de
tumor HPV status, bepaald met CISH, geassocieerd worden met rookgedrag, alcoholgebruik
en de 2j-OS. De tumor HPV status, bepaald met p16-IHC, kon geassocieerd worden met
rookgedrag en alcoholgebruik. De 2j-OS en 3j-OS dat terug gevonden werd voor deze
studiepopulatie bedroeg 73,6% en 68,1%, respectievelijk. Deze verschilt licht met hetgeen in
de literatuur terug gevonden wordt voor orofaryngeale kankerpatiënten, behandeld met
(chemo)radiotherapie. De HPV status werd bepaald aan de hand van 2 technieken: CISH en
p16-IHC. Hoewel het aantal HPV positieven in deze studie lager ligt dan in de meeste andere
gepubliceerde studies, bevestigt deze studie dat de CISH techniek leidt tot hogere HPV
positiviteit in vergelijking met de p16 IHC techniek. Onze huidige studie heeft verschillende
beperkingen waarvan de grootste de grootte van de studie populatie is. Het feit dat er weinig
tot geen voorgaand genetisch onderzoek uitgevoerd werd naar overleving vormt een tweede
beperkende factor. De keuze van de genen en de SNPs werd hierdoor voornamelijk gebaseerd
op associatiestudies naar kankerpredispositie. Daarnaast is deze studie een retrospectieve
studie, waarbij de patiënt-gerelateerde factoren, zoals rookgedrag en alcoholgebruik,
gebaseerd was op vragenlijsten die de patiënten zelf moesten invullen. Naar de toekomst toe
zou deze studie uitgevoerd moeten worden op een groter aantal patiënten. Wanneer de
patiëntengroep uit Maastricht toegevoegd kan worden aan deze studie, levert dit al een
verdubbeling op van de studiepopulatie.
46
Referentielijst
[1] Epidemiology and Clinical Aspects of HPV in Head and Neck Cancers (Anil K. Chaturvedi)
[2] Babu JM, Prathibha R, Jijith VS, Hariharan R, Radhakrishna Pillai M (2011). A miR-centric view of headand neck cancers. Biochimica et Biophysica Acta-Reviews on Cancer 1816(1): 67-72.
[3] www.kankerregister.org/media/docs/stK_publicatie.pdf
[4] www.cijfersoverkanker.nl/selecties/Dataset_2/img50f6ac9b16348
[5] Argiris A, Karamouzis MV, Raben D, Ferris RL (2008). Head and neck cancer. Lancet 371(9625): 16951709.
[6] Ang KK, Sturgis EM (2012). Human Papillomavirus as a Marker of the Natural History and Response to
Therapy of Head And Neck Squamous Cell Carcinoma. Seminars In Radiation Oncology 22(2): 128-142.
[7] Howard JD, Chung CH (2012). Biology of Human Papillomavirus-Related Oropharyngeal Cancer. Seminars
in Radiation Oncology 22(3): 187-193.
[8] Marur S, Forastiere A (2008). Head And Neck Cancer: Changing Epidemiology, Diagnosis And Treatment.
Mayo Clinic Proceedings 83(5): 489-501
[9] Haas SL, Ye WM, Lohr JM (2012) Alcohol consumption and digestive tract cancer. Current Opinion in
Clinical Nutrition and Metabolic Care 15(5): 457-467.
[10] Chung CH, Gillison ML (2009). Human Papillomavirus in Head And Neck Cancer: Its Role in Pathogenesis
and Clinical Implications. Clinical Cancer Research 15(22): 6758-6762.
[11] Thomas J, Primeaux T (2012). Is p16 immunohistochemistry a more cost-effective method for identification
of human papillomavirus-associated head and neck squamous cell carcinoma? Annals Of Diagnostic Pathology
16(2): 91-99.
[12] http://www.cancer.gov/cancertopics/pdq/treatment/oropharyngeal/Patient/page1
[13] Ang KK, Harris J, Wheeler R, Weber R, Rosenthal DI, Nguyen-Tân PF, Westra WH, Chung CH, Jordan
RC, Lu C, Kim H, Axelrod R, Silverman C, Redmond KP, Gilison ML (2010). Human Papillomavirus and
Survival of Patients with Oropharyngeal Cancer. The New England Journal Of Medicine 363(1): 24-35.
[14] Granata R, Miceli R, Orlandi E, Perrone F, Cortelazzi B, Franceschini M, Locati LD, Bossi P, Bergamini C,
Mirabile A, Mariani L, Olmi P, Scaramellini G, Potepan P, Quattrone P, Ang KK, Licitra L (2012). Tumor stage,
human papillomavirus and smoking status affect the survival of patients with oropharyngeal cancer: an Italian
validation study. Annals of Oncology 23(7): 1832-1837.
[15] Vu HL, Sikora AG, Fu SB, Kao J (2010). HPV-induced oropharyngeal cancer, immune response and
response to therapy. Cancer Letters 288(2): 149-155.
[16] www.who.int/vaccine_research/diseases/viral_cancers/en/index3.html
[17] Zur Hausen H (2000). Papillomaviruses causing cancer : Evasion from host-cell control in early events in
carcinogenesis. Journal Of The National Cancer Institute 92(9) : 690-698.
[18] Vozenin MC, Lord HK, Hart D, Deutsch E (2010). Unravelling the biology of human papillomavirus (HPV)
related tumors to enhance their radiosensitivity. Cancer Treatment Reviews 36(8): 629-636.
[19] Laborde RR, Janus JR, Olsen SM, Wang VW, Garcia JJ, Graham RP, Moore EJ, Olsen KD, Kasperbauer
JL, Price DL, Berres M, Halling G, Smith DI (2012). Human papillomavirus in oropharyngeal cancer carcinoma:
Assessing virus presence in normal tissue and activity in cervival metastasis. Laryngoscope 122(12): 2707-2711.
[20] Chan PKS, Picconi MA, Cheung TH, Giovannelli L, Park JS (2012). Laboratory and clinical aspects of
human papillomavirus testing. Critical Reviews In Clinical Laboratory Sciences 49(4): 117-136.
[21] Hoffmann M, Tribius S, Quabius ES, Henry H, Pfannenschmidt S, Burckhardt C, Gorogh T, Halec G,
Hoffmann AS, Kahn T, Rocken C, Haag J, Waterboer T, Schmitt M (2012). HPV DNA E6*I mRNA expression
and p16(INK4A) immunohistochemistry in head and neck cancer – How valid is p16(INK4A) as surrogate
marker? Cancer Letters 232(1): 88-96.
[22] Park WS, Ryu J, Cho KH, Choi MK, Moon SH, Yun T, Chun BS, Lee GK, Ahn HJ, Lee JH, Vermeer P,
Jung YS (2012). Human papillomavirus in oropharyngeal squamous cell carcinoma in korea: Use of G1 cycle
markers as new prognosticators. Head And Neck-Journal For The Sciences And Specialities Of The Head And
Neck 34(10): 1408-1417.
[23] Snow AN, Laudadio J (2010). Human Papillomavirus Detection in Head and Neck Cancer. Advances In
Anatomic Pathology 17(6): 394-403.
[24] Fakhry C, Westra WH, Cmelak SLA, Ridge JA, Pinto H, Forastiere A, Gillison ML (2008). Improved
survival of patients with human papillomavirus-positive head and neck squamous cell carcinoma in aprospective
clinicl trial. Journal of the national cancer institute 100(4): 261-269.
[25] Gillison ML, Koch WM, Capone RB, Spafford M, Westra WH, Wu L, Zahurak ML, Daniel RW, Viglione
M, Symer DE, Shah KV, Sidranksy D (2000). Evidence for a causal association between human papillomavirus
and a subset of head and neck cancers. Journal of the national cancer institute 92(9): 709-720.
[26] Furness S, Glenny AM, Worthington HV, Pavitt S, Oliver R, Clarkson JE, Macluskey M, Chan KKW
(2011). Interventions for the treatment of oral cavity and oropharyngeal cancer: chemotherapy. Cochrane
47
database of systematic reviews 4: Art. Nr.:CD006386. doi: 10.1002/14651858.CD006386.
[27] Pang E, Delic NC, Hong A, Zhang M, Rose BR, Lyons JG (2011). Radiosensitization of oropharyngeal
squamous cell carcinoma cells by humanpapillomavirus 16 oncoprotein E6*I. International journal of radiation
oncology biology physics 79(3): 860-865.
[28] Lassen P, Eriksen JG, Hamilton-Dutoit S, Tramm T, Alsner J, Overgaard J (2009). Effect of HPVAssociated p16(INK4A) Expression on Response to Radiotherapy and Survival in Squamous Cell Carcinoma of
The Head and Neck. Journal Of Clinical Oncology 27(12): 1992-1998.
[29] Venuti A, Paolini F (2012). HPV detection methods in head and neck cancer. Head and neck pathology
6(suppl 1): S63-74.
[30] Schace AG, Liloglou T, Risk JM, Filia A, Jones TM, Sheard J, Woolgar JA, Helliwell TR, Triantafyllou A,
Robinson M, Sloan P, Harvey-Woodworth C, Sisson D, Shaw RJ (2011). Evaluation of Human Papilloma Virus
Diagnostic Testing in Oropharyngeal Squamous Cell Carcinoma: Sensitivity, Specificity, and Prognostic
Discrimination. Clinical cancer research 177(19): 626-6271.
[31] Jordan RC, Lingen MW, Perez-Ordonez B, He X, Pickard R, Koluder M, Jiang B, Wakely P, Xiao WH,
Gillison ML (2012). Validation of Methods for Oropharyngeal Cancer HPV Status Determination in US
Cooperative Group Trials. American Journal Of Surgical Pathology 36(7): 945-954.
[32] Bishop JA, Ma XJ, Wang HW, Luo YL, Illei PB, Begum S, Taube JM, Koch WM, Westra WH (2012).
Detection of Transcriptionally Active High-risk HPV in Patients With Head and Neck Squamous Cell
Carcinoma as Visualized by a Novel E6/E7 mRNA In Situ Hybridization Method. Amercian Journal Of Surgical
Pathology 36(12): 1874-1882.
[33] Lassen P, Overgaard J (2012). Scoring and classification of oropharyngeal carcinoma based on HPV-related
p16-expression. Radiotherapy and Oncology 105(2): 269-270.
[34] Robinson M, Schache A, Sloan P, Thavaraj S (2012). HPV Specific Testing: A Requirement for
Oropharyngeal Squamous Cell Carcinoma Patients. Head and Neck Pathology 6(1): 83-90.
[35] Christensen BC, Avissar-Whiting M, Ouellet LG, Butler RA, Nelson HH, McClean MD, Marsit CJ, Kelsey
KT (2010). Mature MicroRNA Sequence Polymorphism in MIR196A2 Is Associated with Risk and Prognosis of
Head and Neck Cancer. Clinical Cancer Research 16(14): 3713-3720.
[36] Ryan BM, Robles Al, Hariss CC (2010). Genetic variation in microRNA networks: the implications for
cancer research. Nature reviews cancer 10(6): 389-402.
[37] Wang JB, Wang QW, Liu H, Shao N, Tan BX, Zhang GY, Wang K, Jia YB, Ma W, Wang NN, Cheng YF
(2012). The association of miR-146a rs2910164 and miR-196a2 rs11614913 polymorphisms with cancer risk: a
meta-analysis of 32 studies. Mutagenesis 27(6): 779-788.
[38] Liu Z, Li G, Wei S, Niu J, El-Naggar AK, Sturgis EM, Wei Q (2010). Genetic Variants in Selected PreMicroRNA Genes and the Risk Of Squamous Cell Carcinoma of the Head and Neck. Cancer 116(20): 47534760.
[39] Gong J, Tong Y, Zhang HM, Wang K, Hu T, Shan G, Sun J, Guo AY (2012). Genomde-Wide Identification
of SNPs in MicroRNA Genes and the SNP Effects on MicroRNA Target Binding and Biogenesis. Human
Mutation 33(1): 254-263.
[40] Slaby O, Bienertova-Vasku J, Svoboda M, Vyzula R (2012). Genetic Polymorphisms and microRNAs: new
direction in molecular epidemiology of solid cancer. Journal of Cellular and Molecular Imaging 16(1): 8-21.
[41] Gong J, Tong Y, Zhang HM, Wang K, Hu T, Shan G, Sun J, Guo AY (2012). Genomde-Wide Identification
of SNPs in MicroRNA Genes and the SNP Effects on MicroRNA Target Binding and Biogenesis. Human
Mutation 33(1): 254-263.
[42] Guan XX, Sturgis EM, Song XC, Liu ZS, El-Naggar AK, Wei QY, Li GJ (2013). Pre-microRNA variants
predict HPV16-positive tumors and survival in patients with squamous cell carcinoma of the oropharynx. Cancer
letters 330(2): 233-240.
[43] Zhang XF, Yang HS, Lee J, Kim E, Lippman SM, Khuri FR, Spitz MR, Lotan R, Hong WK, Wu XF
(2010). MicroRNA-related genetic variations as predictors for risk of second primary tumor and/or recurrence in
patients with early-stage head and neck cancer. Carcinogenesis 31(12):2118-2123.
[44] Orosos Z, Szanvi I, Csejtei A, Gerlinger I, Ember I, Kiss I (2013). Association of pre-miR-146a rs2910164
Polymorphism with the Risk of Head and Neck Cancer. Anticancer research 33(1): 341-346.
[45] Permuth-Wey J, Thompson RC, Nabors LB, Olson JJ, Browning JE, Madden MH, Chen YA, Egan KM
(2011). A functional polymorphism in the pre-miR-146a gene is associated with risk and prognosis in adult
glioma. Journal of neuro-oncology 105(3): 639-646.
[46] Zhang M, Jin M, Yu J, Zhang S, Wu Y, Liu H, Liu H, Chen B, Li Q, Ma X, Chen K (2012). Associations of
miRNA polymorphisms and female psychological characteristics with breast cancer risk in Chinese population.
European journal of cancer care 21(2): 274-280.
[47] Poulogiannis G, Luo FJ, Arends MJ (2012). RAS signalling in the colorectum in health and disease. Cell
communication and adhesion 19(1): 1-9.
[48] Oh JS, Kim JJ, Byun JY, Kim IA (2010). Lin28-Let7 modulates radiosensitivity of human cancer cells with
48
activation of K-RAS. International journal of radiation oncology biology physics 76(1): 5-8.
[49] Johnson SM, Grosshans H, Shingara J, Byrom M, Jarvis R, Cheng A, Labourier E, Reinert KL, Brown D,
Slack FJ (2005). RAS is regulated by the let-7 MicroRNA family. Cell 120(5): 635-647.
[50] Smits KM, Paraniape T, Nallur S, Wouters KAD, Weijenberg MP, Schouten LJ, van den Brandt PA,
Bosman FT, Weidhaas JB, van Engeland M (2011). A Let-7 MicroRNA SNP in the KRAS 3’UTR Is Prognostic
in Early-Stage Colorectal Cancer. Clinical cancer research 17(24): 7723-7731.
[51] Chin LJ, Ratner E, Leng S, Zhai R, Nallur S, Babar I, Muller RU, Straka E, Su L, Burki EA, Crowell RE,
Patel R, Kulkarni T, Homer R, Zelterman D, Kidd KK, Zhu Y, Christiani DC, Belinsky SA, Slack FJ, Weidhaas
JB (2008). A SNP in a let-7 microRNA complementary site in the KRAS 3′ untranslated region increases nonsmall cell lung cancer risk. Cancer Research 68(20): 8535–8540.
[52] Ratner E, Lu L, Boeke M, Barnett R, Nallur S, Chin LJ, Pelletier C, Blitzblau R, Tassi R, Paranjape T, Hui
P, Godwin AK, Yu H, Risch H, Rutherford T, Schwartz P, Santin A, Matloff E, Zelterman D, Slack FJ,
Weidhaas JB (2010) A KRAS-variant in ovarian cancer acts as a genetic marker of cancer risk. Cancer Res
70(16): 6509-6515.
[53] Christensen BC, Moyer BJ, Avissar M, Ouellet LG, Plaza SL, McClean MD, Marsit CJ, Kelsey KT (2009).
A let-7 microRNA-binding site polymorphism in the KRAS 3’UTR is associated with reduced survival in oral
cancers. Carcinogenesis 30(6): 1003-1007.
[54] Revenkova E, Eijpe M, Heyting C, Gross B, Jessberger R (2001). Novel meiosis-specific isoform of
mammalian SMC1. Molecular and cellular biology 21(20): 6984-6998.
[55] Cobbe N, Heck MMS (2000). SMCs in the world of chromosome biology – From prokaryotes to higher
eukaryotes. Journal of structural biology 129(2-3): 123-143.
[56] http://www.ensembl.org/index.html
[57] http://www.ncbi.nlm.nih.gov/
[58] http://encodeproject.org/cgi-bin/hgPcr
[59] http://www.restrictionmapper.org/
[60] http://www.invitrogen.com/site/us/en/home/Products-and-Services/Applications/Diagnostics-ClinicalResearch/Clinical-Pathology/CP-Misc/SPOT-Light-HER2-CISH-Kit/CISH-Technology-Overview.html
[61] Singhi AD, Westra WH (2010). Comparison of Human Papillomavirus In Situ Hybridization and p16
Immunohistochemistry in the Detection of Human Papillomavirus-Associated Head and Neck cancer Based on a
Prospective Clinical Experience. Cancer 116(9): 2166-2173.
[62] Perisanidis C, Wrba F, Brandstetter A, Kornek G, Mitchell D, Seemann R, Selzer E, Ewers R, Filipits M
(2013). Impact of epidermal growth factor receptor, mesenchymal-epithelial transition factor, and insulin-like
growth factor receptor 1 expression on survival of patients with oral and oropharyngeal cancer. British journal of
oral & maxillofacial surgery 51(3): 234-240.
[64] Loo SW, Geropantas K, Wilson P, Martin WMC, Roques TW (2013). Target Volume Definition for
Intensity-modulated Radiotherapy after Induction Chemotherapy and Patterns of Treatment Failure after
Sequential Chemoradiotherapy in Locoregionally Advanced Oropharyngeal Squamous Cell Carcinoma. Clinical
oncology 25(3): 162-170.
49
Bijlage A : Patiëntengegevens
1. Patiënt-gerelateerde gegevens
Code
RT066
RT071
RT080
RT081
RT083
RT084
RT103
RT134
RT144
RT148
RT163
RT169
RT171
RT189
RT225
RT234
RT245
RT249
RT256
RT273
RT274
RT275
RT286
RT301
RT311
RT316
RT380
RT406
RT426
RT427
RT494
RT512
RT514
RT528
RT541
RT554
RT562
RT564
Geslacht Leeftijd
v
m
v
m
m
m
m
m
m
v
m
m
m
v
m
m
m
m
m
v
m
m
v
m
v
m
m
m
m
m
m
m
m
v
m
m
m
v
37
66
54
47
77
54
58
63
70
68
41
55
58
61
48
49
58
62
41
73
63
66
48
47
64
60
66
41
48
73
58
70
55
52
60
53
68
66
Type
roken
current
current
former
former
former
former
former
never
current
current
former
current
former
current
current
former
current
never
current
never
current
former
former
current
current
current
former
never
never
former
former
former
former
current
former
never
former
never
Packyears
30
25
60
23
7
4
53
0
63
13
20
8
28
44
50
28
50
0
25
0
54
45
60
38
50
12
55
0
0
40
60
23
35
135
0
28,5
0
Type
alcoholgebruik
never
current
current
former
current
current
current
never
current
never
former
current
current
current
current
current
current
current
current
current
current
former
current
current
former
former
current
current
current
former
current
current
current
current
current
never
former
current
Drinks/week
0
35
50
40
12
8
35
0
70
0
70
21
6
12
28
60
40
5
44
11
7
60
30
45
12
60
10
2
25
14
10
24
7
35
2
0
130
8
Code
RT575
RT579
RT585
RT596
RT600
RT608
RT614
RT620
RT657
RT664
RT672
RT679
RT681
RT688
RT692
RT697
RT700
RT727
RT736
RT737
RT745
RT748
RT772
RT780
RT791
RT799
RT808
RT814
RT833
RT847
RT854
RT855
RT862
RT879
Geslacht Leeftijd
m
m
m
m
v
m
m
m
m
m
v
m
v
m
m
m
m
m
v
m
m
m
m
m
m
m
v
m
m
m
m
m
v
v
68
67
54
68
73
66
52
50
78
43
56
62
66
52
61
50
67
59
57
55
62
57
71
66
56
70
61
55
57
60
51
64
54
38
Type
roken
former
former
former
former
current
former
current
current
former
former
former
former
current
current
former
former
former
former
current
current
current
former
former
former
current
former
current
former
former
current
current
current
current
current
Packyears
28,7
10
22
37,5
37,5
30
37,5
40
22,5
50
30
27
38
18
25
7,5
80
39,5
45
25
50
10
10
20
123
40
22,5
12
41,3
21
35
60
37,5
10
Type
alcoholgebruik
former
current
current
former
current
current
former
current
current
current
current
current
current
current
current
current
former
current
current
current
current
current
current
current
current
current
current
current
former
current
current
current
current
never
Drinks/week
75
1
21
60
7
1
56
12
1
60
21
2
1
7
3
20
100
50
14
70
20
2
7
70
110
4
28
28
45
28
45
15
3
0
2. Klinische gegevens
Code
RT066
RT071
RT080
RT081
RT083
RT084
RT103
RT134
RT144
RT148
RT163
RT169
RT171
RT189
RT225
RT234
RT245
RT249
RT256
RT273
RT274
RT275
RT286
RT301
RT311
RT316
Subsite
tonsil
multiple subsites:
mondbodem
posterior wall
maxilla
multiple subsites:
superior wall
tonsil
superior wall
base of tongue
multiple subsites:
multiple subsites:
tonsil
multiple subsites:
tonsil
multiple subsites:
tonsil
tongbasis
uvula
soft palate
tongrand
base of tongue
tongbasis en
tongrand
Tstage
4
3
3
4
4
4
1
1
4
4
3
3
1
3
2
4
4
3
4
1
1
3
2
4
2
Nstage
2
3
0
2
0
2
2
2
0
3
2
2
2
2
2
2
1
2
2
2
1
2
0
2
0
Chemo
Heelkunde Halsevidement
ja
ja
nee
ja
nee
ja
nee
ja
ja
ja
ja
ja
ja
ja
nee
ja
ja
ja
nee
ja
ja
nee
ja
ja
nee
nee
nee
ja
nee
ja
nee
ja
ja
nee
nee
nee
nee
ja
nee
ja
nee
nee
nee
nee
nee
ja
nee
ja
nee
ja
3
3
ja
nee
Type RT
Status
ja
ja
ja
ja
nee
ja
ja
ja
ja
nee
ja
ja
ja
ja
ja
ja
nee
ja
nee
ja
ja
ja
ja
nee
nee
3DRT
IMRT
3DRT
IMRT
3DRT
IMRT
IMRT
3DRT
IMRT
IMRT
IMRT
IMRT
3DRT
IMRT
3DRT
IMRT
IMRT
IMRT
IMRT
3DRT
3DRT
IMRT
3DRT
IMRT
3DRT
dead
dead
dead
dead
dead
alive
dead
alive
alive
dead
dead
dead
alive
dead
alive
dead
dead
dead
dead
alive
dead
dead
alive
dead
dead
ja
IMRT
dead
Code
RT380
RT406
RT426
RT427
RT494
RT512
RT514
RT528
RT541
RT554
RT562
RT564
RT575
RT579
RT585
RT596
RT600
RT608
RT614
RT620
RT657
RT664
RT672
RT679
RT681
Subsite
Tstage
Nstage
Chemo
Heelkunde Halsevidement
tonsil
lateral wall
lateral wall
tonsil
tonsil en weke
verhemelte
tonsil
tonsil
soft palate
tonsil
lateral wall
tonsil
tongbasis
base of tongue
tonsil
soft palate
tonsil
tonsil
base of tongue
tonsil
tonsil
tonsil
lateral wall
posterior wall
tonsil
tonsil
2
2
1
3
2
2
2
2
0
2
nee
ja
nee
nee
nee
ja
nee
nee
ja
nee
2
3
3
4
3
1
1
2
3
1
2
3
2
3
2
3
2
1
2
4
0
2
0
2
2
2
2
2
2
3
1
0
2
1
3
0
2
0
3
2
nee
nee
ja
ja
ja
nee
nee
nee
ja
ja
nee
nee
ja
ja
ja
nee
nee
nee
ja
ja
ja
ja
nee
nee
nee
ja
nee
nee
nee
nee
ja
nee
nee
ja
nee
ja
ja
ja
nee
nee
Type RT
Status
ja
ja
ja
nee
ja
3DRT
IMRT
IMRT
IMRT
3DRT
dead
dead
alive
alive
alive
ja
ja
nee
ja
ja
ja
ja
ja
ja
nee
ja
nee
ja
nee
ja
ja
ja
nee
nee
nee
3DRT
3DRT
IMRT
IMRT
IMRT
IMRT
3DRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
dead
dead
alive
alive
alive
dead
alive
alive
alive
dead
alive
dead
alive
alive
dead
alive
alive
alive
alive
alive
Code
RT688
RT692
RT697
RT700
RT727
RT736
RT737
RT745
RT748
RT772
RT780
RT791
RT799
RT808
RT814
RT833
RT847
RT854
RT855
RT862
RT879
Subsite
Tstage
Nstage
Chemo
tonsil
tonsil
tonsil
base of tongue
tonsil
posterior wall
posterior wall
base of tongue
tonsil
tonsil
base of tongue
tonsil
base of tongue
posterior wall
base of tongue
tonsil
tonsil
tonsil
tonsil
tonsil
tonsil
4
2
2
2
3
3
3
2
2
2
3
2
2
2
1
2
1
4
4
4
2
2
2
2
2
1
1
2
1
2
0
2
2
1
0
2
2
1
2
2
2
2
ja
nee
ja
ja
ja
ja
ja
nee
nee
nee
nee
nee
nee
nee
ja
ja
nee
ja
ja
ja
ja
Heelkunde Halsevidement
nee
nee
ja
nee
nee
nee
nee
nee
nee
nee
nee
nee
nee
nee
nee
ja
nee
nee
nee
nee
nee
nee
ja
ja
ja
nee
nee
nee
nee
ja
nee
ja
nee
nee
nee
ja
ja
nee
nee
ja
ja
ja
Type RT
Status
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
IMRT
dead
alive
dead
alive
dead
alive
alive
alive
alive
alive
alive
alive
alive
alive
dead
dead
alive
alive
alive
alive
alive
Bijlage B : Gebruikte RFLP protocols
1. Reeds geoptimaliseerde protocols binnen de onderzoeksgroep
Drosha rs639174 (G>A)
PCR
- Primers
Forward : Drosha/98361F
Reverse: Drosha/98831R
5'-GATTACAGGAGGTAAGCCGA-3'
(270)
5'-GAGCAAGAGTTTATACTAGAATA-3' (271)
- PCR mix: Standard
- PCR conditions: Touchdown 70°C
- PCR fragment size: 493 bp
Digest
- Enzyme: TatI
- Incubation: 65°C for 4 hours
Digest volume
30 µl
dH2O
17,9 µl
PCR product
10 µl
Buffer tango
2 µl
TatI (0,5 U)
0,1 µl
Expected digest product:
GG: 493 - GA: 493/273/220 -AA: 273/220
Drosha rs3805500 (T>C)
PCR
- Primers
Forward Drosha/69198F
5’-CTGATGATCTCAGTGATAAGTC-3’
(262)
Reverse: Drosha/69554R
5’-ACTGGAACAGATAATTCAGCAC-3’
(261)
- PCR mix: Standard
- PCR conditions: Touchdown starting by 64°C
- PCR fragment size: 378 bp
Digest
- Enzyme: ApoI
- Incubation: 50°C for 2-3 hours
Digest volume
30 µl
dH2O
19,5 µl
PCR product
7 µl
BSA
0,3 µl
NEB3
3 µl
ApoI (2U)
0,2 µl
Expected digest product:
CC: 378 - CT: 378/272/106 - TT: 272/106
KRAS rs61764370 (T>G)
PCR
- Primers : 293/294
- PCR mix : standard + 10% DMSO
- PCR conditions : touchdown program 72°C
PCR fragment size : 762bp
Digest
Enzyme : TfiI Fastdigest
Enzyme : TfiI
Incubation : 37°C for 20-30 minutes
Incubation : 37°C for 3 hours
Digest volume
30 µl
Digest
30 µl
volume
dH2O
17 µl
dH2O
PCR product
10 µl
PCR product
10 µl
Fastdigest Green buffer
2 µl
Buffer O
2 µl
1 µl
TfiI (Fast)
Expected digest product :
TT :
83 / 101 / 135 / 167 / 276
TG:
83 / 101 / 135 / 167 / 276 / 411
GG : 83 / 101 / 167 / 411
17.75 µl
0.25 µl (2.5U)
TfiI (10U/µl)
2. Geoptimaliseerd protocol binnen deze studie
XPO5 rs2227301 (G>A)
PCR
- Primers
Forward (446) 5'- TATATCCTGAACTTCACTGAC -3'
Reverse (447) 5'- CACTGTCCCTTCGTGGAC -3'
- PCR mix: Standaard
- PCR conditions: Algemeen 56°C / Touchdown 58°C
- PCR fragment size: 542 bp
Digest
- Enzyme: BsrI
- Incubation: 65°C (3u)
Digest volume
30 µl
dH2O
17,6 µl
PCR product
9 µl
NEB3
3 µl
BsrI (2U)
0,4 µl
Expected digest product:
GG : 265 / 277 - GA : 265 / 277 / 542 - AA : 542
Bijlage C : Gebruikte HRM protocols
1. Geoptimaliseerde protocols binnen de onderzoeksgroep
KRAS rs61764370 (T>G)
PCR-HRM
- Mix : AB Meldoctor reagentia:
Reagent
Concentration
dNTP mix
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1,2
primer 290
5 µM
1.2
primer 292
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.95
20
- PCR conditions :
95°
10 min
95°
15 sec - 60° 1 min
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
- PCR fragment size : 107 nt
: 40 cycli
Drosha rs10520985 (C>T)
PCR-HRM
- Mix : AB Meldoctor reagentia :
Reagent
Concentration
dNTP mix
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1.2
primer 280
5 µM
1.2
primer 282
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.95
20
PCR mix: Standard HRM mix + 1.5µl DMSO per sample
- PCR conditions :
95°
10 min
95°
15 sec - 62° 1 min
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
- PCR fragment size: 162bp
: 40 cycli
2. Geoptimaliseerde protocols binnen de studie
XPO5 rs699937 (C>T)
PCR-HRM
- Mix : AB Meldoctor reagentia :
Reagent
dNTP mix
Concentration
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1.2
primer 448
5 µM
1.2
primer 450
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.95
20
PCR conditions :
95°
10 min
95°
15 sec - 60°
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
1 min
PCR fragment size : 173 nt
Drosha rs17410035 (C>A)
PCR-HRM
- Mix : AB Meldoctor reagentia :
Reagent
Concentration
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1,2
primer 420
5 µM
1.2
primer 422
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.95
dNTP mix
20
: 40 cycli
PCR conditions :
95°
10 min
95°
15 sec - 60° 1 min : 40 cycli
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
PCR fragment size : 96 nt
SMC1B rs3747238 (A>G)
PCR-HRM
- Mix : AB Meldoctor reagentia :
Reagent
dNTP mix
Concentration
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1.8
primer 451
5 µM
1.2
primer 454
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.35
20
PCR conditions :
95°
95°
10 min
15 sec - 58°
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
PCR fragment size : 100 nt
1 min :
40 cycli
Pre-microRNA196a2 rs11614913 (C>T)
PCR-HRM
- Mix : AB Meldoctor reagentia :
Reagent
Concentration
dNTP mix
Amount (µl)
10mM
0.4
buffer
10X
2
MgCl2
25 mM
1,2
primer 411
5 µM
1.2
primer 412
5 µM
1.2
AmpliTaq
5 U/µl
0,05
HRM dye
20X
1
DNA
25 ng/µl
1
H2O
-
11.95
20
PCR conditions :
95°
95°
10 min
15 sec - 60°
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
PCR fragment size : 123 nt
1 min :
40 cycli
Pre-microRNA146a rs2910164 (G>C) – spike in HRM
Step 1:discrimination between homozygous and heterozygous genotypes
Reagent
(Meltdoctor) Concentration
dNTP mix
Amount (µl)
Reagent
(Syto9)
concentration Amount (µl)
10 mM
0.4
dNTPmix
10 mM
0.4
buffer
10X
2
Buffer
10X
2
MgCl2
25 mM
1,2
MgCl2
25 mM
1.2
primer 416
5 µM
1.2
Primer 416
5 µM
1.2
primer 417
5 µM
1.2
Primer 417
5 µM
1.2
AmpliTaq
5 U/µl
0,05
Amplitaq
5 U/µl
0.05
HRM dye
20X
1
Syto9
50 µM
0.6
DNA
25 ng/µl
1
DNA
25 ng/µl
1
H2O
-
11.95
H2O
-
12.35
20
PCR conditions :
95°
10 min
95°
15 sec - 60°
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
PCR fragment size : 132 nt
20
1 min
: 40 cycli
Step2: only the homozygous genotypes are being used, discrimination between GG and CC
genotypes
Reagent
(Meltdoctor)
dNTP mix
Concentration Amount (µl)
Reagent
(Syto9)
concentration
Amount
(µl)
10 mM
0.4
dNTPmix
10 mM
0.4
buffer
10X
2
Buffer
10X
2
MgCl2
25 mM
1,2
MgCl2
25 mM
1.2
primer 416
5 µM
1.2
Primer 416
5 µM
1.2
primer 417
5 µM
1.2
Primer 417
5 µM
1.2
AmpliTaq
5 U/µl
0,05
Amplitaq
5 U/µl
0.05
HRM dye
20X
1
Syto9
50 µM
0.6
25 ng/µl
1
DNA
25 ng/µl
1
25 ng/µl
1
25 ng/µl
1
Spike-in
DNA
-
10.95
H2O
-
11.35
DNA
Spike-in DNA
H2O
20
20
PCR conditions :
95°
10 min
95°
15 sec - 60°
95°
10 sec
60°
1 min
95°
15 sec
60°
15 sec
PCR fragment size : 132 nt
1 min
: 40 cycli
Bijlage D : Resultaten SNP analyse
Code
RT066
RT071
RT079
RT080
RT081
RT083
RT084
RT103
RT131
RT134
RT141
RT144
RT148
RT163
RT165
RT169
RT171
RT176
RT189
RT225
RT232
RT234
RT243
RT245
DROSHA
rs639174
GA
GG
GG
GG
GG
GA
GG
GG
GA
GG
GG
GA
GA
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GA
rs3805500
CT
TT
TT
TT
CT
TT
CT
TT
CT
CT
CT
TT
TT
TT
TT
CT
CT
CC
TT
CT
TT
CT
TT
CT
PRE-MICRO
RNA
rs10520985 rs17410035 146a
196a2
CC
CC
/
CC
CT
CA
GC
CT
CT
CC
GG
CT
CT
CC
GG
TT
CT
CC
GG
CC
CT
CA
GG
CT
CT
CC
GG
CT
TT
CA
CC
CT
CT
CA
GG
CT
CC
CC
GG
CT
CC
CC
GC
CT
TT
CA
GG
CC
TT
AA
GC
CT
CC
CC
GG
CC
TT
CA
GC
CT
CT
AA
GC
TT
CT
CA
GG
TT
CC
CA
/
TT
CT
CC
GC
TT
CC
CC
GG
CT
TT
CA
GG
TT
CC
CC
GG
CC
TT
CA
GG
CC
CT
CC
GC
CC
KRAS
XPO5
SMC1B
rs61764370 rs2227301 rs699937 rs3747238
TT
GG
CC
AA
TT
GG
CC
AA
TT
GA
CT
AA
TG
GA
CT
AG
TT
GG
CC
AG
TT
GG
CC
AA
TT
GA
CT
AG
TT
GA
CT
AG
TT
GA
CT
AG
TT
GA
CT
GG
TT
GA
CT
GG
TT
GG
CT
AG
TT
GG
CT
AG
TG
GG
CC
AG
GG
GG
CC
AA
TT
GG
CC
AG
TT
GA
CT
AA
TT
GA
/
/
TT
GA
CT
AG
TT
GG
CC
AG
TT
GG
CT
AA
TT
GA
CT
GG
TT
GA
CT
AG
TT
GA
CT
GG
Code
RT246
RT249
RT256
RT258
RT267
RT273
RT274
RT275
RT286
RT301
RT304
RT305
RT311
RT316
RT329
RT352
RT380
RT406
RT416
RT426
RT427
RT494
RT495
RT503
RT512
RT514
RT519
DROSHA
rs639174
GG
GG
AA
GA
GG
GG
GG
GG
GA
GA
GA
GA
GG
GA
GG
GA
GG
GA
GG
GA
GG
GG
GA
GG
GG
AA
GA
rs3805500
TT
TT
CC
CT
TT
TT
CT
TT
TT
CC
CT
CT
CT
CT
TT
CC
CT
CT
TT
CT
TT
TT
CT
CT
CT
TT
CT
PRE-MICRO
RNA
rs10520985 rs17410035 146a
196a2
TT
CC
GC
CC
CT
CA
GC
TT
CC
CC
GC
CC
CT
CC
CC
CC
TT
AA
GG
TT
CT
CC
CC
CT
CT
CA
GG
CC
TT
AA
GC
CT
CT
CA
GG
CT
CC
CA
GC
CT
CT
CC
GC
CT
CT
CA
GG
CC
CT
CA
GG
CT
TT
CC
GG
CT
CC
CA
GC
CT
CC
CA
GC
CT
CT
CA
GG
CT
CT
CA
GG
CT
TT
CA
GG
CT
CT
CC
GC
CC
TT
AA
GG
CT
TT
CA
GG
CT
CC
CC
GC
CT
CT
CA
GC
CC
TT
CC
GC
CC
CC
CA
GC
CC
CC
CC
GG
CT
KRAS
XPO5
SMC1B
rs61764370 rs2227301 rs699937 rs3747238
TT
GA
CT
GG
TT
GG
CC
AG
TT
GG
CC
AA
TT
GG
CC
AG
TT
GG
CC
AA
TT
GG
CC
AG
TT
GA
CT
AG
TG
GG
CT
GG
TT
GG
CC
AA
TT
GG
CT
AG
TT
GG
CC
AG
TT
GG
CC
AG
TT
GG
CT
AG
TT
GA
CT
AA
TG
GG
CC
AG
TT
GA
CT
AG
TT
GG
CC
AG
TT
AA
TT
AG
TT
GA
CT
AG
TT
GA
CT
AA
TT
GA
CT
GG
TT
GG
CC
GG
TG
GG
CC
AA
TT
GG
CC
AG
TT
GG
CC
AG
TG
GG
CT
GG
TT
GG
CT
AG
Code
RT528
RT531
RT532
RT541
RT543
RT554
RT562
RT564
RT575
RT579
RT584
RT585
RT586
RT590
RT592
RT593
RT596
RT597
RT600
RT608
RT614
RT618
RT620
RT636
RT657
RT664
RT672
DROSHA
rs639174
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GA
GG
GG
GG
GG
GG
GG
GA
GA
GA
GG
GA
GG
GG
rs3805500
CT
TT
TT
TT
CT
CT
CC
CT
TT
TT
TT
CT
TT
CT
TT
TT
CT
CT
TT
TT
CT
CT
CT
CT
CT
CT
TT
PRE-MICRO
RNA
rs10520985 rs17410035 146a
196a2
CT
CA
GG
CC
CT
CC
GG
CC
CT
CA
GG
TT
TT
CA
GC
CT
CT
CA
GC
CT
CC
CC
GC
CC
CC
/
GC
CC
CC
CC
GG
CT
/
CC
GC
CT
TT
AA
GC
TT
CT
CA
GC
CC
CT
CC
GC
CT
CT
CC
GG
TT
CT
CA
GC
CT
/
CC
GG
CT
TT
AA
GC
CT
CT
CC
GC
TT
CC
CC
GG
TT
TT
CA
GG
CT
CT
/
GG
CC
CT
CC
GC
CT
CC
CC
GC
CC
CC
CC
GG
CT
CT
CA
GG
CT
CC
CC
GG
CT
CC
CA
GG
CC
CC
CC
GG
TT
KRAS
XPO5
SMC1B
rs61764370 rs2227301 rs699937 rs3747238
TT
GG
CC
AG
TG
GG
CT
AA
TT
GG
CC
AG
TT
GG
CT
AG
TT
GA
TT
AA
TT
GA
CT
AA
TT
GG
CT
AG
TT
GA
TT
AG
TT
GA
CT
GA
TT
GA
CT
GA
TG
GG
CC
GA
TG
GA
CT
GG
TT
GA
CT
GA
TT
GG
CC
AA
TT
GA
CT
AA
TT
GG
CC
AA
TG
GG
CC
GA
TT
GA
CT
GA
TT
GG
CC
GA
TT
GG
CC
GG
TT
GA
CT
GA
TT
GA
TT
GA
TG
GG
CC
GA
TT
GG
CC
AA
TG
GG
CT
GA
TG
GG
CT
GA
TT
GG
CC
GA
Code
RT679
RT681
RT688
RT692
RT694
RT700
RT703
RT709
RT727
RT736
RT737
RT740
RT745
RT748
RT759
RT760
RT772
RT780
RT783
RT791
(op)
RT793
RT799
RT808
RT814
RT824
RT833
DROSHA
rs639174
GG
GG
GA
GG
GA
GG
GG
GG
GG
GG
GA
GG
GG
GG
GA
GG
GG
GG
GG
/
GA
GA
GG
GG
GG
GA
rs3805500
CT
TT
CC
TT
CT
TT
CT
TT
TT
TT
CT
CT
CT
CT
CT
TT
TT
TT
CT
/
CT
CC
TT
CT
CT
TT
PRE-MICRO
RNA
rs10520985 rs17410035 146a
196a2
CT
/
GC
CT
CT
/
GG
TT
CC
CC
GG
CC
TT
/
GG
CC
CT
CA
GG
CT
CT
CA
GC
CT
CT
CC
GC
CC
CC
CC
GG
CT
CT
CC
GG
CT
/
AA
CC
CC
CT
CA
GC
TT
CT
CC
GG
CT
CT
/
GC
CC
CT
CC
GC
CC
CT
CA
GC
CT
CT
/
GG
TT
CC
AA
GG
CC
CT
/
GG
CT
CT
/
GG
/
/
/
/
/
CT
CC
CC
CT
CC
CC
CA
CC
CA
CC
CC
CA
GG
GC
GG
GC
GC
GG
CC
CC
CT
CC
CC
CC
KRAS
XPO5
SMC1B
rs61764370 rs2227301 rs699937 rs3747238
TG
GG
CT
AA
TG
GG
CC
GA
TT
GG
CC
AA
TT
GA
CT
GA
TT
GG
CT
AA
TT
GG
CC
AA
TT
GG
/
GA
TG
GG
CC
GA
TT
GG
CT
GA
TT
GA
CT
AA
TT
GA
CT
GA
TT
GG
CT
AA
TT
AA
TT
GG
TT
GG
CC
GA
TT
GG
CC
AA
TT
GA
CT
GA
TG
AA
TT
GA
TT
GG
CC
GA
TT
GG
CC
GA
/
/
/
/
TG
TT
TG
TT
TT
TT
GG
GG
GA
GA
GG
GG
CC
CC
CT
CT
CC
CC
AA
GA
AA
GA
AA
AA
Code
RT846
RT847
RT854
RT855
RT860
RT862
RT879
DROSHA
rs639174
GA
GA
GA
GG
GA
AA
GG
rs3805500
CT
TT
CT
CT
CT
CC
CT
PRE-MICRO
RNA
rs10520985 rs17410035 146a
196a2
CT
CA
GG
TT
CC
/
GC
CT
CT
CA
GC
CT
CT
CA
GG
CT
TT
CA
GG
CT
CC
CC
GG
CC
CC
CC
GC
CT
KRAS
XPO5
SMC1B
rs61764370 rs2227301 rs699937 rs3747238
TT
GG
CC
GG
TT
GA
TT
GA
TT
GG
CC
AA
TG
GA
CT
GA
TT
GA
CT
GA
TT
GA
CT
GA
TT
GG
CC
AA
Bijlage E : Resultaten Analyses
1. Associatie microRNA genotypes met overleving volgens het recessief model
miRNA
genotypes
Drosha
rs639174
GG+GA
AA
Sterfte
Totaal
(eender
aantal
welke
patiënten oorzaak)
p
Sterfte
(kanker
progressie of
ziekte
gerelateerd)* p
Sterfte,
herval of metastase
p
67
3
29
2
0,456
11
0
0,561
34
2
0,376
rs10520985
CC
CT+TT
56
12
25
6
0,805
10
1
0,387
29
6
0,750
rs3805500
TT+TC
CC
64
6
27
4
0,003
10
1
0,435
32
4
0,050
rs17410035
CC+CA
AA
55
7
27
3
0,799
10
0
0,267
29
4
0,699
XPO5
rs2227301
GG+GA
AA
67
3
30
1
0,883
10
1
0,604
35
1
0,682
rs699937
CC+CT
TT
65
5
30
1
0,410
10
1
0,927
34
2
0,759
premicroRNA
146a
rs2910164
GG+GC
CC
66
3
29
1
0,929
10
1
0,301
33
2
0,425
196a2
rs11614913
CC+CT
TT
60
10
27
4
0,487
10
1
0,538
32
4
0,473
25
miRNA
genotypes
SMC1B
rs3747238
AA+AG
GG
Sterfte
Totaal
(eender
aantal
welke
patiënten oorzaak)
62
10
27
5
p
0,515
Sterfte
(kanker
progressie of
ziekte
gerelateerd)* p
10
2
KRAS
rs61764370
TT+TG
70
31
11
GG
0
0
0
* Totaal aantal patiënten voor analyse naar DSS toe bedroeg 64
Sterfte,
herval of metastase
0,911
32
5
p
0,361
36
0
26
2. Associatie tumor HPV status met microRNA genotypes
HPV+ (CISH)
HPV- (CISH)
p
OR
HPV+ (p16)
HPV- (p16)
p
OR
Drosha
rs639174
GG
GA+AA
10
3
35
19
1*
0,408
0,553
14
5
31
13
1
0,543
0,692
rs10520985
CC
CT+TT
6
7
15
37
1
0,238
0,473
7
12
13
32
1
0,532
0,696
rs3805500
TT
TC+CC
7
6
23
31
1
0,465
0,636
7
12
23
24
1
0,374
1,643
rs17410035
CC
CA+AA
6
5
23
25
1
0,692
0,767
11
6
17
24
1
0,112
0,386
XPO5
rs2227301
GG
GA+AA
7
6
32
22
1
0,723
1,247
10
9
28
19
1
0,606
1,326
rs699937
CC
CT+TT
5
8
23
31
1
0,786
1,187
8
11
19
28
1
0,9
0,933
HPV+ (CISH)
HPV- (CISH)
p
OR
HPV+ (p16)
HPV- (p16)
p
OR
146a rs2910164
GG
GC+CC
7
6
26
27
1
0,757
0,830
10
9
23
23
1
0,847
0,900
196a2 rs11614913
CC
CT+TT
4
9
22
32
1
0,510
1,547
9
10
16
31
1
0,315
0,573
SMC1B
rs3747238
AA
AG+GG
3
11
14
41
1
0,755
1,252
6
14
11
37
1
0,540
0,694
16
3
36
11
1
0,496
0,614
pre-microRNA
KRAS
rs61764370
TT
10
43
1
TG+GG
3
11
0,830
1,173
*1 = referentie. / De resultaten uit deze tabel werden bekomen via logistieke regressie