Moleculaire biologische technieken
Hand-out
Juli 2010
In deze hand-out de meeste teksten. Van de (eigen) animaties zijn enkele belangrijke frames
weergegeven. Als je de links wilt weten moet je de module zelf starten en daar de links opzoeken.
De vragen zijn open gelaten, zodat je zelf de antwoorden erbij kunt schrijven.
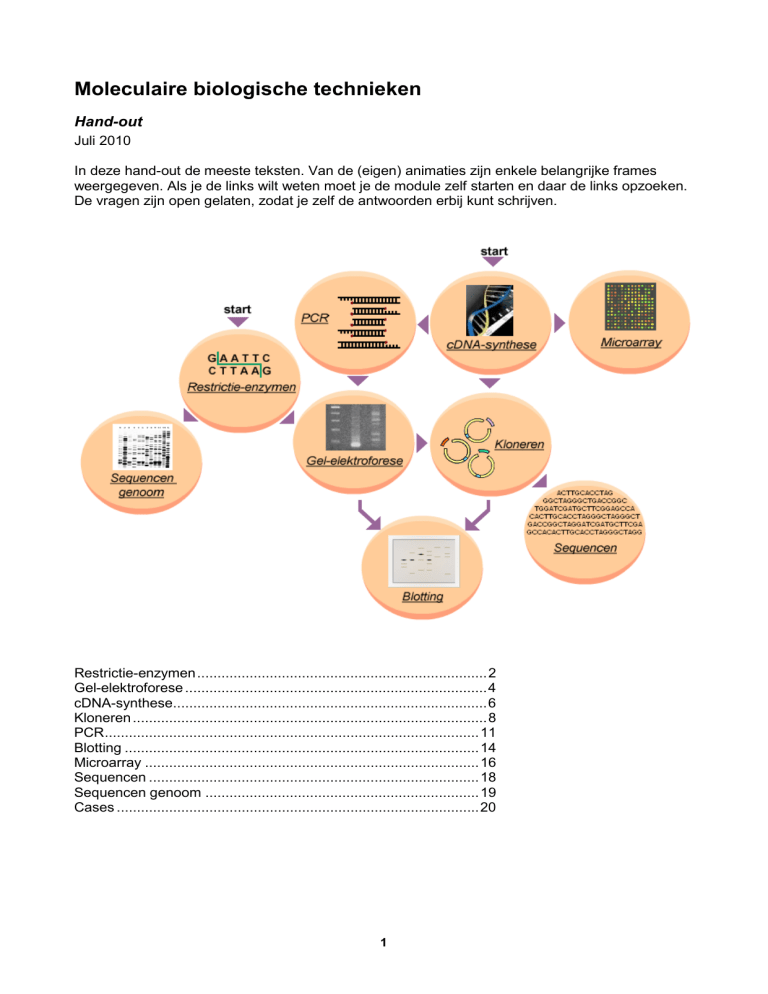
Restrictie-enzymen ........................................................................ 2
Gel-elektroforese ........................................................................... 4
cDNA-synthese.............................................................................. 6
Kloneren ........................................................................................ 8
PCR............................................................................................. 11
Blotting ........................................................................................ 14
Microarray ................................................................................... 16
Sequencen .................................................................................. 18
Sequencen genoom .................................................................... 19
Cases .......................................................................................... 20
1
Restrictie-enzymen
———————————————————————————————————————————
Uitgangsmateriaal: DNA
———————————————————————————————————————————
Eindproduct:
DNA-fragmenten, geknipt op specifieke plaatsen
———————————————————————————————————————————
Gebruik:
- verschillen aantonen in DNA
- kloneren
———————————————————————————————————————————
Principe
Restrictie-enzymen knippen een dubbelstrengs DNA-streng op een specifieke plaats. Ze komen
uit bacteriën, en heten daarnaar: zo komt EcoR I uit E(scherichia) coli.
Sommige restrictie-enzymen geven rechte uiteinden ('blunt ends'). Andere geven ongelijke einden
('sticky ends') die ook weer makkelijk aan elkaar binden. Er zijn heel veel verschillende restrictieenzymen, waarvan er hieronder vijf staan.
Not I, EcoR I en Hind III knippen ongelijk af en er onstaat een ‘sticky end’. Hae III en Alu I knippen
recht af en er ontstaat een ‘blunt’ einde. Restrictie-enzymen werken op vaste sequenties, dus zijn
de 'sticky' uiteinden voor één restrictie-enzym altijd gelijk. Hierdoor is het mogelijk vreemd DNA
aan elkaar te 'plakken', dus ook om vreemde genen in het DNA van een bepaald organisme te
brengen. Deze organismen worden dan transgeen.
Toepassingen
DNA-fingerprinting
Er ontstaan bij restrictie een vast aantal fragmenten met een vaste grootte.
Dit geeft voor elk individu een vast en uniek patroon, dat zichtbaar gemaakt kan worden met gelelektroforese (zie aldaar).
Dit patroon noemt men ook wel de DNA-fingerprint, en wordt gebruikt bij politieonderzoek.
Bekijk ook de animatie RFLP’s (Restriction Fragment Length Polymorphisms)
Kloneren
Doordat de restrictie-enzymen op vaste sequenties werken, zijn de losse uiteinden altijd gelijk.
Hierdoor is het mogelijk vreemd DNA aan elkaar te 'plakken', dus ook om vreemde genen in het
DNA van een bepaald organisme te brengen. Dit worden dan transgene organismen.
Links
Animatie van EcoR I:
http://highered.mcgrawhill.com/olcweb/cgi/pluginpop.cgi?it=swf::535::535::/sites/dl/free/0072437316/120078/bio37.swf::R
estriction%20Endonucleases
Extra links:
Simpeler: http://www.dnalc.org/ddnalc/resources/restriction.html
Kort over drie verschillende restrictie-enzymen: http://www.sinauer.com/cooper5e/animation0406.html
Overzicht van alle restrictie-enzymen: http://www.genscript.com/cgi-bin/products/enzyme.cgi?op=all_ez
Toepassing: RFLP's (Restriction Fragment Length Polymorphisms, interne animatie)
2
Vragen Restrictie-enzymen
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Elk restrictie-enzym knipt altijd in een specifieke sequentie. Kun je hierdoor ook DNA's van
verschillende organismen koppelen?
A. Ja, want DNA van alle organismen bestaat uit dezelfde bouwstenen
B. Nee, want ieder DNA heeft een andere sequentie
2. Restrictie-enzymen zijn afkomstig van bacteriën, en ontlenen hun naam daaraan. Bacteriën
gebruiken restrictie-enzymen om vreemd DNA af te breken. Hoe zullen bacteriën voorkomen
dat het eigen DNA ook wordt afgebroken?
A. Eigen DNA heeft deze sequenties niet.
B. Eigen DNA is beschermd door modificaties
C. De restrictie-enzymen bevinden zich niet in de kern.
3. Je kunt de ontstane fragmenten scheiden op grootte. Geef aan waarbij (vrijwel) alle
fragmenten DNA gelijk zijn:
A. Lichaamscellen van één individu
B. Lichaamscellen van verschillende individuen van één soort
C. Lichaamscellen van individuen van verschillende soorten
4. Van plasmide pJH is bekend waar de restrictie-enzymen EcoR I, Hind
III en Not I knippen.
A. Welke fragmenten ontstaan er als alleen Not I wordt gebruikt?
B. Als deze alledrie worden gebruikt ontstaan er fragmenten
van 400, 500, 600, 700 en 800 nucleotiden. Welke zijn er gebruikt als er
fragmenten van 400 en 1300 nucleotiden ontstaan?
3
Gel-elektroforese
———————————————————————————————————————————
Uitgangsmateriaal: een mengsel van (verschillende) DNA-fragmenten (zie ook: restrictie-enzym)
———————————————————————————————————————————
Eindproduct:
een streepjespatroon in een gel
———————————————————————————————————————————
Gebruik:
- vrijwel altijd uitgevoerd na behandeling met restrictie-enzym of PCR
- vergelijken van grootte en hoeveelheid DNA
———————————————————————————————————————————
Principe
De basis van deze techniek is een gel die poreus is zodat grote moleculen zich daar
langzaam door kunnen verplaatsen. In deze gel worden aan één zijde een aantal
uitsparingen gemaakt (slotjes). Hierin komt straks het mengsel fragmenten.
Als referentie gebruik je ook een mengsel DNA-fragmenten met
een bekende lengte. Omdat de oplossingen met DNA kleurloos
zijn wordt er een kleurstof aan toegevoegd om de gelelektroforese te kunnen volgen.
Doe het mengsel in één van de slotjes. De andere slotjes kun
je vullen met andere DNA-mengsels. In één slot in het midden
van de gel doe je het DNA-mengsel met bekende lengte.
Je doet de hele gel in een bak met een buffer. Zet een
potentiaalverschil over de gel. DNA is negatief geladen en
beweegt daarom naar de positieve pool. De kleurstofmoleculen zijn klein en gaan dus het snelst. Als deze beneden
is, is de gel klaar.
Het DNA moet je nog zichtbaar maken. Dit doe je met een stof
die hecht aan DNA (meestal ethidium-bromide) en het DNA
zichtbaar maakt onder UV-licht.
Aan de hand van de fragmenten DNA met bekende lengte (marker) kun je aflezen wat de lengte
van de gevonden fragmenten is.
Hieronder zie je een foto van een gel onder UV-licht, waarbij met EtBr gekleurd is..
4
Toepassingen
Genen opsporen
Door gel-elektroforese worden de fragmenten van elkaar gescheiden. Met Southern blotting kun je
specifieke sequenties zoeken, en zo de fragmenten DNA vinden die een bepaald gen bevatten.
DNA-fingerprinting
Er ontstaan bij restrictie een vast aantal fragmenten met een vaste grootte. Dit geeft voor elk
individu een vast en uniek patroon, dat zichtbaar gemaakt kan worden met gel-elektroforese. Dit
patroon noemt men ook wel de DNA-fingerprint, en wordt gebruikt bij politieonderzoek.
NB Gel-elektroforese wordt eigenlijk altijd na een bewerking met restrictie-enzymen of PCR
gedaan, en deze combinatie wordt weer bij heel veel technieken gebruikt.
Links
Bij het boek 'Life', van Purves (let op: aan het einde komt de techniek 'blotting'):
http://www.sumanasinc.com/webcontent/anisamples/majorsbiology/gelelectrophoresis.html
Extra links:
Zelf een 'virtueel' experimentje uitvoeren: http://learn.genetics.utah.edu/units/biotech/gel/
Resultaat bij verschillende restrictie-enzymen bekijken:
http://www.wiley.com/legacy/college/boyer/0470003790/animations/agarose/agarose.htm
Vragen Gel-elektroforese
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Je hebt hetzelfde gen geïsoleerd bij 3 verschillende mensen (I, II en III). Je
behandelt dit met een restrictie-enzym, en na gel-elektroforese zie je dit:
A. Welke verklaring heeft dit verschil?
A. Alleen III heeft een werkend gen
B. Persoon III is heterozygoot
B. Betekent dit ook dat er door persoon I en II een ander eiwit gemaakt wordt?
2. Waarom bewegen de DNA-fragmenten bij gel-elektroforese van de negatieve naar de
positieve pool?
A. Vanwege een positieve lading op de basen A en C
B. Vanwege de negatieve lading van de fosfaatgroepen
C. Alle ongeladen deeltjes bewegen van een negatieve pool af
3. Bij de gel zie je dat de bovenste bandjes feller oplichten dan de onderste. Wat betekent dit?
A. Er zijn veel meer grotere fragmenten
B. Er zijn veel meer kleinere fragmenten
C. Kleinere fragmenten lichten minder op
5
cDNA-synthese
—————————————————————————————————————————————
Uitgangsmateriaal: weefsel, cellen of totaal cellulair RNA
———————————————————————————————————————————
Eindproduct:
dubbelstrengs DNA, kopie van originele mRNA
———————————————————————————————————————————
Gebruik:
- zoeken naar specifieke mRNA's
- eukaryotisch DNA geschikt maken voor bacteriën (kloneren)
———————————————————————————————————————————
Principe
Deze techniek heeft twee stappen:
1. mRNA isoleren uit weefsel
Een stukje weefsel wordt gelyseerd (membranen opgelost). Met behulp
van een RNA-isolatie-kit kan hier het RNA uitgehaald worden. Het
resultaat is een mengsel van RNA's, waarvan het meeste rRNA en tRNA is.
Het meeste RNA is nu nog rRNA en tRNA. Het mRNA wordt gescheiden
van de rest door middel van affiniteits-chromatografie. Hierbij wordt de
oplossing door een kolom bolletjes geleid waaraan oligo-dT-staarten
zitten. Deze hechten aan de poly-A-staarten van het mRNA. Op deze
manier wordt het mRNA 'ingevangen'.
2. hiermee cDNA maken
Als primer wordt aan de poly-A-staart een korte T-nucleotide-sequentie
gehecht. Bij toevoegen van reverse transcriptase wordt de
complementaire DNA-streng hieraan gesynthetiseerd. Het
oorspronkelijke mRNA wordt gedegradeerd met RNase H, zodat er
enkelstrengs DNA ontstaat. DNA-polymerase kan hier weer
dubbelstrengs DNA van maken: het cDNA (c=complementair).
Toepassingen
Kloneren van eukaryotisch DNA
Eukaryotische genen hebben introns die bij het maken van werkzaam mRNA worden verwijderd.
Prokaryoten zoals bacteriën doen dat niet, dus zal dit DNA daar geen werkend mRNA opleveren.
Men gebruikt daarom het cDNA voor het eiwit dat de bacterie moet produceren.
cDNA-bibliotheek of -bank
cDNA kan worden ingebouwd in plasmiden en in bacteriën worden ingebracht (kloneren). De
verzameling klonen met al het verschillende cDNA van één weefseltype vormen de bibliotheek van
het weefsel. Het vertegenwoordigt niet het genoom, maar alleen de coderende delen van de
genen die in dit weefseltype mRNA produceren.
NB cDNA-synthese wordt vaak gevolgd door een PCR.
6
Vragen cDNA-synthese
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Het uitgangsmateriaal is mRNA uit een weefsel, het resultaat is DNA. Verschilt dit DNA van
het oorspronkelijke DNA waar het gen voor dit eiwit zat?
A. Ja, het is langer
B. Ja, even lang maar complementair
C. Ja, het is veel korter
D. Nee
2. Wat ontbreekt er nog aan dit DNA om er met RNA-polymerase weer mRNA van te kunnen
maken?
3. Van een weefsel wordt van alle mRNA's cDNA gemaakt. Op deze manier ontstaat een cDNA'bibliotheek'. Waarvoor is deze bibliotheek kenmerkend?
A. Individu
B. Weefsel
C. Eiwitten
7
Kloneren
—————————————————————————————————————————————
Uitgangsmateriaal: - fragmenten DNA waaronder één coderend voor een bepaald gen
- bacteriën met plasmiden met daarop:
· een gen voor resistentie tegen een bepaald antibioticum
· een gen waardoor je de bacteriën kunt kleuren
———————————————————————————————————————————
Eindproduct:
een aantal kolonies bacteriën waarvan enkele dit gen hebben
———————————————————————————————————————————
Gebruik:
- om de DNA-volgorde van een gen te bepalen
- om meer van een bepaald gen te krijgen
- om het daardoor gecodeerde eiwit te produceren
- gentherapie
- fundamenteel onderzoek
———————————————————————————————————————————
Principe
Bij kloneren worden bepaalde genen in een bacterie gebracht. Als de bacteriën zich
vermenigvuldigen, wordt ook het gen vermenigvuldigd.
Om het gen in een bacterie te brengen, is een 'vector' nodig: iets dat het gen bevat en
door de bacterie makkelijk wordt opgenomen. Plasmiden zijn zulke vectoren: kleine
ringetjes DNA die zich zelfstandig kunnen vermenigvuldigen in een bacterie.
Plasmiden komen voor bij bacteriën, maar ook bij sommige eukaryoten zoals gist.
Uitgebreider:
Kloneren heeft een aantal stappen:
1. DNA met restrictie-enzym bewerken
Het DNA met het gewenste gen wordt geïsoleerd en in stukken geknipt. Meestal
eindig je met een mengsel met verschillende fragmenten DNA, waarvan een
aantal het gewenste gen bevatten.
2. Gen in het plasmide inbouwen
Sommige plasmiden bouwen niets in, sommige bouwen een verkeerd stukje in.
3. Transformatie
De plasmiden worden opgenomen door de bacteriën. Dit noemt men
transformatie. Sommige bacteriën nemen niets op, en sommige bacteriën nemen
een verkeerd plasmide op.
4. Bacteriën met het juiste gen vinden
- recombinante bacteriën vinden
Om de bacteriën vinden die een plasmide hebben ingebouwd, gebruik
je een plasmide met een gen voor resistentie tegen een antibioticum.
Alle bacteriën die het plasmide hebben ingebouwd, zijn nu ook resistent.
Op een voedingsbodem met dit antibioticum raak je alle bacteriën
zonder nieuw plasmide kwijt.
- bacteriën met recombinante plasmiden vinden
Om de bacteriën te vinden met plasmiden met een nieuw stukje DNA
heeft het plasmide nog een eigenschap: een gen voor ß-galactosidase.
Dit zit precies op de plek waar het nieuwe gen ingebouwd gaat worden.
ß-galactosidase is een enzym waardoor je de bacteriën blauw kunt
kleuren. Bij plasmiden met een ingebouwd stukje DNA werkt dit gen dan
niet meer. Bacteriën die witte kolonies vormen hebben dus
recombinante plasmiden. Blauwe kolonies hebben een plasmide
opgenomen dat geen extra DNA had ingebouwd. Deze stap wordt ook
wel eens overgeslagen.
8
- bacteriën met het juiste gen vinden
Om te bepalen welke kolonies het juiste gen bevatten gebruik je blotting (zie aldaar). In
het kort:
- maak een afdruk van de kolonies op een papier,
- denatureer het DNA,
- vindt het gewenste DNA met een radioactieve of met fluorescerende probe,
- maak een afdruk van dit papier op een foto, en
- vergelijk de foto met de kolonies.
De kolonies die een afdruk geven bestaan uit bacteriën met het juiste gen.
1. DNA met restrictie-enzym bewerken
Je hebt een aantal cellen met DNA met het gewenste gen. Eerst maak je het DNA vrij uit de
cellen. Vervolgens knip je het DNA in stukken met een restrictie-enzym. Dit mag niet in het
gewenste DNA zelf knippen!
Een restrictie-enzym knipt het DNA op vaste plaatsen. Aan het einde heb je dus een aantal
stukken van verschillende lengte, waarvan één het gewenste gen bevat.
Alle stukken hebben een einde waarmee ze makkelijk binden aan DNA dat geknipt is met
hetzelfde restrictie-enzym.
2. Gen in het plasmide inbouwen
De plasmiden die gebruikt worden bevatten twee speciale genen:
- één voor resistentie tegen een antibioticum, en
- één voor een blauwe kleur van de kolonie.
Ze worden geknipt met hetzelfde restrictie-enzym als gebruikt is voor het DNA met het in te
bouwen gen. Dit moet 1× knippen, namelijk precies in het gen voor de blauwe kleur.
Nu voeg je de oplossing van DNA toe. Dit bevat meestal ook nog stukjes DNA zonder het
gewenste gen.
Sommige plasmiden bouwen het goede DNA in. Er zijn er ook die niets inbouwen (zelfsluiters), of
een verkeerd stukje DNA.
De stukken zijn nog niet definitief aan elkaar verbonden. Om dit te doen voeg je
ligase toe.
3. Transformatie
Plasmiden laten opnemen door bacterie gaat in drie stappen:
1. Voeg de plasmiden toe.
2. Maak de celwanden permeabel (doorlatend) voor de plasmiden, door hem een beetje te
beschadigen.
Dit kan op twee manieren:
- Door rond 0°C te incuberen met bijvoorbeeld CaCl2, en daarna heel kort naar 42°C te
verhitten (hitteschok). Zie ook http://www.dnalc.org/ddnalc/resources/transformation2.html
- Door korte schokken te geven van 100 – 200 Volt (elektroporatie).
3. Geef de bacteriën even de tijd om 'bij te komen'.
9
De beschadigde celwanden moeten herstellen. Na deze herstelperiode zijn de bacteriën
gereed om op te laten groeien en te testen.
Er zijn weinig bacteriën die een plasmide opnemen, ondanks alle voorbereidingen. Over het
algemeen zijn dit maar enkele procenten van het totaal.
4. Bacteriën met het juiste gen vinden
Je groeit de bacteriën op een agar-gel, waarin de bacteriën zich niet kunnen
verplaatsen. Hierin zit het antibioticum, zodat de bacteriën zonder nieuw
plasmide doodgaan. Eén enkele bacterie vormt een kolonie van vele miljoenen
bacteriën die je met het blote oog kunt zien. Bacteriën van één kolonie hebben
hetzelfde plasmide.
Sommige plasmiden waren zelfsluiters en hebben een intact gen voor ßgalactosidase. Je moet dus verder met de witte kolonies.
Van de witte kolonies weet je alleen dat ze een recombinant plasmide hebben
ingebouwd. Om erachter te komen welke bacteriën het juiste gen hebben maak
je gebruik van een andere techniek: Southern blot hybridisatie. Kijk daar voor
meer details.
agar-voedingsbodem met
kolonies van bacteriën
Toepassingen
Productie speciale stoffen (eiwitten)
Met behulp van bacteriën kun je nu grote hoeveelheden van de stof maken waarvoor het gen
codeert, bijvoorbeeld insuline.
Gentherapie
Door midden van kloneren beschikt men over grote hoeveelheden van één gen. Bij de eerste
toepassing werden witte bloedcellen met behulp van genetisch gemanipuleerde virussen voorzien
van het gen voor een belangrijke immuuncel-receptor en teruggeplaatst. Virussen brengen van
nature hun DNA-materiaal in gastcellen in.
Vragen kloneren
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Geef aan welke eigenschappen de bacteriën hebben na kloneren:
Ja, zonder nieuw
Plasmide ingebouwd:
Nee
DNA
Ja, met nieuw
DNA
Blauwe kolonies
Resistent tegen het
antibioticum
2. Alleen bacteriën met plasmiden zonder nieuw DNA kleuren blauw. Gewoonlijk kleurt ongeveer
de helft van de kolonies blauw. Als je de bacteriën zou kweken op een medium zonder
antibioticum, hoeveel kleuren er dan nog blauw?
A. Nog steeds ongeveer de helft
B. Veel minder dan de helft
C. Veel meer dan de helft
10
PCR (polymerase chain reaction)
———————————————————————————————————————————
Uitgangsmateriaal: - een enkel fragment ds-DNA met een specifieke sequentie
- een primer voor het begin van de sequentie
- een primer voor het begin van de complementaire sequentie
———————————————————————————————————————————
Eindproduct:
een groot aantal identieke DNA-fragmenten met de specifieke sequentie
———————————————————————————————————————————
Gebruik:
- vermeerderen van DNA bij weinig uitgangsmateriaal
- vermeerderen van kleine stukjes DNA uit een lang stuk DNA (chromosoom)
———————————————————————————————————————————
Principe
Bij deze techniek wordt een oplossing van DNA, losse nucleotiden, primers en DNA-polymerase
cyclisch verhit en afgekoeld. Na een aantal cycli is er een groot aantal fragmenten specifiek DNA
gevormd. Dit deel wordt bepaald door de primers, die binden aan het begin en (op de
complementaire string) het eind van deze sequentie.
Elke cyclus kent 3 stappen:
1. denaturatie bij 95° C - dubbelstrengs DNA splitsen in enkelstrengs DNA
Denaturatie vindt plaats bij temperaturen boven 80° C.
2. annealing bij 55° C - binden van de primers
De primers binden het snelst bij temperaturen onder de 65° C.
3. elongatie bij 70° C - polymerisatie van het enkelstrengs DNA
De bacteriën waaruit de polymerases komen leven in heetwaterbronnen van ongeveer 70°C.
Het polymerase werkt het beste bij die temperatuur.
Verhitten tot 94 - 96°C splitst het DNA.
Na afkoeling tot 50-65° C binden de primers optimaal aan het DNA.
Verhitten tot 70° C zorgt dat het polymerase optimaal actief is.
Na één cyclus zijn er twee moleculen dsDNA, waarvan één streng aan één kant na de gewenste
sequentie stopt.
Na twee cycli zijn er vier moleculen dsDNA, die echter allemaal nog extra stukken bevatten.
11
Na de derde cyclus zijn er twee moleculen dsDNA die precies de gewenste code bevatten. Elke
volgende cyclus groeit dit aantal snel, en na 10 cycli zijn dit er meer dan 1000. Het aantal
fragmenten met extra DNA neemt ook toe, met maar 2 per cyclus (na 10 cycli nog maar 20).
Uiteindelijk is het merendeel dus het gewenste DNA.
Tegenwoordig gebeurt dit in een apparaat dat automatisch de temperatuur verhoogt en verlaagt.
Eén cyclus duurt maar een paar minuten, en zo kan zeer makkelijk gedurende bijvoorbeeld een
nacht veel DNA gemaakt worden.
Bedenk wat dit voor eisen stelt aan de uitgangsoplossing. Bij het onderdeel 'vragen' wordt daar
verder op ingegaan. Daar zal ook worden gevraagd om uit te rekenen hoeveel gewenste
fragmenten er zijn na een bepaald aantal cycli.
Toepassingen
Materiaal vermeerderen
Bij sporenonderzoek kan de politie van een beetje speeksel of een enkele haar DNA winnen en dit
met PCR vermenigvuldigen. Er worden dan een aantal specifieke gebieden vermenigvuldigd;
meestal zijn 5 van deze gebieden voldoende om de dader te kunnen aanwijzen.
NB
Om specifieke genen te vermenigvuldigen blijft kloneren (zie aldaar) de beste methode. Bij PCR
maakt polymerase heel soms een foutje, maar als er veel cycli zijn kan dit een groot probleem
opleveren in het eindresultaat.
Links
Goede uitleg van de procedure: PCR (interne animatie)
Extra links:
Hoe snel je al een groot aantal kopieën maakt: http://www.dnalc.org/ddnalc/resources/pcr.html
Bij het boek 'The Cell - a molecular approach' van Cooper & Hausman:
http://www.sinauer.com/cooper/4e/animations0409.html
12
Vragen PCR
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Het polymerase dat bij PCR gebruikt wordt is afkomstig van organismen in heetwaterbronnen.
Waarom is hittebestendige polymerase handig?
A. Het moet polymeriseren bij 70° C
B. Het mag niet denatureren bij 95° C
2. In de animatie van PCR stond naast het te vermeerderen DNA alleen nog de primer en het
polymerase aangegeven. Wat heb je nog meer nodig?
3. Bereken hoeveel fragmenten van elke soort na elke cyclus aanwezig zijn.
fragment
cyclus
0
1
0
0
1
2
3
4
5
6
7
8
9
10
13
0
Blotting
———————————————————————————————————————————
Uitgangsmateriaal:
- een gel met eiwitten of DNA- of RNA-fragmenten, of een plaat met
bacterie-koloniën
- een probe voor de gezochte sequentie / het gezochte eiwit
———————————————————————————————————————————
Eindproduct:
een foto met de plaats waar de gezochte fragmenten of eiwitten zitten
———————————————————————————————————————————
Gebruik:
aantonen van specifieke DNA- of RNA-fragmenten, of eiwitten, in een
verzameling irrelevante fragmenten
———————————————————————————————————————————
Principe
Edward Southern aan de Edinburgh University ontwikkelde een methode voor detectie van DNAfragmenten in een gel met behulp van gelabelde complementaire sequenties. Dit noemde men
Southern blot hybridisatie. 'Hybridisatie' omdat de techniek is gebaseerd op het feit dat
complementaire sequenties met elkaar kunnen hybridiseren.
Later werd de techniek aangepast om ook te gebruiken met RNA, en dit noemde men dan
Northern blot hybridisatie. Op dezelfde manier ging men ook eiwitten aantonen met behulp van
antilichamen, en dit noemde men Western blot hybridisatie.
Hier zal alleen de Southern blot hybridisatie worden behandeld.
Uitgangsmateriaal is meestal DNA-fragmenten in een gel (van de elektroforese). Er wordt een
afdruk van de gel gemaakt op een nylon membraan met nitrocellulose. Het DNA hecht aan de
nitrocellulose en wordt daarna met een 'probe' gelabeld. Deze probe heeft meestal een radioactief
label en is specifiek voor een bepaalde DNA-sequentie.
Hierna kan een foto-afdruk worden gemaakt, waarop staat aangegeven waar de gezochte DNAsequentie zich bevond op het originele materiaal.
•
•
•
•
•
•
•
•
•
•
•
Je maakt een bak met alkalische (basische) oplossing en legt daarin een spons.
Op de spons leg je de gel.
Hierop leg je een nylon membraan met nitrocellulose.
Hierop leg je absorberend materiaal (een stapel papieren handdoekjes), en een gewicht
om het goed aan te drukken.
De basische oplossing wordt via de spons door de gel en het nylon membraan met
nitrocellulose naar het absorberend materiaal gezogen.
De basische oplossing denatureert het DNA (dit wordt
enkelstrengs) en neemt het mee. DNA kan niet door het
nylon membraan met nitrocellulose, maar hecht daaraan.
Na een tijdje heb je een afdruk van het DNA op de gel
gekregen op je nylon membraan.
De DNA-bandjes zijn nu niet zichtbaar. Als je ze kon zien
zaten ze nu hier. Je knipt een hoekje af van het nylon vel om
te kunnen onthouden wat boven en onder is. Je kunt
daarnaast nog ergens een potloodstreepje zetten om aan te
geven aan welke kant het DNA gehecht zit.
Daarna gebruik je een radioactieve probe voor de
gewenste DNA-sequentie. Je doet hierbij het nylon
membraan met DNA in een plastic bakje of zakje
samen met de probe.
De probe maak je bijvoorbeeld met een PCR voor het
gewenste stuk DNA, in aanwezigheid van radioactieve
dNTP's (bijvoorbeeld dCT³²P). Je krijgt zo kleine stukjes
radioactief DNA.
Voordat je de probe gebruikt verhit je deze, zodat deze
enkelstrengs wordt. Nu kan het met het DNA op het
14
•
•
nylon membraan hybridiseren.
Na een tijdje spoel je de overmaat weg.
Je legt een fotografisch gevoelige film over je membraan. Daar
waar de probe is gehecht zal er een donkere plek op de film
verschijnen. Dat zijn dan de bandjes met de gewenste
sequentie.
Toepassingen
Genen opsporen
Door te zoeken in het hele DNA van een individu (met restrictie-enzymen en gel-elektroforese)
kunnen op de uiteindelijke gel bepaalde genen worden aangetoond.
Juiste klonen vinden
Bij kloneren kun je de juiste kolonies vinden: de kolonies die bacteriën met het juiste gen hebben.
Hiervoor kun je het nitrocellulose-papier direct op de kolonies drukken: bacteriën uit de kolonies
blijven hieraan plakken.
Daarna los je de cellen op en denatureer je het DNA in een alkalische oplossing, waarna de
procedure hetzelfde is.
Links
Bij het boek 'The Cell: A Molecular Approach' van Cooper & Hausman:
http://www.sinauer.com/cooper/4e/animations0411.html
Extra links:
Eveneens, maar dan 'Kolonie hybridisatie': http://www.sinauer.com/cooper/4e/animations0412.html
Korte animatie: Southern blot (interne animatie)
Van de Universiteit in Hong Kong: http://ihome.cuhk.edu.hk/~z045513/virtuallab/animation/southern.html
Vragen Blotting
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Waarom incubeer je de probe niet direct met de gel?
A. Dat spoelt het DNA weg
B. Dat beschadigt het DNA
C. De probe hecht aan de gel
2. Analoog aan Southern blotting is er ook Western blotting, waarbij eiwitten worden gelabeld.
DNA-probes zullen hier niet werken. Waarvan kunnen dan de probes gemaak zijn?
15
Microarray
———————————————————————————————————————————
Uitgangsmateriaal:
- cellen van (ziek) weefsel waarvan je wilt weten welke genen actief zijn
- ter vergelijking: cellen van (gezond) weefsel waarvan je weet welke
genen actief zijn
- drager met aangehechte verschillende fragmenten DNA van genen
———————————————————————————————————————————
Eindproduct:
matrix waarin te zien is welke genen actief zijn in beide weefsels
———————————————————————————————————————————
Gebruik:
- opsporen van specifieke genetische afwijkingen (kanker)
- vaststellen van het stadium waarin het weefsel is
- vaststellen van het type weefsel
———————————————————————————————————————————
Principe
Om een microarray-assay te doen voer je de volgende 4 stappen uit:
1. cDNA-bank maken van beide weefsels, met fluorescerende labels
Van het te onderzoeken weefsel wordt mRNA geisoleerd. Daarna wordt er via cDNA-synthese
een cDNA-bank gemaakt. Hierbij wordt van elk mRNA uit de cel het complementaire DNA
gesynthetiseerd. Bij deze synthese worden fluorescerende nucleotiden gebruikt.
Als het gaat om het vergelijken van weefsels, wordt hetzelfde gedaan voor gezond weefsel,
waarbij een ander fluorescerend label wordt gebruikt.
2. DNA-hybridisatie op een chip
De techniek hiervoor is afgeleid van Southern Blotting. Het
fluorescerende DNA wordt gehybridiseerd met DNA op een
glasplaat, plastic drager of een siliconen chip.
Een stukje van minstens 16 nucleotiden is uniek voor elk genoom. Synthetisch wordt van elk
gen een stukje van ongeveer 20 nucleotiden gemaakt en op zijn plaats op de
glasplaat gebracht.
Dit gebeurt met een techniek die veel lijkt op die van een printer, en in sommige
gevallen wordt er zelfs gebruik gemaakt van een inktjet printer. Op de animatie zijn
180 fragmenten te zien, maar op een chip zijn dit er vaak 100× zoveel.
Vervolgens kunnen de geïsoleerde en gelabelde DNA-fragmenten worden
gehybridiseerd met het DNA op de chip. De fragmenten van het te onderzoeken weefsel
fluoresceerden rood. De fragmenten van het te vergelijken weefsel fluoresceerden groen.
Zonder fluorescentiemicroscoop is er nu nog niets te zien op de chip.
3. Tabel maken van activiteit van de genen
Nadat de gelabelde fragmenten DNA zijn gehybridiseerd, kan met behulp
van een fluorescentiemicroscoop het resultaat bekeken worden. Als er een
biochip is gebruikt, die veel te klein is om goed te bestuderen, wordt met
een speciale techniek het patroon omgezet in een tabel. Daar waar van
het zieke weefsel DNA is gehybridiseerd wordt het vakje meestal rood
aangegeven, daar waar van gezond weefsel DNA is gehybridiseerd groen,
en waar beide zijn gehybridiseerd wordt het vakje geel.
4. Trekken van conclusies
Nu kunnen er conclusies getrokken worden uit de arrays. Als het gaat om het weefsel te
karakteriseren, is er nu een standaardplaat met daarop precies aangegeven welke mRNA's er
in dit weefseltype worden gevormd.
Als het gaat om het vergelijken van gezond en ziek weefsel, kan men nu precies zien waarin
het zieke weefsel verschilt: welke mRNA's hier wel gevormd worden maar normaal niet en
andersom. Hieraan kan men ook het type van de ziekte vaststellen: sommige ziektebeelden
zijn gelijk maar kunnen door verschillende genetische afwijkingen worden veroorzaakt.
16
Hiernaast zie je een 'expression chip', waarbij ook de intensiteit
fluorescentie wordt meten, dus de mate van expressie van de genen.
Slots zijn dan behalve zwart, groen, geel of rood, ook oranje, groengeel,
en meer of minder helder. Hier is kankerweefsel uit de pancreas getest.
Toepassingen
Microarray expression analysis
Door weefsel te vergelijken voor en na een behandeling kan de
(veranderde) expressie van bepaalde genen worden gevolgd, om te bepalen of een
behandeling aanslaat.
Hierbij wordt ook de mate van expressie gemeten.
Microarray Comparative Genomic Hybridization
Vergelijken van ziek en gezond weefsel, waarbij ook de mate van expressie wordt gemeten.
Mutation microarray analysis
Dit gebeurt met een array van DNA-fragmenten van maar één gen, met in elk vakje een
andere versie (mutatie), die maar één of enkele nucleotiden verschilt.
Hierbij zoek je of een persoon een bepaalde mutatie heeft.
Links
Simpele maar effectieve animatie: http://www.bio.davidson.edu/courses/genomics/chip/chipQ.html
Extra links:
Mooie animatie: http://gslc.genetics.utah.edu/units/biotech/microarray
Nog meer info: http://www.ncbi.nlm.nih.gov/About/primer/microarrays.html
In het Nederlands: http://www.watisgenomics.nl/genomics/genomics/i000837.html
Vragen microarray
De vragen verschijnen in willekeurige volgorde, dus de nummering kan verschillen.
1. Stel dat een gen normaal gesproken niet tot expressie komt in een bepaald type weefsel, en
ook niet in tumorwefsel van hetzelfde weefseltype. Welke kleur heeft de plek van dit gen in de
microarray?
Rood / groen / geel / donker
2. In bepaald tumorweefsel maakt een gen door een puntmutatie een verkeerd mRNA aan,
waardoor er geen werkend eiwit kan worden gevormd. In normaal weefsel wordt wel een goed
eiwit gevormd. Welke kleur heeft de plek van dit gen in de vergelijkende microarray?
Rood / groen / geel / donker
3. In ziek weefsel, met slechts één defect gen, zie je vaak toch meerdere verschillen: er zijn
meedere rode plekken waar een normaal inactief gen nu actief is, en ook meerdere groene
plekken waar een anders actief gen nu inactief is. Hoe komt dit?
A. Tumoren ontstaan pas als er meerdere genen defect zijn
B. Eén enkel gen kan meerdere nadere genen reguleren
C. cDNA kan aan verschillende fragmenten op de microarray-plaat hechten
17
Sequencen (onder constructie)
———————————————————————————————————————————
Uitgangsmateriaal:
grote hoeveelheid identieke fragmenten DNA
———————————————————————————————————————————
Eindproduct:
nucleotidevolgorde (sequentie) van het fragment
———————————————————————————————————————————
Gebruik:
opsporen (punt)mutaties, en stap in bepalen hele genoom
———————————————————————————————————————————
Principe
Je kunt fragmenten met behulp van enzymen met de hand sequencen (fragmenten tot 300 bp).
Tegenwoordig kun je fragmenten tot 800 baseparen lang automatisch laten sequencen. Vanwege
de kans op fouten wordt deze bewerking vaak 8-10× op hetzelfde fragment uitgevoerd.
Dit gebeurt door PCR uit te voeren met deze fragmenten in aanwezigheid van een geringe
hoeveelheid gelabelde nucleotiden. Behalve dat deze gelabeld zijn, ontbreekt er een OH-groep
aan deze nucleotiden (zogenaamde dideoxy-nucleotiden), waardoor volgende nucleotiden niet
binden en de elongatie stopt. Zo ontstaan er een groot aantal incomplete fragmenten van
verschillende lengte, met elk één gelabeld nucleotide.
Door deze door een kolom te laten gaan waar ze afhankelijk van grootte uit komen, kan de
nucleotidevolgorde bepaald worden. Zie bij 'links' voor goede animaties.
Toepassingen
Sorry, nog niet klaar.
Links
Goede uitleg van de procedure:
http://www.wiley.com/college/pratt/0471393878/student/animations/dna_sequencing/index.html
Extra links:
http://www.sinauer.com/cooper/4e/animations0408.html
18
Sequencen genoom
———————————————————————————————————————————
Uitgangsmateriaal:
compleet DNA (genoom) van één individu
———————————————————————————————————————————
Eindproduct:
nucleotidevolgorde (sequentie) van heel genoom van dat individu
———————————————————————————————————————————
Gebruik:
genomics (genoomonderzoek)
———————————————————————————————————————————
Principe
Een genoom kan niet in één keer worden gesequenced: het bestaat uit 3 miljard baseparen. Het
sequencen van fragmenten geeft problemen bij het bepalen in welke volgorde de fragmenten in
het genoom zitten.
Meestal wordt daarom gebruik gemaakt van de shortgun-methode. Hierbij worden op 10 tot 12
verschillende manieren het genoom in 6 miljoen fragmenten van ongeveer 500 nucleotiden
verdeeld. Doordat er overlappende fragmenten zijn kan nu wel de volgorde worden bepaald.
Stappen:
1. DNA fragmenteren
Je begint met 10 tot 12 verschillende oplossingen van het DNA van
hetzelfde genoom. In de animatie stelt de kleur de sequentie voor. De
sequentie is echter nog niet bekend.
Aan elke oplossing voeg je restrictie-enzymen toe, die het DNA in stukken
van ongeveer 500 baseparen knippen. In elke oplossing een andere
combinatie, zodat er in elke oplossing verschillende stukken ontstaan.
2. fragmenten sequencen
Vervolgens bepaal je van elk fragment de sequentie.
3. de sequentie van het hele genoom bepalen
Door te kijken naar overlappende stukken kun je uiteindelijk de
sequentie van het genoom bepalen. Dit zijn echter zoveel fragmenten
dat dit heel lang zou duren als het met de hand gedaan moest worden.
Deze stap wordt door een computer uitgevoerd.
Omdat er in het genoom zichzelf herhalende sequenties zijn, is het moeiljk van het genoom van
een soort (zoals de mens) voor 100% de sequentie te bepalen. Van het euchromatine, dat weinig
repeterende delen bevat, is 99% bekend; van het totaal nu 93%.
Toepassingen
Onderzoek
Als het hele genoom bekend is kan beter onderzoek gedaan worden naar:
1. De genregulatie
Door te zoeken naar de plaatsen die voor eiwitten coderen, kan men wellicht nieuwe eiwitten
ontdekken, of ontdekken hoe bepaalde eiwitten worden gereguleerd
2. Genetische verwantschappen
Door genomen te vergelijken kan men genetische verwantschappen (en verschillen) tussen
soorten vinden, en waar die precies in zitten. Zo kan men ook zien welke genen wellicht uit
andere kunnen zijn ontstaan, en in welke aspecten nauw verwante soorten van elkaar
verschillen.
Vragen
1. Stel dat het genoom van de mens voor 100% bekend wordt. Is jouw genoom dan ook voor de
volle 100% bekend?
19
Cases
Er is vooralsnog nog maar één casus beschikbaar.
Casus Borstkanker
Borstkanker
Borstkanker
Erfelijkheidsfactoren
Borstkanker ontstaat meestal bij toeval.
Echter in 5-10% van de gevallen is er
sprake van erfelijke aanleg.
Het betreft dan mutaties voor bepaalde
eiwitproducerende genen.
De bekendste zijn BRCA1, BRCA2 en
TP53.
Deze aanleg erft dominant over, dat wil
zeggen dat als je als vrouw het gen hebt,
je ook een verhoogde kans hebt, en je
dochters ook 50% kans hebben om de
aanleg te hebben.
Dragers ontwikkelen niet altijd
borstkanker: de kans hierop is dan
verhoogd.
Ook ontwikkelt de kanker zich sneller als
je ze eenmaal hebt.
BRCA1 en BRCA2 zijn eiwitten die helpen
bij het herstel van DNA-schade.
TP53 codeert voor het eiwit p53, dat de
celcyclus reguleert.
Het remt een eiwitcomplex, dat ervoor
zorgt dat de cel de S-fase in kan gaan.
Omschrijving casus
Casus
Kansen
Bij een vrouw wordt borstkanker
vastgesteld.
Haar oma (moeder van haar vader) is al
een tijd geleden overleden aan
borstkanker.
Haar tante, zus van haar vader, is ook
kortgeleden voor borstkanker behandeld.
Als de oma de erfelijke vorm had, is er
50% kans dat ze dit doorgeeft.
Als haar tante een dochter was van haar
oma, had deze dus 50% kans.
Ze maakt zich zorgen dat het misschien in
de familie zit, en dat haar dochters ook de
aanleg kunnen hebben.
Ze vraagt daarom om een genetisch
onderzoek, dat moet uitwijzen of ze een
erfelijke vorm heeft.
Hoewel mannen minder snel borstkanker ontwikkelen, kunnen zij wel drager
zijn en het gen doorgeven aan hun
kinderen, weer met 50% kans.
Als haar oma de erfelijke vorm had is de
kans 50% dat haar vader drager was, en
50% kans dat deze het doorgeeft aan
haar.
Zij heeft dan 25% kans op de aanleg.
20
Stappenplan
1. Je hebt een patiënt, van wie je wilt vaststellen of ze een erfelijke vorm van borstkanker heeft. Je neemt
een monster van het weefsel (biopt). Van welk weefsel neem je dit biopt?
B. van het kankergezwel
C. van gezond weefsel
2. Je hebt een patiënt, van wie je wilt vaststellen of ze een erfelijke vorm van borstkanker heeft. Je neemt
een monster van het weefsel (biopt).
Bekend is:
- De genen met vaak voorkomende mutaties (BRCA1, BRCA2 en TP53). Je weet van deze genen de
sequenties van alle exonen.
- De meestvoorkomende mutaties. Dit zijn vaak puntputaties, en je weet van elke mutatie precies welke
nucleotide in welk exon is veranderd.
Welke stappen ga je achtereenvolgens uitvoeren?
3. Je hebt een patiënt, van wie je wilt vaststellen of ze een erfelijke vorm van borstkanker heeft. Je neemt
een monster van het weefsel (biopt). Je weet de genen waar vaak mutaties in voorkomen, en ook de
exonen waarin die mutaties voorkomen. Een voorbeeld is een puntmutatie T G in een exon van gen
BRCA2:
Welk restrictie-enzym zal deze puntmutatie kunnen aantonen?
4. Je weet:
- de genen waar vaak mutaties in voorkomen,
- de exonen waarin die mutaties voorkomen,
- welke (punt)mutaties dit precies zijn.
Je zoekt bij elke mutatie een restrictie-enzym waarvan de knip-site is veranderd. Je isoleert DNA, voert
een PCR uit voor de betreffende exonen en bewerkt met de restrictie-enzymen. Doe je dit voor elke
mutatie apart?
21
Je zoekt bij elke mutatie een restrictie-enzym waarvan de knip-site is veranderd. Je isoleert DNA, voert een
PCR uit voor de betreffende exonen en bewerkt het met de restrictie-enzymen.
5. Wat voor einden moeten de restrictie-enzymen opleveren?
A. ‘sticky’ ends
B. ‘blunt’ ends
C. maakt niet uit
6. Zullen er na gel-elektroforese meer bandjes te zien zijn of juist minder?
A. meer
B. minder
C. kan allebei
7. Vervolgens voer je een gel-elektroforese uit. Stel dat een normaal patroon er uitziet zoals de meest
rechtse hieronder. Hoe kan een patroon van een gemuteerd gen er dan uitzien?
8. Vervolgens voer je voor elke mutatie een gel-elektroforese uit. Je vindt op deze manier geen enkele
afwijking van het normale patroon. Was het dan toeval dat zoveel vrouwen uit deze familie borstkanker
kregen?
A. Ja, zeker
B. Waarschijnlijk wel
C. Waarschijnlijk niet
22











