Samenvatting Biochemie 28 juni 2013
Hoofdstuk 1 Algemene inleiding Biochemie
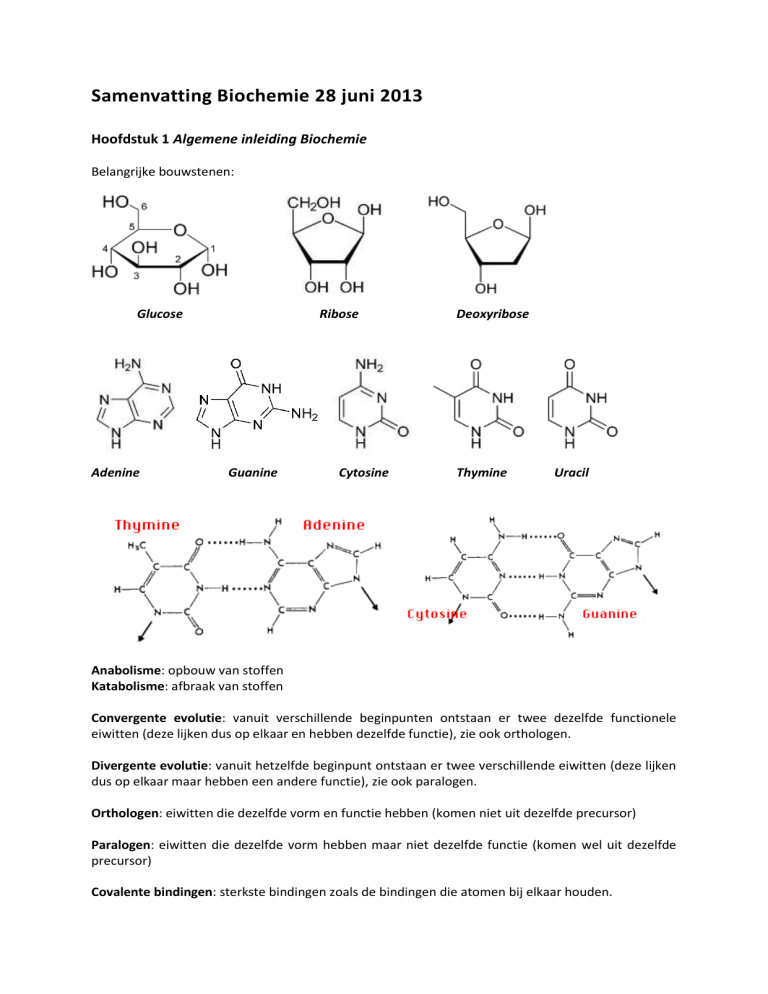
Belangrijke bouwstenen:
Glucose
Adenine
Ribose
Guanine
Cytosine
Deoxyribose
Thymine
Uracil
Anabolisme: opbouw van stoffen
Katabolisme: afbraak van stoffen
Convergente evolutie: vanuit verschillende beginpunten ontstaan er twee dezelfde functionele
eiwitten (deze lijken dus op elkaar en hebben dezelfde functie), zie ook orthologen.
Divergente evolutie: vanuit hetzelfde beginpunt ontstaan er twee verschillende eiwitten (deze lijken
dus op elkaar maar hebben een andere functie), zie ook paralogen.
Orthologen: eiwitten die dezelfde vorm en functie hebben (komen niet uit dezelfde precursor)
Paralogen: eiwitten die dezelfde vorm hebben maar niet dezelfde functie (komen wel uit dezelfde
precursor)
Covalente bindingen: sterkste bindingen zoals de bindingen die atomen bij elkaar houden.
Niet-covalente bindingen: zwakkere bindingen maar cruciaal voor biochemische processen.
Elektrostatische interacties
Waterstofbruggen; in principe ook (zwakke) elektrostatische interacties.
Van der Waals interacties
Hydrofobe interacties
Wetten van de thermodynamica:
De totale energie van het systeem en zijn omgeving is constant.
De totale entropie van een systeem en zijn omgeving neemt altijd toe.
Entropie (S): de mate van willekeurigheid/disorder in een systeem.
Enthalpie (H): hitte die loopt van het systeem naar zijn omgeving.
Δ𝑆𝑜𝑚𝑔𝑒𝑣𝑖𝑛𝑔 = −
Δ𝐻𝑠𝑦𝑠𝑡𝑒𝑒𝑚
𝑇
Δ𝐺 = −𝑇Δ𝑆𝑡𝑜𝑡𝑎𝑎𝑙 = Δ𝐻𝑠𝑦𝑠𝑡𝑒𝑒𝑚 − 𝑇Δ𝑆𝑠𝑦𝑠𝑡𝑒𝑒𝑚
De vrije energie verandering (Gibbs free energy, Δ𝐺) moet negatief zijn wil het proces
spontaan verlopen.
Er is een negatieve vrije energie verandering alleen wanneer de totale entropie van het
universum toeneemt.
Hoofdstuk 2 Structuur van aminozuren en eiwitten
4 niveaus om de structuur van eiwitten te bekijken:
Primaire structuur: aminozuurvolgorde
Secundaire structuur: interacties tussen naburige aminozuren (α-helix en (anti-)parallelle βsheet).
Tertiaire structuur: interacties tussen verder gelegen aminozuren (disulfidebindingen).
Quaternaire structuur: interacties tussen meerdere subunits.
Ureum en een klein beetje β-mercaptoethanol zorgen voor denaturatie en daarna herstel van
eiwitten.
Glutamaat is één van de aminozuren die zorgt voor de vorming van α-helices.
Valine is één van de aminozuren die zorgt voor de vorming van β-sheets.
Glycine past bij elke structuur omdat deze geen hinderde zijgroep heeft.
Hoofdstuk 3 Eiwitten experimentele technieken
De purificatie van eiwitten is een essentiële eerste stap in het begrijpen van hun functie.
De eerste stap is het controleren van de aanwezigheid van het eiwit in het mengsel.
Dit wordt gedaan door bijvoorbeeld in het geval van enzymen de enzymactiviteit te
bepalen.
Vervolgens wordt de cel gefractioneerd en gecentrifugeerd waardoor de zwaardere
onderdelen onderop komen te liggen.
Het supernatant wordt opnieuw gecentrifugeerd met een grotere kracht tot nieuw
supernatant en pallet (differentiële centrifugatie).
Specifieke activiteit: de ratio van de enzymactiviteit en de hoeveelheid eiwit in het mengsel.
Het purificeren van eiwitten kan op basis van verschillende eigenschappen worden gedaan:
Oplosbaarheid
Uitzouten (salting out): door het verhogen van de zoutconcentratie kan het eiwit niet
meer opgelost blijven waardoor deze neer zal slaan.
Grootte
Dialyse: door middel van een semipermeabel membraan kunnen kleine moleculen
wal vanuit het mengsel naar een bufferoplossing en grotere moleculen niet (wordt
gebruikt om het zout bij uitzouten te verwijderen).
Onderscheid tussen twee verschillende eiwitten wordt echter niet gemaakt.
Gelfiltratie chromatografie: door middel van een poreuze gel kunnen moleculen van
boven naar onderen zakken.
Grote moleculen gaan sneller omdat een kleiner volume maar voor ze
beschikbaar is, kleine moleculen blijven in de openingen zitten en komen
langzamer naar beneden.
Lading
Ion-uitwisseling chromatografie: eiwitten met een netto positieve lading gaan
langzamer door een kolom met carboxylgroepen dan negatief geladen eiwitten.
Kation-uitwisseling: positief geladen eiwitten binden aan anionisch
vulmateriaal.
Bindingsaffiniteit
Affiniteitschromatografie: bepaalde eiwitten hebben een hoge affiniteit voor een
bepaalde stof waarmee de kolom kan worden gevuld. De overige stoffen zullen
hierdoor sneller naar beneden gaan dan het eiwit.
Affiniteit kan ook worden verkregen door een affiniteitstag toe te voegen
(bijvoorbeeld His-tag).
High-pressure liquid chromatografie: door een fijnverdeeld kolommateriaal is er druk
nodig om de te onderzoeken stof door de kolom te laten gaan.
Er is echter wel meer interactie tussen de stoffen.
Het bekijken van de effectiviteit van het purificeren kan door het meten van de specifieke activiteit
(deze zou moeten toenemen) en het bepalen van het aantal verschillende eiwitten:
Dit laatste kan worden gedaan door middel van gelelektroforese – SDS-polyacrylamide gel
elektroforese (SDS-PAGE).
Het elektrische veld is zo gericht dat de eiwitten van de negatieve naar de positieve
pool migreren (van boven naar beneden)
De eiwitten worden voorzien van een lading door middel van SDS (deze lading is niet
van belang voor de snelheid waarmee het eiwit van boven naar beneden gaat).
Eiwitten worden gedenatureerd en disulfidebindingen worden verbroken.
Er wordt gescheiden op basis van massa (boven blijven de grootste eiwitten hangen.
De massa en de relatieve mobiliteit hebben een lineair verband.
Het verschil tussen 10 aminozuren kan met SDS-PAGE worden aangegeven.
Eiwitten kunnen ook op basis van hun iso-elektrisch punt (pI) worden gescheiden.
Op dit punt is hun relatieve mobiliteit gelijk aan 0 omdat de netto lading gelijk is aan
0.
Er wordt een gel gemaakt met aan de ene kan een lage pH en aan de andere kan een
hoge pH. De waarde van de pH neemt dus langzaam toe.
De bovenstaande technieken kunnen ook tegelijk worden uitgevoerd als tweedimensionale
elektroforese:
Een sample wordt eerst gescheiden door een horizontale iso-elektrische focusing en
vervolgens door SDS-PAGE.
Verticaal is er geselecteerd op basis van massa en horizontaal op basis van het isoelektrische punt.
Ultracentrifugatie: hiermee kunnen biomoleculen worden gescheiden en hun massa worden
bepaald.
Edman degradatie: met deze techniek kan de eiwitsequentie worden bepaald:
Hiervoor moet eerst de aminozuren die aanwezig zijn worden bepaald door middel van
ionuitwisselingschromatografie.
Daarna wordt telkens vanaf de N-terminus een aminozuur eraf gehaald en geanalyseerd.
Deze vrijgekomen deeltjes kunnen worden geanalyseerd met high-pressure liquid
chromatografie.
Slechts eiwitten van 50 aminozuren lang kunnen worden geanalyseerd. Het kan ook met
grotere eiwitten maar dan moet dit eiwit worden gesplitst.
Chemische klievingen:
Cyanogeenbromide: carboxylzijde van methionine residuen
Enzymatische klievingen:
Trypsine: carboxylzijde van lysine en arginine residuen
Chymotrypsine: carboxylzijde van tyrosine, tryptofaan, phenylalanine, leucine en
methionine.
Trombine: carboxylzijde van arginine en aminozijde van glycine (dus tussen arginine en
glycine).
Carboxypeptidase A: aminozijde van C-terminale aminozuur (niet arginine, lysine of proline).
Antilichamen (immunoglobuline, Ig) voor specifieke eiwitten kunnen worden gemaakt.
Epitopen: bepaalde groepen op een target molecuul waarom een antilichaam zich kan hechten.
Eiwitten kunnen worden gedetecteerd en gekwantificeerd met het gebruik van enzyme-linked
immunosorbent assay (ELISA):
Hierbij wordt gebruik gemaakt van een enzym dan covalent gebonden is aan een specifiek
antilichaam dat een bepaald target antigeen herkent.
Wanneer het antigen aanwezig is wordt het antilichaam-enzym complex hieraan
geboden met additie van een substraat waardoor er een gekleurd product ontstaat.
Er kan gebruik worden gemaakt van indirecte (voor het aantonen van de aanwezigheid van
een antilichaam) en sandwich ELISA (voor het aantonen van de aanwezigheid van een
antigen).
Western blotting: hiermee kunnen eiwitten worden gedetecteerd na gelelektroforese:
Een polymere sheet wordt gedrukt tegen de gel (van SDS-PAGE) waardoor er eiwitten
worden overgebracht op de sheet.
Een antilichaam dat specifiek is voor het eiwit waarin men geïnteresseerd is wordt
aan de sheet toegevoegd.
Een tweede antilichaam (gelabeld met een radioactief element/fluorescerend label)
wordt toegevoegd.
Massaspectrometrie: hiermee kan de massa (m/z verhouding, mass-to-charge ratio) van een eiwit
nauwkeurig worden bepaald.
Het bestaat uit 3 onderdelen: ionbron (hierin wordt het sample in geïoniseerde toestand
gebracht), mass analyser en een detector.
MALDI: matrix-assisted laser desorption/ionization
ESI: electrospray ionization
MALDI-TOF: time-of-flight
Met massaspectrometrie kan ook de eiwitsequentie worden bepaald.
De driedimensionale structuur van eiwitten kan worden bepaald met röntgentechnieken en NMR.
Hoofdstuk 8 Enzymen concepten en kinetiek
Enzymen werken door overgangstoestanden te stabiliseren/faciliteren. Bovendien zorgen ze er
slechts voor dat het evenwicht sneller wordt bereikt).
De activeringsenergie wordt verlaagd.
Cofactor: de katalytische activiteit van een enzym hangt hiervan vaak af, heet co-enzym wanneer het
een klein organisch molecuul betreft.
Halo-enzym: enzym met cofactor.
Apo-enzym: enzym zonder cofactor.
′
′
𝐾𝑒𝑞
=𝑒
−
Δ𝐺 °
𝑅𝑇
=
[𝐶][𝐷]
[𝐴][𝐵]
Actieve site: het gebied dat het substraat bindt (en cofactor wanneer deze nodig is).
Dit gedeelte bevat de residuen (katalytische groepen) die nodig zijn voor het verbreken of
maken van bindingen.
Het binden van het substraat door een groot aantal zwakke bindingen zorgt ervoor dat vrije
energie wordt afgegeven (bindingsenergie); hierdoor neemt de activeringsenergie af
Enzymen hebben de grootste affiniteit voor de stof wanneer deze zich in de
overgangsfase bevindt, maar deze wordt snel omgezet.
Het product heeft een lagere affiniteit waardoor deze zal worden losgelaten.
𝐸+𝑆
k1
k1
k2
𝐸 + 𝑃 met 𝐾𝑀 =
𝐸𝑆
𝑘−1 +𝑘2
𝑘1
=
[𝐸][𝑆]
[𝐸𝑆]
[𝑆]
Michaelis-Menten vergelijking: 𝑉0 = 𝑉𝑚𝑎𝑥 [𝑆]+𝐾
𝑀
Dus 𝐾𝑀 is gelijk aan de substraatconcentratie waarbij de reaction rate gelijk is aan de helft
van zijn maximale waarde (dit is dus een mate voor de affiniteit van het enzym voor het
substraat: kleine 𝐾𝑀 staat voor een grote affiniteit, maar alleen wanneer de vorming van het
ES-complex veel sneller is dan de vorming van het product P).
De 𝐾𝑀 en 𝑉𝑚𝑎𝑥 kunnen worden bepaald door het
gebruik van de volgende vergelijking:
1
𝐾
1
1
Lineweaver-Burk plot: 𝑉 = 𝑉 𝑀 ∙ 𝑆 + 𝑉
0
𝑚𝑎𝑥
𝑚𝑎𝑥
De fractie van het aantal gevulde actieve sites kan worden weergegeven met de volgende
vergelijking:
𝑓𝐸𝑆 =
𝑉
𝑉𝑚𝑎𝑥
[𝑆]
= [𝑆]+𝐾
𝑀
Enzymen kunnen geïnhibeerd worden door specifieke moleculen:
Competitieve inhibitor: vermindert de mate van katalyse door het reduceren van de
proportie enzymmoleculen gebonden aan het substraat.
Geen verandering van de 𝑉𝑚𝑎𝑥 maar de 𝐾𝑀 wordt groter (kleinere affiniteit).
Niet-competitieve (noncompetitive) inhibitor: inhibitor en substraat kunnen tegelijk binden
aan een enzymmolecuul op verschillende sites. Het vermindert dus de concentratie van het
functionele enzym dat gebonden is aan het substraat.
Vermindering van de 𝑉𝑚𝑎𝑥 maar de 𝐾𝑀 blijft gelijk.
𝑉
𝑎𝑝𝑝
𝑉𝑚𝑎𝑥 = 𝑚𝑎𝑥
[𝐼]
1+
𝐾𝑖
Hoofdstuk 9 Enzymen katalytische mechanismen
Enzymen volgen over het algemeen de volgende strategieën bij het katalyseren van specifieke
reacties:
Covalente katalyse: de actieve site bevat een reactieve groep (vaak een sterk nucleofiel)
Algemene Zuur-Base katalyse
Katalyse bij benadering
Metaalion katalyse
Proteasen klieven eiwitten door hydrolysereacties (additie van water aan een peptidebinding).
De actieve site van chymotrypsine bevat een reactieve serine (195) die een centrale rol speelt in de
hydrolyse van peptidebindingen.
De katalytische triade van chemotrypsine bestaat uit aspartaat (102), histidine (57) en serine
(195).
Dit wordt bewerkstelligd door een oxyanion holte waarin het alkoxide-ion wordt
gestabiliseerd.
Naburige aminozuren van serine 195 zorgen hiervoor (bijvoorbeeld glycine 193).
De specificiteitspocket (S1-pocket) van chymotrypsine bevat voornamelijk hydrofobe
aminozuren omdat deze graag in de buurt van grote hydrofobe zijstaarten zitten (deze klieft
chymotrypsine dan ook).
Sommige enzymen zijn specifieker en hebben hierdoor meerdere pockets die ook aminozuren over
een groter gebied herkennen.
Trypsine klieft na lange positief geladen zijgroepen (arginine en lysine) waardoor de S1-pocket
bestaat uit aspartaat 189.
De zijketens bepalen dus welke peptidebindingen worden doorbroken.
De hydrolyse werkt vervolgens weer door de katalytische triade.
De katalytische triade werkt ook met threonine of een primaire aminogroep zoals lysine of de Nterminus in plaats van het histidine en aspartaat.
Er zijn ook andere klassen proteasen die niet werken met een reactieve serine.
Cysteïne proteasen, aspartyl proteasen, metalloproteasen.
De volgende eigenschappen hebben alle 3 de bovenstaande klassen proteasen:
1. Ze activeren water of een ander nucleofiel
2. Ze polariseren de peptide carbonylgroep
3. Ze stabiliseren het tetrahedrale intermediair
Veel enzymen bevatten metaalionen door de
stabiliserende werking
Carbonic anhydrase bevat een zinkion waarmee
water wordt gecoördineerd. Daarnaast ook nog 3
histidine residuen.
1. Het zinkion faciliteert het afgeven van
een proton van het watermolecuul (tot
vorming van een hydroxide-ion).
2. Het CO2-substraat bindt aan de actieve
site en wordt zo gepositioneerd dat het
kan reageren met het hydroxide-ion.
3. Het hydroxide-ion valt het CO2-molecuul
aan zodat HCO3 ontstaat.
4. De katalytische site wordt hersteld door het afgeven van HCO3 en het binden van
een nieuw watermolecuul.
Restrictie endonucleases (restrictie enzymen) knippen stukken DNA:
Deze enzymen herkennen bepaal de basensequenties
Er wordt gekliefd door in-line displacement van 3’-zuurstof van het fosforatoom door
magnesium-geactiveerd water.
Vaak wordt host-DNA beschermd door methylgroepen te plaatsten op de basen.
Hoofdstuk 4 DNA, RNA, and the Flow of Genetic Information
DNA-synthese heeft de volgende karakteristieken:
De reactie heeft alle 4 de geactiveerde precursors nodig (dATP, dGTP, dCTP en TTP) en een
Mg2+-ion.
De nieuwe DNA-keten wordt direct geassembleerd op de DNA-template.
DNA-polymerase heeft een primer nodig om te beginnen met synthetiseren.
Veel DNA-polymerasen zijn in staat op fouten te herstellen door het verwijderen van
verkeerde nucleotiden.
RNA-polymerase heeft de volgende componenten nodig:
Een template (geprefereerd wordt dubbelstrengs DNA)
Geactiveerde precursors: alle 4 de ribonucleoside trifosfaten (ATP, GTP, CTP en UTP)
Een divalente metaalion: dit is of Mg2+ of Mn2+
RNA-polymerase heeft geen primer nodig in tegenstelling tot DNA-polymerase.
Promoter site: bepaalde plekken op het DNA waarom het RNA-polymerase bindt en vanuit daar kan
bepalen waar de transcriptie begint. In eukaryoten is dit vaak de CAAT en TATA box.
Na de transcriptie wordt er bij eukaryoten een cap (5’-einde) en een poly(A)-staart (3’-einde) aan het
mRNA toegevoegd.
Transfer RNA (tRNA) bindt aan het aminozuur (met een esterbinding) met adenosine monofosfaat.
Hieraan wordt dat het tRNA gekoppeld.
Het tRNA heeft een herkennings-site waar het codon wordt herkend.
De beginsite bij eukaryotisch mRNA voor de translatie is het eerste AUG triplet vanaf de 5’-zijde (bij
de cap).
Het blijkt dat eukaryotisch mRNA discontinu is, namelijk dat het bestaat uit intronen en exonen. De
intronen worden tijdens de bewerking van mRNA verwijderd. De exonen zorgen uiteindelijk voor de
translatie naar het eiwit.
Intronen beginnen bijna altijd met GU en eindigen met AG die vooraf wordt gegaan door een
pyrimidinerijk stuk mRNA (dus C of U).
Hoofdstuk 28 DNA Replication, Repair, and Recombination
DNA-polymerase heeft een template en een RNA-primer nodig.
De exonuclease zorgt voor de controle en correctie van het gesynthetiseerde DNA en is dus
een onderdeel van het DNA-polymerase.
DNA-polymerase heeft 2 metaalionen waarvan er één de primer en dNTP coördineert en één
alleen de dNTP.
De OH-groep van de primer wordt geactiveerd en dNTP kan eraan worden
gebonden. Dit is het begin van de DNA-replicatie.
De RNA-primer wordt geproduceerd door primase (soort RNA-polymerase).
Doordat DNA dubbelstrengs is, ontstaan er een leading strand en lagging strand (deze wordt
gevormd door Okazaki fragmenten).
Dit komt doordat de DNA-synthese altijd van 5’ naar 3’-richting plaatsvindt.
Verschillende enzymen die een rol spelen bij de DNA-replicatie:
DNA-polymerase III (prokaryoten) en δ (eukaryoten): zorgt voor de replicatie.
Door exonuclease kan proofreading worden gedaan aan het 3’-uiteinde van het
gesynthetiseerde DNA.
DNA-polymerase II (prokaryoten) en β (eukaryoten): zorgt voor de DNA-reparatie.
DNA-polymerase I (prokaryoten) en α (eukaryoten): zorgt ervoor dat de RNA-primers worden
verwijderd en deze worden opgevuld met de juiste nucleotiden.
DNA-ligase: verbindt de uiteinden van DNA van Okazaki fragmenten.
De 3’-hydroxylgroep van de deoxyribose wordt verbonden aan de fosfaatgroep met
ATP als energiebron.
Helicase: het separeren van de DNA-strengen met ATP als energiebron.
Helicase bestaat uit 6 identieke subunits die door ATP en ADP een
conformatieverandering ondergaan (zie ook ATP-synthase).
Topoisomerase: zorgen ervoor dat de supercoil in het DNA-molecuul worden weggehaald
zodat deze kan worden gerepliceerd.
Type I topoisomerase: katalyseert de relaxatie van het supercoiled DNA (geen ATP
nodig).
Type II topoisomerase: katalyseert de additie van negatieve supercoils aan het DNA
(ATP nodig).
Dit wordt gedaan door het DNA te klieven (bij type I door tyrosine), passeren en
resealen.
Telomerase: draagt de informatie die nodig is om de telomeersequentie te synthetiseren en
doet dit ook.
Supercoiling:
Lk (linking number): het aantal keer dat een DNA-streng draait in een rechtshandige draai.
Tw (twist): maat van helische winding van de DNA-streng rond elkaar.
Wr (writhe): coiling van de as van de dubbele helix (supercoiling).
Lk = Tw + Wr
Natuurlijk zijn de meeste DNA-moleculen negatief (rechtshandig) supercoiled.
Supercoiling ontstaat door het teveel of te weinig draaien van het DNA-molecuul.
DNA-polymerase werkt met het zogeheten trombone model zodat ook de lagging strand dicht tegen
het enzym zit zodat er niet telkens een grote afstand hoeft te worden afgelegd om een nieuw stuk te
synthetiseren.
Hoofdstuk 29 RNA Synthesis and Processing
Spliceosomen: eiwitten die mRNA precursors splitsen zodat enkel de exonen overblijven.
Het bestaat uit twee transesterficatie reacties.
RNA-synthese kan beginnen zonder primer. Deze begint namelijk met of pppG of pppA.
RNA-polymerase backtracken waardoor fouten kunnen worden gecorrigeerd.
-subunits in RNA-polymerase herkennen de promoter-sites in het DNA.
Het enzym is langzamer dan DNA-polymerase.
Het stopsignaal bestaat uit een GC-rijk gedeelte gevolgd door een AT-rijk gebied. Door een
palindromische GC deel kan er een haarspeld worden gevormd. Er wordt vervolgens nog een korte
staart van uracil basen toegevoegd waardoor het RNA-DNA complex uitelkaar valt.
Hoofdstuk 30 Protein Synthesis
Het verbinden van een aminozuur aan tRNA wordt gekatalyseerd door aminoacyl-tRNA synthetase
onder invloed van ATP.
Het aminozuur wordt verbonden aan de CCA-terminus.
Er wordt aan proofreading gedaan om fout gebonden aminozuren te vermijden.
Het eerste aminozuur (methionine) wordt gebracht door het eiwit initiation factors (IF1, IF2 en IF3)
en daarna door elongation factor Tu (EG-Tu)
Het tRNA-aminozuur complex wordt opgeschoven van de A-site naar de P-site door de
conformatieverandering onder invloed van GTP/GDP van EF-G (translocase).
Release factor (RF) koppelt het eiwit af van het ribosoom nadat er een stopcodon is aangetroffen.












