Faculteit Bio-ingenieurswetenschappen
Adacemiejaar 2013-2014
Optimalisatie en validatie van een TaqMan Low Density
Array Card voor de moleculaire detectie van seksueel
overdraagbare aandoeningen
Jessie Calleeuw
Promotor: Prof. Dr. Sc. Van Landschoot Anita
Tutor: Dr. Reynders Marijke en Dr. Sc. Descheemaeker Patrick
Masterproef voorgedragen tot het behalen van de graad van
Master of Science in de industriële wetenschappen: Biochemie
Faculteit Bio-ingenieurswetenschappen
Adacemiejaar 2013-2014
Optimalisatie en validatie van een TaqMan Low Density
Array Card voor de moleculaire detectie van seksueel
overdraagbare aandoeningen
Jessie Calleeuw
Promotor: Prof. Dr. Sc. Van Landschoot Anita
Tutor: Dr. Reynders Marijke en Dr. Sc. Descheemaeker Patrick
Masterproef voorgedragen tot het behalen van de graad van
Master of Science in de industriële wetenschappen: Biochemie
“De auteur en de promotor geven de toelating deze scriptie voor consultatie beschikbaar te
stellen en delen van de scriptie te kopiëren voor persoonlijk gebruik. Elk ander gebruik valt
onder de beperkingen van het auteursrecht, in het bijzonder met betrekking tot de verplichting
de bron uitdrukkelijk te vermelden bij het aanhalen van resultaten uit deze scriptie.”
“The author and the promoter give the permission to use this thesis for consultation and to
copy parts of it for personal use. Every other use is subject to the copyright laws, more
specifically the source must be extensively specified when using the results from this thesis”.
6 juni 2014
Auteur Calleeuw Jessie
Promotor Prof. Dr. Sc Van Landschoot Anita
Woord vooraf
Deze scriptie is dankzij de steun en hulp van vele mensen tot stand gekomen.
Graag wil ik mijn promotoren, Dr. Marijke Reynders en Dr. Sc. Patrick Descheemaeker,
bedanken voor de zeer goede begeleiding en hun vrijgemaakte tijd om deze scriptie mede tot
stand te brengen.
Daarnaast dank ik ook de collega’s van het labo moleculaire microbiologie, Sofie, Ellen,
Thomas, Charlotte, Liselotte en Sarah voor hun hulpvaardigheid tijdens het onderzoek.
Ook Dr. Sc. Anita Van Landschoot, die mij heeft begeleid tijdens het academiejaar, wil ik
graag bedanken.
Tot slot wil ik ook mijn ouders, partner, familie en vrienden bedanken. Zij hebben mij
gesteund toen het moeilijker ging, mij de moed gegeven om door te zetten tot de eindmeet in
zicht was en geholpen elk op hun manier.
Bedankt aan iedereen,
Jessie
Abstract
Seksueel overdraagbare aandoeningen komen frequent voor in onze maatschappij. Het
ziektebeeld gepaard gaande met een onbehandelde soa kan zeer ernstig zijn. Daarom is
vroegtijdige diagnostiek gewenst. De klassieke bacteriologische diagnostiek is weinig
gevoelig waardoor moleculaire detectiemethoden worden aangewend. De vaakgebruikte
commerciële kits kunnen echter nog altijd onvoldoende gevoelig zijn en ze zijn vaak beperkt
in hun aantal op te sporen parameters.
Het doel van dit onderzoek is een brede in-huis screeningsmethode te ontwikkelen waarbij
Neisseria gonorrhoeae, Mycoplasma genitalium, Ureaplasma urealyticum, Ureaplasma
parvum, Haemophilus ducreyi, Herpes Simplex Virus II, Herpes Simplex Virus I,
Trichomonas vaginalis, Chlamydia trachomatis, Mycoplasma hominis en Treponema
pallidum simultaan kunnen worden opgespoord met een TaqMan Low Density Array Card
(TAC) op basis van Real-Time PCR. Tijdens dit onderzoek wordt de nadruk gelegd op de
eerste zes pathogenen, de overige vijf worden behandeld in het gelijklopende thesisonderwerp
“Ontwikkeling van een TaqMan Array Card voor de simultane detectie van seksueel
overdraagbare pathogenen” door Jeroom Pieters.
Tijdens de ontwikkeling werden in de eerste plaats speciesspecifieke targetgenen geselecteerd
waarop primer- en probesequenties werden gegenereerd. Daarna werden deze geoptimaliseerd
en met bijkomende targets op de TAC geplaatst door middel van lyofyliseren. Uit de
resultaten na validatie is gebleken dat een aantal targets een slechte efficiëntie hadden of
kruisreactie vertoonden. Deze targets zullen bij een volgende versie van de TAC niet meer
aanwezig zijn. Daarnaast is gebleken dat de TAC een gevoeliger methode is dan de huidige
BD ProbeTec-methode en ze een bredere screening zal uitvoeren. De specificiteit is hoog
door de meerdere targets per pathogeen en de methode is meer gecontroleerd dankzij de
interne staalcontrole, inhibitie- en extractiecontrole en TAC-controle. Ook bewaarcondities en
matrix geïnduceerd effect werden nagegaan maar dit is tot op heden nog niet afgerond.
Seksueel overdraagbare aandoeningen, TAC, Real-Time PCR, validatie
Abstract
Sexually transmitted diseases are common in our society. The pathology associated with
untreated STDs can be very serious. Therefore, early diagnosis is desired. The classic
bacteriological diagnosis has a low sensitivity whereby molecular detection methods are used.
However, the frequently used commercial kits are not always sufficiently sensitive and they
tend to be restricted in their number of detectable pathogens.
The purpose of this research is to develop a broader in-house screening method where
Neisseria gonorrhoeae, Mycoplasma genitalium, Ureaplasma urealyticum, Ureaplasma
parvum, Haemophilus ducreyi, Herpes Simplex Virus II, Herpes Simplex Virus I,
Trichomonas vaginalis, Chlamydia trachomatis, Mycoplasma hominis and Treponema
pallidum will be detected simultaneously with a TaqMan Low Density Array Card (TAC)
based on Real-Time PCR. During this study, the focus will be on the first six pathogens, the
remaining five are treated in a parallel study "Ontwikkeling van een TaqMan Array Card voor
de simultane detectie van seksueel overdraagbare pathogenen" by Jeroom Pieters.
The first stage during the development was the selection of species-specific target genes to
generate primers and probes. These were then optimized and placed on the TAC by
lyophilizing with additional targets. The results after validation have shown that a number of
targets had a poor efficiency or cross-reaction was shown. These targets will not be present in
the next version of the TAC. In addition, it has been found that the TAC is a more sensitive
method than the current BD ProbeTec method and it will perform a wider screening. The
specificity is high because of the multiple targets per pathogen and the method is more
controlled through the internal controls. These controls include sample control,
inhibition/extraction control and TAC control. Also, storage conditions and matrix induced
effect were examined, but this has not been completed to date.
Sexually transmitted diseases, TAC, Real-Time PCR, validation
Inhoudsopgave
1
Inleiding ........................................................................................................................................ 13
2
Literatuurstudie ............................................................................................................................. 15
3
2.1
Seksueel overdraagbare aandoeningen .................................................................................. 15
2.2
De geselecteerde pathogenen ................................................................................................ 18
2.2.1
Neisseria gonorrhoeae .................................................................................................. 18
2.2.2
Herpes Simplex Virus II ................................................................................................ 20
2.2.3
Mycoplasma genitalium................................................................................................. 22
2.2.4
Ureaplasma urealyticum ............................................................................................... 25
2.2.5
Ureaplasma parvum ...................................................................................................... 26
2.2.6
Haemophilus ducreyi ..................................................................................................... 27
2.3
Extractie van nucleïnezuren .................................................................................................. 30
2.4
Real-Time PCR ..................................................................................................................... 32
2.4.1
Primers en probes .......................................................................................................... 35
2.4.2
Detectie via non-specific labels ..................................................................................... 37
2.4.3
Detectie via sequence specific probes ........................................................................... 41
2.4.4
ViiaTM7 Real-Time PCR system.................................................................................... 45
2.5
Gelelektroforese .................................................................................................................... 47
2.6
TaqMan Low Density Array Card ......................................................................................... 48
2.7
BD ProbeTec ......................................................................................................................... 49
Materiaal en methoden .................................................................................................................. 50
3.1
Materiaal................................................................................................................................ 50
3.1.1
Vircell controle DNA .................................................................................................... 50
3.1.2
Bacteriële culturen ......................................................................................................... 52
3.1.3
Klinische monsters ........................................................................................................ 52
3.1.4
Primers en probes .......................................................................................................... 53
3.2
Extractie van nucleïnezuren .................................................................................................. 56
3.2.1
3.3
Real-Time PCR Gene Expression Assays ............................................................................. 57
3.3.1
3.4
Werkwijze ..................................................................................................................... 56
Werkwijze ..................................................................................................................... 57
Real-Time PCR ..................................................................................................................... 58
3.4.1
Werkwijze Real-Time PCR met SYBR Select MasterMix en smeltcurveanalyse ........ 58
3.4.2
Werkwijze Real-Time PCR met TaqMan Fast Virus 1-step MasterMix ...................... 59
3.4.3
Werkwijze Real-Time PCR TAC-STD met Fast Virus 1-step MasterMix ................... 60
3.5
Gelelektroforese .................................................................................................................... 62
3.5.1
4
Werkwijze ..................................................................................................................... 62
3.6
Primer/probeselectie en optimalisatie uniplex PCR .............................................................. 64
3.7
Validatie TaqMan Low Density Array Card ......................................................................... 66
3.7.1
Matrices ......................................................................................................................... 66
3.7.2
Bepaling aanvaardbare performantiekenmerken ........................................................... 68
3.7.3
Praktische voorbereiding ............................................................................................... 74
Resultaten en discussie .................................................................................................................. 77
4.1
Selectie primers en probes ..................................................................................................... 77
4.1.1
Herpes Simplex Virus II ................................................................................................ 77
4.1.2
Haemophilus ducreyi ..................................................................................................... 77
4.1.3
Mycoplasma genitalium................................................................................................. 78
4.1.4
Ureaplasma urealyticum en Ureaplasma parvum......................................................... 78
4.1.5
Neisseria gonorrhoeae .................................................................................................. 78
4.2
Optimalisatie uniplex PCR .................................................................................................... 79
4.2.1
Functionaliteit primers .................................................................................................. 79
4.2.2
Efficiëntie PCR.............................................................................................................. 80
4.2.3
Specificiteit aan de hand van smeltcurveanalyse .......................................................... 83
4.2.4
Gelelektroforese ............................................................................................................ 86
4.2.5
Specificiteit in aanwezigheid van vreemd DNA ........................................................... 87
4.2.6
Functionaliteit probes .................................................................................................... 87
4.2.7
Specificiteit probes ........................................................................................................ 88
4.2.8
Gevoeligheid probe gebaseerde Real-Time PCR-reacties............................................. 90
4.2.9
Detectie genomisch DNA .............................................................................................. 92
4.2.10
Samenstelling TAC-STD array card ............................................................................. 95
4.3
Validatie TAC ....................................................................................................................... 98
4.3.1
Selecteren matrices ........................................................................................................ 98
4.3.2
Detectielimiet ................................................................................................................ 98
4.3.3
Herhaalbaarheid en reproduceerbaarheid .................................................................... 100
4.3.4
Matrix geïnduceerd effect............................................................................................ 103
4.3.5
Efficiëntie TaqMan Low Density Array Card ............................................................. 105
4.3.6
Specificiteit .................................................................................................................. 108
4.3.7
Juistheid ....................................................................................................................... 109
4.3.8
Bewaarcondities .......................................................................................................... 111
4.3.9
Vergelijking van methoden ......................................................................................... 111
4.3.10
Interne controles .......................................................................................................... 112
5
Besluit.......................................................................................................................................... 115
6
Bibliografische lijst ..................................................................................................................... 116
7
Bijlagen ....................................................................................................................................... 122
Lijst met figuren
Figuur 1: N. gonorrhoeae, foto met behulp van EM (Kenneth, 2012) .................................... 18
Figuur 2: Herpes Simplex Virus, vereenvoudigde figuur van de opbouw (Wagner,2003) ..... 20
Figuur 3: M. genitalium, foto met behulp van TEM (Taylor-Robinson & Jensen,2011) ........ 22
Figuur 4: U. urealyticum, foto met behulp van TEM (BacMap,2013) .................................... 25
Figuur 5: H. ducreyi, foto met behulp van TEM (BacMap,2013) ........................................... 28
Figuur 6: Schematische voorstelling van het principe van de QIA Symphony SP van Qiagen
(Descheemaeker,2013) ............................................................................................................. 31
Figuur 7: Éen cyclus tijdens PCR (polymerasekettingreactie,2014)........................................ 32
Figuur 8: Grafische representatie van Real-Time PCR-data (AB,2011) ................................. 34
Figuur 9: Spectrum en moleculaire structuur van SYBR Green (SYBR Green, 2013) ........... 37
Figuur 10: Denaturatiecurve waarbij fluorescentie daalt in functie van de temperatuur (Van
Gool,1997)................................................................................................................................ 38
Figuur 11: Smeltcurveanalyse van organisme A, B en combinatie organisme A en B (naar
MeltingCurveAnalysis,2013) ................................................................................................... 39
Figuur 12: Smeltcurveanalyse waarbij SNP wordt aangetoond (HighResolutionMelt,2014) . 39
Figuur 13: Proximal quenching principe (Vandesompele,2009) ............................................. 41
Figuur 14: FRET quenching principe (Vandesompele,2009) .................................................. 41
Figuur 15: Principe hybridization probes (LightCycler Real-time PCR,2014) ....................... 42
Figuur 16: Hydrolyse TaqMan-probe door hitte stabiel DNA-polymerase (Asuragen,2012) . 43
Figuur 17: Hydrolyse TaqMan MGB-probe door hitte stabiel DNA-polymerase (Kutyavin et
al.,2000) .................................................................................................................................... 44
Figuur 18: Werkingsprincipe van de ViiaTM7 Real-Time PCR-system (Nollet,2013) .......... 46
Figuur 19: Taqman Low Density Array Card (Steg et al.,2006;Zheng et al.,2006) ................ 48
Figuur 20: Temperatuurstemplate PCR en smeltcurveanalyse ................................................ 59
Figuur 21: Temperatuurstemplate uniplex PCR-reactie........................................................... 60
Figuur 22: Temperatuurstemplate TAC-STD array card PCR-reactie..................................... 61
Figuur 23: Amplificatiecurve met de 4 primerparen van N. gonorrhoeae............................... 80
Figuur 24: Amplificatiecurve verdunningsreeks N. gonorrhoeae primerpaar 307-308........... 84
Figuur 25: Smeltcurve verdunningsreeks N. gonorrhoeae primerpaar 307-308 ..................... 84
Figuur 26: Smeltcurve verdunningsreeks N. gonorrhoeae primerpaar 310-311 ..................... 85
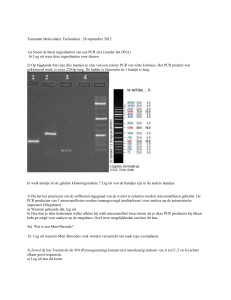
Figuur 27: Scheiding op agarosegel van alle primerparen van N. gonorrhoeae ...................... 86
Figuur 28: Amplificatiecurve U. parvum en S. aureus ............................................................ 88
Figuur 29: Multicomponent plot U. parvum en S. aureus ....................................................... 89
Figuur 30: Kruisreactie primerpaar 313-314 probe 315 met N. meningitidis .......................... 89
Figuur 31: Multicomponent plot Gene expression assay met humaan bloedstaal ................... 93
Figuur 32: Kruisreactie NG Omp AVK ................................................................................. 109
Figuur 33: Resultaten inhibitie/extractiecontrole ................................................................... 113
Figuur 34: Resultaten staalcontroles ...................................................................................... 114
Lijst met tabellen
Tabel 1: Overzicht van de belangrijkste soa’s en hun etiologisch agens (De Schrijver, 2004) 17
Tabel 2: Run-type met data collecion point (AB,2011) ........................................................... 45
Tabel 3: Gegevens Vircell controle DNA ................................................................................ 51
Tabel 4: Gegevens geselecteerde primers ................................................................................ 53
Tabel 5: Gegevens geselecteerde probes .................................................................................. 55
Tabel 6: Targetgenen per organisme ........................................................................................ 64
Tabel 7: Overzicht planning precisie in verschillende matrices .............................................. 70
Tabel 8: Samenstelling mixen kruisreactie .............................................................................. 71
Tabel 9: Mogelijks kruisreactieve stalen .................................................................................. 72
Tabel 10: Samenstelling en opmaak Vircell controle DNA mix 1 .......................................... 74
Tabel 11:Samenstelling en opmaak Vircell controle DNA mix 2 ........................................... 75
Tabel 12: Samenstelling mengstaal mix 1 en mengstaal mix 2 ............................................... 76
Tabel 13: Gemiddelde Ct-waarden van ieder organisme met bijhorende efficiëntie ............... 81
Tabel 14: Geselecteerde primerparen ....................................................................................... 83
Tabel 15: Resultaten geconcentreerde urines ........................................................................... 91
Tabel 16: Invloed van verschillende matrices op de PCR-reactie ........................................... 92
Tabel 17: Resultaten Gene expression assay ........................................................................... 93
Tabel 18: Resultaten Gene expression assay met klinische stalen........................................... 94
Tabel 19: Samenstelling TAC-STD array card ........................................................................ 96
Tabel 20: Bepaling limit of detection ....................................................................................... 99
Tabel 21: Herhaalbaarheid en reproduceerbaarheid ............................................................... 100
Tabel 22: Verschil in resultaat bij verschillende matrices ..................................................... 104
Tabel 23: Invloed van matrix op kruisreactie......................................................................... 104
Tabel 24: De efficiëntie voor ieder targetgen per organisme ................................................. 106
Tabel 25: Kwaliteitscontroles met BD ProbTec en TAC-STD array card ............................. 110
Tabel 26: Resultaat met BD ProbeTec-methode .................................................................... 112
1
Inleiding
Seksueel overdraagbare aandoeningen komen frequent voor in onze maatschappij. De
symptomen van een onbehandelde soa kunnen verregaande gevolgen met zich meebrengen.
Daarom is het van belang om soa’s in een vroeg stadium te diagnosticeren zodat een gepaste
behandeling kan worden gestart. De klassieke bacteriologische diagnostiek kan hiervoor
aangewend worden, maar deze is weinig gevoelig en niet altijd even evident. Daarom wordt
er gebruikgemaakt van moleculaire detectiemethoden die een hogere gevoeligheid kennen. De
moleculaire detectiemethode aangewend in het AZ Sint-Jan Brugge-Oostende is een
commerciële kit, de BD ProbeTec-methode, die echter onvoldoende gevoeligheid biedt en die
beperkt is tot het detecteren van twee soa’s, namelijk N. gonorrhoeae en C. trachomatis.
Daarnaast wordt er ook getest op Herpes Simplex Virus als hier specifieke vraag naar is.
Andere soa’s worden tijdens routinetesten niet opgespoord.
Het doel van het huidige onderzoek is het ontwikkelen van een brede en gevoelige in-huis
screeningsmethode waarbij Neisseria gonorrhoeae, Mycoplasma genitalium, Ureaplasma
urealyticum, Ureaplasma parvum, Haemophilus ducreyi, Herpes Simplex Virus II, Herpes
Simplex Virus I, Trichomonas vaginalis, Chlamydia trachomatis, Mycoplasma hominis en
Treponema pallidum simultaan kunnen worden opgespoord met een TaqMan Low Density
Array Card (TAC). Tijdens dit onderzoek wordt de nadruk gelegd op de eerste zes
pathogenen, de overige vijf worden behandeld in het gelijklopend thesisonderwerp
“Ontwikkeling van een TaqMan Array Card voor de simultane detectie van seksueel
overdraagbare pathogenen” door Jeroom Pieters. Tijdens de bachelorproef werd er reeds
onderzoek verricht naar speciesspecifieke targetgenen voor deze pathogenen waarbij voor
ieder organisme minimum twee targetgenen werden gezocht. Specifieke primers en probes
worden ontwikkeld op basis van deze targetgenen. Hierna volgt de optimalisatie van de
primers en probes.
De primers worden als eerste geoptimaliseerd met de SYBR Select MasterMix. Daarna
worden de probes geoptimaliseerd met de TaqMan Fast Virus 1-step MasterMix. Uit deze
resultaten zullen dan de twee gevoeligste primer-probe-paren per organisme geselecteerd
worden om op de TAC-STD Array card te plaatsen via lyofyliseren. De overige wells worden
aangevuld met interne controles en extra targets om de specificiteit te verhogen. De
daaropvolgende stap is de validatie van de TAC-STD Array card.
13
Een vooropgesteld validatieplan wordt gevolgd waarbij de volgende performantiekenmerken
zullen worden nagegaan:
- Limit of detection
- Herhaalbaarheid en reproduceerbaarheid
- Matrix geïnduceerd effect
- Efficiëntie
- Specificiteit
- Juistheid
- Bewaarcondities
- Vergelijking van methoden
Daarnaast zal ook de cut-off waarde bepaald worden bij de interne controles zodat er kan
worden nagegaan wanneer bijvoorbeeld de staalname onvoldoende is.
Aan de hand van de bekomen resultaten kan dan een besluit worden genomen of de
screeningsmethode al dan niet gevoeliger is en ter vervanging kan dienen van de huidig
aangewende methode.
Dit onderzoek is opgebouwd als volgt:
Als eerste wordt een literatuurstudie verricht naar de behandelde soa’s en wordt er van ieder
pathogeen een korte achtergrond geschetst. Ook de methoden die aangewend worden tijdens
het onderzoek worden nader toegelicht in deze literatuurstudie. In ‘Materiaal en methoden’
worden de gebuikte materialen en de gevolgde methoden beschreven. Hierna volgen de
resultaten van de optimalisatie en de validatie waarna een algemeen besluit kan worden
genomen betreffende de nieuw ontwikkelde screeningsmethode.
14
2
Literatuurstudie
2.1
Seksueel overdraagbare aandoeningen
Een seksueel overdraagbare aandoening (soa) of sexually transmitted disease (STD) is een
term die wordt gebruikt voor een scala aan klinische aandoeningen die veroorzaakt worden
door pathogenen die verworven en doorgegeven worden door hoofdzakelijk seksueel contact
(CDC,2010).
Een pathogeen dat een soa veroorzaakt, wordt overgedragen via bepaalde
lichaamsvloeistoffen, zoals sperma, bloed, vaginaal vocht of bij contact tussen slijmvliezen
die zich onder andere bevinden in de anus, penis, vagina en mond. In bepaalde gevallen
kunnen soa’s overgedragen worden via verticale transmissie1 tijdens de bevalling van een
besmette vrouw. Zo kunnen hiv, hepatitis B, hepatitis C en syfilis reeds overgedragen worden
tijdens de zwangerschap van moeder op foetus. Daarnaast kunnen hiv, hepatitis B, hepatitis C,
syfilis, de genitale Mycoplasma’s en Ureaplasma’s, chlamydia, herpes simplex virus en
gonorroe de baby infecteren tijdens zijn of haar passage door het geboortekanaal. Een soa kan
niet worden doorgegeven via speeksel, hoestbuien, insectenbeten of een onhygiënische wcbril. Ook bijvoorbeeld zwembaden vormen geen risico.
Wanneer men besmet is met een soa, kan dat al dan niet gepaard gaan met klachten, zoals:
-
al dan niet purulente afscheiding uit de genitaliën en/of anus;
-
symptomen gelijkaardig aan een blaasontsteking;
-
zweertjes, wratjes of blaasjes op of rond de genitaliën;
-
jeuk aan of rond de genitaliën;
-
gezwollen klieren in de liezen;
-
pijn aan de voortplantingsorganen.
Als er effectief een soa wordt vastgesteld, moet die worden behandeld ook al zijn er geen
klachten of symptomen. Zonder de juiste behandeling kan een soa aanleiding geven tot
ernstiger problemen (zie 2.2). De meeste soa’s zijn te genezen, andere zijn onder controle te
houden met de correcte antimicrobiële behandeling (vaak een combinatietherapie) die
specifiek is voor de betreffende soa.
1
Overdracht van het pathogeen van moeder op kind in de baarmoeder (in utero), tijdens de geboorte of door
borstvoeding.
15
Momenteel komen soa’s opnieuw frequenter voor. Redenen hiervoor zijn bijvoorbeeld de
terugval van het “safe-seksgedrag” (De schrijver, 2004). Aangezien meerdere soa’s te
behandelen zijn, neemt niet iedereen de gevaren die met deze pathogenen gepaard gaan even
serieus, waardoor vaker de protectieve voorbehoedsmiddelen achterwege worden gelaten.
Een andere reden is ook het taboe rond soa’s. Ze zijn vaak moeilijk bespreekbaar, zeker
binnen relaties waar maar één van beide partners besmet is. Als de soa wordt verzwegen, kan
de partner ook besmet worden zonder dat hij of zij er enig besef van heeft. Als dit verzwegen
wordt tegenover losse sekspartners, dan worden meerdere mensen geïnfecteerd tegen beter
weten in. Geregeld worden meerdere mensen besmet met één of meerdere soa’s die al dan
niet voor onaangename symptomen kunnen zorgen en bijkomende co-infecties met zich
meebrengen die de gezondheid in het gedrang brengen.
Het frequent voorkomen van soa’s ligt niet alleen aan de mensen die onverantwoord
promiscue gedrag vertonen, maar ook situeert er zich actueel in België een probleem ter
hoogte van de analytische beperkingen van de huidige detectiemethoden om de pathogenen
op te sporen. De actuele (vaak commerciële) analysemethoden detecteren over het algemeen
maar één of een beperkt aantal pathogenen, voornamelijk Neisseria gonorrhoeae en
Chlamydia trachomatis, al dan niet aangevuld met Mycoplasma genitalium. Hier is zeker
plaats voor de verbetering van de diagnostiek als men weet dat de meest voorkomende soa’s
zich niet enkel tot die pathogenen beperken. De belangrijkste soa’s met hun respectievelijk
etiologisch agens worden weergegeven in tabel 1. Zoals aangehaald in de inleiding zullen een
aantal van die pathogenen verder toegelicht worden.
16
Tabel 1: Overzicht van de belangrijkste soa’s en hun etiologisch agens (De Schrijver, 2004)
Aandoening
Infectueus agens
Chlamydia infectie
Chlamydia trachomatis
Condyloma acuminata/ genitale wratten
Humaan papilloma virus (HPV)
Donovanose / granuloma inguinale
Calymmatobacterium granulomatis
Gonorroe
Neisseria gonorrhoeae
Herpes genitalis
Herpes simplex virus (HSV-I en HSV-II)
Hepatitis B
Hepatitis B-virus
Hiv en aids
Human immunodefieciency virus (HIV-I en
HIV-II)
Lymphogranuloma venereum
Chlamydia trachomatis serovars L1 L2 L3
Scabies
Sarcoptes scabiei
Phthiriasis / schaamluizen
Phthirius pubis
Syfilis
Treponema pallidum
Trichomonase
Trichomonas vaginalis
Ulcus molle
Haemophilus ducreyi
Nongonococcal urethritis
Chlamydia
trachomatis,
Ureaplasma
urealyticum en parvum, HSV I en HSV II,
adenovirus, CMV, Mycoplasma hominis en
M. genitalium, Trichomonas vaginalis en
candida species
Genitale
infecties
(Cervicitis, Neisseria
gonorrhoeae,
Chlamydia
endomyometritis, salpingitis, tubo-avariële trachomatis, Mycoplasma hominis en M.
abcessen, PID)
genitalium, Ureaplasma urealyticum en U.
parvum, Trichomonas vaginalis, HSV I en
HSV II, CMV, anaerobe bacteriën,
Gardnerella
vaginalis,
Streptococci
(agalactiae,
pneumonia,
pyogenes),
enterobacteriaceae
17
2.2
De geselecteerde pathogenen
De pathogenen die behandeld worden tijdens dit onderzoek zijn vijf bacteriële stammen en
één virus, namelijk N. gonorrhoeae, M. genitalium, U. urealyticum, U. parvum, H. ducreyi en
Herpes Simplex Virus II. Deze zes pathogenen worden hieronder kort besproken.
2.2.1 Neisseria gonorrhoeae
Situering
Taxonomische indeling:
Rijk: Bacteria
Stam: Proteobacteria
Klasse: Beta Proteobacteria
Orde: Neisseriales
Familie: Neisseriaceae
Geslacht: Neisseria
Soort: Neisseria gonorrhoeae
Neisseria gonorrhoeae is een Gram-negatieve,
aerobe bacterie (diplococcen) die de seksueel
overdraagbare
aandoening
gonorroe
veroorzaakt. De bacterie is genoemd naar zijn
ontdekker, Albert Neisser.
Meerdere Neisseria species koloniseren het
menselijk lichaam, waarvan de meeste
commensale microflora betreffen, zoals N.
cinera of N. lactamica. De twee pathogene
stammen binnen het genus Neisseria zijn N.
Figuur 1: N. gonorrhoeae, foto met behulp van EM
gonorrhoeae en N. meningitidis. In dit
(Kenneth, 2012)
onderzoek zal de nadruk gelegd worden op N. gonorrhoeae, aangezien dat een soa is. N.
meningitidis is een bacterie die een ernstige bacteriële meningo-encephalitis veroorzaakt, wat
in het huidige onderzoek niet aan de orde is.
N. gonorrhoeae is enkel terug te vinden na seksueel contact of bij direct contact met een
besmet persoon. De bacterie is zeer gevoelig voor veranderingen in haar leefomgeving en
groeit dus best in een stabiele omgeving waardoor het menselijk lichaam een goede gastheer
is. Naast een stabiele leefomgeving biedt het menselijk lichaam ook bloed of hemoglobine
alsook verschillende vitamines en aminozuren, zodat de bacterie goed kan groeien
(Kenneth,2012).
18
Het natuurlijk afweermechanisme van het menselijk lichaam kan de bacterie niet volledig
elimineren doordat ze slecht wordt gefagocyteerd door de aanwezigheid van het
polysaccharidekapsel en de aanwezige pili of fimbriae (zie figuur 1).
Ziektebeeld
N. gonorrhoeae-infectie bij mannen brengt frequent een ontsteking teweeg van de muceuze
membranen van het voorste deel van de urethra. Die infectie veroorzaakt de afscheiding van
etter, kan moeilijk of pijnlijk urineren veroorzaken en gaat gepaard met een karakteristieke
roodheid, zwelling, branderig warm gevoel en pijn in dit voorste deel van de urethra.
Alternatieve infecties omvatten proctitis (na anorectale besmetting), pharyngitis (na orale
besmetting) en ophthalmitis. Maar de infectie kan ook asymptomatisch verlopen waardoor
deze niet of laattijdig gediagnosticeerd wordt en waardoor de risico’s op laattijdige
complicaties verhogen. De man kan daardoor onbewust drager zijn van gonorroe en de ziekte
verspreiden via onbeschermd seksueel contact.
N. gonorrhoeae-infectie bij vrouwen geeft veelal aanleiding tot een ontsteking van de
endocervix. Dat gaat gepaard met vaginale afscheiding en eventueel met klachten gelijkend
aan die van een blaasontsteking. Bij zowat vijftig percent van de geïnfecteerde vrouwen
verloopt de infectie asymptomatisch, wat dezelfde nadelen met zich meebrengt als bij
mannen. Hiernaast zal de kans op PID2 (pelvic inflammatory disease) stijgen. N. gonorrhoeae
was de eerste oorzaak van PID (CDC,2010; JM & Squifflet,2011; Boyce & Mitchell,1985)
maar sinds de ontdekking en het gebruik van penicilline in de eerste helft van de twintigste
eeuw, is het aantal gonorroe-gevallen op significante wijze afgenomen. Momenteel komt
gonorroe ook duidelijk minder frequent voor in vergelijking met andere soa’s gelinkt aan PID,
zoals Mycoplasma genitalium en Chlamydia trachomatis (zie tabel 1).
Bij pasgeboren baby’s kunnen er zich, via direct contact met de geïnfecteerde moeder,
ooginfecties ontwikkelen die het hoornvlies definitief beschadigen (ophthalmia neonatorum).
Ook kan gonorroe in zeldzame gevallen van gedissemineerde infectie (DGI = disseminated
gonococcal infection) een fatale afloop hebben bij pasgeboren baby’s, met een beeld van
sepsis met secundaire infecties: artritis, huidinfectie, tenosynovitis, endocarditis, meningitis
en “gonococcal scalp abcessen”.
2
PID: Ernstige ontstekingen van de vrouwelijke voortplantingsorganen, zoals onder andere de ovaria, eileiders
en uterus wat littekenweefsel en verklevingen veroorzaakt met infertiliteit tot gevolg.
19
Problemen bij diagnostiek
Detectie van N. gonorrhoeae kan ten gevolge van inadequate of te beperkte specificiteit van
de uitgevoerde detectietechniek verwarring met zich meebrengen door vals positieve reacties
met klinisch verwaarloosbare commensale Neisseria species (Boyce & Mitchell,1985). Die
commensale flora komen voor op dezelfde plaatsen van het menselijk lichaam en ze hebben
een gelijkaardige morfologie. Men dient er dus zeker van te zijn dat de gebruikte
detectiemethode specifiek is voor N. gonorrhoeae, gezien de mogelijk zware gevolgen onder
andere in het kader van verkrachting of kindermisbruik.
2.2.2 Herpes Simplex Virus II
Situering
Virus classificatie:
Groep: Groep 1 (dsDNA)
Orde: Herpesvirales
Familie: Herpesviridae
Subfamilie: Alphaherpesvirinae
Geslacht: Simplexvirus
Soort: Herpes Simplex Virus II
Herpesvirussen zijn grote en complexe
dsDNA-virussen die geëvolueerd zijn
over minstens 400 miljoen jaar.
Ondanks
de
verschillen
in
aminozuursequentie
zijn
ongeveer
veertig
genen
vergelijkbaar
en
geconserveerd
gebleven
in
alle
herpesvirussen
in
zoogdieren
(Norberg,2010).
De
genetische
informatie van die virussen komt voor
als een dubbelstrengig, lineair DNAmolecule aanwezig in een icosahedraal
Figuur 2: Herpes Simplex Virus, vereenvoudigde figuur van de
opbouw (Wagner,2003)
capside (zie figuur 2).
De capside wordt omgeven door een lipide dubbellaag die de envelop wordt genoemd. Tussen
de envelop en de capside bevindt zich het tegument wat onder andere zorgt voor de
aanhechting van de envelop en de capside. Op de envelop bevinden zich glycoproteïne spikes
die dienen als herkenningselement.
20
Ze binden specifiek met receptoren die zich bevinden op het oppervlak van een gastheercel.
Een viruspartikel moet namelijk specifiek binden met een gastheercel om deze te invaderen en
te infecteren (Norberg,2010). Het virus wordt verspreid via direct contact, seksueel contact of
via verticale transmissie tijdens de geboorte.
Ziektebeeld
Meer dan veertig jaar geleden werd HSV I onderscheiden van HSV II. Hierbij werd HSV II
hoofdverantwoordelijk geacht voor de genitale herpes en HSV I voor herpes labialis.
Wetenschappelijk onderzoek heeft echter uitgewezen dat die scheiding nog moeilijk te maken
is en beide virussen verantwoordelijk kunnen zijn voor zowel genitale herpes als herpes
labialis.
Een virale infectie van HSV II gaat meestal gepaard met genitale laesies en genitale zweren
(GUD = genital ulcer disease3). In die genitale zweren zijn de viruspartikels in overvloed
aanwezig maar ook asymptomatische infectie komt frequent voor. Ruw geschat maken
ongeveer veertig percent van de geïnfecteerde mensen asymptomatische infecties door. Die
personen kunnen het virus overdragen, zelfs na meerdere jaren. Ook is er sprake van vaginale
afscheiding, baarmoederhalsontsteking en bekkenpijn bij vrouwen (Biswas et al.,2011). In
bepaalde gevallen kan er zelfs sprake zijn van neurologische klachten, zoals zenuwpijn,
gevoelsstoornissen ter hoogte van de onderste ledematen, hoofdpijn en dergelijke.
De eerste acute symptomatische fase kan ofwel een primaire infectie betreffen bij een persoon
die nog nooit in contact kwam met het virus ofwel betreft het een klinische manifestatie bij
een persoon die reeds van tevoren was geïnfecteerd. De eerste symptomen verschijnen in
geval van primo-infectie ongeveer zeven dagen na de transmissie van het virus. De
symptomen duren enkele weken en verdwijnen dan spontaan maar het virus zal latent in het
lichaam aanwezig blijven. Opflakkering treedt op wanneer er sprake is van een lage
weerstand van de gastheer. Die opflakkeringen zijn frequenter bij HSV II dan bij HSV I.
Het virus kan ook ernstiger symptomen veroorzaken, zoals meningitis en zware neonatale
sepsis (gekenmerkt door een multi-orgaaninfectie met viremie) waaraan een hoge mortaliteit
gelinkt is. Ook zullen de genitale laesies een grote risicofactor vormen in geval van aanwezige
hiv (human immunodeficiency virus) doordat de infectie de kans op parallelle hiv-Ibesmetting meer dan tweemaal zal verhogen (Biswas et al.,2011). Er wordt verondersteld dat
de infectie met HSV II de genitale mucosa beschadigt en zo gevoelige plaatsen of een zekere
permeabiliteit creëert voor een hiv-virus, wat leidt tot een verhoogde vatbaarheid voor dit
virus bij hiv-negatieve personen.
3
Genital ulcer disease: Zwerende, erosieve, pustulaire of vesiculaire genitale laesis veroorzaakt door
voornamelijk HSV, Treponema pallidum en Haemophilus ducreyi.
21
Problemen bij diagnostiek
De actueel gebruikte diagnostische methoden zijn niet erg gevoelig en tegelijkertijd weinig
specifiek.
Enerzijds zijn de klassieke virale celcultuur en de HSV I- en HSV II-antigeen detectietest de
meest gebruikte tests om het virus rechtstreeks op te sporen en simultaan te typeren.
Anderzijds wordt HSV-serologie gebruikt om onrechtstreeks een voorgaand contact met HSV
aan het licht te brengen door middel van antistofbepaling.
Als HSV niet wordt gedetecteerd in urogenitale monsters, wil dat niet zeggen dat HSV
afwezig is, omdat de virale afscheiding intermittent is. Een controletest is dus noodzakelijk
om de resultaten te vergelijken.
2.2.3 Mycoplasma genitalium
Situering
Taxonomische indeling:
Rijk: Bacteria
Stam: Firmicutes
Klasse: Mollicutes
Orde: Mycoplasmatales
Familie: Mycoplasmataceae
Geslacht: Mycoplasma
Soort: Mycoplasma genitalium
Mycoplasma genitalium is een kleine
intracellulaire bacteriesoort die de genitaliën
koloniseert. Ze heeft een karakteristieke
flesvorm (zie figuur 3) waarbij het
binnendringen van de bacterie wordt
bemiddeld door de gespecialiseerde
tipstructuur. Deze soort is de kleinst
zelfreplicerende bacterie en is regressief
geëvolueerd door genoomreductie vanuit
Gram-positieve
bacteriële
voorouders,
namelijk clostridia (Taylor-Robinson &
Jensen,2011).
Figuur 3: M. genitalium, foto met behulp van TEM
(Taylor-Robinson & Jensen,2011)
22
M. genitalium werd voor het eerst geïsoleerd in 1981 uit twee mannen met non-gonococcal
urethritis (NGU) (Haggerty & Taylor,2011). NGU is een ontsteking van de urethra die niet
door gonorroe is veroorzaakt. Intussen is Mycoplasma genitalium algemeen gekend als een
pathogeen dat NGU veroorzaakt, net zoals ook Chlamydia trachomatis, Ureaplasma
urealyticum en Trichomonas vaginalis (Kim et al.,2011).
Ziektebeeld
Mycoplasma genitalium is een pathogeen dat ook regelmatig voorkomt bij vrouwen met PID
(Haggerty & Taylor,2011) en kan zowel symptomatisch als asymptomatisch voorkomen. PID
omvat infecties van de vrouwelijke voortplantingsorganen, zoals infectie van de uterus, de
cervix, de ovaria en de tuba. Die infecties worden veroorzaakt door verscheidene types van
bacteriën, waaronder de pathogenen die verantwoordelijk zijn voor gonorroe en chlamydia.
Ze zullen zich uitbreiden naar de buikholte en daar ernstige complicaties tot gevolg hebben,
waaronder mogelijke verklevingen van darmlissen, abcessen en obstructie van de tuba.
Hoewel Chlamydia trachomatis en Neisseria gonorrhoeae vaak PID’s veroorzaken, zou tot
zeventig percent van de infecties een andere nog te identificeren oorsprong hebben. M.
genitalium is geassocieerd met PID, maar is onafhankelijk van een chlamydia- of gonorroebesmetting. Door deze associatie zal de risicofactor voor PID bij een individu veel hoger
liggen als het individu besmet is met M. genitalium.
Cervicitis (= baarmoederhalsontsteking) kan eveneens veroorzaakt worden door M.
genitalium. Cervicitis is ook een veelvoorkomend antecedent voor PID (Haggerty &
Taylor,2011). Dus kunnen we ervan uitgaan dat M. genitalium non-gonococcal en nonchlamydiale PID zal veroorzaken. Wetenschappelijk bewijs hiervoor werd reeds geleverd
door onderzoek bij muizen. Dat toont aan dat de pathogenen stijgen van de lower genital tract
naar de upper genital tract waarbij morfologische veranderingen in eileidercellen met cilia
worden gedetecteerd. Ook bij vrouwen met een eileiderontsteking worden deze morfologische
veranderingen waargenomen in de eileidercellen. Verder is ook aangetoond dat M. genitalium
zich kan vasthechten aan menselijke spermatozoa. Hierdoor is er een hogere kans dat de
bacterie meegevoerd zal worden naar de vrouwelijke upper genital tract. Via PCR
(polymerase chain reaction) is er een onafhankelijk verband aan te tonen tussen M.
genitalium en PID. Aanvullend observeerde men in de PEACH4-studie dat vijftien percent
van de vrouwen met PID ook positief test voor M. genitalium in de baarmoederhals en het
baarmoederslijmvlies.
4
PID Evaluation and Clinical Health study
23
PID kan resulteren in langdurige problemen voor de maternale reproductiviteit, zoals
infertiliteit, buitenbaarmoederlijke zwangerschap en chronisch bekkenpijn. Of M. genitalium
al dan niet kan resulteren in gynaecologische problemen is niet duidelijk. Zoals hierboven
beschreven kan M. genitalium ook asymptomatisch voorkomen, wat de kans op ‘silent’ PID
doet toenemen.
Sommige studies linken M. genitalium met vaginale afscheiding en urinebuisinfectie, maar
die symptomen zijn minder uitgesproken dan in geval van gonococcen-infectie. Bij
asymptomatische besmetting verhoogt de kans op een te late behandeling, waardoor de ziekte
blijft aanslepen. Ook is er aangetoond dat PID in combinatie met M. genitalium gelijkaardige
symptomen heeft als PID in associatie met C. trachomatis maar de symptomen worden vaak
later in het ziekteproces opgemerkt, waardoor er al meer schade aangericht kan zijn (Haggerty
& Taylor,2011). PID in combinatie met N. gonorrhoeae zal ernstiger symptomen veroorzaken
die sneller merkbaar zijn, waardoor deze bacteriële infectie sneller effectief kan worden
behandeld.
PID in combinatie met M. genitalium wordt behandeld met antibiotica5 waarbij rekening moet
worden gehouden met de toenemende macrolide-resistentie van M. genitalium. Die resistentie
kan leiden tot foute en inadequate behandeling en bijgevolg persisterende of recidiverende
infecties, wat resulteert in chronische ontstekingen en secundaire infertiliteit.
Bij genitale infectie in mannen, en dus voornamelijk een beeld van urethritis, ziet men dat de
spermakwaliteit duidelijk afneemt, met niet enkel een effect op de mobiliteit van de
spermacellen maar tevens op de structuur, de viabiliteit en de concentratie.
Problemen bij diagnostiek
M. genitalium is een moeilijk aantoonbare intercellulaire bacterie zonder celwand. Ze is niet
aan te tonen met klassieke celkweektechnieken door de plaats van voorkomen in de cel en
doordat de cellen zeer klein zijn (< 1 µm). Momenteel worden nucleic acid amplification tests
(NAATs) aanbevolen om dit pathogeen te detecteren (Haggerty & Taylor,2011; Ross &
Jensen,2006).
5
Ofloxacin, levofloxacin, ceftriaxone plus doxycycline of cefoxitin en probenecid plus doxycycline.
24
2.2.4 Ureaplasma urealyticum
Situering
Taxonomische indeling:
Rijk: Bacteria
Stam: Firmicutes
Klasse: Mollicutes
Orde: Mycoplasmatales
Familie: Mycoplasmataceae
Geslacht: Ureaplasma
Soort: Ureaplasma urealyticum
Ureaplasma urealyticum, vroeger Tmycoplasma genoemd, is een genitaal
Mycoplasma
dat
de
genitaliën
koloniseert en infecties kan veroorzaken.
Het is een Gram-negatief unicellulair
organisme zonder celwand en het
produceert kolonies die onzichtbaar zijn
voor het blote oog (zie figuur 4). De
bacterie wordt verspreid via seksueel
contact, maar kan ook verspreid worden
via speeksel, besmette naalden en bloed.
Figuur 4: U. urealyticum, foto met behulp van TEM
Daarom
wordt
U.
urealyticum
(BacMap,2013)
voornamelijk geclassificeerd als een
bacteriële infectie en niet als klassieke overdraagbare aandoening, ook al wordt ze vaak
verspreid via seksueel contact (Ureaplasma urealyticum,2013) en wordt ze teruggevonden bij
zowat zeventig percent van de seksueel actieve populatie.
U. urealyticum kan als gevaarlijk beschouwd worden, aangezien de symptomen vaak
nauwelijks merkbaar zijn. Hierdoor kan de bacterie lange tijd onopgemerkt in het lichaam
verblijven en de aanleiding geven tot bepaalde ziektes, zoals urethritis.
Ziektebeeld
U. urealyticum wordt herkend als een pathogeen voor NGU sinds de jaren 1950. Volgens
sommige artsen wordt de bacterie beschouwd als een natuurlijk voorkomend organisme
(kolonisator van mucosa) in het menselijk lichaam, terwijl ze volgens andere artsen een niet te
verwaarlozen pathogeen is. Dit komt door tegenstrijdige studies die beschrijven dat U.
urealyticum al dan niet een causatief agens is voor NGU (Kim et al.,2011).
25
Naast NGU wordt U. urealyticum ook duidelijk geassocieerd met andere medische
problemen, zoals onvruchtbaarheid, doodgeboorte, vroeggeboorte, neonatale pneumonia,
bronchopulmonary dysplasia (BPD6), chorioamnionitis7 en neonatale meningitis al dan niet
gepaard gaande met sepsis.
De symptomen zijn niet altijd meteen merkbaar. Enkele vaak voorkomende maar vrij vage
klachten zijn een brandend gevoel en pijn tijdens het urineren, vaak urineren, ontsteking van
de genitaliën, genitale afscheiding, bloedverlies en pijn in de onderbuik.
Mannen ondervinden vaak een ontsteking van de bijbal en zwelling van de testikels. Vrouwen
ondervinden mogelijk ernstige bekkenpijn vanwege PID die hiermee gepaard gaat.
Problemen bij diagnostiek
Er zijn gevoelige methoden nodig, in de eerste plaats om U. urealyticum op te sporen en een
onderscheid te kunnen maken met U. parvum. Klassieke methoden komen daarvoor niet in
aanmerking, methoden op basis van PCR zijn hiervoor beter geschikt.
2.2.5 Ureaplasma parvum
Situering
Taxonomische indeling:
Rijk: Bacteria
Stam: Firmicutes
Klasse: Mollicutes
Orde: Mycoplasmatales
Familie: Mycoplasmataceae
Geslacht: Ureaplasma
Soort: Ureaplasma parvum
In 1954 werd een kleine Mycoplasma-bacterie beschreven die door Maurice C. Shepard
gevonden was in het menselijk urogenitale kanaal. In 1962 werd dit bevestigd door Denys K.
Ford. De bacterie is een zelf replicerende entiteit die groeit bij een lage pH. Net zoals bij U.
urealyticum is het een Gram-negatief unicellulair organisme zonder celwand en het
produceert kolonies die onzichtbaar zijn voor het blote oog.
6
BPD = longziekte met ontsteking en littekenweefsel bij te vroeggeboren baby’s.
Chorioamnionitis = infectie van de membranen (placentaweefsels) en het vruchtwater. Kan leiden tot
bacteriëmie bij de moeder en tot vroeggeboorte en ernstige infecties bij de pasgeboren baby
7
26
Binnen U. urealyticum werden er veertien serotypes8 geïdentificeerd. Binnen deze serotypes
worden twee verschillende groepen erkend, namelijk biovar 1 en biovar 2. Nu wordt
Ureaplasma urealyticum biovar 1 actueel U. parvum genoemd. In deze groep vallen de
serotypes 1, 3, 6 en 14. De andere serotypes vallen onder Ureaplasma urealyticum biovar 2
die nu U. urealyticum wordt genoemd (Katsura et al.,2010).
Beide soorten kunnen via conventionele cultuurmethoden niet van elkaar worden
onderscheiden. Daarvoor zijn gevoeligere methoden nodig, zoals PCR.
Ziektebeeld
U. parvum wordt geassocieerd met verschillende ziektes, zoals urethritis, chorioamnionitis,
infecties bij chirurgische ingrepen, neonatale meningitidis, PID en dergelijke. De bacterie kan
onvruchtbaarheid veroorzaken door ontsteking van de placenta en infectie van de
vruchtwaterzak. Bij bepaalde ziektes bij pasgeboren baby’s wordt de bacterie teruggevonden,
zoals bij chronische longziekte, bronchopulmonary dysplasia, pneumonia en dergelijke. Ze
wordt ook teruggevonden bij kinderen met het respiratory distress syndrome (RDS) (Katsura
et al.,2010).
Problemen bij diagnostiek
Bij U. parvum treden dezelfde problemen op als bij U. urealyticum (zie 2.2.4).
2.2.6 Haemophilus ducreyi
Situering
Taxonomische indeling:
Rijk: Bacteria
Stam: Proteobacteria
Klasse: Gamma Proteobacteria
Orde: Pasteurellales
Familie: Pasteurellaceae
Geslacht: Haemophilus
Soort: Haemophilus ducreyi
8
Een serotype of serovar is een specifiek subtype van een bepaald micro-organisme, ingedeeld op basis van de
specifieke antigenen aanwezig op het celmembraan.
27
H.
ducreyi
is
een
Gram-negatieve
streptococcus bacil (zie figuur 5) die
verantwoordelijk is voor de seksueel
overdraagbare
aandoening
“ulcus
molle”, ook wel “weke sjanker” of
“chancroïd” genoemd. Deze bacterie
wordt uitsluitend verspreid via seksueel
contact en de natuurlijke gastheer is het
menselijk lichaam. Buiten het menselijk
lichaam zal de bacterie vlug afsterven.
Dat maakt het moeilijk om de bacterie
verder te kunnen bestuderen in vitro
(Alfa,2005).
Figuur 5: H. ducreyi, foto met behulp van TEM
(BacMap,2013)
Ziektebeeld
Chancroïd gaat gepaard met pijnlijke open zweren op de genitaliën en soms met gezwollen
lymfeklieren in de lies. Die zweren verschijnen klassiek vanaf twee à drie dagen na
besmetting, maar ze kunnen ook uitgesteld voorkomen tot één maand na infectie (Kemp et
al.,2011). Zowel mannen als vrouwen worden geïnfecteerd al zullen de symptomen bij
vrouwen vaak subklinisch voorkomen.
Globaal gezien is H. ducreyi de meest voorkomende oorzaak voor GUD. Ze komt ook vaak
voor samen met syfilis. Deze co-infectie moet worden gedetecteerd, zodat de gepaste
antibioticakuur kan worden gestart. Naast GUD zal H. ducreyi een verhoogd risico op
besmetting met en transmissie van hiv met zich meebrengen. De genitale zweren van
chancroïd zijn namelijk geassocieerd met seksuele transmissie van het hiv-virus (EMBLEBI,2013).
Problemen bij diagnostiek
Chancroïd komt vooral voor in ontwikkelingslanden, zoals Centraal-Afrika, Azië en LatijnsAmerika. Aangezien de bacterie nauwelijks voorkomt in Westerse landen en GUD
veroorzaakt kan worden door andere vaak voorkomende soa’s, zoals HSV I, HSV II en
Treponema pallidum, is de kans op een verkeerde diagnose zeer reëel. De zweren veroorzaakt
door H. ducreyi zijn namelijk zeer gelijkaardig aan die van primaire syfilis. Behandeling met
antibiotica kan de infectie met H. ducreyi genezen, al moet men rekening houden met de
toenemende resistentie.
28
Detectie van de ziekte gebeurt via klinisch onderzoek, microbieel nazicht van de bacteriële
flora ter hoogte van de zweren en via klinische opsporing en vervolgens via een biopsie van
de gezwollen lymfeknoppen. Bloedtesten zijn niet voorhanden. De detectiemethode is dus vrij
ongevoelig bij gebrek aan informatie omtrent specifiek vermoeden van deze infectie, gezien
de fragiliteit van de kiem9 en de multibacteriële omgeving in de liezen, waardoor de groei van
H. ducreyi niet wordt opgemerkt10. Hierdoor is een betere detectiemethode gewenst. Die moet
minder gevoelig zijn voor de pre-analytische fase die een klinisch monster doorloopt en
minder afhankelijk zijn van het replicatievermogen van dit micro-organisme, wat het geval is
bij de klassieke bacteriële kweek.
9
De kiem is erg gevoelig voor pre-analytische omgevingsfactoren en heeft nood aan specifieke groeifactoren.
Dit door bacteriële overgroei door huidflora en andere bacteria.
10
29
2.3
Extractie van nucleïnezuren
Het doel van een nucleïnezuurextractie (DNA/RNA-isolatie) is het opzuiveren en
concentreren van nucleïnezuren (NZ) uit micro-organismen aanwezig in een klinisch monster,
zodat dit kan gebruikt worden bij verdere moleculaire technieken, zoals Real-Time PCR. Er
werd gebruikgemaakt van het QIASymphony SP-toestel van Qiagen waarbij de NZ-extractie
aan de hand van magnetische silica beads bestaat uit volgende stappen:
-
lyseren van de cellen
guanidiumisothiocyanaat;
-
degraderen van de eiwitten met proteïnase K en SDS;
-
binden van de vrijgekomen nucleïnezuren aan magnetische silica-micropartikels bij
en
denatureren/inactiveren
van
de
eiwitten
met
hoge natriumchlorideconcentratie;
-
wegwassen van de onzuiverheden;
-
elueren van de nucleïnezuren in buffer met laag natriumchlorideconcentratie.
Guanidium isothiocyanaat (GITC) wordt toegevoegd om de cellen te lyseren. Het is een sterk
chaotroop agens dat naast eiwitten in de cel en celmembraan, ook mogelijks aanwezige
DNase en RNase zal denatureren zodat hun activiteit verloren gaat. Dat zorgt ervoor dat de
meeste cellen spontaan lyseren. Het is een noodzakelijke stap tijdens de NZ-extractie om de
vrijgekomen nucleïnezuren te beschermen tegen afbraak van die enzymen.
Een vaak gebruikt detergens is sodium dodecyl sulfaat (SDS). Het detergens zal de eiwitten
denatureren en de eigen lading van de eiwitten maskeren met een negatieve lading waardoor
proteïnase K makkelijker zal kunnen inwerken om de eiwitten af te breken.
Proteïnase K is een endolytisch protease dat wordt gebruikt voor de afbraak van eiwitten in
nucleïnezuurbereidingen. Het splitst de peptidebindingen aan het carboxyluiteinde van
alifatische, aromatische of hydrofobe aminozuren. Proteïnase K werkt tot op hoge
temperaturen zonder zelf te denatureren (boven 65 °C zal dit enzym denatureren). Het heeft
ook een brede pH range (pH 4,0 tot 12,5 met optimum = 7,5 tot 8,0) en is ongevoelig voor de
denaturerende eigenschap van SDS (Kruhøffer et al.,2010).
De selectieve extractie van de nucleïnezuren gebeurt aan de hand van magnetische silicamicropartikels. Het mechanisme werkt als volgt (zie figuur 6):
30
De vrijgekomen nucleïnezuren worden gebonden op magnetische silica-micropartikels, die
vooraf aan het staal zijn toegevoegd, door middel van gevormde kationbruggen bij een hoge
natriumchlorideconcentratie. De magnetische silica-micropartikels worden vervolgens
aangetrokken door een magnetische (roer)staaf die wordt beschermd door een beschermhuls
(rod cover) die in het gelyseerde staal gedompeld is. De staaf met zijn beschermhuls wordt
overgebracht in een ander recipiënt met wasbuffer waarna de magnetische staaf wordt
verwijderd uit zijn beschermhuls en de magnetische silica-partikels loslaten van de
beschermhuls. Dit zal meerdere malen gebeuren om onzuiverheden weg te wassen, aangezien
deze onzuiverheden een inhiberend effect kunnen teweegbrengen in verdere moleculaire
technieken, zoals Real-Time PCR.
De laatste stap is de elutie van nucleïnezuren in een elutiebuffer met laag
natriumchloridegehalte waarbij de kationbruggen worden verbroken. Hierbij worden de
nucleïnezuren gescheiden van de magnetische silica-micropartikels en wordt een zuiver staal
verkregen na afzonderen van de magnetische silica-micropartikels (Descheemaeker,2013).
Het extract is nu klaar voor verder gebruik.
Figuur 6: Schematische voorstelling van het principe van de QIA Symphony SP van Qiagen (Descheemaeker,2013)
Tijdens extractie wordt altijd een interne controle meegenomen in het staal wat verder wordt
besproken bij 3.2.1.
31
2.4
Real-Time PCR
PCR (= Polymerase Chain Reaction) is een efficiënte methode om DNA-moleculen te
vermenigvuldigen op gecontroleerde wijze (= amplificeren) en zo het doelwit-DNA tot
detecteerbare proporties te brengen. De te amplificeren sequenties (= de targets) zijn de
regio’s die uniek en geconserveerd zijn binnen de genetische variabiliteit van ieder (micro-)
organisme.
Bij een conventionele PCR zal er enkel een kwalitatieve bepaling plaatsvinden, terwijl bij
Real-Time PCR ook een accurate, gevoelige en vlugge kwantitatieve bepaling mogelijk is
(Pestana et al.,2010).
Om een PCR-reactie uit te voeren, zijn verscheidene basiselementen nodig: template-DNA,
primers, buffer, MgCl2, dNTPs en hitte-stabiel DNA-polymerase. Verscheidene cycli worden
doorlopen (40 à 45) bij een PCR-reactie waarbij iedere cyclus bestaat uit drie stappen (zie
figuur 7):
1. Denaturatie (= melting)
Het template-DNA wordt gedenatureerd bij 95 °C.
2. Aanhechting (= annealing)
Primers hechten zich aan de gedenatureerde strengen waarna DNA-polymerase
kan aanhechten. Dit gebeurt bij een temperatuur die een vijftal graden onder de
smelttemperatuur van de primers ligt.
3. Synthese (= elongation)
Hittestabiel DNA-polymerase zal de streng verlengen in de 5’-3’-richting bij de
optimale temperatuur van het DNA-polymerase.
Figuur 7: Éen cyclus tijdens PCR (polymerasekettingreactie,2014)
32
De drie verschillende stappen tijdens een PCR-cyclus gebeuren in veel PCR-reacties op drie
verschillende temperaturen, wat een drie-staps-PCR wordt genoemd. Tijdens het onderzoek
werd echter gebruikgemaakt van een twee-staps-PCR, wat wil zeggen dat de annealing van de
primers en de elongatie beide op dezelfde temperatuur gebeuren, namelijk 60 °C (zie 3.4).
Binnen een korte tijd worden (exponentieel) exacte kopieën aangemaakt van de
targetsequentie die in overvloed aanwezig zullen zijn. Het geamplificeerde DNA is
beschikbaar voor verdere analyse. Theoretisch gezien zal de hoeveelheid amplificatieproduct
tijdens iedere cyclus verdubbelen, maar in de realiteit zal de geamplificeerde hoeveelheid een
plateauwaarde bereiken na een aantal cycli door uitputting van bepaalde basiselementen.
Bij conventionele PCR zal de analyse van het geamplificeerde DNA gebeuren na het
beëindigen van de PCR-reactie aan de hand van bijvoorbeeld agarosegelelektroforese van het
amplicon in aanwezigheid van ethidiumbromide. Dat resulteert in een kwalitatieve bepaling
en een semi-kwantitatieve bepaling als men de intensiteit van de bekomen bandjes vergelijkt.
Bij Real-Time PCR is het mogelijk om de amplicons al tijdens een vroeg stadium van de
PCR-reactie te meten, namelijk in de exponentiële fase waar er wel een kwantitatieve relatie
is met het inputmateriaal. Hier zal dus naast een kwalitatieve bepaling ook een kwantitatieve
bepaling plaatsvinden. Om deze meting te realiseren, wordt er gebruikgemaakt van
fluorescente probes of fluorescente intercallerende stoffen die tijdens de reactie een meetbaar
signaal genereren dat proportioneel is met de hoeveelheid van het geamplificeerde product
(Pestana et al., 2010).
De standaard grafische weergave van een PCR-reactie is de amplificatiecurve zoals
weergegeven in figuur 8 (AB,2011).
In weergave A van figuur 8 wordt Rn (= normalized reporter signal) weergegeven tegenover
het PCR-cyclusnummer. Rn is de fluorescentie van de reporterkleurstof gedeeld door de
fluorescentie van de passieve referentiekleurstof11. Op deze grafiek wordt de baseline
aangeduid welke als volgt wordt verkregen: Tijdens de PCR-reactie kunnen veranderende
reactiecondities de fluorescentie beïnvloeden. Hierdoor verkrijgen we een achtergrondsignaal.
Dit achtergrondsignaal komt voor tijdens de initiële cycli van de PCR-reactie die voorafgaat
aan de significante accumulatie van het amplicon. Het achtergrondsignaal wordt dan gebruikt
om de baseline fluorescentie te bepalen van de reactieplaat. Eenmaal het amplificatiesignaal
boven deze achtergrond uitkomt, worden meer accurate en betrouwbare resultaten bekomen
van de fluorescentiemetingen.
11
De interne referentiekleurstof wordt gebruikt om de reporterkleurstof te normaliseren tijdens de data-analyse,
wat noodzakelijk is om fluorescentie-fluctuaties door veranderingen in concentratie of volume door
pipetteerfouten te corrigeren.
33
Op weergave B van figuur 8 wordt de deltaRn (= Rn minus de baseline) weergegeven ten
opzichte van het PCR-cyclusnummer. Hierop kan de threshold aangeduid worden. De
threshold is de numerieke waarde die toegekend wordt aan iedere run welke een statistisch
significant punt boven de baseline weergeeft. De intersectie tussen de amplificatiecurve en de
thresholdlijn wordt de threshold cycle (Ct) genoemd. Kort samengevat geeft de Ct-waarde het
punt weer waar het signaal wordt gedetecteerd boven het achtergrondsignaal van de reactie.
Vanaf dit punt zal de reactie zich ook in de exponentiële fase bevinden.
De Ct-waarde kan worden beïnvloed door verschillende factoren. Daarom kunnen Ct-waarden
van verschillende PCR-reacties met verschillende condities of reagentia niet vergeleken
worden. De factoren die de Ct-waarde kunnen beïnvloeden zijn de mastermix componenten,
de beginconcentratie van het target-DNA, de ROX passieve referentiekleurstof en de
efficiëntie van de PCR-reactie.
Figuur 8: Grafische representatie van Real-Time PCR-data (AB,2011)
Naast de bekomen amplificatiecurve wordt ook een multicomponentplot weergegeven waarbij
de fluorescentie wordt uitgezet in functie van het aantal cycli. Beide dienen bekeken te
worden om een goede interpretatie te geven van het resultaat (zie 4 Resultaten en discussie).
34
2.4.1 Primers en probes
Tijdens de PCR-reactie zijn forward en reverse primers nodig zodat amplificatie kan
plaatsvinden. Primers zijn kleine enkelstrengige DNA-fragmenten die complementair zijn aan
de 3’- en 5’-uiteinden van het gewenste DNA-fragment, waarbij de forward primer bindt aan
de anti-sense12-streng van het template-DNA en de reverse primer aan de sense13-streng van
het template-DNA. Om die primers synthetisch aan te maken, moet de gensequentie van de
flankerende regio’s van het te amplificeren DNA-fragment gekend zijn en moet er zo goed
mogelijk voldaan worden aan de volgende voorwaarden (Pestana et al.,2010):
-
De primers moeten specifiek zijn voor de gewenste doelwitsequentie en er een stabiele
duplex mee vormen. Primer Blast NCBI werd gebruikt om de specificiteit van alle
primers na te gaan waarbij geen enkele bekende sequentie in de databank meer dan 98
% Query Coverage en Max Identity mag vertonen om kruisreactie te voorkomen.
-
Als de primer te kort is, stijgt de kans dat de primer bindt op verschillende sequenties
wat nadelig is voor de specificiteit. Is de primer echter te lang, is dit ten voordele van
de specificiteit maar nadelig voor de PCR-reactie, aangezien er zich secundaire
structuren kunnen voordoen in de primersequentie waardoor de primer niet meer kan
binden met de gewenste DNA-sequentie. Een goede primerlengte die vaak wordt
gebruikt is tussen de 18 en 24 baseparen lang maar dit wordt hoofdzakelijk bepaald
door de smelttemperatuur ervan.
-
De smelttemperatuur14 voor de forward en reverse primer moet ongeveer gelijk zijn en
ligt meestal tussen 52 °C en 58 °C, afhankelijk van de lengte van de primer (tijdens
het onderzoek is dit 60 °C). Secundaire structuren zoals hairpins, self dimers en cross
dimers moeten minimaal zijn. Om deze parameters na te gaan, werd gebruikgemaakt
van drie gelijkaardige programma’s: IDT OligoAnalyzer, uMELT en SeqMan.
-
De verhouding van de baseparen, AT=GC, moet evenredig zijn en verspreid liggen ten
opzichte van elkaar. Hierbij worden repeats vermeden daar deze mispriming
veroorzaken. Het GC % ligt best tussen de 40 % en 60 % voor een optimale PCRreactie. Meer dan drie G’s of C’s worden het best vermeden in de laatste vijf basen aan
het 3’-uiteinde van de primer, omdat deze zogenaamde GC clamp formation ervoor zal
zorgen dat de primer niet volledig kan dissociëren van het template-DNA.
12
Afleesrichting komt niet overeen met het mRNA, de complementaire streng wel.
Afleesrichting komt overeen met het mRNA.
14
Smelttemperatuur is de temperatuur waarbij 50 % van de strengen gedissocieerd zijn.
13
35
-
De baseparing aan het 3’-uiteinde van de primer is cruciaal, aangezien het DNApolymerase hier moet aanhechten en de verlenging hier start. Bij slechte
complementariteit zal het DNA-polymerase de ketenverlenging niet starten.
-
Ook de primerconcentratie moet in acht worden genomen. Wanneer deze te hoog is,
zal de kans op mispriming stijgen waardoor er zich aspecifieke amplificatie zal
voordoen. Als de concentratie te laag is, zal dit inefficiënte PCR-reacties veroorzaken.
De gebruikte probes die ook worden aangehaald in punt 3.1.4 zijn net als de primers
sequentiespecifiek en worden gesynthetiseerd op basis van de sequenties die worden
afgebakend door de geselecteerde forward en reverse primers. Ook de probes moeten aan
bepaalde voorwaarden voldoen vooraleer ze gebruikt kunnen worden in een PCR-reactie
(Pestana et al.,2010):
-
De probe bestaat gemiddeld uit een 20 à 30-tal baseparen.
-
De smelttemperatuur moet een tiental graden Celcius hoger liggen dan de
smelttemperatuur van de primers.
-
De GC-verhouding ligt tussen de 30 à 80 %.
-
De probe wordt zo dicht mogelijk bij de primer geplaatst zonder ze te overlappen om
zo vlugger een detectiesignaal te genereren.
-
De verhouding van de baseparen, AT=GC, moet evenredig zijn en verspreid liggen ten
opzichte van elkaar.
-
Probedimeervorming, mismatchen tussen probe en target en G op het 5’-uiteinde moet
vermeden worden.
36
2.4.2 Detectie via non-specific labels
De meest gebruikte fluorescerende kleurstof voor niet-specifieke kwantitatieve detectie is
SYBR Green. Dit is een asymmetrische cyanine kleurstof, die fluorescent wordt als het
intercaleert met DNA. Het resulterende DNA-kleurstofcomplex absorbeert blauw licht bij een
golflengte van λmax = 497 nm en zendt groen licht uit met een golflengte λmax = 520 nm
(Vanderstraeten,2010) (zie figuur 9).
Figuur 9: Spectrum en moleculaire structuur van SYBR Green (SYBR Green, 2013)
Bij voorkeur zal SYBR Green intercaleren in de kleine groeve van dsDNA. Het kan ook
reageren met enkelstrengig DNA of RNA maar dat zal in veel mindere mate gebeuren en ook
een veel lagere fluorescentie weergeven.
Als SYBR Green intercaleert met dsDNA, zal de intensiteit van de fluorescente emissie
verhogen. Als er tijdens de PCR-reactie meer dsDNA wordt gevormd, zal het signaal dat
SYBR Green veroorzaakt ook verhogen. Op die manier kan een kwantitatieve bepaling
plaatsvinden.
Het nadeel van deze methode is dat SYBR Green zal intercaleren met eender welke
dubbelstrengige DNA-molecule. Dus aspecifieke amplificatie en primerdimeren worden ook
gedetecteerd wat een vertekend signaal teruggeeft. Om de specificiteit van het amplicon te
controleren, wordt vaak gebruikgemaakt van een smeltcurveanalyse na PCR-reactie bij deze
detectiemethode. Dit is een kinetische analyse van de thermische denaturatie van
dubbelstrengig DNA tot complementaire enkelstrengige DNA-moleculen door
temperatuursverhoging.
37
Naarmate de temperatuur stijgt, zal het dubbelstrengig DNA meer dissociëren wat leidt tot
een daling van de fluorescentie door vrijkomen van de SYBR Green-moleculen (SYBR Green
zal een factor 1000 meer fluoresceren bij intercalleren aan dubbelstrengig DNA). Op basis
hiervan wordt het denaturatieproces gevolgd. De denaturatiecurve verloopt sigmoïdaal (zie
figuur 10) en wordt gekarakteriseerd door het smeltpunt, Tm, van de dubbelstrengige DNAmolecule. De Tm-waarde wordt bepaald door de thermische stabiliteit van het dubbelstrengig
DNA die afhangt van en rechtstreeks evenredig is met het GC %. Het smeltpunt is geen
traditioneel smeltpunt zoals dat van ijs, maar het is hier de temperatuur waarbij 50 % van het
DNA gedenatureerd is (Van Gool,1997).
Figuur 10: Denaturatiecurve waarbij fluorescentie daalt in functie van de temperatuur (Van Gool,1997)
Bij een uniplex PCR-reactie zal ieder amplicon dezelfde Tm-waarde hebben afhankelijk van
zijn grootte en basesamenstelling. Afhankelijk van de intensiteit en de bijhorende
smelttemperatuur kan dan bepaald worden of het gewenste amplicon werd gedetecteerd en of
er al dan niet contaminatie of primerdimeren aanwezig zijn. Bij een multiplex PCR-reactie zal
er een verscheidenheid zijn aan amplicons waarbij de smeltcurveanalyse kan worden gebruikt
ter differentiatie (zie figuur 11). Een goed signaal is een unieke welgedefinieerde piek op de
smeltcurve, waarbij de afgeleide van de fluorescentie wordt uitgezet in functie van de
temperatuur. Op de linker grafiek is de fluorescentie uitgezet in functie van de temperatuur,
op de rechter grafiek is de afgeleide van de fluorescentie uitgezet in functie van de
temperatuur waarbij de typische smeltpiek van ieder organisme wordt bekomen.
38
Figuur 11: Smeltcurveanalyse van organisme A, B en combinatie organisme A en B
(naar MeltingCurveAnalysis,2013)
De informatie bekomen door smeltcurveanalyse kan ook gebruikt worden om single
nucleotide polymorphisms (SNP)15 op te sporen. DNA met een hoger GC % door een SNP zal
een hogere smelttemperatuur hebben dan DNA met een hoger AT % (zie figuur 12). Om
SNP’s op te sporen wordt gebruikgemaakt van High Resolution Melting (HRM) wat eigenlijk
een smeltcurveanalyse is met zeer hoge gevoeligheid (door de trage temperatuursstijging
tijdens de analyse) en waarbij het proces in real-time wordt gevolgd.
Met die gevoelige techniek worden ook mutaties en epigenetische verschillen in
dubbelstrengig DNA opgespoord en kan onderscheid worden gemaakt tussen homozygote
wildtype allelen, heterozygote wildtype allelen en homozygote mutante allelen.
Figuur 12: Smeltcurveanalyse waarbij SNP wordt aangetoond (HighResolutionMelt,2014)
15
SNP is een variatie in de DNA-sequentie wanneer één nucleotide in het genoom verschillend is ten opzichte
van de andere gelijke species of van het gepaarde chromosoom.
39
SYBR Green is aanwezig in de SYBR Select MasterMix die gebruikt wordt tijdens het
onderzoek en hierin zijn volgende componenten aanwezig (AB,2013):
-
SYBR Green
-
DNA-polymerase met hot start-mechanisme
-
UDG (Uracil-DNA glycosylase) of UNG (Uracil-N-glycosylase)
-
ROX kleurstof passieve referentie
-
dNTP blend met dUTP/dTTP
-
geoptimaliseerde buffercomponenten
Het DNA-polymerase is aanwezig in een inactieve toestand wat flexibiliteit toelaat in de
reactie-opzet. Zo is het vooraf mengen van PCR-reagentia mogelijk op kamertemperatuur
zonder kans op vroegtijdige start van de PCR-reactie. Het DNA-polymerase wordt
gereactiveerd na twee minuten bij 95 °C.
Het hitte labiel UNG zal kruiscontaminatie met reeds geamplificeerd DNA minimaliseren
alvorens de PCR-reactie start.
Tijdens de PCR-reactie worden dUTP’s als bouwsteen gebruikt in plaats van dTTP’s. De
amplicons zijn dus opgebouwd uit de basen A, G, C en U. De uracil glycosidebinding in de
amplicons wordt herkend door UNG, waarbij UNG de glycosidische binding zal verbreken en
de base vervolgens wordt verwijderd. Als een PCR-reactie wordt opgestart, zal het templateDNA aanwezig zijn alsook UNG die in de mastermix aanwezig is. Alvorens de PCR-reactie
start, zal een eerste stap plaatsvinden bij een lagere temperatuur van 50 °C, zodat UNG
werkzaam is om eventuele aanwezige amplicons van voorgaande PCR-reacties af te breken.
De amplicons worden gesplitst in kleinere stukken en kunnen niet meer geamplificeerd
worden doordat ze nu te klein zijn voor binding met de primers. Het template-DNA blijft
intact, want het bevat geen dUTP ingebouwde bouwstenen. Ook eventueel aanwezig RNA zal
niet afgebroken worden, want RNA bevat als bouwsteen UTP in plaats van dUTP.
Na deze stap zal de reactie opgewarmd worden naar 95 °C om het polymerase te activeren en
het UNG te inactiveren.
Als interne referentie tijdens de Real-Time PCR-reactie is de ROX kleurstof passieve
referentie aanwezig in de mastermix. Die interne referentie wordt gebruikt om de
reporterkleurstof (SYBR Green) te normaliseren tijdens data-analyse, wat noodzakelijk is om
fluorescentie-fluctuaties door veranderingen in concentratie of volume door pipetteerfouten te
corrigeren.
40
2.4.3 Detectie via sequence specific probes
Een probe is een oligonucleotide die een quenching molecule (= quencher) bevat op het 3’uiteinde en/of een fluorofoor (= reporter) op het 5’-uiteinde. Een fluorofoor is een molecule
die licht van een bepaalde golflengte emitteert na absorptie van licht met een specifieke maar
kortere golflengte waarbij de emissiegolflengte altijd hoger is dan de absorptiegolflengte. Een
quencher is een molecule die de energie van de fluorofoor grotendeels zal capteren waardoor
fluorescentie van de fluorofoor zal worden onderdrukt. Dit proces gaat door wanneer de
quencher en de reporter zich in elkaars nabijheid bevinden en wordt proximal quenching
genoemd. Een fluorofoor kan gecombineerd worden met verscheidene quenchers, maar het
absorptiespectrum van de quencher moet een goede overlap hebben met het emissiespectrum
van de fluorofoor om een optimale quenching te bekomen.
Wanneer de quencher de opgenomen energie omzet in warmte (zie figuur 13), zal er geen
fluorescentie plaatsvinden door de quencher waardoor een beter signal-to-noise verhouding
wordt bekomen. Er wordt gesproken van een dark quencher of black hole quencher welke
tijdens dit onderzoek gebruikt worden (Vandesompele,2009).
Figuur 13: Proximal quenching principe (Vandesompele,2009)
De opgenomen energie door de quencher kan ook geëmitteerd worden als fluorescentie van
een hogere golflengte (zie figuur 14). Het effect wordt fluorescence resonance energy transfer
genoemd (FRET) en er wordt gesproken van FRET quenching in dit geval
(Vandesompele,2009).
Figuur 14: FRET quenching principe (Vandesompele,2009)
41
Twee soorten probes worden onderscheiden namelijk hydrolysis probes en hybridization
probes.
Hybridization probes zijn twee probes die, aangrenzend aan elkaar, binden aan de
targetsequentie. De ene heeft een 3’-uiteinde gelabeld met een reporter terwijl de tweede
probe een 5’-uiteinde heeft gelabeld met een quencher. Wanneer de probes niet gebonden zijn
aan de targetsequentie, zal het fluorescentiesignaal van de reporterkleurstof gedetecteerd
worden bij een bepaalde golflengte. Als de probes echter beide hybridizeren met de
targetsequentie tijdens de annealingsfase in de PCR-reactie, bevinden de fluoroforen van
beide probes zich op korte afstand van elkaar wat energietransfer tussen reporter en quencher
toelaat (zie figuur 15). De quencher zal op zijn beurt licht emitteren wat als
fluorescentiesignaal wordt gedetecteerd bij een bepaalde golflengte afhankelijk van de
quencher (Vandesompele,2009;Teo et al.,2002).
Figuur 15: Principe hybridization probes (LightCycler Real-time PCR,2014)
Bij hydrolysis probes wordt gebruikgemaakt van één enkelstrengig molecule complementair
aan de targetsequentie. Een vaak gebruikte probe is de TaqMan probe of double-dye probe
genoemd. Aan het 5’-uiteinde bevindt zich de reporter (=fluorofoor) en aan het 3’-uiteinde
bevindt zich de quencher. Bij excitatie van de reporter wordt de energie via FRET
doorgegeven naar de quencher en erdoor gecapteerd waarbij als reporter vaak FAM (= 6carboxyfluoresceïne) wordt gebruikt en als quencher TAMRA (= tetramethylrhodamine) of
BHQ (= black hole quencher). Tijdens de annealingsfase van de PCR-reactie zal de probe
binden aan de targetsequentie. De fluorescentie van TAMRA wordt gedetecteerd met een
verschillende golflengte als van FAM waardoor het achtergrondsignaal van FAM laag zal
zijn. Wanneer het DNA-polymerase begint met de elongatiefase, zal het de probe vanaf het
5’-uiteinde wegduwen en afbreken met zijn 5’-3’-exonucleaseactiviteit. De probe wordt
gehydrolyseerd waardoor de reporter en quencher zich niet meer in elkaars nabijheid
bevinden en de fluorescentie van de reporter niet meer wordt gecapteerd door de quencher
(zie figuur 16). Dit leidt tot een irreversibele verhoging in fluorescentie van FAM en een
verlaging van het TAMRA-signaal.
42
De probes worden zo dicht mogelijk bij de primers geplaatst waardoor fluorescentie vrijwel
onmiddellijk plaatsvindt als de elongatie start op de bewuste strengen. Naast TAMRA wordt
nu voornamelijk een BHQ gebruikt. Hierbij zal de gecapteerde fluorescentie omgezet worden
naar warmte en wordt er geen signaal gedetecteerd. Bij hydrolyse van de probe zal enkel het
fluorescentiesignaal van de reporter irreversibel verhogen en zal het achtergrondsignaal
minimaal zijn (Vandesompele,2009).
Figuur 16: Hydrolyse TaqMan-probe door hitte stabiel DNA-polymerase (Asuragen,2012)
Tijdens het onderzoek wordt gebruikgemaakt van een TaqManMGB-probe, wat een variant is
van de TaqMan-probe. Bij die probe is er op het 3’-uiteinde een Minor Groove Binder (MGB)
toegevoegd die de smelttemperatuur van de probe zal stabiliseren (verhogen) waardoor een
kortere probe kan worden gebruikt (zie figuur 17).
43
Die MGB is een chemische modificatie die specifiek bindt in de minor groove van
dubbelstrengig DNA waardoor de binding zeer specifiek is en een hoge affiniteit heeft
(Vandesompele,2009).
Dat maakt de probe beter geschikt om een single nucleotide polymorfhism (SNP) te detecteren
en het zal de specificiteit sterk doen toenemen. De probes zijn hierdoor zeer geschikt voor
zowel kwalitatieve als kwantitatieve detectie.
Figuur 17: Hydrolyse TaqMan MGB-probe door hitte stabiel DNA-polymerase (Kutyavin et al.,2000)
In de TaqMan Fast Virus 1-step MasterMix gebruikt tijdens het onderzoek bevinden zich
volgende componenten (AB,2011):
-
AmpliTaq® Fast DNA Polymerase;
-
thermostabiel MMLV enzyme (= reverse transcriptase);
-
dNTP’s (dATP, dGTP, dCTP en dTTP);
-
RNaseOUT™ Recombinant Ribonuclease Inhibitor;
-
geoptimaliseerde buffercomponenten voor maximale gevoeligheid. Deze oplossing
blijft vloeibaar bij -20 °C opslagtemperatuur.
44
RNaseOUT™ Recombinant Ribonuclease Inhibitor is een niet-competitieve inhibitor en
wordt gebruikt om RNA-degradatie door RNase A, RNase B en RNase C te vermijden. Het is
van toepassing bij cDNA-synthese, Real-Time PCR en in vitro transcriptie en translatie
(LifeTechnologies,2014).
2.4.4 ViiaTM7 Real-Time PCR system
In het lab wordt er gebruikgemaakt van het ViiaTM7 Real-Time PCR system (Viia7 genoemd)
(AB,2011). Dit toestel maakt gebruik van fluorescente reagentia tijdens de PCR-reactie met
volgende toepassingen:
-
kwantitatieve detectie van nucleïnezuursequenties via Real-Time analyse;
-
kwalitatieve detectie van de targets via post-PCR-analyse (= eindpuntanalyse);
-
kwalitatieve analyse van de PCR-producten via smeltcurve-analyse die post-PCR
plaatsvindt.
Het toestel verzamelt ruwe fluorescente data gedurende verschillende tijdstippen tijdens de
PCR-reactie (=data collection points) welke afhankelijk zijn van het type run die wordt
uitgevoerd (zie tabel 2).
Tabel 2: Run-type met data collecion point (AB,2011)
Run type
Standaardcurve
Real-time
runs
Relatieve standaardcurve
Comparatieve CT
Smeltcurve
Post-PCR
runs
(eindpunt)
Genotypering
Aanwezigheid/afwezigheid
Data collection point
Data worden verzameld
tijdens iedere stap van de
PCR
1. Voor de PCR
2. Tijdens de PCR
3. Na de PCR
Ongeacht het type run zal een data collection point bestaan uit drie fases:
-
Excitatie: Alle wells van de reactieplaat in het toestel worden belicht waardoor de
fluoroforen in iedere aanwezige reactie zullen exciteren.
-
Emissie: Het toestel bevat een optisch gedeelte dat de residuele fluorescentie vanuit de
wells van de reactieplaat verzamelt.
45
-
Collectie: Er wordt een digitale representatie geassembleerd van de verzamelde
fluorescentie over een bepaald tijdsinterval wat dan kan worden geanalyseerd via de
aanwezige software van het toestel.
Het principe kan voorgesteld worden zoals op figuur 18.
Figuur 18: Werkingsprincipe van de ViiaTM7 Real-Time PCR-system (Nollet,2013)
Het licht van een halogeenlamp wordt door een excitatiefilter gestuurd en door een
dichroïsche beam splitter gericht op de PCR-reactieplaat die op zijn plaats wordt gehouden
door de thermische blok van het systeem. Die blok wordt omgeven door optische elementen
om het fluorescente licht te collecteren, het licht afkomstig van plaatsen verschillend van de
wells te onderdrukken en om verdamping en condensatie tijdens de PCR-reactie te
voorkomen. Het excitatielicht zal de aanwezige fluorescerende kleurstoffen in iedere
gebruikte reactiewell doen exciteren waardoor emissie zal plaatsvinden. Dit geëmitteerde
fluorescentielicht wordt dan door een serie optische lenzen en een corresponderende
emissiefilter gestuurd waarna het wordt gedetecteerd door een charge coupled device camera
(CCD-camera). De bekomen gegevens worden doorgestuurd naar de pc ter verwerking
(Nollet,2013).
46
2.5
Gelelektroforese
Onder elektroforese verstaat men het migreren van een deeltje met een bepaalde lading in een
viskeus midden onder invloed van een elektrisch veld. Als het viskeus midden een gel is,
spreekt men van gelelektroforese wat een scheidingstechniek is waarmee nucleïnezuren en
eiwitten bijvoorbeeld op grootte worden gescheiden. Nucleïnezuren dragen een negatieve
lading (bij neutrale pH) door de ionisatie van de fosfaatgroep. Ieder nucleotide levert een
bijdrage waardoor de scheiding afhankelijk is van het aantal ladingen en dus van de grootte
van het fragment.
De twee meest gebruikte gelen zijn polyacrylamidegelen en agarosegelen.
Polyacrylamidegelen zijn vooral bruikbaar voor kleinere DNA- of eiwitfragmenten door hun
kleinere poriegrootte maar worden vaak vervangen door agarosegelen omwille van de
toxiciteit en de cancerogeniciteit van de bestanddelen om een polyacrylamidegel te maken.
Voor grotere fragmenten, zoals DNA-moleculen, wordt gebruikgemaakt van agarosegelen die
een groter scheidingsgebied hebben. De negatieve DNA-fragmenten zullen naargelang hun
grootte migreren met een bepaalde snelheid van de negatieve pool naar de positieve pool
waarbij de kleinste fragmenten het snelst migreren. Door gebruik te maken van
baseparenladders kan de grootte van die DNA-fragmenten ingeschat worden.
Om de gescheiden nucleïnezuren zichtbaar te maken, wordt een intercalerend fluorochroom
toegevoegd voor of na de elektroforese waardoor het bandenpatroon na de elektroforese
zichtbaar wordt, wat zich leent tot kwalitatieve identificatie.
47
2.6
TaqMan Low Density Array Card
Een TaqMan Low Density Array Card, of kort gezegd TAC, is een 384 wells reactieplaat
opgedeeld in 8 zones die per staal 48 individuele uniplex Real-Time PCR-analyses mogelijk
maakt (zie figuur 19). Hierdoor kunnen verscheidene pathogenen en controles simultaan
geanalyseerd worden (Kodani et al.,2011). Analyses gebaseerd op PCR hebben doorgaans het
nadeel dat bij elke verandering van een bestaande primer of bij nieuwe primers en probes, de
volledige assay moet gerevalueerd worden. Of als er nieuwe informatie beschikbaar is in
verband met de gensequentie van de bewuste pathogenen die geanalyseerd worden, kan het
zijn dat de gebruikte primers en probes moeten aangepast of vervangen worden. Dit is een
duur en moeilijk proces. TAC biedt daarvoor een oplossing. Het is namelijk een gesloten
systeem met geïntegreerde uniplex Real-Time PCR wat de flexibiliteit geeft om nieuwe
primers en probes toe te voegen zonder dat de andere geïntegreerde primers en probes dienen
gerevalueerd te worden. Een ander voordeel is dat de primers en probes vooraf toegewezen
zijn aan de wells door middel van lyofyliseren, waardoor het staal maar één keer moet
toegevoegd worden samen met bijhorende mastermix. Het monster zal zich dan verspreiden
over die wells, ook wel spiked samples genoemd. Dit alles zal de gevoeligheid van die
analyse verhogen en een specifieke simultane Real-Time PCR-analyse mogelijk maken.
Figuur 19: Taqman Low Density Array Card (Steg et al.,2006;Zheng et al.,2006)
48
2.7
BD ProbeTec
De BD ProbeTecTM ET geamplificeerde DNA-analysen maken een directe, kwalitatieve
detectie mogelijk van Chlamydia trachomatis en Neisseria gonorrhoeae (CT/GC) DNA in
endocervicale en mannelijke urethrale uitstrijkjes en mannelijke en vrouwelijke urinemonsters
die worden afgenomen bij symptomatische of asymptomatische patiënten.
De techniek maakt gebruik van de homogene Strand Displacement Amplification (SDA)technologie voor een directe en kwalitatieve detectie waarbij fluorescente energietransfer
wordt gebruikt om de aanwezigheid van Chlamydia trachomatis en Neisseria gonorrhoeae te
detecteren. Het BD ProbeTec ET-instrument detecteert iedere minuut over een tijdsduur van
60 minuten fluorescente signalen (ruwe data) van de microwells waarin de reacties
plaatsvinden. Het signaal wordt gerelateerd aan de hoeveelheid amplificatieproduct in de
microwell. Een verhoging in het signaal wijst op een verhoging van de hoeveelheid
amplificatieproduct en wijst dus op een positieve reactie. Een negatieve reactie zal geen
amplificatieproduct teweegbrengen en dus zal er geen verandering in signaal plaatsvinden.
De aan- of afwezigheid van CT en/of GC wordt bij deze analyse bepaald aan de hand van de
MOTA16-score. Dit is een beoordeling voor de omvang van het bekomen signaal en wordt
gerelateerd aan vooraf gedetermineerde cut off values om een besluit te nemen over het al dan
niet aanwezig zijn van het pathogeen (Skovgaard et al.,2012)
16
Method Other Than Acceleration
49
3
Materiaal en methoden
3.1
Materiaal
3.1.1 Vircell controle DNA
Gezuiverd microbieel DNA van vijf verschillende organismen met gekende concentratie
(uitgedrukt als kopieën genomisch DNA/µl) werd aangekocht bij de firma Vircell (zie tabel
3). Aan de hand van dat referentiemateriaal werd tijdens de optimalisatie van de verschillende
uniplex PCR’s de functionaliteit van de primers en probes, de gevoeligheid en eventuele
kruisreactie tussen de verschillende detectiemethoden uitgetest. Alsook werd de limit of
detection (LOD) tijdens de validatie van de TaqMan Low Density Array Card (TAC) met dat
referentiemateriaal bepaald.
50
Tabel 3: Gegevens Vircell controle DNA
Amplirun Mycoplasma genitalium DNA control
LOT: 12MBC085002
1x Vircell MGE DNA control
1x 500 µl Vircell control reconstitution solution
Concentration (copies/µl)
LOT: 12MBC085102
LOT: 12MBCR003
14500
Amplirun Neisseria gonorrhoeae DNA control
LOT: 12MBC075002
1x Vircell NGO DNA control
LOT: 12MBC075102
1x 500 µl Vircell control reconstitution solution
Concentration (copies/µl)
LOT: 12MBCR003
12920
Amplirun Haemophilus ducreyi DNA control
LOT: 12MBC021001
1x Vircell HDU DNA control
1x 500 µl Vircell control reconstitution solution
Concentration (copies/µl)
LOT: 12MBC021101
LOT: 12MBCRS002
12670
Amplirun Ureaplasma urealyticum DNA control
LOT: 13MBC112001
1x Vircell UUR DNA control
1x 500 µl Vircell control reconstitution solution
Concentration (copies/µl)
LOT: 13MBC112101
LOT: 12MBCRS003
15230
Amplirun Herpes Simplex Virus II DNA control
LOT: 12MBC024002
1x Vircell HSV2 DNA control
1x 500 µl Vircell control reconstitution solution
Concentration (copies/µl)
LOT: 12MBC024102
LOT: 12MBCRS003
19300
51
3.1.2 Bacteriële culturen
Voor het testen van de specificiteit werd gebruikgemaakt van DNA geïsoleerd uit 42
verschillende bacteriële culturen, namelijk: Staphylococcus aureus, Salmonella enteritidis,
Salmonella groep C, beta hemolytische streptococcen groep C, Streptococcus pneumoniae,
Campylobacter jejuni, Shigella sonnei, Klebsiella pneumoniae, Citrobacter koseri,
Salmonella groep B, Sphingomonas paucimobilis, Enterococcus faecalis, Morganella
morganii, Actinomyces israellii, Escherichia coli, Yersinia enterocolitica, Clostridium
difficile, Shigella boydi, Candida tropicalis, Bacteriodes fragilis, Enterococcus faecium,
Neisseria meningitidis, Salmonella groep D, vancomycine resistente enterococcen,
Streptococcus agalactiae, Vibrio, Campylobacter coli, Enterobacter cloacae, Klebsiella
oxytoca, Enterobacter aerogenes, Proteus vulgaris, Streptococcus oralis, Mycobacterium
smegmatis, Serratia liquefaciens, Serratia liquefaciens, Serratia marcescens, Citrobacter
freundii, Shigella flexneri, Staphylococcus epidermidis, Lactobacillus paracasei,
Streptococcus pneumoniae (zie bijlage 1 “Bacteriële stammen (negatief referentiemateriaal)”).
3.1.3 Klinische monsters
Naast het Vircell controle DNA werd als positief referentiemateriaal gebruikgemaakt van een
reeks klinische monsters (vaginale, cervicale, rectale en urethra wissers, urines, peritoneum,
ulcus, anaal secreet en biopt) correct geïdentificeerd door middel van een referentietechniek.
Chlamydia en/of gonorroe positieve monsters werden bekomen via het AZ Sint-Jan BruggeOostende AV17. Positieve monsters voor Ureaplasme urealyticum, Ureaplasma parvum,
Neisseria gonorrhoeae, Mycoplasma genitalium, Chlamydia trachomatis, Treponema
pallidum en Trichomonas vaginalis werden bekomen via het Sint-Augustinus Ziekenhuis
Antwerpen, Az Damiaan Oostende, Labo Microbiologie Universiteit Brussel en het Instituut
voor Tropische Geneeskunde Antwerpen (zie bijlage 2 “klinische monsters (positief
referentiemateriaal)”). Deze monsters werden gebruikt voor het uittesten van de efficiëntie, de
herhaalbaarheid, reproduceerbaarheid en kruisreactie van de verschillende PCR-reacties.
17
Identificatie aan de hand van de BD Probetec-methode.
52
3.1.4 Primers en probes
De gegevens van de geselecteerde primers voor de specifieke detectie van de verschillende
organismen zijn terug te vinden in tabel 4. De primers zijn aangekocht bij de firma IDT
(Integrated DNA Technologies) aan een concentratie van 100 µM.
Tabel 4: Gegevens geselecteerde primers
Naam
Tm
MW (Da)
Forward
Reversed
304 NGPorA1 Fw
305 NGPorA1 Rv
55,2 °C
56,6 °C
6716,4
5803,8
Primerpaar 2
Forward
Reversed
307 NGPorA2 Fw
308 NGPorA2 Rv
54,0 °C
55,2 °C
6700,4
6716,4
Primerpaar 3
Forward
Reversed
310 NGOmp Fw
311 NGOmp Rv
54,6 °C
56,5 °C
7342,8
7394,9
Primerpaar 4
Forward
Reversed
313 NGOpaD Fw
314 NGOpaD Rv
57,7 °C
55,6 °C
5477,6
6994,6
Herpes Simplex Virus II
Primerpaar 1
(totaal)
Forward
Reversed
249 HSV tot1 Fw
250 HSV tot1 Rv
59,2 °C
60,2 °C
7058,6
6486,3
Primerpaar 2
(II)
Forward
Reversed
258 HSV2#1 Fw
259 HSV2#1 Rv
61,6 °C
59,4 °C
7129,7
6100
Primerpaar 3
(Routine)
Forward
Reversed
015 HSV2 Fw
016 HSV2 Rv
55,8 °C
55,2 °C
5741,8
5803,8
Forward
Reversed
261 HDp27 Fw
262 HDp27 Rv
53,6 °C
52,2 °C
7129,7
7016,6
Forward
Reversed
264 HDhdA Fw
265 HDhdA Rv
54,9 °C
53,6 °C
6814,5
7882,2
Forward
282 MG pdhD Fw
54,9 °C
8251,4
Neisseria gonorrhoeae
Primerpaar 1
Haemophilus ducreyi
Primerpaar 1
Primerpaar 2
Mycoplasma genitalium
Primerpaar 1
53
Primerpaar 2
Ureaplasma urealyticum
Primerpaar 1
Primerpaar 2
Haemophilus ducreyi
Primerpaar 3
Primerpaar 4
Ureaplasma parvum
Primerpaar 1
Reversed
283 MG pdhD Rv
55,6 °C
7745,1
Forward
Reversed
285 MGMgPa Fw
286 MGMgPa Rv
55,9 °C
54,1 °C
7417,9
10076,6
Forward
Reversed
288 UUureD Fw
289 UUureD Rv
61,6 °C
55,1 °C
6167
7956,3
Forward
Reversed
291 UUure Fw
292 Uuure Rv
52,4 °C
54,9 °C
7888,2
7318,8
Forward
Reversed
264 HDHdA Fw
265 HDHdA Rv
54,9 °C
53,6 °C
6814,5
7888,2
Forward
Reversed
319 Hdtox Fw
320 Hdtox Rv
55,2 °C
56,2 °C
7641,1
7736,1
Forward
Reversed
326 UP Fw
327 UP Rv
54,1 °C
55,1 °C
7120,7
7956,3
54
De gegevens van de geselecteerde MGB-probes zijn terug te vinden in tabel 5. De probes zijn
aangekocht bij de firma Thermo Fisher (voormalig Life Technologies) aan een concentratie
van 100 µM.
Tabel 5: Gegevens geselecteerde probes
Naam
MW (Da)
Neisseria gonorrhoeae
p306_NGPorA1 MGB
p315_NGOpaD MGB
6559
7348
p018,2_HSV2 MGB
p251_HSV tot MGB
7461
6583
p263_HDp27 Rev MGB
7106
p287_MGMgPa MGB
p284_MGpdhD MGB
7180
8483
p290_UUureD MGB
p293_UUure MGB
6810
6896
p328_UP MGB
6788
p315.2_NGOpaD MGB
6740
Herpes Simplex Virus II
Haemophilus ducreyi
Mycoplasma genitalium
Ureaplasma urealyticum
Ureaplasma parvum
Neisseria gonorrhoeae
55
3.2
Extractie van nucleïnezuren
3.2.1 Werkwijze
Er wordt gebruikgemaakt van twee extractieprotocollen op het QiaSymphony-toestel:
pathogen complex 400 en pathogen complex 800. Dit houdt in dat er een volume van 400 µl
respectievelijk 800 µl staal wordt geëxtraheerd en geëlueerd in een vooraf bepaald volume
van 60 µl, 85 µl of 110 µl. Om rekening te houden met het doodvolume van het toestel, wordt
een staalvolume van 550 µl, respectievelijk 1000 µl aangeboden aan het toestel.
Tijdens de extractie wordt een interne controle toegevoegd aan ieder staal, namelijk SHV ( =
Seal Herpesvirus, Phocine Herpes virus 1). SHV is een DNA-virus zonder
sequentiegelijkenissen met het menselijk genoom, menselijke virussen of andere pathogenen
die bij mensen voorkomen en dient ter controle voor een correcte DNA-extractie en eventuele
inhibitie van het staal tijdens amplificatie.
Voorbereiding staal:
Stap 1:
Homogeniseren van het staal.
Stap 2:
Afhankelijk van het protocol wordt 550 µl of 1000 µl staal in een 2 ml tube
gepipetteerd.
Stap 3:
2 ml tube zonder dop wordt in het elutie(thermo)blok geplaatst waarin het
eluaat wordt opgevangen na extractie. Dat blok wordt op de voorziene plaats
gezet in de QIA Symphony waarna die ter controle wordt gescand.
Stap 4:
De tube met het staal wordt in het rek gestoken waarna het rek in de QIA
Symphony wordt opgeladen. Ook het rek met de interne controles wordt
opgeladen. Hierna worden de parameters; zoals staalplaats, soort tube…
ingelezen of handmatig ingevuld en het gewenste protocol wordt aangeduid. Na
het instellen worden alle verbruiksgoederen en reagentia gescand om na te gaan
of alles nog in voldoende mate aanwezig is en of alles juist geplaatst is.
Stap 5:
Start de extractie.
56
3.3
Real-Time PCR Gene Expression Assays
Drie gene expression assays werden besteld bij de firma Life Technologies (Cat no
Hs04260456_s1, Hs04260457_s1, Hs04260458_s1) voor de specifieke amplificatie van
humaan genomisch DNA (AB,2010).
In iedere tube bevinden zich volgende elementen:
-
twee ongelabelde primers (1x finale concentratie van 900 nM per primer uit 20x
stockconcentratie van 18 µM per primer)
-
één 6-FAM™ dye-labeled TaqMan® MGB probe (1x finale concentratie van 250 nM
uit 20x stockconcentratie van 5 µM)
-
één 6-VIC® dye-labeled TaqMan MGB probe (1x concentratie van 250 nM uit 20x
stockconcentratie van 5 µM)
3.3.1 Werkwijze
Stap 1:
Extraheer het staal, zodat het kan gebruikt worden om een PCR-reactie uit te
voeren.
Stap 2:
Pipetteer het volgende in een microcentrifuge tube van 1,5 ml:
PCR-reactie mix component
Volume voor één reactie (µl)
20x TaqMan® Gene Expression Assay
4x TaqMan Fast Virus 1-step MasterMix
RNase vrij water
1,0
5,0
9,0
Stap 3:
Inverteer de tube een aantal keer om te homogeniseren.
Stap 4:
Kort afcentrifugeren (short spin).
Stap 5:
Breng 15 µl in de PCR-reactieplaat en voeg 5 µl DNA-template (geëxtraheerd
staal) toe.
Stap 6:
Sluit de plaat af.
Stap 7:
Vortex kort de plaat, centrifugeer deze af en plaats de plaat in het toestel.
Stap 8:
Start de PCR-reactie (Fast template).
Uit deze drie assays zal de gevoeligste worden uitgekozen om op de TAC-STD array card te
plaatsen als staalcontrole. Hiernaast zullen nog twee targetgenen gebruikt worden voor de
staalcontrole, namelijk RNaseP en 18S-Hs99999901_s1.
57
3.4
Real-Time PCR
3.4.1 Werkwijze Real-Time PCR met SYBR Select MasterMix en smeltcurveanalyse
De primers worden bewaard bij -30 °C en dienen eerst ontdooid te worden voor gebruik. De
mastermix wordt bewaard bij 4 °C en is klaar voor gebruik.
Samenstelling SYBR Select MasterMix:
-
SYBR Green
-
DNA-polymerase met hot start-mechanisme
-
UDG (Uracil-DNA glycosylase) of UNG (Uracil-N-glycosylase)
-
ROX kleurstof passieve referentie
-
dNTP mengsel met dUTP/dTTP
-
geoptimaliseerde buffercomponenten
Werkwijze:
Stap 1:
De primers worden verdund van 100 µM naar 20 pmol/µl: 20 µl primer + 80 µl
H2O (RNase vrij).
Stap 2:
De mix met SYBR Select MasterMix wordt gemaakt in een Sarstedt-tube en bestaat
voor één PCR-reactie uit:
Reagentia
Hoeveelheid (µl)
Concentratie in oplossing
Forward primer (20 pmol/µl)
0,30
300 nM
Reverse primer (20 pmol/µl)
0,30
300 nM
SYBR Select MasterMix 2x
10
1x
H2O (RNase vrij)
4,4
Stap 3:
Maak deze mix voor het gewenste aantal reacties + 1.
Stap 4:
Homogeniseer de mix door de Sarstedt-tube te inverteren of kort te vortexen.
Stap 5:
Centrifugeer de tube (short spin), zodat het volledige volume onderaan de tube zit.
Stap 6:
Verdeel de mix uit in de reactieplaat of een strip per 15 µl.
Stap 7:
Voeg aan iedere well 5µl van het gewenste DNA toe van het geëxtraheerde staal
(extractieprotocol complex 400).
Stap 8:
Sluit de reactieplaat of de strips goed af, vortex kort en voer een short spin uit.
Stap 9:
De reactieplaat of wells worden nu ingezet in het ViiaTM7 Real-Time PCR system
met een vooropgestelde template die de cyclus uit figuur 20 doorloopt.
58
Figuur 20: Temperatuurstemplate PCR en smeltcurveanalyse
3.4.2 Werkwijze Real-Time PCR met TaqMan Fast Virus 1-step MasterMix
De primers, probes en mastermix worden bewaard bij -30 °C en dienen eerst ontdooid te
worden voor gebruik. Die mastermix is 4x geconcentreerd en is zeer viskeus, de tube moet 2
tot 3x geïnverteerd worden om een homogene oplossing te bekomen na ontdooien.
Samenstelling TaqMan Fast Virus 1-step MasterMix:
-
AmpliTaq® Fast DNA-Polymerase
-
thermostabiel MMLV enzyme (= reverse transcriptase)
-
dNTP’s (dATP, dGTP, dCTP en dTTP)
-
RNaseOUT™ Recombinant Ribonuclease Inhibitor
geoptimaliseerde buffercomponenten voor maximale gevoeligheid
-
Werkwijze:
Stap 1:
De probes worden verdund van 100 µM naar 20 pmol/µl: 5 µl probe + 20 µl H2O
(RNase vrij).
Stap 2:
De mix met de TaqMan Fast Virus 1-step MasterMix (4x) wordt gemaakt in een
Sarstedt-tube en bestaat voor één PCR-reactie uit:
Reagentia
Forward primer (100 µM/ml)
Reverse primer (100 µM/ml)
Probe (20 pmol/µl)
H2O (RNase vrij)
Taqman Fast Virus 1-step
MasterMix (4x)
Hoeveelheid (µl)
Concentratie in oplossing
0,2
0,2
0,25
4,4
5
1000 nM
1000 nM
250 nM
1x
Stap 3:
Maak deze mix voor het gewenste aantal reacties + 1.
Stap 4:
Homogeniseer de mix door de Sarstedt-tube te inverteren of kort te vortexen.
59
Stap 5:
Centrifugeer de tube (short spin), zodat het volledige volume onderaan de tube zit.
Stap 6:
Verdeel de mix uit in de reactieplaat per 10 µl.
Stap 7:
Voeg aan iedere well 10 µl van het gewenste DNA toe van het geëxtraheerde staal
(extractieprotocol complex 400).
Stap 8:
Sluit de reactieplaat goed af, vortex kort en voer een short spin uit.
Stap 9:
De reactieplaat of wells worden nu ingezet in het ViiaTM7 Real-Time PCR system
met een vooropgestelde template die de cyclus uit figuur 21 doorloopt.
Bij iedere plaat dient ook een positieve controle en negatieve controle / blanco meegenomen
te worden per targetorganisme.
Figuur 21: Temperatuurstemplate uniplex PCR-reactie
3.4.3 Werkwijze Real-Time PCR TAC-STD met Fast Virus 1-step MasterMix
De Fast Virus 1-step MasterMix wordt bewaard bij -30 °C en dient ontdooid te worden voor
gebruik. Deze mastermix is 4x geconcentreerd en zeer viskeus. De tube moet 2 tot 3x
geïnverteerd worden om een homogene oplossing te bekomen na ontdooien.
60
Stap 1:
Haal een TAC-STD array card uit de koelkast en laat deze aanpassen aan de
kamertemperatuur.
Stap 2:
De TaqMan Fast Virus 1-step MasterMix wordt uitverdeeld per 26 µl in een strip
van 8 wells.
Stap 3:
Aan iedere well wordt 78 µl van het gewenste geëxtraheerde staal toegevoegd
(extractie protocolcomplex 800).
Stap 4:
Pipetteer 5x op en neer om de oplossingen te homogeniseren.
Stap 5:
Uit iedere well wordt 100 µl geladen op de TAC-STD array card.
Stap 6:
Centrifugeer de kaart 2x 2 minuten aan 1200 rpm (= 314 rcf) en seal hierna de
kaart.
Stap 7:
De slots aan de bovenkant van de TAC worden afgeknipt.
Stap 8:
Breng de kaart in de array houder van het ViiaTM7 Real-Time PCR system en
start de PCR-reactie met de vooraf opgestelde STD-template die de cyclus uit
figuur 22 doorloopt.
Het gepipetteerde volume in de TAC-STD array card verdeelt zich over alle laantjes en de 48
wells. In iedere well zal er één µl aanwezig zijn waarmee de PCR-reactie zal plaatsvinden.
Het resterend volume bevindt zich in de verbindingslaantjes.
Figuur 22: Temperatuurstemplate TAC-STD array card PCR-reactie
61
3.5
Gelelektroforese
3.5.1 Werkwijze
DNA-fragmenten (amplicons met een grootte van 90-120 bp) bekomen na PCR-reactie
worden gescheiden op een 3 % agarosegel.
Bereiding gel:
Stap 1:
Plak de randen van de gelhouder goed af met plakband, zodat het gel niet kan
lekken en plaats het kammetje in de houder.
Stap 2:
Breng 3,0 gram agarose in 100 ml 0,5x TBE-buffer (Tris/Boraat/EDTA18) en
verwarm dit in de microgolfoven tot de vloeistof net niet kookt.
Stap 3:
Meng goed en kijk of alle agarose is opgelost. Er mag geen vaste agarose meer
aanwezig zijn in de vloeistof.
Stap 4:
Laat licht afkoelen in een warmwaterbad tot de erlenmeyer waarin de vloeistof
zich bevindt, kan worden vastgehouden.
Stap 5:
Breng 10 µl SybrSafe in het vloeibare gel en meng.
Stap 6:
Giet het gel vanuit één hoek in de afgeplakte houder om een homogene gel te
bekomen.
Stap 7:
Laat het gel hard worden (30 min – 1 uur).
Stap 8:
Verwijder het plakband en het kammetje en plaats de houder met het gel in het
elektroforesetoestel.
Stap 9:
Voeg TBE-buffer toe in het elektroforesetoestel tot het gel volledig onderstaat.
Voorbereiding en laden staal:
Stap 1:
Meng 10 µl van ieder staal met 3 µl laadbuffer (= broomfenol loading dye) en
homogeniseer goed.
Stap 2:
Meng 3 µl 100bp-ladder met 3 µl laadbuffer en homogeniseer goed.
Stap 3:
Laad de stalen en de basepaarladder op het gel.
18
ethyleendiaminetetra-azijnzuur
62
Elektroforese starten:
De negatieve pool van het toestel wordt aangesloten op de kant van de geladen stalen en de
positieve pool op de andere zijde. Het voltage wordt ingesteld op 100 mVolt en de tijd op 30
minuten waarna de spanning wordt aangelegd. Tijdens de elektroforese moet tijdig
gecontroleerd worden of de (gekleurde) laadbuffer niet van het gel migreert. Als de stalen na
30 minuten ver genoeg gemigreerd zijn, wordt de spanning afgezet en kan het gel
geïnterpreteerd worden onder uv-licht. Aan de hand van de basepaarladder op het gel kan de
grootte van de gemigreerde stalen geschat worden. Hiervan wordt een foto genomen en de
opname wordt gedigitaliseerd.
63
3.6
Primer/probeselectie en optimalisatie uniplex PCR
Per organisme zijn targetgenen geselecteerd om specifieke primers en probes te ontwikkelen.
De behandelde organismen tijdens dit onderzoek zijn in tabel 6 in vet gedrukt.
Tabel 6: Targetgenen per organisme
Organisme
Afkorting
Target
Herpes simplex virus
HSV TOTAL DNA-polymerasegen
HSV1
glycoproteïne G-gen
glycoproteïne D-gen
HSV2
Haemophilus ducreyi
HD
27 kDa H. ducreyi-specific protein
Mycoplasma genitalium
MG
dihydrolipoamide dehydrogenase
major surface protein MgPa
Ureaplasma urealyticum
UU
ureD subunit van het urease gen
Ureaplasme parvum
UP
ureD subunit van het urease gen
Neisseria gonorrhoeae
NG
porine pseudogen
outer membrane protein
opacity gene
Treponema pallidum
TP
47 kDa membrane lipoprotein
DNA polymerase I gene
Trichomonas vaginalis
TV
2-kb repetitief sequentie
beta-tubulin protein.
Mycoplasma hominis
MH
membrane protein translocase
elongation factor Tu (EF-TU)
Chlamydia trachomatis
CT
cryptische plasmide
16S
outer membrane protein
polymorphic membrane protein H gene
64
Na selectie van de targetgenen, worden tijdens de optimalisatie de volgende onderdelen
doorlopen:
Deel 1: Uittesten van de geselecteerde primers (zie 3.1.4 tabel 4) met SYBR Select
MasterMix in uniplex formaat (detectie via niet-selectief label, geen probe aanwezig).
-
De functionaliteit en specificiteit van de primers worden met het Vircell controle DNA
(zie 3.1.1 tabel 3) als referentiemateriaal getest.
-
10-voudige verdunningsreeksen van de verschillende vircellcontroles worden
opgesteld. Minstens drie verdunningen in de reeks moeten aanleiding geven tot een
positieve reactie. Per pathogeen worden de bekomen Ct-waarden per primerpaar
uitgezet ten opzichte van de concentratie van het inputmateriaal in een grafiek waaruit
de efficiëntie en gevoeligheid per primerpaar kan worden afgeleid.
-
De smeltcurves van de bekomen resultaten worden opgesteld en vergeleken.
-
Gelelektroforese kan toegepast worden op het geamplificeerde materiaal ter
confirmatie of bij afwijkende smeltcurves.
-
De specificiteit van de primerparen wordt getest met DNA van bacteriële stammen die
niet positief zijn voor de geselecteerde pathogenen (0,5 en 1 MF suspensies, zie 3.1.2).
Als kruisreactie voorkomt, wordt dit verder onderzocht.
-
De primers worden eventueel aangepast en de meest geschikte primerparen worden
geselecteerd om te testen in combinatie met de respectievelijke probes.
Deel 2: Uittesten van de geselecteerde primers en probes met de TaqMan Fast Virus 1-step
MasterMix (detectie met sequentie-specifieke probe).
-
De geselecteerde primers en probes (zie 3.1.4 tabel 4 en 5) worden getest met TaqMan
Fast Virus 1-step MasterMix in combinatie met de verschillende vircellcontroles in
uniplex PCR-reacties.
-
De specificiteit van de primers en probes wordt getest met dezelfde bacteriële
stammen als in deel 1. De dubbelzinnige resultaten hiervan uit deel 1 worden hier
opnieuw getest. Ook positieve stalen vanuit verschillende labs worden getest en de
resultaten worden vergeleken.
Deel 3: de Gene expression (GEx) assay, besproken in 3.3.
-
De assay om genomisch humaan DNA op te sporen wordt getest. De PCR-reacties
worden ingezet met en zonder de reverse transcriptase stap om eventueel verschil
tussen beide PCR-reacties aan te tonen.
65
3.7
Validatie TaqMan Low Density Array Card
3.7.1 Matrices
Soa’s kunnen opgespoord worden in verschillende matrices of een staalname kan gebeuren
via een (urogenitale) wisser die al of niet in een transportmedium naar het lab gebracht wordt.
Tijdens de validatie van de TAC-STD array card wordt de invloed van volgende matrices op
de Real-Time PCR-reacties uitgetest:
19
-
1x TE-buffer (pH 8): Dit is een medium waarin getransporteerde urogenitale wissers
kunnen uitgedrukt worden. De 100x stockoplossing bestaat uit 1M Tris (=
(hydroxymethyl)aminomethaan), wat een pH-buffer is, en 0,1M EDTA
(ethyleendiaminetetra-azijnzuur), wat een chelerend agens is. De TE-buffer wordt
verdund tot 1x TE-buffer met moleculair grade water wat een finale samenstelling
geeft van 0,01M Tris en 0,001 EDTA.
-
E-swab (= Elution Swab): Dit is een collectie- en transportmedium voor afname van
klinische specimens die aerobe en anaerobe bacteriën bevatten. Dit medium bestaat uit
anorganische fosfaatbuffer, natriumchloride, kaliumchloride, calciumchloride,
magnesiumchloride, monokaliumfosfaat, dinatriumfosfaat, natriumthioglycollaat en
gedestilleerd water.
-
Preserve Cyt: Dit is een gebufferd methanol gebaseerd transportmedium waarin
cervicale wissers voor de detectie van HPV (humaan papillomavirus) en soa’s
ingebracht worden.
-
UTM (= Universal transport medium): Dit is een collectie- en transportmedium voor
virussen, Chlamydia, Mycoplasma en Ureaplasma. Het bestaat uit een gemodificeerde
Hank’s gebalanceerde zoutoplossing waaraan BSA (= bovin serum albumin), cysteïne,
gelatine, sucrose en glutaminezuur is toegevoegd. De pH wordt gebufferd met HEPES
(= 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid) buffer en fenolrood wordt
gebruikt voor pH-indicatie. Vancomycine, amphotericine B en colistine zijn
geïncorporeerd in het medium om groei van competitieve bacteriën en gisten te
inhiberen. De aanwezigheid van sucrose fungeert als een cryoprotectant 19 wat helpt bij
het bewaren als het staal in dit medium wordt ingevroren.
-
First voided urine: Dit is de eerste ochtendurine.
Bescherming tegen beschadiging bij bevriezing
66
-
Weefselbiopt (chronische ulcera, klierbiopt, huidbiopt, placenta…): Er wordt
gebruikgemaakt van de gentleMACS-dissociator (Miltenyi,2014) om een celsuspensie
te bekomen van placenta. Voor dit toestel zijn specifieke tubes ontworpen, namelijk
gentleMACS™ C-Tubes, die voor een zachte en efficiënte dissociatie van het weefsel
zorgen waardoor een celsuspensie wordt verkregen. Placenta wordt versneden in
kleine stukken, waarna het in de gentleMACS™ C-Tubes wordt overgebracht (tussen
20 – 4000 mg weefsel, afhankelijk van de weefselconsistentie en het gentleMACSprotocol). Er wordt een fosfaatbuffer pH 6,8 toegevoegd tot ongeveer 10 ml, wat het
maximaal volume is dat aanwezig mag zijn in de tube. De tubes worden op de
gentleMACS-dissociator geplaatst om de stukjes placenta te versnijden waardoor de
cellen zullen loskomen in de fosfaatbuffer. De celsuspensie (fosfaatbuffer + cellen)
wordt hierna overgebracht naar een propere tube en een kleine hoeveelheid hiervan
wordt naar de Unicel DxH 800 (Beckman Coulter) gebracht, waar een celtelling
uitgevoerd wordt. Afhankelijk van de telling wordt de celsuspensie doorverdund tot
ongeveer 1.000.000 cellen/ml.
-
Serum: De vloeistof die overblijft als bloed stolt en wordt afgecentrifugeerd, is serum.
De samenstelling is vergelijkbaar met bloedplasma maar bij serum zijn de
coagulatiefactoren (stollingseiwitten, zoals fibrogeen, kunnen interfereren met de
metingen) grotendeels verwijderd. Dit wordt verkregen van de dienst serologie in het
AZ Sint-Jan en wordt samengebracht in één volume, zodat er één samenstelling wordt
verkregen van het serum.
-
Vol bloed: werd niet getest in dit eindwerk.
Er wordt nagegaan of alle matrices negatief zijn voor alle te testen pathogenen. Ze worden
getest met de uniplex PCR-reacties alvorens te valideren op de TAC-STD array card. TEbuffer, E-swab, Preserve Cyt en UTM zijn steriele transportmedia die niet op voorhand
moeten getest worden. Het first voided urine, het serum en de placenta worden wel getest
omdat deze media niet noodzakelijk steriel zijn en er kans is dat de organismen die we
wensen te spiken hierin al aanwezig zijn. Dit zorgt voor vals-positief resultaat tijdens de
validatie en moet vermeden worden.
Als de te testen media negatief zijn, worden deze uitverdeeld per 900 µl in Sarstedt-tubes van
1,5 ml om mengstaal mix 1 en mengstaal mix 2 (zie 3.7.3 tabel 12) hierin te spiken. Die stalen
worden geëxtraheerd met het Complex 800 protocol en getest op de TAC-STD array card om
het matrix geïnduceerd effect na te gaan.
67
3.7.2 Bepaling aanvaardbare performantiekenmerken
De performantiekenmerken worden als volgt gedefinieerd:
Detectielimiet (= analytische sensitiviteit)
“Limit of detection (LOD) is de laagste concentratie of hoeveelheid van een analiet waar meer
dan 95 % van de tests positieve resultaten geven na seriële verdunningen van internationaal
referentiemateriaal, gekalibreerd referentiemateriaal of een monster (Raymaekers et
al.,2011).”
De LOD wordt bepaald op basis van:
-
verdunningen om en bij de LOD, tien maal herhalen over minstens vier verschillende
runs.
-
(verdunningsreeksen in iedere matrix)
-
(QCMD-gegevens)
Het Vircell controle DNA worden samengevoegd volgens de samenstelling in tabel 10 en 11
(zie 3.7.3) en hiervan worden de verdunningsreeksen gemaakt als volgt:
De Vircellcontroles werden doorverdund tot 200 kopieën/µl per organisme. Volgens de
werkwijze van de TAC-STD array card (3.4.3) wordt er 78 µl staal gebruikt dus zullen er
(200x78) 15600 kopieën aanwezig zijn. Die 78 µl wordt gehomogeniseerd met 26 µl
mastermix en 100 µl hiervan wordt aangebracht op de TAC-STD array card. In ieder cupje
van de TAC-STD array card kan 1 µl dus zullen er in ieder cupje ongeveer 156 kopieën
aanwezig zijn. We verdunnen door naar 100 kopieën/µl, 50 kopieën/µl, 25 kopieën/µl, 4
kopieën/µl en 2 kopieën/µl:
Stock
mix1-1
mix1-2
mix1-3
mix1-4
mix1-5
200
15600
156
100
7800
78
50
3900
39
25
1950
19,5
4
312
3,12
2
156
1,56
Het totale volume van iedere verdunning is 2 ml. De gebruikte volumes vertrekkende vanuit
de stockoplossing om de verdunningen te bekomen zijn de volgende:
mix1-1
volume mix 1000
volume H2O 1000
mix1-2
mix1-3
mix1-4
mix1-5
500
1500
250
1750
20
1980
10
1990
68
Het totale volume van de stockoplossing die gebruikt wordt, is 1780 µl. Met deze waarde kan
dan het aantal kopieën per ml staal berekend worden (kopieën in 78 µl x 1,78 ml):
Stock
mix 1-1 mix 1-2 mix 1-3 mix 1-4 mix 1-5
27768
13884
6942
3471
555,36
277,68
Op deze berekeningen zit een fout door de veralgemening van 200 kopieën per µl. In tabel 10
en 11 uit 3.7.3 zien we dat dit niet overal exact 200 kopieën zijn maar deze veralgemening is
een goede benadering waarmee zal worden verder gewerkt.
De precisie
“De precisie is de mate van overeenstemming van de individuele testresultaten binnen een
enkele run (intra-assay precision of herhaalbaarheid) en vanuit de ene run naar de andere
(inter-assay precision of reproduceerbaarheid). Dit wordt gekenmerkt door de
standaardafwijking van de metingen en de correlatiecoëfficiënt (Raymaekers et al.,2011).”
De herhaalbaarheid (zie tabel 7) is een maat voor de spreiding van meetwaarden die onder
dezelfde omstandigheden worden uitgevoerd. Daaronder verstaat men: door dezelfde persoon,
met dezelfde methode, met dezelfde meetapparatuur en hulpstoffen en binnen een kort
tijdsbestek (één dag). Binnen dit kort tijdsbestek worden de zes stalen van mengstaal mix 1 en
mengstaal mix 2 (zie 3.7.3 tabel 12) in TE-buffer driemaal geëxtraheerd waarna de drie
extracten worden samengevoegd per verdunning. Iedere verdunning wordt driemaal getest
met de TAC-STD array card binnen dezelfde dag. De resultaten van de herhaalbaarheid
moeten voldoen aan %CV20 < 5 %.
De reproduceerbaarheidsmetingen (zie tabel 7) worden onder verschillende omstandigheden
uitgevoerd, dit wil zeggen door verschillende personen of met verschillende meetapparaten,
verspreid over een langere periode (dag twee, drie en vier). Het restant van het extract uit dag
één wordt één keer getest met de TAC-STD array card op dag twee. Iedere verdunning van
mengstaal mix 1 en mengstaal mix 2 (zie 3.7.3 tabel 12) wordt bijkomend geëxtraheerd en
getest op dag drie en vier. De resultaten van de reproduceerbaarheid moeten voldoen aan
%CV < 10 %.
Matrix geïnduceerd effect
De drie sterkste verdunningen van mengstaal mix 1 en mengstaal mix 2 (zie 3.7.3 tabel 12),
dit zijn de drie laagste concentraties die nog positief resulteren, worden herhaald in de
verschillende matrices en dit in drievoud.
20
Variatiecoefficiënt
69
Dat gebeurt door de verdunningsreeks in TE-buffer 1 op 10 door te verdunnen in de
respectievelijke matrix (zie tabel 7), deze te extraheren en te detecteren in drievoud met de
TAC-STD array card. Onder ideale omstandigheden is er geen effect van de matrix op de
LOD.
Tabel 7: Overzicht planning precisie in verschillende matrices
Lineariteit
“De lineariteit wordt gedefinieerd als de bepaling van het lineaire bereik van de
kwantificering. Dit wordt gekenmerkt door de regressiecoëfficiënt na lineaire regressie
(Raymaekers et al.,2011).”
De lineariteit wordt geverifieerd door een seriële dilutiereeks van een positief staal te
analyseren (10-voudige dilutiereeks met minstens 3 dilutiestappen) in duplicaat. De resultaten
worden onderworpen aan lineaire regressie-analyse met een ideale regressiecoëfficiënt van
één.
De resultaten moeten voldoen aan:
Regressiecoëfficiënt R2 > 0,95 over de 4 diluties
Efficiëntie van de PCR 0,9 ≤ E ≤ 1,1 ( -3,3 ≤ slope ≤ -3,6)
Analytische specificiteit (interferentie studie)
“De analytische specificiteit wordt gedefinieerd als het vermogen van de methode om
negatieve resultaten te verkrijgen in concordantie met negatief resultaat verkregen door de
referentiemethode (Raymaekers et al.,2011).”
70
De resultaten voor de analytische specificiteit moeten voldoen aan volgende voorwaarden:
-
98 % concordantie met het referentiemateriaal (alle negatieven zijn negatief).
-
Geen kruisreactiviteit in de monsters met andere micro-organismen die simultaan in
hetzelfde monster aanwezig kunnen zijn.
-
Geen enkel bekende sequentie in de NCBI databank mag 100 % “Query Coverage” en
“Max Identity” vertonen voor de primers en probes.
Om kruisreactie te testen, wordt op de TAC-STD array card ieder positief monster getest voor
alle parameters waaruit onderlinge kruisreactie binnen het soa-panel moet volgen. Minstens
twintig soa-negatieve monsters, mogelijke kruisreactieve stalen21, moeten ook negatief testen.
Nauw verwante species en kiemen die urogenitaal frequent voorkomen, worden ook getest op
eventuele kruisreactie.
De bacteriële stammen die hiervoor worden gebruikt, worden samengebracht in vijf
verschillende mixen (zie tabel 8) die per mix worden geëxtraheerd en getest op de TAC-STD
array card.
De samenstelling is als volgt:
Tabel 8: Samenstelling mixen kruisreactie
Staalnummer
Identificatie
Mix
137542621
137540619
137540321
137541520
137542508
140009719
140009922
140009142
Salmonella enteritidis
Salmonella groep C
beta hemolytische strep groep C (dysgalactiae)
Streptococcus pneumoniae
Campylobacter jejuni
Shigella sonnei
Klebsiella pneumoniae
Citrobacter koseri
Mix 1
Mix 1
Mix 1
Mix 1
Mix 1
Mix 1
Mix 1
Mix 1
142024517
142023128
142025608
144303918
146335567
148854813
148854329
Salmonella groep B
Sphingomonas paucimobilis
Enterococcus faecalis
Morganella morganii
Actinomyces israellii
Escherichia coli
Yersinia enterocolitica
Mix 2
Mix 2
Mix 2
Mix 2
Mix 2
Mix 2
Mix 2
21
Stalen die positief testen voor virussen van dezelfde familie, stalen die geënt zijn met potentieel kruisreactief
referentiemateriaal of stalen die besmet zijn met pathogenen die dezelfde kliniek veroorzaken en dus aanwezig
kunnen zijn in het te analyseren monster.
71
148855303
Clostridium difficile
Mix 2
153309325
154088428
155497115
155499908
168719417
171096216
169238519
171316411
Shigella boydi
Candida tropicalis
Bacteriodes fragilis
Enterococcus faecium
Salmonella groep D
Vancomycine-resistente enterococcen
Streptococcus agalactiae
Vibrio (genus)
Mix 3
Mix 3
Mix 3
Mix 3
Mix 3
Mix 3
Mix 3
Mix 3
166524807
165591714
167261314
Campylobacter coli
Enterobacter cloacae
Klebsiella oxytoca
Mix 4
Mix 4
Mix 4
168467805
173788113
161891616
161889320
168998315
Enterobacter aerogenes
Proteus vulgaris
Streptococcus oralis
Mycobacterium smegmatis
Serratia liquefaciens
Mix 4
Mix 4
Mix 4
Mix 4
Mix 4
171048015
154464116
163596024
160094218
137427714
Serratia marcescens
Citrobacter freundi
Shigella flexneri
Staphylococcus epidermidis
Lactobacillus paracasei
Mix 5
Mix 5
Mix 5
Mix 5
Mix 5
176632005
176515619
Streptococcus pneumoniae
Streptococcus pneumoniae
Mix 5
Mix 5
Mogelijke kruisreactieve stalen (zie tabel 9) die apart werden getest op de TAC-STD array
card zijn de volgende:
Tabel 9: Mogelijks kruisreactieve stalen
Staalnummer
Identificatie
137542222
144304812
158225915
158139610
173047716
168718012
ATCC 13077
Staphylococcus aureus
Staphylococcus aureus
Neisseria meningitidis
Neisseria meningitidis
Neisseria meningitidis
Neisseria meningitidis
Neisseria meningitidis
72
Juistheid
“De juistheid wordt gedefinieerd als de mate van overeenstemming van een gemeten of
berekende hoeveelheid tot zijn werkelijke waarde en kan worden geschat door analyses van
referentiematerialen of vergelijkingen van de resultaten met die bekomen van een
referentiemethode. Dit wordt gekenmerkt door het percentage van overeenstemming met de
referentiemethode (Raymaekers et al.,2011).”
De minimale vereisten zijn hier het testen van 20 stalen (7 positieve, 6 laag positieve en 7
negatieve) in parallel met de TAC-STD array card. Bijkomende validatie-experimenten zijn
noodzakelijk om het mogelijke matrix-geïnduceerd effect na te gaan. Op zijn minst moeten 9
monsters (3 positieve, 3 laag positieve en 3 negatieve) getest worden voor ieder bijkomende
matrix (zie 3.7.1).
Bij goede resultaten voor juistheid moeten alle kwaliteitscontroles correct gedetecteerd
worden en alle stalen die positief zijn met de referentietechniek, moeten ook positief zijn met
de TAC-STD array card. De gebruikte kwaliteitscontrole, QCMD 2014 Neisseria
gonorrhoeae DNA EQA programme A, bestaat uit 10 gevriesdroogde stalen, 6 urines en 4
swabs met verschillende concentraties aan N. gonorrhoeae of negatieve stalen. De
gevriesdroogde stalen werden gereconstitueerd door toevoeging van 4 ml molecular grade
water en getest met de huidige routinetest BD Probetec CT/NG test en de TAC-STD array
card.
Bewaarcondities monstermateriaal
De verdunningsreeks van mengstaal mix 1 en mengstaal mix 2 (zie 3.7.3 tabel 12) wordt
opgemaakt in de verschillende matrices en die worden bewaard gedurende verschillende
dagen onder verschillende condities:
Van iedere verdunningsreeks worden per matrix en per mix vier reeksen opgemaakt. De
eerste reeks wordt getest op dag 0 (zie matrix geïnduceerd effect). De overige drie reeksen
worden vier dagen bewaard op kamertemperatuur (25 °C) om staaltransport na te bootsen. Op
dag vier wordt de eerste reeks geëxtraheerd en getest met de TAC-STD array card. Reeks 2 en
3 worden nu vier bijkomende dagen bewaard in de koelkast (4 °C). Op de achtste dag wordt
reeks 2 geëxtraheerd en getest, reeks 3 wordt nu zeven bijkomende dagen bewaard in de
diepvries (-30 °C). Op dag 15 wordt reeks 3 ontdooid, geëxtraheerd en getest.
De bedoeling hiervan is het nabootsen van de transportcondities en de bewaartijden in het lab.
Onder ideale omstandigheden zou er geen achteruitgang van het monstermateriaal mogen
plaatsvinden.
Bewaarcondities reagentia
Niet van toepassing, bewaarcondities van de firma worden overgenomen.
73
Vergelijking van methoden
Alle resultaten bekomen met de referentiemethode moeten ook positief zijn met de te
valideren methode: CT/NG BD ProbeTec vs TAC-STD array card.
Om dit na te gaan, worden volgende testen uitgevoerd:
-
De verdunningsreeksen van mengstaal mix 1 en mengstaal mix 2 in TE-buffer worden
getest met de BD-methode.
-
Minimum 10 NG stalen die positief zijn met de BD-methode worden hertest met de
TAC-STD array card.
-
Minimum 20 stalen die negatief zijn met de BD-methode worden hertest met de TACSTD array card.
3.7.3 Praktische voorbereiding
Aankoop Vircell controle DNA
Tien verschillende vircellcontroles werden aangekocht met gekende DNA-concentratie. Deze
zijn weergegeven in tabel 3 onder punt 3.1.1.
Opmaak van Vircell controle DNA mixen
Om de detectielimiet te bepalen van alle individuele Real-Time PCR-reacties worden 2 mixen
gemaakt van verschillende Vircell controle DNA’s (samenstelling zie tabel 10 en 11). Per mix
worden vijf verschillende vircellcontroles verdund en samengevoegd, zodat er in de finale
mix nog ongeveer 200 kopieën per µl van ieder organisme aanwezig zijn. De benodigde
volumes voor het opmaken van deze mixen worden weergegeven in tabellen 10 en 11.
Tabel 10: Samenstelling en opmaak Vircell controle DNA mix 1
Organisme
Kopieën/µl
Verdunnings-
Volume Vircell
Volume H2O
Kopieën/µl
Kopieën/µl
Stock
factor
(µl)
(µl)
na verdunning
in mix
HSV II
19300
19
40
720
1015
203,158
HD
12670
12,5
40
460
1013
202,72
MG
14500
14,5
40
540
1000
200
UU
15230
15
40
560
1015
203,066
NG
12920
12,5
40
460
1033
206,72
74
Tabel 11:Samenstelling en opmaak Vircell controle DNA mix 2
Organisme
Kopieën/µl
Verdunnings-
Volume Vircell
Volume H2O
Kopieën/µl
Kopieën/µl
Stock
factor
(µl)
(µl)
na verdunning
in mix
TVA
10000
10
40
360
1000
200
CT
12680
12,5
40
460
1014
202,88
MHO
17100
17
40
640
1005
201,176
HSV I
12117
12
40
440
1009
201,95
300
1323
66,15
TPA
1323
/
22
100
Aankoop QCMD-kwaliteitscontroles (voor zover beschikbaar)
NgDNA14A
CTDNA14A
HSVDNA14
NgDNA14B
CTDNA14B
Scheduled: FEB 2014
Scheduled: FEB 2014
Scheduled: MAR 2014
Scheduled: JUN 2014
Scheduled: JUN 2014
Verzamelen positieve klinische stalen
Klinische stalen die sterk positief zijn voor de verschillende organismen worden verzameld.
Er wordt nagegaan of het staal positief is voor één enkel organisme met de verschillende
uniplex PCR-reacties en de lading ervan wordt ingeschat aan de hand van de Ct-waarde.
Stalen met een Ct-waarde < 25 komen in aanmerking.
Van de verzamelde stalen die in aanmerking komen, worden mengstalen (zie tabel 12) en
verdunningsreeksen opgemaakt in verschillende matrices voor de evaluatie van het extractieen amplificatieproces.
22
Ander volume doordat veel minder kopieën per µl aanwezig zijn in het aangekochte Vircell controle DNA.
75
Tabel 12: Samenstelling mengstaal mix 1 en mengstaal mix 2
Organisme
Mix 1
HSV I (klinisch staal)
HSV II (kwaliteitscontrole)
Mix 2
X
X
(HD)
MG (kweek)
X
UU (kweek en klinisch staal)
X
UP (kweek en klinisch staal)
X
NG (kweek)
X
(TP)
TV (klinisch staal)
X
MH (kweek)
X
CT (klinisch staal)
X
HD en TP zijn niet aanwezig in de mixen doordat sterk positief klinische stalen niet
beschikbaar waren.
76
4
Resultaten en discussie
4.1
Selectie primers en probes
Vals positieve resultaten voor de detectie van Neisseria gonorrhoeae door bepaalde
commerciële testen (COBAS AMPLICOR (NG/CT) Test (Roche Diagnostics)) zijn een
significant probleem, vooral bij de STD-screening in relatief laag prevalente populaties. De
lage positief predictieve waarde van de gebruikte test maakt het noodzakelijk alle positieve
resultaten te confirmeren door een ander, meer specifieke test. Daarom werd voor deze studie
het principe aangenomen om voor alle te detecteren pathogenen simultaan meerdere targets
per pathogeen (minstens twee) op te sporen, wat de specificiteit en de positief predictieve
waarden van de test verhoogt en tevens minder vatbaar maakt voor het vals negatief
rapporteren van varianten die kunnen optreden in de natuur.
De resultaten van de literatuurstudie beschreven in de bachelorproef was de basis voor het
selecteren van minstens twee species specifieke targetgenen en in voorkomend geval ook het
selecteren van de reeds beschreven primers en probes. De sequenties en details van de
hieronder geselecteerde primers en probes en de wijzigingen ten opzichte van de sequenties
beschreven in de literatuur worden niet weergegeven wegens confidentialiteit.
4.1.1 Herpes Simplex Virus II
Voor HSV II was er reeds een gevalideerde multiplex voorhanden in het lab op basis van de
sequenties beschreven door van Doornum et al (JCM 2003, 41;576-580) gelegen in het
glycoproteïne D-gen. Na NCBI genbankanalyse werd zowel in de forward als in de reversed
primers een degeneratie toegevoegd om alle varianten op te pikken. De beschreven FAMTamra probe werd aan het 3’-uiteinde ingekort en omgevormd tot een MGB-probe.
Verder werden niet gepubliceerde sequenties bekomen via Dr. M. Curran (Addenbrooks
Hospital, Cambridges University, UK) voor het specifiek opsporen van HSV II en het
opsporen van HSV zonder onderscheid te maken tussen zowel HSV I als HSV II. Deze
sequenties zijn gelegen in het HSV DNA-polymerasegen.
4.1.2 Haemophilus ducreyi
Uit de literatuur bleek dat vooral het 16S rRNA-gen, het p27-gen en het hemolytic
cyrtotoxine-gen de meest voorkomende doelwitgenen zijn voor de specifieke detectie van
HD. De Primers en probes beschreven door Orle et al (JCM 1996) werden aangepast door
Suntoke et al (STI 2009) en amplificeren een 439 bp-fragment.
77
Deze PCR werd niet weerhouden daar de meest efficiënte Real-Time PCR-reactie een zo kort
mogelijk fragment amplificeert. Op basis van de hemi-nested PCR beschreven door TekleMichael et al (2001) die specifiek het p27-gen detecteert, werden eigen primers en probe
ontwikkeld met behulp van het Primer Express programma (Life Technologies). Bijkomende
sequenties die specifiek het hemolytic cytotoxin (HhdA) gen detecteren, werden bekomen via
het PHA reference lab (Cambridge), initieel gebaseerd op een CDC23 protocol.
4.1.3 Mycoplasma genitalium
Het MgPa (major surface protein MgPa)-gen en het 16S rRNA-gen zijn de meest gebruikte
doelwitsequenties in MG-qPCR. Echter wegens het gebrek aan specificiteit met de 16S rRNA
primers ontwikkelde Müller et al. (2012) een kwantitatieve Real-Time Rotor-Gene PCR
(qPCR) assay met het pdhD gene van MG als target, die codeert voor het dihydrolipoamide
dehydrogenase. De beschreven probe werd terug omgevormd tot een MGB-probe. Als
alternatief targetgen werd gekozen voor MgPa-target daar dit in vele studies gebruikt werd,
Müller et al. (2012) beschreef echter een zeker varaibiliteit in het MgPa-gen. Door het
invoeren van een degeneratie in de primers wordt deze variabiliteit grotendeels opgevangen.
4.1.4 Ureaplasma urealyticum en Ureaplasma parvum
Van Cutsem et al (J Mol Diag 2011) beschreef een gemodifieerde PCR voor de detectie en
differentiatie van UU en UP, gebaseerd op de sequenties gepubliceerd door Xuan Cao et al
(Diag Microb Infect Dis 2007) gelegen in de ureD subunit van het ureasegen van UP en UU.
Een alternatief targetgen, buiten het ureasegen, werd niet gevonden. K. Mallard, et al (J
Microbiol Meth 2005) beschreef ook een Real-Time PCR ter differentiatie van UU en UP
gelegen in het UreB subunit van het ureasegen, deze diende als basis voor de tweede target
voor de detectie van UU en UP.
4.1.5 Neisseria gonorrhoeae
NG vertoont nauwe verwantschap met N. meningitidis en andere commensale Neisseria
species (N. subflava, N. cinerea, N. lactamica…). Een multitarget aanpak is hier dan ook
uiterst belangrijk.
Hjelmevoll (2012) heeft zijn PhD studie gewijd aan het specifiek detecteren van NG wat
resulteerde in meerdere publicaties waaruit bleek dat het opsporen van het pseudogen (PorA)
aanleiding gaf tot de meest specifieke en efficiëntste PCR.
23
CDC = Center for Disease Control and Prevention
78
Een ander veelgebruikt targetgen, het outer membrane protein gen (OMP), blijkt toch wat
kruisreactiviteit te vertonen met andere commensale Neisseria species en werd daarom niet
gekozen als targetgen.
De outer membrane opacity (opa) genen zijn een aantrekkelijke keuze als targetgenen daar
alle stammen van NG deze opa-proteinen tot expressie brengen. Iedere stam bevat multiple
opa gene kopieën (11-13), commensale Neisseria species bevatten echter homologe
sequenties waardoor de keuze van de primers en probes zeer zorgvuldig moet gebeuren.
Geraats-Peters (JCM,2005) beschreef een PCR-reactie die specifiek NG opa-gen detecteert.
Later bleek echter dat er een Australisch NG variant bestaat die 5 nucleotiden mismatch
vertoont ten opzichte van de oorspronkelijk 19 nucleotide tellende probe. Om deze variant
ook op te pikken worden beide probes uitgetest in de huidige studie.
4.2
Optimalisatie uniplex PCR
4.2.1 Functionaliteit primers
In eerste instantie werden alle aangekochte primerparen getest op hun functionaliteit. Alle
primers vermeld in tabel 4 (zie 3.1.4) werden besteld bij de firma IDT (Integrated DNA
Technologies) en werden geleverd aan een concentratie van 100 µM. Deze werden
doorverdund naar 20 pmol/µl als werkoplossing. De functionaliteit van de verschillende
primerparen werd getest in een Real-Time PCR-reactie met SYBR Green MasterMix waarbij
de finale concentratie van de primers 250 nM bedroeg. Dit gebeurde met de respectievelijke
Vircell controle DNA’s in duplicaat. Voor alle primerparen, behalve voor HD, werd een
goede amplificatiecurve bekomen (zie figuur 23) waaruit kon besloten worden dat de primers
functioneel waren en kunnen gebruikt worden voor verdere evaluatie.
Primerpaar HD261-262 (p27-gen) leverde een positief resultaat op met het HD Vircell
controle DNA terwijl primerpaar HD264-265 (HhdA-gen) niet functioneel bleek. Deze
primers werden opnieuw aangekocht in de veronderstelling dat er misschien een fout was
gebeurd in de productie ervan. Ook met dit nieuwe lot primers werd er een negatief resultaat
bekomen. Deze PCR werd verlaten en er werd op zoek gegaan naar een alternatief targetgen.
In de weinige literatuur omtrent dit organisme werd een primerpaar op basis van het DNApolymerasegen teruggevonden (Liu et al.,2005). Dit primerpaar werd ook aangekocht en
getest maar bleek opnieuw niet functioneel in combinatie met het Vircell controle DNA.
Volgens Ricotta et al (J. bact 2011) kan HD taxonomisch opgesplitst worden in twee
verschillende species. Een mogelijke reden voor het falen van voorgaande PCR-reacties is dat
het aangekochte HD Vircell controle DNA behoort tot één van de twee mogelijke verwante
species en dat de beschreven PCR het andere species als target heeft.
79
Daar HD uiterst zelden voorkomt in onze contreien en hoofdzakelijk beschreven wordt in
Centraal-Afrika, Azië en Latijns-Amerika (zie 2.2.6), besluiten we om geen andere
functionele PCR te ontwikkelen voor dit pathogeen.
Figuur 23: Amplificatiecurve met de 4 primerparen van N. gonorrhoeae
(Van links naar rechts: 313-314 ; 310-311 ; 307-308 ; 304-305)
4.2.2 Efficiëntie PCR
Per pathogeen werden meerdere primerparen getest op hun functionaliteit. De volgende stap
in het optimalisatieproces was per pathogeen en per targetgen de meest gevoelige, efficiëntste
PCR te kiezen die finaal op de TAC zal gespot worden.
Er werden 10-voudige verdunningsreeksen gemaakt van de verschillende Vircell controle
DNA’s en deze werden met de overeenkomstige primerparen in drievoud ingezet in een RealTime PCR-reactie met SYBR Select MasterMix, gevolgd door smeltcurveanalyse. De
bekomen Ct-waarden en de smelttemperaturen voor iedere verdunning werden geregistreerd
en de gemiddelde Ct-waarden per verdunning in functie van de log10 van de concentratie
werden grafisch uitgezet. Uit deze grafische weergave kan de efficiëntie van de betreffende
PCR berekend worden, welke gecorreleerd is met de gevoeligheid van de test (zie tabel 13).
80
Tabel 13: Gemiddelde Ct-waarden van ieder organisme met bijhorende efficiëntie
Herpes Simplex Virus II
HSV249 - 250
Ureaplasma parvum
HSV258-259
HSV15-16
UP326-327
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
1x verdunning
22,81
83,77
22,72
87,69
22,47
80,31
18,60
73,57
10x
26,57
84,20
26,48
87,99
25,94
80,70
22,27
73,57
100x
30,34
84,26
29,97
88,09
29,48
80,73
26,80
73,64
1000x
35,41
84,36
34,71
88,15
34,63
80,80
29,27
73,64
10000x
ND
ND
ND
ND
35,06
80,78
32,76
73,64
Helling
-4,16
-3,92
-3,68
-3,32
R²
0,99
1,00
0,97
0,99
Efficiëntie %
74,02
79,92
88,87
102,42
Neisseria gonorrhoeae
NG304-305
NG307-308
NG310-311
NG313-314
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
1x verdunning
20,44
80,30
20,07
79,17
20,10
83,34
19,09
77,16
10x
23,75
80,51
23,42
79,39
23,45
83,61
22,65
77,34
100x
27,26
80,57
26,63
79,46
26,91
83,61
26,17
77,43
1000x
31,16
80,60
30,59
79,49
30,58
83,61
29,90
77,40
10000x
34,20
80,60
33,30
79,51
34,07
83,68
34,39
77,47
Helling
-3,45
-3,43
-3,51
-3,56
R²
1,00
1,00
1,00
0,99
Efficiëntie %
96,87
96,15
93,20
91,03
81
Mycoplasma genitalium
MG282-283
Ureaplasma urealyticum
MG285-286
UU288-289
UU291-292
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
1x verdunning
23,21
75,90
24,71
73,75
22,65
75,70
22,82
73,95
10x
26,47
76,20
27,79
74,04
26,21
75,66
26,36
73,92
100x
30,08
76,26
31,73
74,11
29,84
75,66
30,32
73,95
1000x
33,84
76,30
35,96
74,17
33,94
75,72
34,61
73,98
10000x
35,10
76,23
37,05
74,26
36,95
75,75
36,95
73,99
Helling
-3,38
-3,67
-3,74
-3,86
R²
0,99
0,99
1,00
0,99
Efficiëntie %
98,10
89,15
85,45
82,92
Haemophilus ducreyi
HD261-262
HD264-265
HD264-265
HD319-320
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
Ct-waarde
Tm-waarde
1x verdunning
26,01
75,28
ND
ND
ND
ND
ND
ND
10x
29,60
75,68
ND
ND
ND
ND
ND
ND
100x
32,91
75,74
ND
ND
ND
ND
ND
ND
1000x
36,16
75,84
ND
ND
ND
ND
ND
ND
10000x
ND
ND
ND
ND
ND
ND
ND
ND
Helling
-3,35
-2,76
R²
1,00
0,96
Efficiëntie %
98,98
171,71
82
Naast de gevoeligheid werd ook de ampliconlengte, kans op vormen van primerdimeren, kans
op vormen van tweedimensionele structuren etc. (zie bijlage 4 “Primers en probes”) in
rekening gebracht. Volgende primerparen werden geselecteerd (tabel 14) voor verdere
evaluatie (ruwe resultaten zie bijlage 5 “Selectie primerparen”):
Tabel 14: Geselecteerde primerparen
Herpes Simplex Virus II
DNA-polymerase
glycoproteïne G-gen
primerpaar 249-250
primerpaar 15-16
Mycoplasma genitalium
pdhD-gen
MgPa-gen
primerpaar 282-283
primerpaar 285-286
Ureaplasma urealyticum
ureD subunit urease-gen
Urease-gen (ureB)
primerpaar 288-289
primerpaar 291-292
Ureaplasma parvum
Urease-gen
primerpaar 326-327
Haemophilus ducreyi
Hemolytic cytotoxin
primerpaar 261-262
Neisseria gonorrhoeae
Por A (pseudogen)
Opa-gen
primerpaar 304-305
primerpaar 313-314
4.2.3 Specificiteit aan de hand van smeltcurveanalyse
De Real-Time PCR-reacties beschreven onder 4.2.2 werden steeds gevolgd door een
smeltcurveanalyse. Hierbij wordt de smeltcurve van iedere PCR bekeken ten opzichte van de
respectievelijke amplificatiecurve (zie figuur 24 en 25).
83
Figuur 24: Amplificatiecurve verdunningsreeks N. gonorrhoeae primerpaar 307-308
Figuur 25: Smeltcurve verdunningsreeks N. gonorrhoeae primerpaar 307-308
84
Bij de amplificatieplot is er een duidelijke correlatie tussen de hoeveelheid input-materiaal
(DNA-concentratie) en de Ct-waarde. Eventuele aspecifieke amplificaties kunnen hier niet uit
afgeleid worden. Door het amplificatieproces te laten volgen door een smeltcurveanalyse,
werd een smeltcurve gegenereerd die onafhankelijk is van de input-concentratie maar die wel
informatie geeft over de specificiteit van het gegenereerde amplicon.
Alle primerparen genereerden één unieke smeltpiek (Tm weergegeven in tabel 13) behalve
primerpaar NG310-311. Er werd bij herhaling een abnormaliteit waargenomen in de
smeltcurve (zie figuur 26) wat niet terug te vinden was in de blanco waardoor vorming van
primerdimeren wordt uitgesloten. Mogelijk is dit te wijten aan aspecifieke amplificatie. Om
hierover uitsluitsel te kunnen geven, werden de amplicons onderworpen aan gelelektroforese
(zie 4.2.4)
Figuur 26: Smeltcurve verdunningsreeks N. gonorrhoeae primerpaar 310-311
85
4.2.4 Gelelektroforese
Gelelektroforese werd toegepast op de bekomen amplicons van N. gonorrhoeae (zie figuur
27):
Figuur 27: Scheiding op agarosegel van alle primerparen van N. gonorrhoeae
In het eerste en laatste laantje bevinden zich de 100bp-ladders om het moleculair gewicht van
de amplicons in te schatten.
Bij alle NG primerparen, inclusief NG310-311; zien we in ieder laantje een mooi te
onderscheiden bandje, wat wil zeggen dat enkel de targetsequentie van het organisme werd
geamplificeerd en geen aspecifieke amplificatie of primerdimeerformatie kon aangetoond
worden. Een mogelijke verklaring kan zijn dat er geen homogene verspreiding van het GC%
is waardoor de kleine piek wordt gegenereerd bij uitsmelten. Verder onderzoek hiernaar werd
niet gedaan wegens gebrek aan tijd en materiaal en aangezien er geen aspecifieke amplificatie
is, vormt dit geen obstakel tijdens het onderzoek.
86
4.2.5 Specificiteit in aanwezigheid van vreemd DNA
Om de specificiteit te testen, werd gebruikgemaakt van DNA geïsoleerd uit bacteriële
stammen vermeld in 3.1.2. Alle bacteriële stammen werden getest met de SYBR Green
MasterMix in combinatie met alle primerparen. De bekomen resultaten zijn terug te vinden in
bijlage 1. Een species-specifieke PCR-reactie met DNA-input afkomstig van een 0,5 McF
(1McF = 1x108 CFU/ml) cultuur resulteert in een Ct-waarde van om en bij de Ct 12. Zo werd
voor de N. gonorrhoeae PCR een Ct-waarde bekomen van 13,55 en 12,45 met een 0,5 McF
NG-input. Bij een zeer specifieke reactie wordt dan ook geen amplificatie verwacht in
aanwezigheid van vreemd DNA.
De meerderheid van de reacties (omtrent 65 %) gaf geen enkele amplificatie, een minderheid
van de reacties (omtrent 34 %) resulteerde in een zeer zwakke amplificatie met een Tm die al
of niet meer dan 2 °C afwijkend was van de beoogde Tm. Deze inefficiënte reacties werden
als niet significant beschouwd. Van de 48 organismen getest met alle primerparen werd echter
in een tiental reacties een zwakke amplificatie bekomen met een Ct-waarde om en bij de LOD
van de species-specifieke PCR-reactie en een Tm identiek (± 1 °C) aan de Tm van het te
detecteren amplicon. Dit kan wijzen op een niet efficiënte aspecifieke amplificatie waar er een
zekere kruisreactie is van de gebruikte primers met het genoom verschillend van het te
detecteren genoom. Deze probleemgevallen zullen verder onderzocht worden aan de hand van
Real-Time PCR met een species-specifieke probe.
Een snelle screening van de invloed van de verschillende matrices werd uitgevoerd door naast
de bacteriële stammen, positieve controles (Vircell controle DNA) en de blanco’s (water,
RNase vrij) ook blanco’s te testen van de broths waarin de bacteriële stammen gesuspendeerd
zijn, dit om na te gaan of de broth interfereert met de PCR-reactie, wat niet het geval blijkt te
zijn.
4.2.6 Functionaliteit probes
Voor de zes te detecteren organismen werden tien primerparen geselecteerd op basis van
efficiëntie en specificiteit (zie tabel 14) door middel van Real-Time PCR in aanwezigheid van
SYBR Green. De volgende stap in het proces van optimalisatie is het invoeren van een
fluorescente detectieprobe en het nagaan als deze probe in combinatie met de geselecteerde
primers leidt tot een sterk en specifiek signaal. Dit gebeurt bij een vaste concentratie aan
primers (1000 nM) en probe (250 nM) samen met de TaqMan Fast Virus 1-step MasterMix.
Deze concentraties en mastermix zullen gebruikt worden op de TAC-STD array card
waardoor in deze fase de concentraties niet gevarieerd worden ter optimalisatie.
87
Een aantal resultaten uit bijlage 1 wijzen op een mogelijke kruisreactie van het primerpaar
met het genoom van een ander, of verwant, organisme wat bevestigd werd bij herhaling. Deze
resultaten worden opnieuw getest met hetzelfde primerpaar en de respectievelijke probe en
geven hierbij een negatief resultaat behalve primerpaar NG313-314 met probe 315 in
combinatie met N. meningitidis en primerpaar UP326-327 met probe 328 in combinatie met S.
aureus. Dit wijst op kruisreactie en wordt verder besproken bij 4.2.7.
Naast deze bacteriële stammen wordt ook een negatieve controle, een positieve controle en
een blanco (water, RNase vrij) meegenomen bij iedere run. Iedere positieve controle is
positief, wat aangeeft dat de probes werken bij hun specifiek organisme en iedere negatieve
controle negatief, wat aangeeft dat er geen contaminatie is. De resultaten zijn opgenomen in
bijlage 1.
4.2.7 Specificiteit probes
Kruisreactie tussen U. urealyticum en U. parvum wordt nagegaan door de primerparen en
probe van U. urealyticum te testen met Vircell controle DNA van U. parvum en omgekeerd.
Uit deze resultaten blijkt geen kruisreactie tussen deze organismen.
Zoals aangehaald in 4.2.6 vindt er wel een zwakke aspecifieke amplificatie plaats tussen de
primer- en probesequentie van U. parvum met het S. aureus genoom hoewel dit niet het geval
was volgens de literatuur waarop de sequentie is gebaseerd (BRON: zie artikel Van Cutsem et
al). Zes S. aureus stammen werden bekomen via de dienst microbiologie van het AZ Sint-Jan
om deze kruisreactie na te gaan (zie bijlage 1). DNA werd geëxtraheerd uit 0,5 McF culturen
van deze S. aureus stammen en ingezet met primerpaar UP326-327 en probe 328. Het
gegenereerde signaal in aanwezigheid van S. aureus DNA resulteert niet in een mooie
exponentiële amplificatiereactie zoals dit het geval is bij een UP positief staal (zie figuur 28).
Dit wordt duidelijker interpreteerbaar bij het bekijken van de multicomponent plot (figuur
29). Hierop is duidelijk te zien dat het UP positief staal een mooie sigmoïdale curve genereert
terwijl de curve van S. aureus betrekkelijk vlak blijft. Om deze aspecifieke reactie weg te
werken, zal op de TAC-STD array card de reversed sequentie van de probe gespot worden.
Figuur 28: Amplificatiecurve U. parvum en S. aureus
88
Figuur 29: Multicomponent plot U. parvum en S. aureus
Ook is er kruisreactie aangetoond tussen de primer- en probesequentie (NG313-314;315) van
N. gonorrhoeae met het genoom van N. meningitidis (zie figuur 30). Hier is er sprake van een
zwak positief resultaat aangezien er in vergelijking met het positief staal exponentiële
amplificatie plaatsvindt. Ook op de multicomponent plot zien we een duidelijke stijging die
weliswaar zwakker is maar toch duidelijk waarneembaar.
Figuur 30: Kruisreactie primerpaar 313-314 probe 315 met N. meningitidis
De gegenereerde amplicons van dit positief signaal werden gesequeneerd en er werd
inderdaad N. meningitidis vastgesteld (zwak positief). Als de sequenties van de gebruikte
primers en probe nogmaals werden geblast, werd er bij de reversed primer complementariteit
gevonden met het N. meningitidis genoom in beperkte mate (95 % Max Identity, 91 % Query
Coverage) en bij de probe (94 % Max Identity en 94 % Query Coverage), bij de forward
primer was dit niet het geval.
89
Theoretisch gezien zou amplificatie niet mogen plaatsvinden maar in de praktijk is dit wel het
geval, echter zeer inefficiënt. Om deze aspecifieke amplificatie te beperken werd de sequentie
van de probe aangepast zodat het aantal mismatches met het N. meningitidis genoom groter
werd. Deze probe werd opnieuw besteld en getest waarna geen kruisreactie meer aanwezig
was (ruwe resultaten zie bijlage 1).
Bij de andere organismen was er geen kruisreactie bij de geteste bacteriële stammen.
4.2.8 Gevoeligheid probe gebaseerde Real-Time PCR-reacties
De gevoeligheid wordt bepaald aan de hand van verdunningsreeksen met Vircell controle
DNA, net zoals bij de primers in punt 4.2.2, en met positieve klinische monsters. De klinische
monsters werden reeds getest in het lab van herkomst en deze resultaten werden vergeleken
met de eigen bekomen resultaten (resultaten zie bijlage 2).
1. Resutlaat verdunningsreeksen
a. LOD
Vircell controle DNA 10x, 100x en 1000x verdund werd in iedere Real-Time PCR-reactie
gedetecteerd met een Ct-waarde van Ct 29 tot Ct 36, afhankelijk van de startconcentratie van
het Vircell controle DNA. Hieruit kunnen geen conclusies getrokken worden betreffende de
LOD. Meer gestandaardiseerde concentraties zullen gebruikt worden om de LOD te bepalen
op de TaqMan Low Density Array Card.
b. Intensiteit fluorescentie
Bij de resultaten van de verdunningsreeksen werd ook de signal to noise ratio van de
bekomen fluorescentie bekeken (zie bijlage 3 “Fluorescentie”). Hoe groter het verschil tussen
begin- en eindgehalte van de fluorescentie (delta Rn), hoe gevoeliger en eenduidiger de
detectie van het organisme. De minimale vereiste signal to noise ratio werd arbitrair
vastgelegd op 1 000 000. Alle primer/probecombinaties resulteerden in een voldoende sterk
signaal behalve de primer/probecombinatie voor M. genitalium (MG282-283;284) die minder
sterke signalen genereerden dan bij de andere organismen. De detectie van het MgPa-gen van
M. genitalium gaf een delta Rn waarde > 2 000 000. De detectie van het pdhD-gen van M.
genitalium gaf een delta Rn waarde < 1 000 000. Hier bleek dat de probe aan zijn 5’-uiteinde
startte met 3 G’s wat de oorzaak was van deze verlaagde intensiteit. Een nieuwe probe werd
gezocht binnen het te amplificeren fragment welke op de TaqMan Low Density Array Card
zal geëvalueerd worden.
90
2. Positieve klinische stalen
De resultaten bekomen bij het analyseren van klinische stalen positief voor Herpes
simplexvirus II en N. gonorrhoeae (NG304-305) tonen dezelfde gevoeligheid aan als de
resultaten bekomen in het lab van herkomst en de resultaten voor U. urealyticum , U. parvum
en N. gonorrhoeae (NG313-314) wijzen op een hogere gevoeligheid (zie bijlage 2). Van H.
ducreyi waren geen klinische stalen ter beschikking dus hiervan zijn enkel de resultaten van
de verdunningsreeks ter beschikking.
Analyse van 12 M. genitalium positieve stalen (AZ Damiaan ziekenhuis Oostende) tonen een
duidelijk lagere gevoeligheid aan ten opzichte van de techniek gebruikt in Oostende. Bij
MG282-283/284 werden 2 van de 12 stalen niet gedetecteerd, bij MG 285-286/287 waren dit
4 van de 12 stalen (steeds stalen met een Ct > 35). De stalen die wel gedetecteerd werden,
hadden een hogere Ct-waarde dan de Ct-waarden bekomen in Oostende (tussen de 1 à 3,5
eenheden hoger). Daar het uitsluitend zwak positieve stalen zijn die niet gedetecteerd werden
kan dit te wijten zijn aan verschillende factoren, zoals staalname, bewaarcondities, transport,
extractieprotocol, mastermix en sequentie van de primers en probes.
De klinische stalen in Oostende worden geconcentreerd alvorens PCR-reactie uit te voeren,
maar verder zijn er weinig details bekend van de gevolgde procedures dus de oorzaak voor
deze mindere gevoeligheid kon niet exact vastgesteld worden. Validatie van de TAC-STD
array card zal grondiger het matrix geïnduceerd effect en bewaarcondities nagaan.
Er werd ook nagegaan als de gevoeligheid kon verbeterd worden door het concentreren van
het staal of als de matrix een invloed heeft op het resultaat:
a. Concentreren urinestalen
Uit drie positieve en drie negatieve urinestalen werd DNA geëxtraheerd uit 550 µl origineel
staal, uit 2 ml staal (geconcentreerd naar 550 µl) en uit 4 ml staal (geconcentreerd naar 550
µl). Dit geeft volgende resultaten (tabel 15):
Tabel 15: Resultaten geconcentreerde urines
Neisseria gonorrhoeae
Klinische stalen
p304-305
p313-314
Nieuwe probe
urine
Staal
2 ml
4 ml
Staal
2 ml
4 ml
Staal
2 ml
4 ml
166033602 NG +
19,79
18,24
17,79
16,08
14,43
13,96
18,16
16,39
15,95
177779801 NG +
25,89
24,92
23,96
22,49
21,42
20,44
25,51
24,61
23,16
142518403 NG +
24,83
22,98
22,03
21,26
19,96
18,55
24,53
22,54
21,35
91
Er is dus een verschil van gemiddeld 3Ct tussen het oorspronkelijk staal en het meest
geconcentreerde staal wat de detectie ten goede kan komen om zwak positieve stalen te
detecteren. Uit praktische overwegingen (beschikbare tubes, beschikbare centrifuges…) wordt
geopteerd om in de toekomst 15 ml urine of PreservCyt transportmedium te concentreren naar
1 ml.
b. Invloed verschillende matrices
Een aantal veel voorkomende matrices worden getest om na te gaan of deze interfereren in de
PCR-reactie. Hiervoor wordt een klinisch staal (staalnummer urine 33650558, positief voor
NG en CT) met Ct-waarde 23,14 bij NG304-305/306 en Ct-waarde 19,42 bij NG313-314/315
gespiked in urine, E-swab en TE-buffer en doorverdund in deze media. De resultaten hiervan
zijn de volgende (tabel 16):
Tabel 16: Invloed van verschillende matrices op de PCR-reactie
NG304-305/probe 306
NG313-314/probe 315
Urine
E-swab
TE-buffer
Urine
E-swab
TE-buffer
10x
100x
1000x
10 000x
27,6
31,59
36,55
NA
27,29
32,13
34,39
NA
27,77
31,91
35,47
NA
24,01
28,09
32,37
34,79
23,93
28,87
32,68
34,81
24,42
28,3
32,94
36,21
100 000x
NA
NA
NA
35,44
NA
NA
Hieruit wordt besloten dat de betreffende media niet interfereren met de PCR-reactie. Dit
wordt verder uitgebreid bestudeerd bij de validatie van de TAC-STD array card.
4.2.9 Detectie genomisch DNA
Een correcte staalname impliceert dat ook humane epitheliale cellen, eventueel witte
bloedcellen, aanwezig zijn in het staal. Door het detecteren van een minimale hoeveelheid
humaan DNA in het staal, kan er een besluit gevormd worden betreffende de correcte
staalname en eventuele bewaarcondities.
Er wordt een controle ingebouwd op de TAC-STD array card om de integriteit van het staal te
beoordelen. Verschillende Gene expression (GEx) assays zijn commercieel beschikbaar bij de
firma Life Technologies. Drie assays werden aangekocht die gebaseerd zijn op de detectie van
een genvrij genomische regio die niet wordt afgeschreven.
92
Alle drie de assays worden getest met twee verschillende runprotocollen (met en zonder
reverse transcriptase stap (zie tabel 17)) op twee verschillende stalen, bloed en urine, waaruit
DNA geëxtraheerd werd. Daarnaast werd ook een staal getest waar geen humaan DNA
aanwezig is ter controle, namelijk Bacteriodes fragilis.
Tabel 17: Resultaten Gene expression assay
Hs04260456
Hs04260457
Hs04260458
Met RT
Zonder RT
Met RT
Zonder RT
Met RT
Zonder RT
Bloedstaal
22,03
21,72
22,91
23,15
21,8
21,96
01660336 (urine)
Bact frag (neg)
Blanco
20,95
NA
NA
21,1
NA
/
20,27
NA
NA
20,11
NA
NA
20,77
NA
NA
21,2
NA
NA
Er is geen significant verschil tussen beide runs. Dit wil zeggen dat het niet coderend gen
inderdaad niet wordt afgeschreven naar mRNA en dat we inderdaad te maken hebben met een
genvrij genomische regio die niet wordt afgeschreven. Daarnaast wordt het bacterieel DNA
van Bacteriodes fragilis niet gedetecteerd, enkel het humaan DNA in bloed en urine. De assay
voldoet dus aan de voorwaade om humaan DNA aan te tonen. Er wordt ook besloten dat assay
Hs04260458_s1 op de TAC-STD array card wordt gezet door de hogere fluorescentie bij het
humane bloedstaal wat goed te zien is op de multicomponent plot (zie figuur 31).
Figuur 31: Multicomponent plot Gene expression assay met humaan bloedstaal
Ter confirmatie werden eerder geteste positieve stalen ook getest met deze assay met beide
runprotocollen (zie tabel 18). Hierbij werden geen significante verschillen waargenomen
tussen de twee amplificatieprotocollen. Ook bij deze resultaten is het duidelijk te zien dat de
derde assay, Hs04260458_s1, een hogere fluorescentie genereert wanneer er wordt gekeken
naar de multicomponent plot, deze curve zal telkens duidelijker tot uiting komen zoals op
voorgaande figuur (zie figuur 31).
93
Tabel 18: Resultaten Gene expression assay met klinische stalen
Met RT
Met RT
Met RT
Zonder RT
Hs04260456
Hs04260457
Hs04260458
Hs04260458
22,89
24,23
Klinische stalen
3272-0922 (CT)
22,53
22,66
Sint Augustinus Antw
3331-0023 (CT)
21,85
21,36
21,6
22,16
bf32440509 (CT)
24,67
24,89
24,95
26,15
ur32021253 (CT)
24,41
23,94
24,01
24,93
3361-1008 (CT)
19
18,77
19,35
19,45
3271-1204 (CT)
21,63
21,23
21,77
22,15
3084-0025 (CT)
26,02
25,71
25,33
25,82
3242-1197 (CT)
30,08
29,15
29,96
30,25
ut30831211 (NG)
20,71
20,97
21,57
21,98
ut33340206 (NG)
28,07
27,55
28,14
29,31
3246-0567 (NG)
25,03
25,39
25,4
26,48
33650558 (NG en CT)
20,64
20,5
20,51
20,67
11111797 (NG)
26,37
26,25
26,7
26,72
11112284 (NG)
26,47
26,2
26,79
27,51
11111355 (NG)
26,17
26,27
26,55
27,13
176465001 (II)
24,62
24,65
25,15
26,53
163113502 (I)
20,94
20,43
21,43
21,66
171170501 (II)
27,74
28,03
28,07
30,01
161794601 (I)
24,44
24,08
24,76
25,82
170452401 (II)
26,49
26,6
26,88
27,97
163114202 (II)
23,23
23,19
23,52
24,41
178306601 (I)
23,76
23,52
24,17
25,12
176298802 (I)
21,61
21,36
21,9
22,66
173870401 (I)
22,82
22,25
22,61
22,79
165952401 (I)
24,61
24,24
24,78
26,01
174460800 (II)
32,2
32,18
32,29
33,75
Volbloed (pos)
25,36
26,58
25,22
26,39
Camp jej (neg)
NA
NA
NA
NA
Blanco
NA
NA
NA
NA
Klinische stalen Brugge
HSV
94
4.2.10 Samenstelling TAC-STD array card
Na de optimalisatie van de primers en probes worden deze gespot op de TAC-STD array card.
De resterende plaatsen worden aangevuld met andere aangekochte of niet gevalideerde
primers en probes teruggevonden in de literatuur (zie tabel 19).
95
Tabel 19: Samenstelling TAC-STD array card
Assay Name
Targetgen
Primer Forward
Primer Reverse
Probe
1 HSV TOTAL #1** DNA polymerasegen
p249_HSVtot1Fw
p250_HSVtot1Rv
p251_HSVtot1MGB
2 HSV1 Bruges**
3 HSV2 Bruges**
glycoproteïne G-gen
glycoproteïne D-gen
p011_HSV1fw
p015_HSV2fw
p013_HSV1rv
p016_HSV2rv
p014,2_HSV1probe
p018_HSV2probe
4 HSV TOTAL #2
DNA polymerasegen
p252_HSVtot2Fw
p253_HSVtot2Rv
p254_HSVtot2MGB
5 HSV1 #1
6 HSV2 #1
DNA polymerasegen
DNA polymerasegen
p255_HSV1#1Fw
p256_HSV1#1Rv
p257_HSV1#1MGB
7 HD P27**
27 kDa H. ducreyi-specific protein
p261_HDp27Fw
p262_HDp27Rv
p263_HDp27RevMGB
8 HD ABI
Ongekend, commercieel beschikbaar via Life Technologies
9 MG pdhD**
10 MG MgPa**
dihydrolipoamide dehydrogenase
major surface protein MgPa
p282_MGpdhDFw
p285_MGMgPaFw
p283_MGpdhDRv
p286_MGMgPaRv
anders ingekort
p287_MGMgPaMGB
11 UU ureD**
ureD subunit van het urease gen
p288_UUureDFw
p289_UUureDRv
p290_UUureDMGB
12 UU ureB**
13 UU ABI
ureB subunit van het urease gen
Ongekend, commercieel beschikbaar via Life Technologies
p291_UUureFw
p292_UUureRv
p293_UUureMGB
14 UP ureD**
ureD subunit van het urease gen
p326_UPFw:
p327_UPRv
p328_UPMBGprobe
15 UP ureD_rev
16 UP ureB
ureD subunit van het urease gen
ureB subunit van het urease gen
p326_UPFw:
p327_UPRv
reversed probe
17 NG PorAHjel**
porine pseudogen
p304_NGPorA1Fw
p305_NGPorA1Rv
p306_NGPorA1MGB
18 NG PorAAVK
19 NG Omp AVK
porine pseudogen
outer membrane protein
p307_NGPorA2Fw
p308_NGPorA2Rv
p309_NGPorA2MGB
p313_NGOpaDFw
p314_NGOpaDRv
p315_2_NGOpaDMGB
20 NG OpaDPDS_2** 5' untranslated region of the opa genes that encode opacity proteins.
21 NG Opa G-P1
opacity gene
22 NG Opa G-P2
opacity gene
23 TP tpp47PDS**
24 TP tpp47 Schields
47 kDa membrane lipoprotein
47 kDa membrane lipoprotein
25 TP tpp47Gay
47 kDa membrane lipoprotein
p243_TPSY767Fw
p244_TPSY834Rv
p245_TPMGB
26 TP polA**
27 TP polA ITG
DNA polymerase I gene
DNA polymerase I gene
p246_TPpolAFw
p247_TPpolARv
p248_ TPpolAMGB
96
28 TV 2kb**
2-kb repetitief sequentie
29 TV repeat
29 TV repeat rev
repaeted segment
repaeted segment
p267_TV2kbFw
p268_TV2kbRv
p269_TV2kbMGB
30 TV Btub
beta-tubulin protein, a major component of the T. vaginalis cytoskeleton.
p273_TVbtFw
p274_TVbtRv
p275_TVbtMGB
32 MH Yid**
a membrane protein translocase
p276_MHyidCFw
p277_MHyidCRv
p278_MHyidCMGB
33 MH tuf**
elongation factor Tu (EF-TU)
p279_MHtufFw
p280_MHtufRv
p281_MHtufMGB
34 CT crPAVK**
cryptische plasmide
p294_CTCP1Fw
p295_CTCP1Rv
p296_CTCP1MGB
35 CT crPAVKrev
36 CT crPBennet
cryptische plasmide
cryptische plasmide
p294_CTCP1Fw
p298_CTCP2Fw
p295_CTCP1Rv
p299_CTCP2Rv
p297_CTCP1RevMGB
p300_CTCP2MGB
37 CT crP PHA
38 CT 16S**
cryptische plasmide
16S
p301_CT16SFw
p302_CT16SRv
probe anders ingkort
39 CT Omp1PVL
40 CT Omp1
outer membrane protein
outer membrane protein
41 CT LGV
polymorphic membrane protein H gene (pmp gene
42 CT LGV ABI
Ongekend, commercieel beschikbaar via Life Technologies
43 CMV Bruges
44 PDV
45 SHV
46 RNAseP ABI
RNAse P
47 Hs04260458_s1
genomic DNA, ongekend, commercieel beschikbaar via Life Technologies
48 Hs99999901_s1
18S controle ABI, ongekend, commercieel beschikbaar via Life Technologies
** In deze studie gevalideerde primer en probesequentie, de niet gemarkeerde zijn varianten op de gevalideerde sequenties of volledig nieuw geselecteerde sequentie
97
4.3
Validatie TAC
4.3.1
Selecteren matrices
De geselecteerde matrices zijn TE-buffer, E-swab, Preserve Cyt, UTM, serum, first voided
urine en weefselbiopt. De niet-steriele matrices (serum, urine en weefsel) werden getest met
de verschillende uniplex PCR-reacties en bleken alle negatief te zijn behalve het first voided
urine en het weefselbiopt.
First voided urine: In totaal werden negen verschillende urines getest op de aanwezigheid van
de te detecteren pathogenen. Één urinestaal bleek positief te zijn voor U. urealyticum. De
overige vijf urinestalen waren negatief en konden gebruikt worden voor het spiken met de
verschillende pathogenen.
Weefselbiopt: Het eerste weefselbiopt was een linker- en rechter amandel die werden
versneden met de gentleMACS-dissociator om een celsuspensie te bekomen. Het celaantal
werd bepaald op de coulter en daarna doorverdund naar 1 000 000 cellen per ml. De suspensie
hiervan testte positief op M. hominis en U. urealyticum. Deze suspensie was dus niet geschikt
om te gebruiken tijdens de validatie.
Een andere suspensie van placenta verkregen via de keizersnede werd gemaakt volgens
hetzelfde principe. Het celaantal werd bepaald en bedroeg 1 156 000 cellen per ml, wat niet
meer moest doorverdund worden. Na testen met de uniplex PCR-reacties bleek de suspensie
negatief te zijn voor alle organismen en was dus geschikt om te gebruiken tijdens de validatie.
4.3.2 Detectielimiet
De detectielimiet werd bepaald aan de hand van de twee Vircell controle DNA mixen (zie
3.7.3 tabel 10 en 11). De vijf verdunningen van Vircell controle DNA mix 1 en Vircell
controle DNA mix 2 werden 10x bepaald op de TAC-STD array card. Uitgaande van deze
resultaten werd de LOD bepaald (ruwe resultaten zie bijlage 6 “Limit of Detecton”). In tabel
20 wordt het aantal positieve testen van de 10 bepalingen per organisme en per targetgen
weergegeven met de bijhorende LOD.
98
Tabel 20: Bepaling limit of detection
Targetgen
Mix 1-1
Mix 1-2
Mix 1-3
Mix 1-4
Mix 1-5
LOD
Gemiddelde
13884
6942
3471
555
277
kopieën/ml
kopieën/ml
kopieën/ml
kopieën/ml
kopieën/ml
kopieën/ml
HD ABI
8/10
8/10
6/10
/
/
6942-3471
32,584
HD P27
10/10
8/10
3/10
/
/
6942-3471
33,281
HSV TOTAL #1
10/10
10/10
10/10
5/10
/
3471-555
32,282
33,565
Ct-waarde
HSV TOTAL #2
10/10
10/10
10/10
6/10
2/4
3471-555
HSV2 #1
10/10
10/10
9/10
2/10
1/4
3471-555
34,5
HSV2 Bruges
10/10
10/10
10/10
7/10
/
3471-555
30,76
MG MgPa
10/10
10/10
10/10
2/10
1/4
3471-555
32,838
MG pdhD
10/10
10/10
10/10
5/10
1/4
3471-555
31,213
NG Omp AVK
10/10
10/10
10/10
6/10
1/4
3471-555
30,627
NG Opa G-P1
10/10
10/10
10/10
10/10
3/4
555-277
33,466
NG Opa G-P224
/
/
/
/
/
/
/
NG OpaD PDS 2
10/10
10/10
10/10
10/10
4/4
< 277
33,375
NG PorAAVK
10/10
10/10
10/10
5/10
/
3471-555
31,594
NG PorA Hjel
10/10
10/10
10/10
5/10
2/4
3471-555
30,876
UU ABI
10/10
10/10
10/10
6/10
2/4
3471-555
31,504
UU ureB
10/10
10/10
10/10
6/10
3/4
3471-555
30,632
UU ureD
10/10
10/10
10/10
4/10
/
3471-555
31,625
Per pathogeen hebben de verschillende targets een gelijkaardige gevoeligheid, behalve voor
N. gonorrhoeae waar OpaD PDS het gevoeligste blijkt. Dit kan te wijten zijn aan het feit dat
de Opa-genen multycopy genes zijn (Geraats-Peters et al.,2005).
Op basis van het Vircell controle DNA wordt een schatting gemaakt van de LOD. Voor alle
organismen ligt de LOD voor de targetgenen tussen de 3471-555 kopieën per ml behalve voor
volgende targets:
NG Opa G-P1: 555-277 kopieën per ml
NG OpaD PDS 2: < 277 kopieën per ml
Beide targetgenen zijn gevoeliger. De targetgenen van H. ducreyi zijn duidelijk minder
gevoelig, de LOD ligt hier tussen de 6942-3471 kopieën per ml. Voor dit organisme zal in de
toekomst een betere target gezocht worden.
24
Australische variant, geeft geen resultaat met NG van hier.
99
Voor alle targetgenen ligt de LOD hoger dan bij een klassieke uniplex Real-Time PCR waar
meestal een LOD van 100 à 200 kopieëen per ml wordt bekomen. Dit kan te wijten zijn aan
het verschil in reactievolume waarbij de TAC-STD array card een reactievolume beschikbaar
is van 1 µl (0,75 µl DNA en 0,25 µl mastermix) en bij de klassieke uniplex PCR een
reactievolume van 50 µl (20 µl DNA en 30 µl mastermix).
4.3.3 Herhaalbaarheid en reproduceerbaarheid
De positieve klinische stalen die gebruikt werden om mengstaal mix 1 en mengstaal mix 2
(zie 3.7.3 tabel 12) te maken in TE-buffer, werden eerst getest met de uniplex PCR-reacties
om een schatting te maken van de lading op basis van de Ct-waarden (zie bijlage 7 “Ctwaarden klinische monsters”). Mengstaal mix 1 en mengstaal mix 2 werden samengesteld op
basis hiervan en doorverdund. Iedere verdunning werd drie keer bepaald binnen eenzelfde run
(herhaalbaarheid) en bijkomend twee keer herhaald in twee bijkomende runs
(reproduceerbaarheid) (zie 3.7.2). De resultaten zijn terug te vinden in bijlage 8
“Herhaalbaarheid en reproduceerbaarheid”.
De herhaalbaarheid en reproduceerbaarheid worden
standaarddeviatie en variatiecoëfficiënt in tabel 21.
samengevat
met
bijhorende
Tabel 21: Herhaalbaarheid en reproduceerbaarheid
-1
10
HSV2 #1
gem
25,27
Std
0,711
% CV
2,8
HSV2 Bruges
gem
19,91
Std
0,480
% CV
2,4
HSV TOTAL # 1
gem
20,68
Std
0,240
% CV
1,2
HSV TOTAL # 2
gem
21,52
Std
1,021
% CV
4,7
Herhaalbaarheid
Reproduceerbaarheid
-3
-4
-5
-1
10
10
10
10
10
10-2
10-3
10-4
Resultaat Herpes Simplex Virus II
-2
10-5
28,67
0,416
1,5
30,50
1,826
6,0
33,03
1,403
4,2
34,94
/
/
25,02
0,791
3,2
28,28
0,685
2,4
30,05
1,458
4,9
33,03
1,009
3,1
34,89
0,064
0,2
23,79
0,690
2,9
26,84
1,096
4,1
29,78
0,156
0,5
33,91
1,427
4,2
19,07
1,428
7,5
23,52
0,655
2,8
26,74
0,793
3,0
29,94
0,655
2,2
33,06
2,409
7,3
24,07
0,120
0,5
27,25
1,341
4,9
30,89
0,188
0,6
33,87
0,400
1,2
20,56
0,262
1,3
23,88
0,363
1,5
27,13
0,965
3,6
30,50
0,566
1,9
34,04
1,061
3,1
25,65
1,506
5,9
28,42
2,702
9,5
33,47
0,428
1,3
34,79
1,910
5,5
21,51
1,162
5,4
25,39
1,190
4,7
28,84
2,011
7,0
33,20
0,481
1,4
34,97
1,123
3,2
100
Resultaat Mycoplasma genitalium
MG MgPa
gem
21,48
Std
0,329
% CV
1,5
MG pdhD
gem
20,35
Std
1,248
% CV
6,1
UP ureB
X
27,13
Std
2,551
% CV
9,4
UP ureD_rev
gem
27,99
Std
0,321
% CV
1,1
UP ureD
gem
28,72
Std
0,326
% CV
1,1
NG Omp AVK
gem
19,22
Std
0,312
% CV
1,6
NG Opa G-P1
gem
18,02
Std
0,652
% CV
3,6
NG Opa G-P2
gem
/
Std
/
% CV
/
NG OpaD PDS_2
gem
17,13
Std
0,569
% CV
3,3
NG PorAAVK
gem
20,62
Std
0,589
% CV
2,9
24,60
0,246
1,0
27,51
0,588
2,1
33,45
0,759
2,3
34,36
0,949
2,8
21,06
0,704
3,3
24,22
0,731
3,0
27,19
0,663
2,4
33,25
0,784
2,4
34,44
0,788
2,3
23,55
0,771
3,3
26,19
1,502
5,7
32,01
32,94
19,79
22,83
0,758
4,796
1,161
1,186
2,4
14,6
5,9
5,2
Resultaat Ureaplasma parvum
25,70
1,258
4,9
31,58
0,832
2,6
33,36
3,449
10,3
32,16
1,783
5,5
34,28
1,711
5,0
34,35
/
/
/
/
/
27,68
1,976
7,1
32,53
1,360
4,2
34,25
1,398
4,1
34,35
/
/
/
/
/
32,32
1,125
3,5
32,67
/
/
/
/
/
/
/
/
28,05
0,253
0,9
31,94
1,019
3,2
34,21
2,169
6,3
/
/
/
/
/
/
33,01
0,488
1,5
34,28
/
/
/
/
28,32
32,18
/
/
0,594
1,212
/
/
2,1
3,8
Resultaat Neisseria gonorrhoeae
34,35
0,129
0,4
/
/
/
/
/
/
23,04
0,466
2,0
26,72
0,684
2,6
29,88
0,431
1,4
36,81
2,639
7,2
18,81
0,607
3,2
22,33
1,136
5,1
26,15
0,926
3,5
29,48
0,679
2,3
34,68
3,083
8,9
20,75
0,802
3,9
24,50
1,397
5,7
27,79
0,667
2,4
32,47
0,709
2,2
17,43
0,962
5,5
20,45
0,810
4,0
24,16
1,094
4,5
27,54
0,590
2,1
31,87
0,964
3,0
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
20,98
0,217
1,0
24,42
0,700
2,9
24,33
0,931
3,8
31,75
1,582
5,0
16,61
0,883
5,3
19,90
1,680
8,4
23,51
1,602
6,8
24,90
1,256
5,0
31,17
1,432
4,6
24,23
0,077
0,3
27,23
0,934
3,4
30,06
1,358
4,5
34,36
1,008
2,9
31,17
0,891
2,9
23,66
0,779
3,3
26,42
1,517
5,7
29,67
1,155
3,9
33,46
1,800
5,4
101
NG PorA Hjel
gem
19,70
Std
0,354
% CV
1,8
UU ABI
gem
Std
% CV
UU ureB
gem
Std
% CV
UU ureD
gem
Std
% CV
23,48
0,496
2,1
26,59
30,48
34,01
19,50
23,24
1,638
0,662
1,016
0,379
0,500
6,2
2,2
3,0
1,9
2,2
Resultaat Ureaplasma urealyticum
26,30
1,296
4,9
30,32
0,543
1,8
34,42
1,652
4,8
25,25
0,526
2,1
29,34
0,277
0,9
33,12
2,504
7,6
33,12
/
/
/
/
/
25,11
0,504
2,0
28,61
1,083
3,8
32,36
2,071
6,4
33,12
/
/
/
/
/
24,19
0,563
2,3
28,44
1,046
3,7
33,89
0,631
1,9
/
/
/
/
/
/
24,60
0,697
2,8
28,29
0,816
2,9
33,06
1,294
3,9
34,45
/
/
/
/
/
23,45
1,050
4,5
28,09
0,104
0,4
31,87
0,592
1,9
33,35
0,654
2,0
/
/
/
23,58
0,883
3,7
27,89
0,291
1,0
31,78
0,447
1,4
33,88
1,334
3,9
/
/
/
De standaarddeviatie is een maat voor de spreiding van de waarden rond het gemiddelde. Hoe
beter de herhaalbaarheid en hoe dichter de waarden bijeen liggen, hoe lager de
standaarddeviatie zal zijn. Aan de hand van die waarde en het gemiddelde werd de
variatiecoëfficiënt berekend. Die moet voldoen aan de vooropgestelde waarde voor
herhaalbaarheid (< 5 % CV) en voor reproduceerbaarheid (< 10 % CV).
De reproduceerbaarheid geeft voor ieder organisme een CV < 10 % behalve bij targetgen MG
pdhD is dit 10,3 %. Bij de hoogste verdunning om en bij de LOD is het normaal dat de CV
wat hoger uitvalt door de stochastische verdeling van het te amplificeren materiaal.
Bij de herhaalbaarheid is te zien dat een aantal targetgenen een CV hebben hoger dan de
grenswaarde van 5 %. Als dit voorvalt bij hoger geconcentreerd materiaal, moet nagegaan
worden of de cycle treshold correct ingesteld werd of als er afwijkende curven bekomen
werden.
In het algemeen zien we bij de targetgenen waar afwijkende %CV bekomen werden, dat de
baseline curve fluctueert (= fluorescentie gemeten in de cycli voor er werkelijk amplificatie
gedetecteerd wordt). Soms is dit minimaal, soms is dit meer uitgeproken bij probes die een
hogere achtergrondwaarde hebben. De delta Rn wordt bekomen door de verhouding van de
gemeten flurescentie (Rn) t.o.v. de fluorescentie van Rox (passive reference dye). Door deze
waarde uit te zetten tov de cycli kan de Ct waarde bepaald worden. Bij een fluctuerende
baseline kan dit tot een lagere Ct leiden, met een grotere %CV tot gevolg.
102
Daar het hier om een kwalitatieve detectie gaat, werd opgemerkt dat de hoger %CV bekomen
bij een lagere verdunning van het input materiaal niet gecorreleerd is met de afwezigeheid van
een Ct-waarde. Met andere woorden, bij de lagere verduningen werd de traget steeds
kwalitatief gedetecteerd, echter soms met een afwijkende Ct waarde. De afwijkende %CV
heeft geen invloed op het kwalitatief resultaat.
Waarom deze baselinefluorescentie fluctueert is niet eenduidig te verklaren en kan mogelijk
iets te maken hebben met de tweedimentionele structuur die de probe kan aannemen in
oplossing. Verdere optimalisatie van de probe zou hierover mogelijks uitsluitsel geven.
Bij de targetgenen van UP en UU werden de metingen uitgevoerd dichter bij de LOD
waardoor hogere Ct-waarden werden bekomen bij de eerste verdunningen. Dit kan een
verklaring zijn voor de hoge CV van het UP ureB targetgen en het UU ABI targetgen.
De reproduceerbaarheid is over het algemeen goed voor alle organismen, de herhaalbaarheid
is kwalitatief gezien goed, maar kwantitatief geeft dit uiteenlopende waarden voor de
aangehaalde targetgenen.
Daarnaast werd opgemerkt dat de gemiddelde Ct-waarden van de sterkst verdunde klinische
stalen (zie tabel 21) hogere gemiddelde Ct-waarden vertonen dan de Ct-waarden bekomen
tijdens de bepaling van de detectielimiet (zie tabel 20). Een mogelijke verklaring hiervoor kan
zijn dat een deel van het aangekochte DNA van de vircellcontroles waaruit een
verdunningsreeks gemaakt werd in molecular grade water, blijft “plakken” aan de wand van
de tubes waarin de LOD verdunningsreeksen gemaakt werden. Bij bepaling van de
herhaalbaarheid en reproduceerbaarheid werd er gebruikgemaakt van mixen van positieve
klinische stalen die naast het geëxtraheerd DNA van het targetorganisme en mastermix ook
nog ander DNA en andere componenten bevatten door het extractieprotocol. Hierdoor zal er
minder van het targetDNA achterblijven in de wells en kunnen latere Ct-waarden bekomen
worden.
4.3.4 Matrix geïnduceerd effect
Mengstaal mix 1 en mengstaal mix 2 (zie 3.7.2 tabel 12) werden doorverdund in verschillende
matrices (zie 3.7.2 tabel 7). Serum en placenta werden nog niet gebruikt wegens tekort aan
TAC-STD array cards. Aan de hand van verdunning 10-3 en 10-4 werd een representatief
resultaat gegenereerd om te kunnen vergelijken met de herhaalbaarheid in TE-buffer.
Verdunning 10-4 is altijd positief tijdens testen van de herhaalbaarheid in TE-buffer (behalve
bij UP), verdunning 10-5 niet.
103
Als de resultaten van de twee verdunningen in de verschillende matrices positief resultaat
leveren, is het niet nodig om verdunning 10-2 en 10-1 te testen aangezien deze verdunningen
nog sterker zijn en dus zeker positief zullen zijn. De resultaten van de geteste matrices zijn
terug te vinden in bijlage 9 “Matrix geïnduceerd effect”.
Wanneer de resultaten van de herhaalbaarheid in TE-buffer en de verschillende matrices
werden vergeleken, bleken deze elkaar (kwalitatief) te bevestigen.
Alle PCR-reacties voor de detectie van NG, HSV II en MG vertonen geen invloed van de
matrices op de gevoeligheidheid van de detectie. Enkel UU en UP vertonen een invloed en
worden weergegeven in tabel 22. Als er meer positieve resultaten zijn in de betreffende matrix
ten opzichte van de TE-buffer, wordt dit weergegeven in een groene kleur. Wanneer er
minder positieve resultaten zijn, wordt dit weergegeven in een rode kleur.
Tabel 22: Verschil in resultaat bij verschillende matrices
UP ureB
-3
UP ureD_rev
-4
Verdunning
10
10
10
TE-buffer
3/3
1/3
E-swab
0/3
Preserve Cyt
-3
-4
UP ureD
-3
UU ABI
-4
-3
UU ureB
10-4
0/3
3/3
2/3
3/3
0/3
3/3
1/3
1/3
3/3
0/3
3/3
2/3
2/3
1/3
3/3
2/3
3/3
0/3
0/3
3/3
1/3
3/3
1/3
3/3
2/3
0/3
0/3
1/3
0/3
1/3
1/3
0/3
0/3
1/3
0/3
3/3
1/3
3/3
3/3
3/3
1/3
10
-3
UU ureD
10-3
10
-4
10
10
10
10
1/3
0/3
1/3
0/3
3/3
1/3
3/3
0/3
2/3
0/3
1/3
0/3
3/3
1/3
0/3
0/3
1/3
0/3
3/3
0/3
3/3
UTM
1/3
0/3
1/3
0/3
2/3
0/3
Urine 2
2/3
0/3
1/3
0/3
3/3
Urine 3
0/3
0/3
0/3
0/3
Urine 5
1/3
1/3
0/3
0/3
10
-4
Ook wordt er bij deze targetgenen kruisreactie waargenomen (zie 4.3.5), wat ook verschilt
van matrix tot matrix (zie tabel 23).
Tabel 23: Invloed van matrix op kruisreactie
UP ureB
-3
UP ureD_rev
-4
Verdunning
10
10
10
TE-buffer
1/3
0/3
E-swab
0/3
Preserve Cyt
-3
-4
UP ureD
-3
UU ABI
-4
-3
UU ureB
10-4
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
2/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
3/3
0/3
0/3
0/3
0/3
0/3
10
-3
UU ureD
10-3
10
-4
10
10
10
10
2/3
1/3
0/3
0/3
1/3
0/3
0/3
0/3
1/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
0/3
2/3
0/3
1/3
UTM
0/3
0/3
0/3
0/3
2/3
0/3
Urine 2
0/3
0/3
1/3
0/3
2/3
Urine 3
0/3
0/3
0/3
0/3
Urine 5
2/3
0/3
0/3
0/3
10
-4
104
Voor sommige van deze zes targetgenen wordt er een minder goed resultaat bekomen, maar
de matrices blijken dan wel weer een positief effect te hebben op kruisreactie.
De verschillende matrices blijken geen kwalitatief effect te hebben op de organismen behalve
op U. urealyticum en U. parvum. Dit kan echter te wijten zijn aan de concentratie van beide
organismen. Er werd namelijk gestart van een hogere Ct-waarde voor beide organismen
waardoor de reacties doorgaan om en bij de LOD. Dit kan van invloed zijn op de resultaten en
zal in de toekomst herbekeken worden. Er kan dus besloten worden dat staalname kan
gebeuren in deze matrices.
4.3.5 Efficiëntie TaqMan Low Density Array Card
De lineariteit werd nagegaan aan de hand van de resultaten van de herhaalbaarheid en
reproduceerbaarheid (zie bijlage 10 “Efficiëntie”): De gemiddelde Ct-waarde per verdunning
werd grafisch uitgezet in functie van de log10 van de concentratie. Uit deze grafische
weergave werd de helling bepaald en de efficiëntie van de betreffende PCR berekend, welke
gecorreleerd is met de gevoeligheid van de test (zie tabel 24). Zoals aangehaald in 3.7.2
moeten de resultaten voldoen aan:
Regressiecoëfficiënt R2 > 0,95 over de (minstens 3) diluties.
Efficiëntie van de PCR 0,9 ≤ E ≤ 1,1
105
Tabel 24: De efficiëntie voor ieder targetgen per organisme
Herpes Simplex Virus II
Targetgen
Helling
R2
Efficiëntie %
HSV2 #1
-2,4504
0,9907
155,92%
HSV2 Bruges**
-3,4397
0,9944
95,31%
HSV TOTAL #1**
-3,3594
0,9997
98,46%
HSV TOTAL #2
-3,4731
0,9871
94,06%
Mycoplasma genitalium
Targetgen
Helling
R2
Efficiëntie %
MG MgPa**
-3,5766
0,9694
90,37%
MG pdhD**
-4,0864
0,9742
75,68%
Ureaplasma parvum
Targetgen
Helling
R2
Efficiëntie %
UP ureB
-2,1742
0,8036
188,36%
UP ureD_rev
-3,0801
0,9772
111,19%
UP ureD **
-3,0272
0,9756
113,96%
Neisseria gonorrhoeae
Targetgen
Helling
R2
Efficiëntie %
NG Omp AVK
-3,8892
0,9936
80,77%
NG Opa G-P1
-3,5963
0,9965
89,70%
NG OpaD PDS_2**
-3,4136
0,9636
96,31%
NG PorAAVK
-3,284
0,9975
101,61%
NG PorAHjel**
-3,6928
0,9975
86,55%
Ureaplasma urealyticum
Targetgen
Helling
R2
Efficiëntie %
UU ABI
-3,626
0,996
88,71%
UU ureB**
-4,2291
0,9945
72,37%
UU ureD**
-3,4778
0,9787
93,88%
**In deze studie geoptimaliseerde primer- en probesequenties
Bij ieder targetgen werd een realtief goede regressiecoëfficiënt waargenomen behalve bij UP
ureB. Voor de helling en daaruit afgeleide efficiëntie zijn er meerdere resultaten die niet
voldoen aan de vooropgestelde waarden. Deze waarden zijn aangeduid in het rood in tabel 24.
De verkregen amplificatiecurves en multicomponent plots zijn echter duidelijk positief, wat
ook belangrijk is bij de kwalitatieve bepaling van de TAC-STD array card.
106
HSV II: Bij targetgen HSV2 #1 en HSV2 Bruges waren alle testen positief tot en met
verdunning 10-4. Bij verdunning 10-5 werd het signaal niet meer gedetecteerd bij iedere run en
bij verdunning 10-6 was alles negatief. De efficiëntie van HSV2 #1 is zeer slecht en zal bij een
volgende versie van de TAC-STD array card weggelaten worden.
De andere twee targetgenen testten voor alles positief tot en met verdunning 10-5, bij een
verdunning verder was het resultaat negatief. Ook de efficiëntie heeft een goede waarde die
valt binnen de vooropgestelde waarden.
MG: Tot en met verdunning 10-5 was alles positief (op één resultaat na bij MG MgPa) voor
beide targetgenen. De efficiëntie van het targetgen MG MgPa ligt wat aan de lage kant maar
toch nog binnen de vooropgestelde waarden, de efficiëntie van het targetgen MG pdhD is veel
te laag. Tijdens de optimalisatie werd er besloten om voor dit targetgen een andere probe op
de TAC-STD array card te plaatsen door de lage fluorescentie. Deze target zal dus nog verder
bekeken worden in de toekomst tijdens de validatie zodat de efficiëntie kan verbeterd worden.
UP: Targetgen UP ureB verkreeg betere resultaten bij verdunning 10-3 dan de andere twee
targetgenen. Maar UP ureB heeft wel de slechtste efficiëntie en als enige een slechte
regressiecoëfficiënt. Ook de andere targetgenen hebben een slechte efficiëntie. Dit kan
toegeschreven worden aan de verdunningen die uitgaan van een hogere Ct-waarde in
vergelijking met de andere organismen. Hierdoor worden resultaten bekomen rond de LOD en
kan de efficiëntie niet goed bepaald worden. Deze verdunningen dienen opnieuw te gebeuren
in de toekomst tijdens de validatie, want nu kan nog geen uitspraak gedaan worden omtrent de
efficiëntie.
NG: Targetgen NG Omp AVK en NG PorAHjel vertonen een minder goede efficiëntie maar
praktisch iedere verdunning was positief, net zoals bij de andere targetgenen. Targetgen NG
OpaG verkreeg geen resultaat aangezien dit een Australische variant is waarvan het DNA niet
aanwezig is.
UU: Targetgen UU ureD heeft een lagere maar goede efficiëntie, de andere targetgenen niet.
Dit targetgen was ook positief tot en met verdunning 10-4, in tegenstelling tot de andere
targetgenen die tot verdunning 10-3 allemaal positief waren. Hier stelt zich hetzelfde probleem
als bij UP en kan nog geen uitspraak worden gedaan omtrent de efficiëntie.
De efficiëntie vertoond uiteenlopende waarden waarvan de meeste binnen de aanvaardbare
grenzen liggen. De uitschieter van HSV2 #1 wordt verwijderd van de TAC-STD array card
door de onaanvaardbaar hoge efficiëntie. Daarnaast kon de efficiëntie van UP en UU nog niet
bepaald worden doordat de resultaten bij de LOD werden bekomen en dus geen accurate
metingen hebben plaatsgevonden om de efficiëntie te bepalen.
107
Targetgen MG pdhD zal nog verder worden bekeken maar de verhoogde %CV (besproken bij
4.3.3) kan ook aan de basis liggen van een verminderde efficiëntie. De andere targets vertonen
een aanvaardbare efficiëntie.
4.3.6 Specificiteit
Tijdens de optimalisatie werd de specificiteit nagegaan van alle Real-Time PCR-reacties ten
opzichte van een reeks met bacteriële stammen die simultaan in hetzelfde klinische monster
kunnen aanwezig zijn. Deze stammen werden samengebracht in verschillende mixen (zie
3.7.2 tabel 8) en getest met de TAC-STD array card. De stammen specifiek voor N.
meningitidis en S. aureus worden elk apart getest omdat hier bij de optimalisatie problemen
waren met kruisreactie (zie 3.7.2 tabel 9).
Er werd geen kruisreactie gedetecteerd bij de 43 verschillende Real-Time PCR-reacties,
behalve voor UU ABI, NG OmpAVK en alle targetgenen van UP.
- NG Omp AVK
De vijf stammen voor N. meningitidis werden als positief gedetecteerd met de NG Omp AVK
PCR (zie figuur 32). Alle andere NG-PCR’s vertoonden geen kruisreactie met N.
meningitidis, S. aureus en de bacteriële stammen. Deze target zal bij een volgende versie van
de TAC-STD array card weggelaten worden.
108
Figuur 32: Kruisreactie NG Omp AVK
- UU ABI
Tijdens het testen van de herhaalbaarheid en reproduceerbaarheid werden vals-positieve
resultaten bekomen bij UU ABI (zie bijlage 8). De targetsequentie is blijkbaar speciesspecifiek maar maakt geen onderscheid tussen UU en UP. Deze PCR werd verkregen via de
firma Applied Biosystems en zal verwijderd worden van de TAC-STD array card.
- UP
Alle targetgenen van U. parvum vertoonden vals-positieve resultaten bij het testen van de
herhaalbaarheid en reproduceerbaarheid (zie bijlage 8). Dit werd toegewezen aan het feit dat
de gebruikte mixen hiervoor bestaan uit positief klinische stalen die niet hoofdzakelijk zijn
getest op U. parvum en hier wel positief kunnen voor zijn aangezien dit organisme vaak
voorkomt.
We kunnen stellen dat als de target NG Omp AVK en UU ABI van de TAC-STD array card
verwijderd worden, de gewenste specificiteit wordt bekomen.
4.3.7 Juistheid
Kwaliteitscontroles voor beide methoden werden gelijktijdig uitgevoerd. Om een goede
juistheid te bekomen, moeten de kwaliteitscontroles een juist resultaat weergeven (zie tabel
25).
109
Tabel 25: Kwaliteitscontroles met BD ProbTec en TAC-STD array card
kwaliteitscontrole
BD
CT
QCMD 14 NG-01
QCMD 14 NG-02
QCMD 14 NG-03
QCMD 14 NG-04
QCMD 14 NG-05
QCMD 14 NG-06
QCMD 14 NG-07
QCMD 14 NG-08
QCMD 14 NG-09
QCMD 14 NG-10
Urine
Urine
Urine
Urine
Urine
Urine
swab
swab
swab
swab
neg
pos (39945)
neg
neg
neg
neg
neg
neg
neg
neg
NG
pos (52086)
neg
neg
pos (31700)
pos (49335)
neg
neg
neg
neg
neg
TAC-STD
CT
neg
pos (33-37)
neg
neg
neg
neg
neg
neg
pos (31-35)
neg
NG
pos (29-34)
neg
neg
pos (32-32 )
pos (29)
neg
neg
pos (39)
neg
neg
Bij de resultaten bekomen met de kwaliteitscontroles in urine werden alle resultaten
bevestigd. Bij de swabs daarintegen werden bij de TAC-STD array card twee extra positieve
resultaten bekomen. Ct-waarde 39 is zeer zwak positief en kan niet gedetecteerd geweest zijn
door de BD ProbeTec-methode, maar Ct-waarde 31-35 is duidelijk positief (meerdere targets
waren hier positief) en deze controle is dan ook duidelijk gemist door de BD ProbeTec
methode. Het finaal rapport moet afgewacht worden om hier een duidelijk oordeel over te
vellen. Door een positieve reactie in meerdere targetgenen zijn we echter overtuigd van de
positiviteit van de TAC-STD array card die een hogere gevoeligheid aangeeft ten opzichte
van de huidige BD ProbeTec methode
Naast de kwaliteitscontroles moeten minstens 20 stalen getest worden (7 positieve, 6 laag
positieve en 7 negatieve) met beide methoden. De resultaten hiervan zijn terug te vinden in
bijlage 11 “Juistheid”.
61 stalen zijn in het dubbel geanalyseerd. De 17 (zwak) positieve stalen werden
geconfirmeerd met de TAC-STD array card. Bijkomstig werden binnen deze set positieve
stalen ook nog positief resultaat bekomen voor 1x MG, 4x MH, 2x UU, 6x UP, 1x TP en 1x
HSV II met de TAC-STD array card. De 38 stalen die negatief waren met de BD ProbeTecmethode werden geconfirmeerd en 21 van de 38 stalen waren positief voor één of meerdere
van de andere parameters op de TAC-STD array card (1x MG, 5x MH, 3x UU, 12x UP, 1x
TV, 2x HSV I en 1x HSV II).
De juistheid werd enkel nog maar geanalyseerd voor N. gonorrhoeae (en C. trachomatis) op
basis van 10 QCMD stalen en 54 klinische stalen. Alle werden correct gedetecteerd door de
TAC-STD array card (100 % specificiteit) met zelf twee zwak positieve resultaten extra
gedetecteerd in de QCMD stalen.
110
Hierdoor verwachten we een betere gevoeligheid voor de TAC-STD array card ten opzichte
van de BD ProbeTec-methode. Voor de andere pathogenen zullen in de toekomst alle
beschikbare QCMD stalen voor de overige organismen geanalyseerd worden met de TACSTD array card, inclusief klinische stalen geanalyseerd door een referentietechniek. Dit
onderdeel is dus nog verder af te werken.
4.3.8 Bewaarcondities
- Kamertemperatuur (25 °C)
De resultaten zijn terug te vinden in bijlage 12 “Bewaarcondities”.
De targetgenen van U. parvum en U. urealyticum vertoonden een minder goede werking ten
opzichte van de resultaten van herhaalbaarheid en reproduceerbaarheid. Er werden meer
negatieve resultaten bekomen. Bij het matrix geïnduceerd effect werd ook een negatieve
invloed waargenomen voor deze targetgenen, dus dit is niet per se te wijten aan de
bewaarcondities.
In urine 3 bleek de bewaarconditie ook een negatieve invloed te hebben. Naast de targetgenen
van U. parvum en U. urealyticum, vertoonden de targetgenen van Herpes Simplex Virus II en
Neisseria gonorrhoeae meer negatieve resultaten.
Hieruit kan besloten worden dat bewaren op kamertemperatuur een negatieve invloed kan
hebben op een urinestaal, wat in rekening moet gebracht worden.
- Koelkast (4 °C)
Nog geen resultaten wegens tekort aan TAC-STD array cards.
- Diepvries (-30 °C)
Nog geen resultaten wegens tekort aan TAC-STD array cards.
4.3.9 Vergelijking van methoden
De BD ProbeTec-methode werd vergeleken met de TAC-STD array card zoals beschreven in
3.7.2. De resultaten hiervan zijn terug te vinden in bijlage 13 “Vergelijking van methoden”.
De verdunningsreeksen die getest werden met de BD ProbeTec-methode, genereerden
volgend kwalitatief resultaat (tabel 26):
111
Tabel 26: Resultaat met BD ProbeTec-methode
Verdunning
NG
-1
+
10
-2
+
10-3
+
-4
+
-5
+
10
10
10
De resultaten werden kwalitatief bevestigd maar kwantitatief kon er geen vergelijking
bekomen worden.
De klinische stalen die hertest zijn met de TAC-STD array card (zie bijlage 13) bevestigden
ieder positief resultaat. Twijfelachtige resultaten met de BD ProbeTec methode bleken
overduidelijk positief met de TAC-STD array card wat te wijten kan zijn aan een hogere
gevoeligheid van de TAC. Daarnaast werden er ook telkens bij de meeste klinische stalen,
extra pathogenen gedetecteerd die met de BD ProbeTec-methode niet kunnen aangetoond
worden.
De negatieve stalen die opnieuw werden getest met de TAC-STD array card, zijn ook
allemaal negatief met een paar uitzonderingen waarbij er toch detectie is maar met een hoge
Ct-waarde, wat ook nog eens duidt op een betere gevoeligheid van de TAC.
Hieruit besluiten we dat de TAC-STD array card gevoeliger is dan de BD ProbeTec-methode,
wat ook werd aangetoond bij de juistheid van de methode (zie 4.3.7). De zwak positieve
kwaliteistcontroles werden nog altijd gedetecteerd net zoals de zwak positief klinische stalen
duidelijk positief zijn bij de TAC-STD array card. Ook worden alle positieve resultaten met
de BD ProbeTec methode bevestigd. De TAC-STD array card kan de huidige methode
vervangen op basis van de goede prestatiekenmerken en de beduidende grotere hoeveelheid
aan informatie die verkregen wordt voor de andere pathogenen.
4.3.10 Interne controles
Op de TAC-STD array card staan drie interne controles.
a) Extractie en inhibitiecontrole: Seal Herpes Virus
SHV wordt met een bepaalde concentratie toegevoegd aan ieder staal tijdens extractie. Dit wil
dan ook zeggen dat SHV met een bepaalde Ct-waarde moet teruggevonden worden indien er
geen inhibitie optreedt. Over 205 runs (zie bijlage 14 “Interne controles”) wordt een
gemiddelde Ct-waarde bekomen van Ct 29,34 met (std=1,04).
112
Bij deze waarde wordt er nog 3,32 Ct bijgerekend wat overeenkomt met ongeveer 1 log10
verschil. Afgerond wordt een Ct-waarde 33 bekomen (zie figuur 34). Dus voor de TAC-STD
array card wordt inhibitie vastgesteld bij SHV > Ct 33. Indien er toch positieve waarden
gedetecteerd worden bij SHV> 33 zal dit wel correct zijn, maar kunnen we mogelijke andere
pathogenen die aanwezig zijn in lagere concentraties, missen.
SHV
100
aantal
80
60
SHV
40
gem
-3SD
20
+3SD
0
24
25
26
27
28
29
30
31
32
33
34
35
Ct
Figuur 33: Resultaten inhibitie/extractiecontrole
b) Staalcontroles
Aan de hand van deze controles kan nagegaan worden of er een voldoende hoeveelheid
celmateriaal aanwezig is om een efficiënte PCR-reactie op uit te voeren. 3 staalcontroles
worden momenteel uitgevoerd op de TAC-STD array card:
HS04260458_s1
Het prefix Hs verwijst naar de species Homo sapiens en het suffix _s staat voor de volgende
definitie: “Een assay waarbij de primers en probes zijn ontworpen binnen een exon.
Dergelijke assays detecteren per definitie genomisch DNA.”
RNAseP
Ribonuclease P gen wordt gebruikt als targetsequentie om humaan DNA op te sporen.
18S-Hs99999901_s1
18S rRNA is eukaryoot. De primers en probe liggen in een enkel exon en de assay zal
genomisch DNA detecteren. Deze controle wordt steeds op de kaart gespot door LifeTech als
kwaliteitscontrole van hun productieproces.
113
Het resultaat van deze drie controles is te zien op figuur 34. RNAseP en HS04260458_s1 zijn
beide bruikbaar ter beoordeling van de integriteit van het staal. 18S-Hs99999901_s1 zal niet
bruikbaar zijn door de te lage Ct-waarden. De voorkeur gaat uit naar HS04260458_s1 wegens
de grotere frequentie aan waarden tussen Ct-waarde 20 en Ct-waarde 23. De Ct-waarden bij
RNAseP zijn meer gespreid zoals te zien aan de minder scherpe piek op figuur 34.
aantal
controles
20
18
16
14
12
10
8
6
4
2
0
Hs04260458
RNAse P
18S-Hs99999901_s1
9 101112131415161718192021222324252627282930
Ct
Figuur 34: Resultaten staalcontroles
Alhoewel het aantal urinestalen nog steeds vrij beperkt is, en een opvolgstudie dit zal moeten
confirmeren, blijkt uit de resultaten dat urines steeds hogere Ct-waarden hebben. Na
concentratie van urinestalen blijkt dat dit een aanleiding geeft tot een verschuiving van
ongeveer 3 Ct. Wanneer stalen (en vooral urinestalen) een Ct-waarde hebben die groter is dan
30, worden deze als “ongeschikt staal” gerapporteerd. Als dit het geval is voor urinestalen, zal
het staal hoogstwaarschijnlijk een midstream urine zijn in plaats van een first voided urine.
Verdere validatie zal nodig zijn om na te gaan als extreme bewaarcondities een invloed
hebben op de staalcontroles waardoor de Ct-waarde groter wordt dan 30.
c) TAC controle
18S-Hs99999901_s1: Het 18S rRNA is eukaryoot. De primers en probes liggen in een enkel
exon en de assay zal genomisch DNA detecteren. Deze controle wordt steeds op de kaart
gespot door LifeTech als kwaliteitscontrole van hun productieproces.
114
5
Besluit
We zijn gestart met het selecteren van speciesspecifieke targetgenen (cfr bachelorproef) voor
de zes behandelde pathogenen. De primers voor deze targetgenen werden eerst getest op
functionaliteit, specificiteit en gevoeligheid met de SYBR Select MasterMix. Op basis van de
gevoeligheid werden dan twee primerparen per organisme geselecteerd. De probes behorend
bij de geselecteerde primerparen werden hiena getest op functionaliteit, specificiteit en
gevoeligheid met de TaqMan Fast Virus 1-step MasterMix. Hier werd kruisreactie
waargenomen bij bepaalde primer-probe combinaties waarna deze werden aangepast. Na deze
aanpassingen was de specificiteit terug 100 % en kon overgegaan worden naar de validatie.
De bedoeling van deze voorafgaande optimalisatie was om op een kostenefficiënte manier
Real-Time PCR’s te selecteren die op de TAC-STD array card kunnen gespot worden. Een
TaqMan Low Density Array Card is namelijk een zeer duur gegeven en door op deze manier
te werken is er een grotere zekerheid van effectief werkende Real-Time PCR’s op de TACSTD array card.
Uit de validatie bleek dat de TAC-STD array Card een aantal targetgenen bevatte die ofwel
kruisreactie vertoonden ofwel zeer inefficiënt bleken te zijn. Die targetgenen zullen
verwijderd worden bij een volgende versie waarna de vrijgekomen wells kunnen aangevuld
worden met nieuwe targets. We bekomen een methode die gevoeliger is dan de BD ProbeTecmethode en een hoge specificiteit handhaaft dankzij meerdere targets per pathogeen.
Daarnaast zullen met deze methode simultaan 11 pathogenen opgespoord worden tegenover
de 2 pathogenen die opgespoord worden met de BD ProbTec-methode. Ook is de methode
zeer gecontroleerd met de drie verschillende interne controles waar er bij andere commerciële
kits vaak maar twee aanwezig zijn.
Tot slot dient nog vermeld te worden dat de validatie nog steeds voortgaand is. Bepaalde
aspecten dienen nog afgewerkt te worden en nieuwe targets die op de volgende versie van de
TAC-STD array card geplaatst worden, dienen opnieuw gevalideerd te worden. Eenmaal de
validatie afgerond is, zal deze methode gebruikt worden in de routine van het AZ Sint-Jan
Brugge-Oostende en zal de BD ProbeTec-methode hierdoor vervangen worden.
115
6
Bibliografische lijst
AB applied biosystems by life technologies. (2010). TaqMan Gene Expression Assay Protocol [pdf].
AB applied biosystems by life technologies. (2011). Applied Biosystems Viia 7 Real-Time
PCR System – Getting Started Guides [pdf].
AB applied biosystems by life technologies. (2011). Applied Biosystems Viia 7 Real-Time
PCR System – User Guide [pdf].
AB applied biosystems by life technologies. (2011). Real-time PCR: Understanding Ct.
[application note]. Life Technologies Corporation.
AB applied biosystems by life technologies. (2011). TaqMan Fast Virus 1-Step MasterMix
[pdf]. Geraadpleegd via
http://tools.lifetechnologies.com/content/sfs/brochures/cms_085892.pdf;
http://tools.lifetechnologies.com/content/sfs/manuals/cms_084608.pdf;
http://tools.lifetechnologies.com/content/sfs/manuals/cms_083989.pdf.
AB applied biosystems by life technologies. (2013). SYBR Select Master Mix – User Guide
[pdf].
Alfa, M. (2005). The laboratory diagnosis of Haemophilus ducreyi. The Canadian Journal of
Infectious Diseases & Medical Microbiology, 16(1), 31.
Asuragen. (2012). AB TaqMan Gene Expression Services. Geraadpleegd in februari 2014 via
http://asuragen.com/
BacMap: an interactive atlas for exploring bacterial genomes – Haemophilus ducreyi. (2013).
Geraadpleegd op 13 februari 2014 via http://wishart.biology.ualberta.ca
BacMap: an interactive atlas for exploring bacterial genomes – Ureaplasma urealyticum.
(2013). Geraadpleegd op 13 februari 2014 via http://wishart.biology.ualberta.ca
Biswas, D., Borkakoty, B., Mahanta, J., Walia, K., Saikia, L., Akoijam, B. S., ... & Zomawia,
E. (2011). Seroprevalence and risk factors of herpes simplex virus type-2 infection among
pregnant women in Northeast India. BMC infectious diseases, 11(1), 325.
116
Boyce, J. M., & Mitchell, E. B. (1985). Difficulties in differentiating Neisseria cinerea from
Neisseria gonorrhoeae in rapid systems used for identifying pathogenic Neisseria
species. Journal of clinical microbiology, 22(5), 731-734.
Cao, X., Wang, Y., Hu, X., Qing, H., & Wang, H. (2007). Real-time TaqMan polymerase
chain reaction assays for quantitative detection and differentiation of Ureaplasma urealyticum
and Ureaplasma parvum. Diagnostic microbiology and infectious disease, 57(4), 373-378.
Centers for Disease Control and Prevention (CDC). (2010). Sexually transmitted diseases
treatment guidelines, 2010. Department of Health and Human Services, Centers for Disease
Control and Prevention.
De Schrijver, K. (2004). De epidemiologie van seksueel overdraagbare aandoeningen in
België. FARMACEUTISCH TIJDSCHRIFT VOOR BELGIE, (1), 13-19.
Descheemaeker, P. (2013). QIAsymphony [SOP]. AZ Sint-Jan Brugge.
EMBL-EBI. Bacteria Genomes – Haemophilus ducreyi. (2013). Geraadpleegd op 13 februari
2014 via http://www.ebi.ac.uk
Geraats-Peters, C. W. M., Brouwers, M., Schneeberger, P. M., van der Zanden, A. G. M.,
Bruisten, S. M., Weers-Pothoff, G., ... & Hermans, M. H. A. (2005). Specific and sensitive
detection of Neisseria gonorrhoeae in clinical specimens by real-time PCR. Journal of
clinical microbiology, 43(11), 5653-5659.
Haggerty, C. L., & Taylor, B. D. (2011). Mycoplasma genitalium: an emerging cause of
pelvic inflammatory disease. Infectious diseases in obstetrics and gynecology, 2011.
High Resolution Melt. (2014). In Wikipedia. Geraadpleegd in april 2014 via
http://en.wikipedia.org/wiki/High_Resolution_Melt
Hjelmevoll, S. O. (2012). Molecular diagnostics and characterization of Neisseria
gonorrhoeae.
JM, F., & Squifflet, J. M. (2011). Pelvic Infections in Women: Belgian guidelines.
Katsura, C., Larsen, R., Pogliano, K. Ureaplasma parvum. (2010). Geraadpleegd op 13
februari 2014 via http://microbewiki.kenyon.ecu
117
Kemp, M., Christensen, J. J., Lautenschlager, S., Vall-Mayans, M., & Moi, H. (2011).
European guideline for the management of chancroid, 2011.International journal of STD &
AIDS, 22(5), 241-244
Kenneth, T. (2012). Pathogenic Neisseriae: Gonorrhea. Todar’s Online Textbook of
Bacteriology. Geraadpleegd op 13 februari 2014 via http://textbookofbacteriology.net
Kim, S. J., Lee, D. S., & Lee, S. J. (2011). The prevalence and clinical significance of
urethritis and cervicitis in asymptomatic people by use of multiplex polymerase chain
reaction. Korean journal of urology, 52(10), 703-708.
Kodani, M., Yang, G., Conklin, L. M., Travis, T. C., Whitney, C. G., Anderson, L. J., ... &
Fields, B. S. (2011). Application of TaqMan low-density arrays for simultaneous detection of
multiple respiratory pathogens. Journal of clinical microbiology, 49(6), 2175-2182.
Kruhøffer, M., Voss, T., Beller, K., Scherer, M., Cramer, J., Deutschmann, T., ... & Lenz, C.
(2010). Evaluation of the QIAsymphony SP Workstation for Magnetic Particle—Based
Nucleic Acid Purification from Different Sample Types for Demanding Downstream
Applications. Journal of the Association for Laboratory Automation, 15(1), 41-51.
Kutyavin, I. V., Afonina, I. A., Mills, A., Gorn, V. V., Lukhtanov, E. A., Belousov, E. S., ...
& Hedgpeth, J. (2000). 3′-minor groove binder-DNA probes increase sequence specificity at
PCR extension temperatures. Nucleic acids research, 28(2), 655-661.
Life Technologies. (2014). RNaseOUT Recombinant Ribonuclease Inhibitor. Geraadpleegd in
maart 2014 via http://www.lifetechnologies.com/order/catalog/product/10777019
LightCycler Real-time PCR. (2014). Geraadpleegd in februari 2014 via
https://dna.utah.edu/LightCycler/Top_LightCycler.html
Liu, A. Y., Jiang, M. J., Yin, Y. P., & Sun, J. F. (2005). Detection of pathogens causing
genital ulcer disease by multiplex polymerase chain reaction. Chinese medical sciences
journal= Chung-kuo i hsueh k'o hsueh tsa chih/Chinese Academy of Medical Sciences, 20(4),
273-275.
Mallard, K., Schopfer, K., & Bodmer, T. (2005). Development of real-time PCR for the
differential detection and quantification of Ureaplasma urealyticum and Ureaplasma
parvum. Journal of microbiological methods, 60(1), 13-19.
118
Melting Curve Analysis.(2013). In Wikipedia. Geraadpleegd in februari 2014 via
http://en.wikipedia.org/wiki/Melting_curve_analysis
Miltenyi Biotec GmbH. (2014). GentleMACS C Tubes [pdf]. Geraadpleegd via
https://www.miltenyibiotec.com/~/media/Images/Products/Import/0001400/IM0001425.ashx
Müller, E. E., Venter, J. M., Magooa, M. P., Morrison, C., Lewis, D. A., & Mavedzenge, S.
N. (2012). Development of a rotor-gene real-time PCR assay for the detection and
quantification of Mycoplasma genitalium. Journal of microbiological methods, 88(2), 311315.
Nollet, F. (2013). Viia7 Life Technologies [SOP]. AZ Sint-Jan Brugge
Norberg, P. (2010). Divergence and genotyping of human α-herpesviruses: An
overview. Infection, genetics and evolution, 10(1), 14-25.
Orle, K. A., Gates, C. A., Martin, D. H., Body, B. A., & Weiss, J. B. (1996). Simultaneous
PCR detection of Haemophilus ducreyi, Treponema pallidum, and herpes simplex virus types
1 and 2 from genital ulcers. Journal of Clinical Microbiology, 34(1), 49-54.
Pestana, E. A., Belak, S., Diallo, A., Crowther, J. R., & Viljoen, G. J. (2010). Real-Time
PCR–The Basic Principles. In Early, rapid and sensitive veterinary molecular diagnosticsreal time PCR applications (pp. 27-46). Springer Netherlands.
Polymerasekettingreactie. (2014). In Wikipedia. Geraadpleegd in februari 2014 via
http://nl.wikipedia.org/wiki/Polymerasekettingreactie
Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., El Housni, H., Descheemaeker, P., ...
& Vankeerberghen, A. (2011). Reflections and proposals to assure quality in molecular
diagnostics. Acta Clinica Belgica,66(1), 33-41.
Ricotta, E. E., Wang, N., Cutler, R., Lawrence, J. G., & Humphreys, T. L. (2011). Rapid
divergence of two classes of Haemophilus ducreyi. Journal of bacteriology, 193(12), 29412947.
Ross, J. D. C., & Jensen, J. S. (2006). Mycoplasma genitalium as a sexually transmitted
infection: implications for screening, testing, and treatment.Sexually transmitted
infections, 82(4), 269-271.
119
Skovgaard, S., Kiellberg Larsen, H., Sand, C., Friis-Møller, A., Schønning, K., Skov Jensen,
J. 3., & Westh, H. (2012). Genital and extra-genital screening for gonorrhoea using the BD
Probetec ET system with an in-house PCR method targeting the porA pseudogene as
confirmatory test. Acta dermato-venereologica, 92(1).
Steg, A., Wang, W., Blanquicett, C., Grunda, J. M., Eltoum, I. A., Wang, K., ... & Johnson,
M. R. (2006). Multiple gene expression analyses in paraffin-embedded tissues by TaqMan
low-density array: Application to hedgehog and Wnt pathway analysis in ovarian
endometrioid adenocarcinoma. The Journal of Molecular Diagnostics, 8(1), 76-83.
Suntoke, T. R., Hardick, A., Tobian, A. A. R., Mpoza, B., Laeyendecker, O., Serwadda, D., ...
& Reynolds, S. J. (2009). Evaluation of multiplex real-time PCR for detection of
Haemophilus ducreyi, Treponema pallidum, herpes simplex virus type 1 and 2 in the
diagnosis of genital ulcer disease in the Rakai District, Uganda. Sexually transmitted
infections, 85(2), 97-101.
SYBR Green. (2013). In Wikipedia. Geraadpleegd op 13 februari 2014 via
http://wikipedia.org/wiki/SYBR_Green
Taylor-Robinson, D., & Jensen, J. S. (2011). Mycoplasma genitalium: from Chrysalis to
multicolored butterfly. Clinical microbiology reviews, 24(3), 498-514.
Tekle-Michael, T., Van Dyck, E., Abdellati, S., & Laga, M. (2001). Development of a
heminested polymerase chain reaction assay for the detection of Haemophilus ducreyi in
clinical specimens. International journal of STD & AIDS, 12(12), 797-803.
Teo, I. A., Choi, J. W., Morlese, J., Taylor, G., & Shaunak, S. (2002). LightCycler qPCR
optimisation for low copy number target DNA. Journal of immunological methods, 270(1),
119-133.
Ureaplasma urealyticum.com. (2013). Geraadpleegd op 13 februari 2014 via
http://www.ureaplasmaurealyticum.com
Van Doornum, G. J. J., Guldemeester, J., Osterhaus, A. D. M. E., & Niesters, H. G. M.
(2003). Diagnosing herpesvirus infections by real-time amplification and rapid
culture. Journal of clinical microbiology, 41(2), 576-580.
Van Gool, A. P. (1997). Van nucleotide tot genoom. Het genetisch elan. Garant.
120
Vancutsem, E., Soetens, O., Breugelmans, M., Foulon, W., & Naessens, A. (2011). Modified
Real-Time PCR for Detecting, Differentiating, and Quantifying Ureaplasma urealyticum and
Ureaplasma parvum. The Journal of Molecular Diagnostics, 13(2), 206-212.
Vanderstraeten, V. (2010). Genetische diagnostiek van 15 volledige doofheidsgenen via
second generation sequencing [afstudeerwerk]. Universiteit Gent, Faculteit Farmaceutische
Wetenschappen.
Vandesompele, J. (2009). qPCR guide. [pdf]. Geraadpleegd via http://www. eurogentec.
com/uploads/qPCR-guide
Wagner, E. (2003). Herpes Simplex Virus general properties. Geraadpleegd op 13 februari
2014 via http://darwin.bio.uci.edu
Zheng, J., Jin, H., Xingang, L., Yuquan, J., Wei, Z., & Daru, L. (2006). Expression analyses
of 27 DNA repair genes in astrocytoma by TaqMan low-density array. Neuroscience
letters, 409(2), 112-117.
121
7
Bijlagen
Alle bijlagen zijn terug te vinden op bijgevoegde CD-ROM wegens de omvang van de
tabellen. Onderstaand overzicht geeft de volgorde van de bijlagen weer.
Bijlage 1: Bacteriële stammen (negatief referentiemateriaal)
Tabblad 1: Bacteriële stammen testing = Bacteriële stammen getest met de in deze
thesis geselecteerde primers en probes.
Tabblad 2: Resultaten met alle primerparen = Bacteriële stammen getest met alle
geselecteerde primers en probes die op de TAC zullen geplaatst worden.
Bijlage 2: Klinische monsters (positief referentiemateriaal)
Tabblad 1: Positieve klinische stalen (1) = Positieve klinische stalen getest met de in
deze thesis geselecteerde primers en probes.
Tabblad 2: Positieve klinische stalen (2) = Positieve klinische stalen getest met de
primers en probes geselecteerd door Pieters Jeroom (zie gelijklopend onderzoek
“Ontwikkeling van een TaqMan Array Card voor de simultane detectie van seksueel
overdraagbare pathogenen”)
Bijlage 3: Fluorescentie
Tabblad 1: Verdunningsreeksen Fluorescentie = Resultaten omtrent de fluorescentie
van de geselecteerde primers en probes in deze thesis.
Bijlage 4: Primers en probes
Tabblad 1: Gegevens = Alle theoretische gegevens omtrent ampliconlengte,
primerlengte, probelengte, GC%, Tm amplicon, dimeervorming …
Bijlage 5: Selectie primerparen
Tabblad 1: Primerparen = Resultaten omtrent de gevoeligheid. Hieruit werden de
gevoeligste primerparen geselecteerd (aangeduid in groen). Onderaan het bestand zijn
de grafieken terug te vinden in verband met de gevoeligheid.
Bijlage 6: Limit of Detection
Tabblad 1: LOD = Resultaten van de limit of detection voor alle targets op de TACSTD array card.
Bijlage 7: Ct-waarden klinische monsters
Tabblad 1: Ct-waarden = De bekomen Ct-waarden van positief klinische stalen om
mengstaal mix 1 en mengstaal mix 2 te maken.
122
Bijlage 8: Herhaalbaarheid en reproduceerbaarheid
Tabblad 1: Reproduceerbaarheid = Resultaten van de runs over twee dagen.
Tabblad 2: Herhaalbaarheid = Resultaten van 3 runs over eenzelfde dag.
Tabblad 3: R en H samen = Resultaten van herhaalbaarheid en reproduceerbaarheid in
één tabel.
Bijlage 9: Matrix geïnduceerd effect
De tabellen zijn gelijkaardig opgemaakt als deze van herhaalbaarheid en reproduceerbaarheid.
Tabblad 1: Eswab herhaalbaarheid = Resultaten van de herhaalbaarheid in Eswab als
matrix.
Tabblad 2: Preserve Cyt herhaal = Resultaten van de herhaalbaarheid in PreservCyt als
matrix.
Tabblad 3: UTM herhaal = Resultaten van de herhaalbaarheid in UTM als matrix.
Tabblad 4: Urine2 herhaal = Resultaten van de herhaalbaarheid in urine als matrix.
Tabblad 5: Urine3 herhaal = Resultaten van de herhaalbaarheid in urine als matrix.
Tabblad 6: Urine 6 herhaal = Resultaten van de herhaalbaarheid in urine als matrix.
Bijlage 10: Efficiëntie
Tabblad 1: R en H samen = De resultaten van herhaalbaarheid en reproduceerbaarheid
worden samen gezet en het gemiddelde wordt berekend (samen met de
standaarddeviatie). Uit deze resultaten wordt de grafiek opgesteld waarvan de helling
wordt gebruikt om de efficiëntie te berekenen van iedere PCR.
Bijlage 11: Juistheid
Tabblad 1: Klinische stalen = Het aantal klinische stalen die in het dubbel zijn getest
om de juistheid na te gaan.
Tabblad 2: QCMD = De geteste kwaliteitscontroles voor NG en CT.
Bijlage 12: Bewaarcondities
Tabblad 1: 25 °C = Resultaten van de geteste bewaarconditie bij kamertemperatuur.
Tabblad 2: 4 °C = Resultaten nog niet bekomen bij 4 °C.
Tabblad 3: -30 °C = Resultaten nog niet bekomen bij -30 °C.
Bijlage 13: Vergelijking van methoden
123
Tabblad 1: Klinische stalen = De klinische stalen die gebruikt zijn om beide methoden
te vergelijken met elkaar.
Tabblad 2: Verdunning met BD = De resultaten van mengstaal mix 1 en mengstaal
mix 2 getest met de BD ProbeTec methode.
Bijlage 14: Interne controles
Tabblad 1: Ruwe data = De ruwa data van alle runs
Tabblad 2: Analyse
Tabblad 3: Grafiek analyse
Tabblad 4: Analyse controles
Tabblad 5: Grafiek analyse controles
124