FACULTEIT BIO-INGENIEURSWETENSCHAPPEN
Academiejaar 2004 – 2005
KLONERING EN GENEXPRESSIE
IN DE SCHIMMEL MYROTHECIUM GRAMINEUM
Manu DE GROEVE
Promotoren :
Prof. dr. ir. Wim Soetaert
Prof. dr. ir. Erick J. Vandamme
Scriptie voorgedragen tot het behalen van de graad van
BIO-INGENIEUR IN DE CEL- EN GENBIOTECHNOLOGIE
De auteur en de promotoren geven de toelating deze scriptie voor consultatie beschikbaar te
stellen en delen ervan te kopiëren voor persoonlijk gebruik.
Elk ander gebruik valt onder de beperkingen van het auteursrecht, in het bijzonder met
betrekking tot de verplichting uitdrukkelijk de bron te vermelden bij het aanhalen van
fragmenten uit deze scriptie.
The author and the promotors give the permission to use this thesis for consultation and to
copy parts of it for personal use.
Every other use is subject to the copyright laws, more specifically the source must be
extensively specified when using results from this thesis.
Manu DE GROEVE
Begeleidster:
ir. Sofie DE MAESENEIRE
Promotoren:
Prof. dr. ir. Wim SOETAERT
Prof. dr. ir. Erick J. VANDAMME
23 mei 2005
WOORD VOORAF
Als nucleotide begon ik vijf jaar geleden aan de studies bio-ingenieur. Door de jaren heen
werd mijn kennis meer en meer uitgebreid zodat ik uiteindelijk als volwaardig gen aan mijn
thesis kon beginnen. In dit laatste jaar werd dit gen dan tot (over)expressie gebracht. Het
resulterende product heeft U nu voor zich liggen. Klaar om gelezen te worden.
Vooreerst zou ik Prof. dr. ir. Erick Vandamme en Prof. dr. ir. Wim Soetaert van het Labo
voor Industriële Microbiologie en Biokatalyse (LIMAB) oprecht willen bedanken om mij
kennis te laten maken met de boeiende wereld van de industriële biotechnologie. Hun inzet
voor de promotie van deze witte biotechnologie is bewonderenswaardig.
Daarnaast wens ik ir. Sofie De Maeseneire minstens evenveel te bedanken voor de goede
begeleiding en samenwerking gedurende de voorbije maanden. Meer in het bijzonder voor het
vertrouwen, het geduld en de vrijheid die ik sinds de eerste dag heb mogen genieten. Het was
telkens weer een plezier om in het labo te werken. Voor mij was het in ieder geval een
verrijkende ervaring.
Verder zou ik zeker Eline Geyselinck nog willen bedanken voor de hulp in het labo tijdens
haar stage. Zij was steeds het zonnetje in huis. Ook zou ik de anderen in het labo willen
bedanken, meer bepaald Marjan voor de genetische tips, Joeri voor de chemische tips, Katja
voor haar eeuwige goedgeluimdheid, Cassandra voor de humor, Marianne voor de technische
hulp en alle anderen die de sfeer op het labo zo goed maken.
Ik zou ook Ann willen bedanken voor de tips, de morele steun en haar eindeloze geduld.
Tenslotte wil ik nog mijn ouders en de ganse familie bedanken voor hun steun en het
aanbieden van een omgeving waarin ik me volledig heb kunnen ontplooien.
Manu De Groeve
Gent, 23 mei 2005
INHOUDSOPGAVE
I. INLEIDING .........................................................................................................................1
II. LITERATUURSTUDIE ....................................................................................................3
1. Industriële biotechnologie van schimmels.....................................................................3
2. De schimmel Myrothecium gramineum .........................................................................4
3. Transformatie methoden voor schimmels......................................................................6
3.1. Protoplast transformatie .........................................................................................7
3.2. Lithiumacetaat behandeling ...................................................................................8
3.3. Elektroporatie .........................................................................................................8
3.4. Biolistische transformatie.......................................................................................8
3.5. Agrobacterium tumefaciens-gemedieerde transformatie .......................................9
4. Opname van het transformerende DNA.......................................................................10
5. Selectiemerkers ............................................................................................................12
5.1. Soorten selectiemerkers........................................................................................12
5.2. Het OMPD gen als selectiemerker .......................................................................13
6. Heterologe eiwitproductie door schimmels .................................................................25
6.1. Structuur van fungale expressiesystemen ............................................................26
6.2. Transcriptionele controle......................................................................................26
6.3. Terminatie van transcriptie...................................................................................29
6.4. Secretie van heterologe eiwitten ..........................................................................30
6.5. Post-translationele modificaties ...........................................................................31
6.6. Reportergenen en expressiestudies.......................................................................31
6.7. Overproductie van heterologe eiwitten ................................................................32
6.8. Toekomstperspectieven ........................................................................................35
III. MATERIALEN EN METHODEN ...............................................................................36
1. Micro-organismen........................................................................................................36
1.1. Fungale stammen..................................................................................................36
1.2. Bacteriële stammen ..............................................................................................36
2. Cultuurmedia en groeicondities ...................................................................................36
2.1. M. gramineum MUCL 39210 en A. nidulans FGSC A722 ..................................36
2.2. E. coli DH5α-F’ ...................................................................................................37
3. Isolatie van nucleïnezuren............................................................................................38
3.1. Isolatie van genomisch DNA ...............................................................................38
3.2. Isolatie van totaal RNA ........................................................................................38
3.3. Isolatie van plasmiden ..........................................................................................39
3.4. Bepalen nucleïnezuurconcentratie en zuiverheid.................................................39
3.5. Concentreren van DNA ........................................................................................40
3.6. Zuiveren van DNA ...............................................................................................40
4. Gelelektroforese ...........................................................................................................40
4.1. Analytische gelelektroforese ................................................................................40
4.2. Extractie van DNA fragmenten uit agarosegel ....................................................41
5. Polymerase Chain Reaction (PCR) ..............................................................................42
5.1. Standaard Taq PCR ..............................................................................................42
5.2. High Fidelity PCR Master kit (Roche).................................................................43
5.3. Reamplificatie PCR..............................................................................................43
5.4. Synthese van cDNA .............................................................................................44
5.5. Kolonie-PCR ........................................................................................................44
5.6. Ligatie van PCR fragmenten in pGEM®-T Vector (Promega) ............................45
6. Restrictiedigest.............................................................................................................46
7. Ligatie ..........................................................................................................................47
8. Sequenering van DNA fragmenten ..............................................................................47
9. Transformatie van E. coli DH5α-F’.............................................................................48
9.1. Aanmaken van competente cellen van E. coli DH5α-F’ .....................................48
9.2. Transformatieprocedure voor E. coli DH5α-F’ ...................................................50
10. Klonering van het OMPD gen van Myrothecium gramineum MUCL 39210............50
10.1. Gedegenereerde PCR .........................................................................................50
10.2. Genomewalking: BD GenomeWalker™ Universal Kit (BD Biosciences) ........51
10.3. Klonering van het volledige OMPD gen............................................................54
11. Transformatie van A. nidulans FGSC A722 ..............................................................55
11.1. Oplossingen gebruikt voor de transformatie ......................................................55
11.2. Media..................................................................................................................55
11.3. Transformatieprotocol ........................................................................................57
12. Ultraviolet mutatie en filtratieaanrijking ...................................................................59
12.1. Ultraviolet mutatie..............................................................................................59
12.2. Filtratieaanrijking ...............................................................................................60
IV. RESULTATEN EN BESPREKING..............................................................................62
1. Klonering van het OMPD gen van Myrothecium gramineum MUCL 39210..............63
1.1. Ontwikkeling van gedegenereerde primers voor OMPD .....................................63
1.2. Gedegenereerde PCR op het OMPD gen .............................................................68
1.3. GenomeWalking naar 5’ en 3’ kant van het OMPD gen .....................................77
1.4. Klonering en sequenering van het volledige OMPD gen.....................................87
1.5. Studie van de DNA- en eiwitsequentie van OMPD .............................................89
2. Complementatie van A. nidulans FGSC A722 ..........................................................106
3. Creatie OMPD-knockout van M. gramineum ............................................................111
3.1. Ultraviolet (UV) mutatie en filtratieaanrijking ..................................................111
3.2. Constructie van een OMPD knock-out vector ...................................................114
V. BESLUIT.........................................................................................................................117
VI. SAMENVATTING .......................................................................................................119
VII. SUMMARY .................................................................................................................120
VIII. LITERATUURLIJST................................................................................................121
IX. BIJLAGE.......................................................................................................................131
1. Alignments .................................................................................................................131
1.1. Alignment cDNA en genomisch DNA van het OMPD gen...............................131
1.2. Alignment OMPD enzym M. gramineum met OMPD enzymen van andere
filamenteuze schimmels ............................................................................................132
2. De genetische code, gedegenereerde nucleotiden en afkortingen van aminozuren...133
I. Inleiding - 1
MANU DE GROEVE
I. INLEIDING
In hun natuurlijke omgeving zijn filamenteuze schimmels in staat een groot aantal
verschillende koolstof- en stikstofbronnen te gebruiken door de secretie van een grote
verscheidenheid aan enzymen. Samen met de capaciteit om deze enzymen in grote
hoeveelheden te secreteren maakt dit van filamenteuze schimmels aantrekkelijke gastheren
voor de productie van eiwitten op grote schaal.
Tot nog toe werden slechts een beperkt aantal fungale gastheren onderzocht voor
recombinante eiwitproductie (Punt et al., 2002). De moderne fungale biotechnologie is
competitief en trekt meer en meer de aandacht van industrieën buiten de traditionele
fermentatie-industrie die geïnteresseerd zijn in toepassingen van enzymen en andere eiwitten.
Het is dan ook niet verwonderlijk dat de ontwikkeling van nieuwe fungale expressiegastheren
als alternatief voor de reeds gepatenteerde de laatste jaren sterk gestegen is.
In het doctoraatsonderzoek waar deze thesis deel van uitmaakt is het de bedoeling een
geschikt transformatie- en expressiesysteem te ontwikkelen voor Myrothecium gramineum.
Deze filamenteuze schimmel werd geselecteerd na screening van 3000 verschillende fungale
soorten en stammen. M. gramineum bleek enkele interessante industriële eigenschappen te
bezitten waaronder hoge enzymproductie en goede groei op verschillende substraten. Er is
echter slechts zeer weinig gekend over deze schimmel wat het onderzoek bemoeilijkt.
Niettemin kon de schimmel reeds met succes getransformeerd worden. Het
transformatiesysteem voor M. gramineum werd geoptimaliseerd met een antibioticumresistentiegen als selectiemerker. Wanneer de schimmel echter zal gebruikt worden voor
enzymproductie in de voedingsindustrie is het gebruik van antibiotica-resistentiemerkers af te
raden.
Daarom werd gezocht naar andere selectiemerkers. Het enzym orotidine-5’-monofosfaat
decarboxylase (OMPD), dat een rol speelt in de pyrimidinesynthese, werd reeds vaak
toegepast als selectiemerker bij fungi en zal ook in dit onderzoek gebruikt worden. Het
gebruik van OMPD als selectiemerker steunt op het complementeren van een auxotrofe
mutant die OMPD negatief is. Zulke mutanten hebben uracil en/of uridine nodig voor groei.
Wanneer deze mutanten het getransformeerde OMPD gen dan tot expressie brengen wordt
hun auxotrofie opgehoffen en kunnen ze opnieuw groeien op medium zonder uracil of
uridine. Op deze manier kan het OMPD gen als selectiemerker gebruikt worden.
Het gebruik van homologe selectiemerkers kan de transformatie-efficiëntie sterk verhogen in
vergelijk met heterologe selectiemerkers. Bovendien kan het homologe OMPD gen ook
gebruikt worden voor de constructie van knock-out vectoren waarmee via transformatie
gedefinieerde OMPD-negatieve mutanten kunnen worden gecreëerd.
MANU DE GROEVE
I. Inleiding - 2
Omdat het OMPD gen meestal als single-copy aanwezig is kan dit gen ook gebruikt worden
als target bij promotorstudies. Voor zulke studies is het immers belangrijk dat de
expressiecassette op een gedefinieerde plaats in het genoom integreert in single-copy. Door de
expressiecassette in het OMPD gen te laten integreren kan de promotoractiviteit betrouwbaar
vergeleken worden tussen verschillende transformanten.
Het doel van deze scriptie is dan ook de klonering van het homologe OMPD gen van
Myrothecium gramineum. De bekomen nucleotiden- en aminozuursequentie zal bestudeerd
worden en vergeleken worden met deze van andere filamenteuze schimmels. De werking van
het OMPD gen van Myrothecium als selectiemerker zal aangetoond worden door
transformatie en complementatie van een OMPD-negatieve mutant van Aspergillus nidulans.
Ook zullen de pogingen tot het creëren van een OMPD-negatieve mutant van Myrothecium
besproken worden. Er werd getracht zulke mutanten te creëren via enerzijds klassieke
mutagenese, terwijl anderzijds ook OMPD knock-out vectoren werden geconstrueerd voor de
constructie van gedefinieerde mutanten via transformatie. Tot slot werd nog een kolonie-PCR
protocol voor Myrothecium gramineum ontwikkeld voor de snelle identificatie van de
gewenste transformanten.
MANU DE GROEVE
II. Literatuurstudie - 3
II. LITERATUURSTUDIE
1. INDUSTRIËLE BIOTECHNOLOGIE VAN SCHIMMELS
Schimmels en gisten worden reeds duizenden jaren door de mens gebruikt als fermented foods
en bij het maken van bier, wijn, brood en kaas. Het zijn de fermentatieproducten van
filamenteuze schimmels die een belangrijke rol hebben gespeeld in de ontwikkeling van de
hedendaagse industriële biotechnologie (Bennett, 1998). Op het einde van de 19de eeuw kon
met Aspergillus oryzae het enzym diastase (amylase) geproduceerd worden en in 1923 begon
Pfizer met de commerciële productie van citroenzuur door A. niger in de Verenigde Staten.
Maar de industriële microbiologie kwam pas echt tot ontwikkeling na de ontdekking van
penicilline in 1929. Deze ontdekking stimuleerde o.a. de zoektocht naar nieuwe antimicrobiële stoffen als ook het onderzoek naar fungale fysiologie, fermentatietechnologie en
industriële stamverbetering. Bijvoorbeeld in het geval van penicilline kon de opbrengst tot
meer dan 4000 maal verhoogd worden door klassieke stamverbetering (Parekh et al., 2000).
Zoals weergegeven in Tabel 1, worden filamenteuze schimmels momenteel vooral gebruikt
voor de industriële productie van primaire metabolieten (citroenzuur), secundaire
metabolieten (antibiotica) en enzymen.
Tabel 1. Industrieel gebruik van filamenteuze schimmels (Bennett, 1998)
Fermentatieproduct
Organismen
Antibiotica en andere farmaceutisch actieve stoffen
Penicillines
Penicillium chrysogenum
Cephalosporine
Cephalosporium acremonium
Cyclosporine
Tolypocladium inflatum
Ergot alkaloïden
Claviceps purpurea
Griseofulvine
Penicillium griseofulvin
Mevalonine
Aspergillus terreus
Enzymen
A. niger, A. oryzae
α-Amylasen
Cellulase
Humicola insolens, Penicillium funiculosum,
Trichoderma viride
Gluco-amylasen
Aspergillus phoenicis, Rhizopus delemar, R. niveus
Glucose oxidase
A. niger
Invertase
A. niger, A. oryzae
Laccase
Coriolus versicolor
Pectinase
A. niger, A. oryzae, Humicola insolens
Proteïnasen
A. oryzae, Aspergillus melleus, R. delemar
Rennine (microbieel)
Mucor miehei, M. pusillus
Organische zuren
Citroenzuur
A. niger
Itaconzuur
A. terreus
Filamenteuze schimmels, zoals Aspergillus en Trichoderma, blinken uit in hun capaciteit om
grote hoeveelheden eiwitten, metabolieten en organische zuren te secreteren in het
groeimedium (Conesa et al., 2001). Deze extracellulaire productie maakt de downstream
processing veel eenvoudiger in vergelijking met intracellulaire productie. Sommige
MANU DE GROEVE
II. Literatuurstudie - 4
filamenteuze schimmels worden reeds tientallen jaren gebruikt in de voedingsindustrie en
hebben de GRAS-status (Generally Recognised As Safe) verworven. Dit maak hen
aantrekkelijk als gastheer voor de synthese van nieuwe producten. Er zijn immers genetische
transformatietechnieken voorhanden waardoor deze filamenteuze schimmels gebruikt kunnen
worden voor heterologe eiwitproductie en de overproductie van homologe eiwitten. De meest
gebruikte schimmels hiervoor zijn Aspergillus nidulans, A. niger, A. oryzae en Trichoderma
reesei (Bennett, 1998). Hoewel met filamenteuze schimmels in industriële
fermentatieprocessen grote hoeveelheden homologe eiwitten kunnen worden geproduceerd, is
de opbrengst van heterologe eiwitten veel lager (Gouka et al., 1997).
Het commercieel gebruik van het fungale secretiesysteem en de limitaties ervan wat betreft
productie van heterologe eiwitten hebben het onderzoek naar de genetica van filamenteuze
schimmels bevorderd (Conesa et al., 2001). Algemeen wordt aangenomen dat de secretie
pathway in filamenteuze schimmels niet veel verschilt van deze in gisten en hogere
eukaryoten. Er zijn wel enkele duidelijke verschillen. Meer bepaald de specifieke groei van
filamenteuze schimmels als mycelium, dat resulteert uit de polaire extensie van de hyfale
uiteinden, wordt niet teruggevonden bij S. cerevisiae of hogere eukaryoten. Bovendien is de
eiwitsecretie capaciteit veel hoger dan bij gist. Met bepaalde Aspergillus en Trichoderma
stammen werden reeds opbrengsten tot 30 g/L extracellulair eiwit bekomen (Durand et al.,
1988; Finkelstein et al., 1989) terwijl slechts enkele gist soorten (Pichia, Hansenula) het g/L
productieniveau halen (Werten et al., 1999; Wyss et al., 1999).
2. DE SCHIMMEL MYROTHECIUM GRAMINEUM
In een preliminair onderzoek werd de BCCM/MUCL (agro)industriële schimmel- en
gistcollectie gescreend naar nieuwe potentiële fungale gastheren (Jonniaux et al., 2004). In
eerste instantie werden 3000 verschillende fungale soorten en stammen fungi van de MUCL
collectie geselecteerd op basis van hun niet-pathogeniciteit en het feit dat er nog geen
transformatiesysteem voor werd gerapporteerd in de wetenschappelijke literatuur of in
patenten. Deze stammen werden gecultiveerd in vloeibare cultuur en op vast medium. In een
eerste ronde werd geselecteerd op basis van groeisnelheid, biomassaproductie, eiwitproductie,
enzymproductie en de morfologie van de stammen. Eén tiende van de 3000 stammen werd
geselecteerd. Deze stammen werden onderworpen aan een tweede ronde van screening in
vloeibare cultuur. Er werden verschillende soorten media gebruikt met elk een bepaald doel:
maximale eiwitproductie, maximale biomassaproductie of enzyminductie. De
screeningscriteria waren dezelfde als bij de eerste ronde. Op basis van statistische analyse
werden 50 stammen geselecteerd die hoge scores behaalden voor een of meerdere criteria in
de verschillende media. In een volgende ronde werden de stammen verwijderd die antibacteriële of anti-fungale verbindingen produceren. De 30 overblijvende stammen werden
vervolgens getest op hun vermogen om vreemd DNA op te nemen en een reportergen tot
expressie te brengen. Als reportergen werd het TAKA-amylase van A. oryzae gebruikt.
II. Literatuurstudie - 5
MANU DE GROEVE
Uiteindelijk bleek Myrothecium gramineum MUCL 39210 (syn. Xepiculopsis graminea)
alle gewenste eigenschappen te bezitten die nodig zijn voor een industrieel interessante
klonerings- en expressiegastheer. Deze eigenschappen zijn onder andere goede
transformeerbaarheid, goede expressie van homologe en heterologe genen, weinig protease
productie en goede fermentatiekarakteristieken. De systematiek van het Myrothecium genus
en enkele biotechnologisch relevante fungi wordt gegeven in Tabel 2.
Tabel 2. Systematiek van het Myrothecium genus en enkele biotechnologisch relevante fungi (Punt et al., 2002)
Fylum
Ascomycota
Subfylum
Klasse (subklasse)
Saccharomycotina Saccharomycetes
Orde
Saccharomycetales
Pezizomycotina
Schizosaccharomycetales
Eurotiales
Eurotiomycetes
Sordariomycetes
Basidiomycota
Zygomycota
Hymenomycetes
(Hymenomycetideae)
Zygomycetes
Diaporthales
Hypocreales
Sordariales
Agaricales
Auriculariales
Aphyllophorales
Mucorales
Familie
Candidaceae
Dipodascaceae
Genus *
Candida (G)
Geotrichum (V)
Yarrowia (V)
Eremotheciacea
Eremothecium/Ashbya (G)
Saccharomycetaceae
Hansenula/Pichia (R)
Kluyveromyces (G)
Saccharomyces (G)
Schizosaccharomycetaceae Schizosaccharomyces (V)
Monascaceae
Trichocomaceae
Valsaceae
Clavicipitaceae
Hypocreaceae
‘Incertae sedis’
Sordariaceae
Agaricaceae
Pleurotaceae
Pluteaceae
Tricholomataceae
Auriculariaceae
Ganodermataceae
Mortierellaceae
Mucoraceae
Monascus (V)
Aspergillus (G,F)
Penicillium (G,F)
Talaromyces (F)
Cryphonectria/Endothia (G)
Tolypocladium (F)
Hypocrea/Trichoderma (R)
Fusarium/Gibberella (F,G)
Acremonium (F)
Myrothecium
Neurospora (V)
Agaricus (V)
Pleurotus (V)
Volvariella (V)
Flammulina (V)
Lentinula (V)
Auricularia (V)
Ganoderma (V)
Mortierella (G)
(Rhizo)mucor (G)
Rhizopus (G)
* Afkortingen: V = humane voeding; G = GRAS-status; F = productie van farmaceutische verbindingen; R =
recombinante eiwitproductie. Soorten waarvoor reeds genetische transformatietechnieken werden beschreven
zijn weergegeven in vet. De systematiek van het Myrothecium genus is weergegeven op een grijze achtergrond
en onderlijnd.
Het genus Myrothecium werd voor het eerst beschreven in 1790 door Tode en omvat enkele
plantenpathogenen waarvan M. roridum de belangrijkste is (Ahrazem et al., 2000). Vele
Myrothecium soorten, waaronder M. cinctum, M. roridum en M. verrucaria, kunnen cellulose
afbreken. Omwille van hun sterke cellulolytische activiteit worden M. verrucaria soorten
gebruikt als standaard test organismen bij de degradatie van textiel door schimmels (Nicot &
Olivry, 1961). M. verrucaria wordt zelfs omschreven als ‘een van de beste cellulose-afbrekers
tot nog toe gekend’ (Domsch et al., 1980). Er werden reeds tal van antibiotica geïsoleerd uit
M. roridum en M. verrucaria, zoals roridines, myrothecines en verrucarines (Mortimer et al.,
1971). Het Myrothecium genus is een kunstmatig genus en is onderhevig aan kritiek. Tulloch
(1972) erkende de moeilijkheid om Myrothecium te onderscheiden van andere genera.
Ahrazem en medewerkers (2000) stelden aan de hand van de analyse van celwand
polysacchariden vast dat Myrothecium een heterogeen genus is. Omdat de polysacchariden in
de celwand van M. gramineum, M. atrum en M. cincutum zo verschillend bleken te zijn van
MANU DE GROEVE
II. Literatuurstudie - 6
deze van andere Myrothecium soorten concludeerden zij dat deze drie species waarschijnlijk
niet thuis horen in dit genus. Tot welk genus ze dan wel behoren werd niet onderzocht.
De meeste Myrothecium soorten zijn cosmopolitisch en zijn geassocieerd met plantenafval in
zowel gematigde als tropische klimaten (Farr et al., 2004). Myrothecium gramineum MUCL
39210, de stam waarmee in dit onderzoek gewerkt wordt, is afkomstig van een afgestorven
takje uit het Royal Chitwan National Park aan de voet van de Himalaya in Nepal en is
beschikbaar bij de Belgian Co-ordinated Collection of Micro-organisms (BCCM™/MUCL).
Ondanks hun ecologisch succes is voor geen enkele Myrothecium soort een seksuele vorm
bekend (Castlebury et al., 2004). M. gramineum behoort tot de fungi imperfecti en heeft dus
een aseksuele reproductiecyclus die mitotisch is. Schimmels produceren sporen om zich voor
te planten, te verspreiden en/of om te overleven onder slechte omstandigheden (Griffin,
1996). De meeste schimmels zijn haploïd tijdens de mitotische groeifase. Door de aseksuele
reproductie via mitotische sporen kan de schimmel zich klonaal voortplanten.
Het vegetatief lichaam van schimmels is een thallus die bestaat uit draden of hyfen die zich
veelvuldig vertakken (Vandamme, 2002). De totale hyfenmassa van een thallus wordt
mycelium genoemd. De groei is apikaal en gebeurt aan de hyfe-uiteinden maar in principe
kan elk stuk weefsel opnieuw uitgroeien tot nieuw mycelium (hyfale extensie). Op vast
substraat groeien schimmels radiaal terwijl in vloeibaar medium zowel filamenteuze groei als
“pellet” groei kan voorkomen (Vandamme, 2002).
Zoals eerder vermeld wordt hier gewerkt met Myrothecium gramineum MUCL 39210. Uit
preliminair onderzoek bleek dat transformanten van deze filamenteuze schimmel gemiddeld
bijna drie keer meer α-amylase (A. oryzae) produceren dan Aspergillus niger transformanten
die onder dezelfde condities werden bekomen en gecultiveerd (Jonniaux et al., 2004).
Bovendien hadden de Myrothecium transformanten ook een betere specifieke productie van
α-amylase (hoeveelheid enzym per gram biomassa) dan de Aspergillus transformanten.
3. TRANSFORMATIE METHODEN VOOR SCHIMMELS
In 1973 rapporteerden Mishra en Tatum voor het eerst de succesvolle genetische
transformatie van een filamenteuze schimmel, namelijk Neurospora crassa (Mishra & Tatum,
1973; Jeenes et al., 1991). De resultaten van deze vroege onderzoeken werden echter niet
algemeen aanvaard tot Case et al. (1979) via Southern analyse kon aantonen dat exogeen
DNA in het genoom van N. crassa was geïntegreerd. Sinds de ontwikkeling van een
protoplast transformatiesysteem voor N. crassa (Case et al., 1979) werden reeds vele
schimmels succesvol getransformeerd, waaronder ook enkele industrieel belangrijke zoals
Aspergillus niger (Buxton et al., 1985), A. oryzae (Gomi et al., 1987) en Trichoderma reesei
(Pentillä et al., 1988).
MANU DE GROEVE
II. Literatuurstudie - 7
De meest gebruikte methode om transformeerbare fungale cellen te bekomen is
protoplastbereiding gebruik makend van celwand degraderende enzymen (Ruiz-Díez, 2002).
Andere methoden zoals de lithiumacetaat behandeling vermijden het gebruik van
protoplasten. Myrothecium gramineum werd reeds succesvol getransformeerd via protoplast
transformatie (Jonniaux et al., 2004).
3.1. Protoplast transformatie
Als startcellen kunnen gekiemde aseksuele sporen, jong mycelium en basidiosporen gebruikt
worden (Ruiz-Díez, 2002). Bij Neurospora soorten worden meestal kiemende macroconidia
gebruikt. Deze zijn hoofdzakelijk multinucleair. Ook mononucleaire microconidia kunnen
gebruikt worden maar zijn moeilijker te bekomen (Rossier et al., 1985). Een alternatief is het
gebruik van jong mycelium (Buxton & Radford, 1983). Na enzymatische behandeling van de
hyfen kunnen de protoplasten gemakkelijk gescheiden worden van het hyfale debris.
Afhankelijk van het species worden kiemende sporen of mycelium gebruikt. Bij
paddenstoelachtigen (Basidiomyceten, Agaricales) is het mogelijk basidiosporen,
dikaryotisch mycelium of vegetatief geproduceerde conidia te gebruiken (Ruiz-Díez, 2002).
Een zeer belangrijke factor bij de bereiding van protoplasten is de keuze van een enzym om
de celwand af te breken. Complexe mengsels van enzymen van slakken (Helicase en
Glusulase) en een enzymconcentraat van de bacterie Arthrobacter luteus (Zymolase 100T)
werden reeds gebruikt (Ruiz-Díez, 2002). De eerste succesvolle transformaties bij N. crassa
werden uitgevoerd met Glusulase. Het meest gebruikte enyzmmengsel echter is dat van
Trichoderma viridae, bekend onder de commerciële naam Novozyme 234 (Riach &
Kinghorn, 1996). Al deze enzymmengsels bevatten een complex mengsel van hydrolytische
enzymen, meer bepaald 1,3-glucanasen en chitinasen (Fincham, 1989). Omdat de werking
van complexe enzymmengsels nogal variabel is opteren sommige onderzoekers voor meer
gedefinieerde enzymmengsels zoals mengsels van cellulase en chitinase (Binninger et al.,
1987). Protoplasten worden osmotisch gestabiliseerd met NaCl (0.6-0.7 M), KCl, MgSO4 (1.2
M), (NH4)2SO4, mannitol (0.8 M), sorbitol (0.8-1.2 M), sucrose of glucose. Sorbitol wordt het
meest gebruikt en blijkt effectief bij de meeste fungi (May, 1992). Protoplasten van
Neurospora, gestabiliseerd in sorbitol, blijven zeer lang leefbaar bij -70°C (Fincham, 1989).
De opname van DNA door protoplasten wordt vergemakkelijkt door het gebruik van calcium
ionen en additie van hoge concentraties van polyethyleen glycol (PEG). Een incubatieduur
van 10-30 minuten bij kamertemperatuur is gewoonlijk voldoende. Voor de transformatie van
protoplasten wordt gewoonlijk tot 5 µg DNA per mL gebruikt bij een densiteit van 108-109
protoplasten per mL (Ruiz-Díez, 2002).
Protoplasten worden geregenereerd op osmotisch gebufferd selectiemedium, dat enkel groei
toelaat van getransformeerde cellen.
MANU DE GROEVE
II. Literatuurstudie - 8
3.2. Lithiumacetaat behandeling
De lithiumacetaat behandeling, die protoplastformatie vermijdt, wordt veel gebruikt bij S.
cerevisiae en werd ook reeds succesvol toegepast bij N. crassa (Dhawale et al., 1984),
Coprinus cinerus (Binninger et al., 1987) en Ustilago violacea (Bej & Perlin, 1989). Telkens
werden kiemende sporen blootgesteld aan het transformerende DNA in de aanwezigheid van
0.1 M lithiumacetaat.
Het toevoegen van een hoge concentratie polyethyleenglycol (PEG) na een initiële periode
van blootstelling aan het DNA, is een stap die zowel bij de lithiumacetaat behandeling als bij
de protoplast transformatie voorkomt (Ruiz-Díez, 2002). Gewoonlijk worden hoeveelheden
tot 10 volumes 40% PEG 4000 gebruikt. PEG wordt samen met CaCl2 en buffer toegevoegd.
PEG zorgt voor het samenklitten van de behandelde cellen en het DNA en bevordert zo de
opname van DNA.
3.3. Elektroporatie
Deze techniek is gebaseerd op de reversibele permeabilisatie van biomembranen door het
aanleggen van kort durende elektrische velden met hoge amplitude. De veranderingen in de
membraan tijdens de elektrische puls laten de opname toe van recombinant DNA.
Elektroporatie wordt gebruikt voor de transformatie van dierlijke cellen, plantprotoplasten,
bacteriën, gisten en filamenteuze schimmels. Er zijn drie procedures voor de transformatie
van filamenteuze schimmels (Ruiz-Díez, 2002). Een eerste procedure is de voorbehandeling
van conidiën met een celwand verzwakkende agent. Een alternatieve methode is de
elektroporatie van protoplasten. Ten slotte kan men het gebruik van protoplasten vermijden
door elektroporatie van gekiemde sporen, zoals beschreven voor A. nidulans (Sánchez &
Aguirre, 1996). Elektroporatie is de meest gebruikte methode voor de transformatie van
filamenteuze schimmels en werd onder andere reeds toegepast op N. crassa, Penicillium
urticae, Leptosphaeria maculans, A. oryzae (Chakraborty et al., 1991), Scedosporium
prolificans (Ruiz-Díez & Martínez-Suárez, 1999), A. nidulans (Sánchez & Aguirre, 1996), A.
niger (Ozeki et al., 1994), Wangiella dermatitidis en A. fumigatus (Kwon-Chung et al., 1998).
3.4. Biolistische transformatie
Niet alle filamenteuze schimmels kunnen efficiënt getransformeerd worden via bovenstaande
methoden. Bovendien kunnen van sommige schimmels geen protoplasten worden bekomen.
Biolistische transformatie kan gebruikt worden voor het introduceren van plasmide DNA in
intacte fungale cellen met dikke celwanden. Bij deze methode worden tungsten partikels
gebonden met DNA en met hoge snelheid in fungale sporen of hyfen gebracht. Deze methode
heeft als nadeel dat er dure gespecialiseerde apparatuur nodig is.
MANU DE GROEVE
II. Literatuurstudie - 9
Biolistische transformatie werd met succes toegepast bij enkele filamenteuze schimmels zoals
N. crassa, Magnaporthe grisea, Trichoderma harzianum (Riach & Kinghorn, 1996), A.
nidulans (Gomes-Barcellos et al., 1998) en Trichoderma reesei (Hazell et al., 2000).
3.5. Agrobacterium tumefaciens-gemedieerde transformatie
De Agrobacterium tumefaciens gemedieerde transformatie is een veel gebruikte methode voor
de transformatie van plantencellen (Michielse et al., 2005). Deze methode is gebaseerd op het
feit dat A. tumefaciens een deel van het Ti plasmide, meer bepaald het T-DNA, kan
overbrengen naar de doelcel. Het DNA wordt als een enkelstrengige DNA molecule
overgebracht naar de doelcel en wordt geassisteerd door de DNA-bindende eiwitten VirD2 en
VirE2 (Zhu et al., 2000). De transformatie van filamenteuze schimmels via Agrobacterium
tumefaciens werd voor het eerst succesvol toegepast door de Groot et al. (1998). Zij toonden
aan dat zowel protoplasten als conidia van Aspergillus awamori efficiënt konden
getransformeerd worden via A. tumefaciens. Er werden transformatie-efficiënties bekomen
die tot 600 maal hoger lagen in vergelijking met de conventionele technieken voor de
transformatie van protoplasten. De meerderheid van de transformanten bevatte één T-DNA
kopij. Het T-DNA werd op een gelijkaardige manier als bij planten meestal willekeurig
geïntegreerd in het genoom. Verscheidene filamenteuze schimmels konden getransformeerd
worden via deze methode, waaronder A. niger, Fusarium venenatum, Trichoderma reesei,
Colletotrichum gloeosporioides, Neurospora crassa en de paddenstoel Agaricus bisporus (de
Groot et al., 1998). De A. tumefaciens gemedieerde transformatiemethode is beschermd door
verschillende patenten.
Recent werden verschillende transformatiemethoden voor Aspergillus giganteus met elkaar
vergeleken (Meyer et al., 2003). A. giganteus werd getransformeerd volgens vier
verschillende methoden: protoplast transformatie, elektroporatie, biolistische transformatie en
Agrobacterium-gemedieerde transformatie. Elektroporatie en biolistische transformatie
bleken niet efficiënt terwijl de conventionele methode via protoplasten tot 55 transformanten
op 108 protoplasten opleverde per µg DNA. Het aantal transformanten kon tot 140 maal
verhoogd worden met A. tumefaciens-gemedieerde transformatie. Verder bleek nog dat
Aspergillus giganteus transformanten slechts één, willekeurige geïntegreerde T-DNA kopij
bevatten terwijl meerder kopijen werden gevonden bij transformanten bekomen via protoplast
transformatie.
Gouka et al. (1999) toonden aan dat wanneer het Ti DNA homologie vertoont met het A.
awamori genoom er ook homologe recombinatie kan optreden. Op deze manier kunnen
transformanten bekomen worden die een of meerdere kopijen van een bepaald gen bevatten
op een gedefinieerde plaats in het genoom. Michielse en medewerkers (2005) vergeleken bij
A. awamori de efficiëntie van homologe recombinatie bij Agrobacterium gemedieerde
transformatie en CaCl2/PEG gemedieerde transformatie. Zij vonden dat de frequentie van
homologe recombinatie bij Agrobacterium gemedieerde transformatie 3 tot 6 maal hoger was
MANU DE GROEVE
II. Literatuurstudie - 10
dan bij CaCl2/PEG transformatie. Bovendien konden voor efficiënte homologe recombinatie
kortere homologe DNA flanken gebruikt worden dan bij de CaCl2/PEG protoplast
transformatie.
4. OPNAME VAN HET TRANSFORMERENDE DNA
In S. cerevisiae en andere gisten worden plasmiden met selectiemerkers voornamelijk via
homologe recombinatie geïntegreerd in het genoom (Hynes, 1996). Dit gebeurt met een
relatief lage frequentie. Het inbouwen van autonoom replicerende sequenties (ARS) laat
replicatie toe van de plasmiden wat resulteert in onstabiele transformanten aan een hoge
frequentie. Door het toevoegen van centromeersequenties aan ARS vectoren kunnen stabiele
transformanten worden bekomen die 1 tot 3 kopijen van het plasmide bevatten.
Bij filamenteuze schimmels wordt het transformerende DNA vooral opgenomen via
ectopische (niet-homologe) integratie in het genoom (Tudzynski & Tudzynski, 1997).
Aangezien de integratie van vector moleculen op verschillende plaatsen in het genoom of in
tandem op één plaats veelvuldig voorkomt, is het mogelijk transformanten met hoge
kopijaantallen (tot honderden kopijen) te bekomen. In tegenstelling tot transformanten met
replicerende vectoren zijn deze mitotisch stabiel.
Een nuttige eigenschap van transformatie van filamenteuze schimmels is dat hoge frequenties
(30-90%) van co-transformatie van vectoren kunnen bekomen worden (May, 1992; Riach &
Kinghorn, 1996). Wanneer cellen aan twee verschillende soorten DNA blootgesteld worden is
er een grote kans dat wanneer één soort molecule wordt opgenomen ook de andere wordt
opgenomen (Fincham, 1989). Niet alle protoplasten kunnen echter even gemakkelijk DNA
opnemen. De meest competente cellen zullen de neiging hebben meerdere DNA moleculen
simultaan opnemen. Bij co-transformatie bevinden de selectiemerker en het tot expressie te
brengen gen zich op verschillende plasmiden. Bij een hoge ratio selecteerbaar plasmide ten
opzichte van niet-selecteerbaar plasmide worden vaak co-transformatie efficiënties van meer
dan 50% bekomen en zelfs tot 95% bij Aspergillus nidulans (Wernars et al., 1987; Jarai,
1997).
De constructie van autonoom replicerende vectoren voor de transformatie van filamenteuze
schimmels is een belangrijk doel omdat deze hoge transformatiefrequenties geven en de
constructie van shuttle vectoren mogelijk maken (Hynes, 1996). De isolatie van een 383 bp
element van Ustilago maydis met gelijkaardige sequenties als de S. cerevisiae ARS sequenties
maakte dit mogelijk voor deze basidiomyceet (Tsukuda et al., 1988). Bij A. nidulans vonden
Gems et al. (1991) een 6 kb lange origin of replication, AMA-1, die zeer hoge frequenties aan
onstabiele transformanten gaf. Deze transformanten bevatten 10-30 vrije plasmiden per
genoom. De AMA-1 sequentie bleek ook functioneel in A. niger en A. oryzae (Gems et al.,
1991), Gaeumannomyces graminis (Bowyer et al., 1994) en Penicillium chrysogenum (Fierro
MANU DE GROEVE
II. Literatuurstudie - 11
et al., 1996). Ook andere autonoom replicerende systemen werden gevonden maar blijken
zeldzaam te zijn in schimmels. Er werden nog geen centromerische sequenties geïsoleerd die
toelaten stabiele replicerende vectoren te gebruiken.
Vele fungi kunnen telomeren toevoegen aan exogeen DNA (Ruiz-Díez, 2000). Onder meer de
gist Cryptococcus neoformans, de dimorfe ascomyceet Histoplasma capsulatum, de
filamenteuze deuteromyceet Fusarium oxysporum (Powell en Kistler, 1990) en de taxol
producerende filamenteuze schimmel Pestalotiopsis microspora (Long et al., 1998) kunnen
om onbekende redenen telomerische repeats toevoegen aan het transformerende DNA.
Lineaire transformatie-vectoren die telomeersequenties bevatten werden gecreëerd in
Fusarium oxysporum en functioneerden met hoge efficiëntie als autonoom replicerende
vector in de plant pathogenen Nectria haematococca en Cryphonectria parasitica, en in F.
oxysporium (Powell en Kistler, 1990). In A. nidulans vertoonden plasmiden met humane
telomerische DNA sequenties autonome replicatie (Aleksenko en Ivanova, 1998). De isolatie
en karakterisatie van telomeren van andere fungi kan leiden tot de ontwikkeling van lineaire
autonoom replicerende vectoren voor fungale transformatie.
In sommige soorten vergroot linearisering van vector DNA sterk de transformatie efficiëntie
en homologe recombinatie. Linearisering bevordert ook dubbele cross-overs wat resulteert in
gen vervanging en/of conversie (Jeenes et al., 1991). Gene replacement wordt vaak gebruikt
voor de constructie van gedefinieerde deletiemutanten om de functie van het uitgeschakelde
gen te achterhalen. Bij genvervanging wordt meestal gebruik gemaakt van een lineaire gene
replacement cassette die bestaat uit een selectiemerker gen geflankeerd door DNA
fragmenten die homoloog zijn aan het doelgen (Michielse et al., 2005). Nadat deze
constructie in de doelcel is binnengebracht kan deze zowel via homologe als niet-homologe
recombinatie (ectopisch) integreren. De wijze waarop vreemde DNA sequenties in het
genoom integreren hangt af van de dominante pathway van de gastheercel om
dubbelstrengige DNA breuken te herstellen (Schaefer, 2001). In S. cerevisiae worden
dubbelstrengige DNA breuken hoofdzakelijk via homologe recombinatie hersteld, terwijl dit
bij planten en dieren vooral niet-homoloog gebeurt. Bij deze hogere eukaryoten is de
frequentie van integratie via illegitieme recombinatie dan ook veel hoger dan gerichte
integratie via homologe recombinatie. Bij gisten zoals S. cerevisiae en Schizosaccharomyces
pombe zijn korte homologe regio’s van 50-100 bp reeds voldoende voor integratie van de
gene replacement cassette in de homologe locus met een efficiëntie van 50-100% (Bahler et
al., 1998; Wach et al., 1994). In filamenteuze schimmels zijn langere homologe flanken van
minstens 1000 bp nodig om efficiënties van 10-30% te bekomen (Asch & Kinsey, 1990;
Hynes, 1996). Naast de homologe recombinatie-efficiëntie van de gastheercel spelen ook
andere factoren een rol in het bepalen van de efficiëntie van gerichte integratie. Zo zijn
ondermeer de lengte van de homologe DNA flanken, het GC-gehalte van deze flanken, de
transcriptionele status van het doelgen en de chromatinestructuur belangrijke factoren
(Michielse et al., 2005).
MANU DE GROEVE
II. Literatuurstudie - 12
De mogelijkheid om het geïntroduceerde DNA gericht te integreren in specifieke locaties in
het genoom is van cruciaal belang bij vele studies.
5. SELECTIEMERKERS
5.1. Soorten selectiemerkers
Er zijn vele verschillende soorten selectiemerkers beschikbaar voor de transformatie van
filamenteuze schimmels. Deze kunnen algemeen in twee grote groepen ingedeeld worden.
Enerzijds zijn er de dominante selectiemerkers zoals resistentiegenen tegen antibiotica, en
anderzijds wild type genen die een gedefinieerde auxotrofe mutant kunnen complementeren
(Jeenes et al., 1991). Bij filamenteuze schimmels wordt, in tegenstelling tot gist, de voorkeur
gegeven aan dominante selectiesystemen omdat deze geen specifiek mutant genotype
behoeven (Tudzynski & Tudzynski, 1997).
De meest gebruikte resistentie systemen zijn de bacteriële hygromycine B en phleomycine
resistentiegenen en het fungale benomyl resistentiegen. Door gebruik van
transformatieplasmiden met deze resistentiegenen kan een grote variëteit aan
schimmelsoorten getransformeerd worden waarvoor geen of slechts weinig auxotrofe
mutanten bestaan (Timberlake & Marshall, 1989). Uit preliminair onderzoek bleek dat bij
Myrothecium gramineum MUCL 39210 de minimale antibioticaconcentraties waarbij geen
groei optreedt de volgende zijn: 500 µg/mL hygromycine B, >100 µg/mL phleomycine en
>400 µg/mL fosfinotricine (Jonniaux et al., 2004).
In het onderzoek waar deze scriptie deel van uitmaakt werd reeds met succes het
hygromycine B-resistentie gen van E. coli gebruikt als selectiemerker voor de transformatie
van Myrothecium gramineum. Deze selectiemerker werd echter enkel gebruikt voor de
optimalisatie van het transformatieprotocol. Het gebruik van antibiotica resistentiegenen is
immers af te raden voor toepassingen in de voedingssector. Als alternatief werd het gebruik
van het acetamidase gen onderzocht.
Het acetamidase gen (amdS) werd voor het eerst als selectiegen gebruikt door Tilburn en
medewerkers (1983) voor de transformatie van Aspergillus nidulans (Ruiz-Díez, 2002).
Omdat acetamide een arme stikstofbron is voor wild type A. nidulans en de meeste andere
schimmels kan het acetamidase gen dienen als selectiemerker voor vele fungale species.
Transformanten die het amdS gen opgenomen hebben en tot expressie brengen kunnen
groeien op media met acetamide als enige stikstofbron. Bovendien reflecteert de graad van
acetamidegebruik het aantal genkopijen en kan dus gebruikt worden voor de selectie van
transformanten met een hoog kopijaantal (Ruiz-Díez, 2002). Omdat Myrothecium gramineum
MUCL 39210 geen groei vertoont op minimaal medium met acetamide als enige stikstofbron
kan in theorie het acetamidase gen gebruikt worden als selectiemerker (Jonniaux et al., 2004).
MANU DE GROEVE
II. Literatuurstudie - 13
Het amdS systeem bleek echter niet efficiënt voor de transformatie van Myrothecium
gramineum.
Deze dominante selectiemerkers hebben als grote voordeel dat ze voor minder goed
bestudeerde schimmels kunnen gebruikt worden. Naast dominante selectiemerkers kunnen
ook auxotrofe merkers gebruikt worden. Deze hebben echter als nadeel dat de geschikte
genotypes moeten gecreëerd worden in de te transformeren schimmel. Het meest gebruikte
systeem is het chloraat resistentie systeem. Mutanten die geen nitraat reductase meer
aanmaken zijn resistent aan chloraat en kunnen getransformeerd worden met het niaD gen
van A. nidulans als heterologe selectiemerker (Daboussi et al., 1989). Deze methode werd
reeds toegepast op de industrieel belangrijke schimmels A. niger en A. oryzae. Verder werden
selenaat resistente mutanten geïsoleerd die ATP sulfurylase negatief zijn en
gecomplementeerd kunnen worden met het sC gen van A. nidulans. (Buxton et al., 1989).
Ook fluoroacetaat selectie voor het bekomen van acetyl-CoA synthetase mutanten bleek
nuttig (Gouka et al., 1993).
Een andere veel gebruikte methode is het gebruik van 5-fluoro-orotinezuur (5-FOA)
resistentie voor de isolatie van OMPD negatieve mutanten die uracil of uridine auxotroof zijn.
Deze mutanten kunnen dan gecomplementeerd worden met het homologe OMPD gen of de
heterologe pyrG (A. nidulans) of pyr4 (Neurospora crassa) genen die coderen voor orotidinemonofosfaat decarboxylase (OMPD) (d’Enfert, 1996).
Omdat het acetamidase systeem niet bleek te werken bij Myrothecium gramineum werd
geöpteerd voor het gebruik van het 5-FOA resistentiesysteem. Hiervoor moeten uracil/uridine
auxotrofe mutanten worden gecreëerd die kunnen gecomplementeerd worden met het gen
coderend voor orotidine-monofosfaat decarboxylase (OMPD). Deze methode zal in detail
toegelicht worden.
5.2. Het OMPD gen als selectiemerker
Het enzym orotidine-monofosfaat decarboxylase (OMPD; EC 4.1.1.23) katalyseert de laatste
stap in de de novo biosynthese pathway van pyrimidines, namelijk de decarboxylatie van
orotidine-5’-monofosfaat (OMP) tot uridine-5’-monofosfaat (UMP) (Kimsey & Kaiser,
1992). De de novo pyrimidine biosynthese pathway bestaat uit zes enzymatische stappen en is
schematisch weergegeven in Figuur 1 (Boldt & Zrenner, 2003).
II. Literatuurstudie - 14
MANU DE GROEVE
O
-
L-Glutamine + 2 ATP + HCO3 + H2O
carbamoyl fosfaat
synthetase II
2 ADP + L-Glutamaat
+ Pi
HN
O
HO
O
N
O
P
O
OH
OH
O
OH
OH
H2N
Uridine-5'-monofosfaat (UMP)
O P OH
O
CO2
Carbamoyl fosfaat
O
orotidine-5'-monofosfaat
decarboxylase (OMPD)
OH
HO
NH2
O
O
L-Aspartaat
L-aspartaat
carbamoyltransferase
HN
O
O
HO
Pi
P
O
N
O
O
OH
HO
NH2
OH
OH
NH
O
OH
O
OH
Orotidine-5'-monofosfaat (OMP)
O
N-Carbamoyl-L-aspartaat
PPi
orotaat
fosforibosyltransferase (OPRT)
dihydroorotase
PRPP
H2O
O
O
OH
OH
O
HN
dihydroorotaat dehydrogenase
HN
O
N
H
O
Dihydroorotaat
NAD+
NADH
N
H
O
Orotaat
Figuur 1. De novo biosynthese pathway van pyrimidines
Mutanten die geen OMPD activiteit vertonen zijn uracil of uridine auxotroof. Zij hebben dus
uracil of uridine nodig om te groeien. Deze auxotrofie is het gevolg van het ontbreken van de
laatste stap in de de novo biosynthese pathway van pyrimidines waardoor geen UMP meer
wordt gevormd. Door deze mutanten te complementeren met een werkend gen voor OMPD
wordt de auxotrofie opgeheven en worden de mutanten opnieuw prototroof. In dit onderzoek
zal gebruik gemaakt worden van het homologe OMPD gen van Myrothecium gramineum als
selectiemerker. Daarvoor moet het homologe OMPD gen gekloneerd worden en moeten er
OMPD negatieve mutanten worden gecreëerd die kunnen gecomplementeerd worden met het
wild type OMPD gen van Myrothecium gramineum.
MANU DE GROEVE
II. Literatuurstudie - 15
5.2.1. Studie van de DNA- en eiwitsequentie van OMPD
De zes enzymatische stappen zijn identiek in Eubacteria, Archaea en eukaryoten. Deze
geconserveerde pathway en de enzymen ervan zijn dan ook bruikbaar voor fylogenetische
studies (Kimsey & Kaiser, 1992; Radford, 1993; Nara et al., 2000; Traut & Temple, 2000).
Vooral OMPD is geschikt voor dit doeleinde. Het is een essentieel biosynthetisch enzym bij
alle organismen. Bovendien hebben bijna alle organismen tot nu toe onderzocht slechts één
OMPD gen. Een uitzondering hierop is een bepaalde Acremonium endofyt die op raaigras
voorkomt en twee verschillende functionele pyr-4 genen bevat (Collett et al., 1995). De
auteurs konden aantonen dat de duplicatie het gevolg was van een interspecifieke hybridisatie
tussen Epichloë typhina en Acromonium lolii.
Bij de hogere eukaryoten is gedurende de evolutie van OMPD op een bepaald moment het
OMPD gen gefuseerd met een tweede gen, coderend voor orotaat fosforibosyltransferase
(OPRT). Dit tweede enzym katalyseert de voorlaatste stap in de de novo biosynthese pathway
van UMP. Het gefuseerde gen codeert dus voor een bifunctioneel enzym, aangeduid als UMP
synthase. De genen zijn zo gefuseerd dat de activiteit van beide enzymen kon bewaard blijven
(Traut & Temple, 2000). De prokaryotische en fungale OMPD enzymen zijn allen
monofunctioneel.
Het gen coderend voor OMPD werd reeds bij meer dan 150 organismen gekloneerd en
gekarakteriseerd, gaande van Escherichia coli tot Homo sapiens. De enzym sequentie blijkt
goed geconserveerd gedurende evolutie, met regio’s van sterk geconserveerde aminozuren
(Radford & Dix, 1988; Benito et al., 1992; Kimsey & Kaiser, 1992; Traut & Temple, 2000).
Kimsey & Kaiser (1992) vergeleken alle tot dan toe gekende OMPD sequenties via multiple
sequence alignments. Zij vergeleken een set van 20 enzymsequenties uit gewervelde dieren,
slijmschimmels, verscheidene fungi en Gram-positieve en Gram-negatieve bacteriën. Er
werden vier homologe regio’s gevonden die gemeenschappelijk waren voor alle 20
onderzochte OMPD enzymen. Rodríguez en medewerkers (1998) bevestigden de vier
geconserveerde regio’s na analyse van 50 OMPD sequenties. Van deze vier regio’s was de
tweede regio het meest geconserveerd met zes strikt geconserveerde aminozuren waaronder
vijf in de volgende consensus sequentie: DxKxxDI(P/G)(S/T/N)T. In deze sequentie is
lysine (K) essentieel voor de katalytische activiteit van het enzym. Kimsey & Kaiser (1992)
vonden in totaal 10 absoluut geconserveerde aminozuren in de vier regio’s. Bovendien bleek
ook de afstand tussen deze vier regio’s relatief geconserveerd in alle 20 OMPD sequenties.
De enige uitzondering hierop is een insertie van 90-100 aminozuren tussen regio twee en drie
bij Trichoderma reesei, Neurospora crassa en Cephalosporium acremonium. De
geconserveerde afstand tussen de vier regio’s wijst erop dat alle 20 OMPD enzymen een
gelijkaardige driedimensionele conformatie hebben.
MANU DE GROEVE
II. Literatuurstudie - 16
Traut en Temple (2000) vergeleken de OMPD aminozuursequenties van meer dan 80 species
en kwamen tot besluit dat 8 aminozuren invariant of zeer geconserveerd waren op een totaal
van gemiddeld 270 aminozuren per enzym. Het motief voor de katalytische site bevat 3
invariante aminozuren: DxKxxD, waarbij lysine (K) essentieel is voor activiteit. OMPD heeft
een opmerkelijke katalytische activiteit. Dit is vanwege de uitzonderlijke stabiliteit van de
carboxylgroep van OMP, aangezien de niet gekatalyseerde decarboxylatie van OMP een t1/2
van 78 miljoen jaar heeft. Bovendien heeft het enzym geen cofactoren. Aan de hand van
kristalstructuren konden de auteurs besluiten dat waarschijnlijk alle OMPD enzymen de α/β
barrel structuur hebben die essentieel is voor de werking van het enzym. Bij de analyse van de
meer dan 80 aminozuursequenties werd verder nog de 100 aminozuren insert gevonden bij
enkele filamenteuze schimmels waaronder Neurospora crassa. Dit insert werd opnieuw enkel
bij fungi (minstens zeven species) gevonden. Het kan gezien worden als een klein domein dat
tegen de centrale α/β barrel kern ligt maar de functie ervan is tot op heden onbekend.
Recent werden de fungale OMPD genen en aminozuursequenties van verschillende
taxonomische groepen (Zygomycetes, Basidiomycetes en Ascomycetes) met elkaar vergeleken
(Quiles-Rosillo et al., 2003). Hieruit bleek dat de gemiddelde lengte van het OMPD enzym
ongeveer 270 aminozuren is bij Zygomyceten, Basidiomyceten en de meeste Ascomyceten,
behalve de Sordariomyceten. In tegenstelling tot de rest van de filamenteuze Ascomyceten
hebben de onderzochte OMPD eiwitten van Sordariomyceten (Neurospora crassa, Hypocrea
jecorina, Cephalosporium acremonium en Sordaria macrospora) een insertie van ongeveer
100 aminozuren in de centrale regio. Bovendien bevatten de OMPD genen van deze
Sordariomyceten geen introns. Alle andere onderzochte OMPD genen bevatten één of twee
introns op relatief geconserveerde posities (zie Figuur 2). Bij de Ascomyceten komt meestal
één sterk geconserveerd intron voor.
Figuur 2. Aantal introns en positie in OMPD coderende genen van verschillende fungi
(Quiles-Rosillo et al., 2003)
MANU DE GROEVE
II. Literatuurstudie - 17
De aanwezigheid van sterk geconserveerde regio’s in de aminozuursequentie van OMPD laat
de isolatie toe van OMPD coderende genen in genetisch slecht bestudeerde organismen.
De meeste OMPD genen van filamenteuze schimmels werden geïsoleerd via heterologe
hybridisatie van een genomische bank. Onder andere de pyrG genen van Aspergillus niger, A.
awamori, A. parasiticus, Mucor circinelloides, Trichoderma reesei, Phycomyces
blakesleeanus, Blakeslea trispora en het pyr4 gen van Claviceps purpurea werden op deze
manier geïsoleerd (zie Tabel 3). Meestal werd het gekende OMPD gen van een verwante
schimmel gebruikt als heterologe probe voor de hybridisatie van een lambda genomische
bank.
Tabel 3. Isolatie van OMPD genen via heterologe hybridisatie van een genomische bank
Organisme
OMPD gen
Heterologe probe
Referentie
Aspergillus niger
pyrG
pyr4 (Neurospora crassa)
Goosen et al (1987)
Phycomyces blakesleeanus
pyrG
pyrG (A. niger)
Diaz-Minguez et al. (1990)
pyr4 (N. crassa)
Gruber et al. (1990)
Trichoderma reesei
pyrG
pyrG (A. nidulans)
Skory et al. (1990)
A. parasiticus
pyrG
pyrG (P. blakesleanus)
Benito et al. (1992)
Mucor circinelloides
pyrG
Smit & Tudzynski (1992)
Claviceps purpurea
pyr4
pyr4 (N. crassa)
pyrG (A. niger)
Gouka et al. (1995)
A. awamori
pyrG
pyrG (M. circinelloides)
Quiles-Rosillo et al. (2003)
Blakeslea trispora
pyrG
Penicillium nalgiovense
pyrG
pyrG (P. chrysogenum)
Fierro et al. (2004)
De heterologe hybridisatie van een genomische bank is echter niet altijd succesvol. Vaak
worden slechts zeer zwakke hybridisatie signalen verkregen die moeilijk te onderscheiden
zijn van de niet-specifieke hybridisatie (Agnan et al., 1997). In zo’n gevallen moet dan een
homologe probe gebruikt worden. Een homologe probe kan gemaakt worden door een deel
van het homologe gen te amplificeren via gedegenereerde PCR (Polymerase Chain Reaction).
Omdat OMPD verschillende geconserveerde aminozuursequenties bevat is het mogelijk
hierop gedegenereerde primers te ontwikkelen. Gedegenereerde primers zijn primers die
promiscue nucleotiden zoals inosine bevatten en/of bestaan uit een mix van enkele tot
duizenden gelijkaardige primers. Omdat de genetische code gedegenereerd is worden de
meeste aminozuren gecodeerd door verschillende codons. Daarom is het niet mogelijk de
exacte nucleotidensequentie af te leiden uit de aminozuursequentie. Door het invoeren van
“gedegenereerde” nucleotiden in de primer sequentie kan men dit probleem omzeilen. In
Bijlage (Tabel 44, p.134) zijn de benamingen en symbolen van de verschillende
gedegenereerde nucleotiden weergegeven.
De OMPD genen van onder andere Aspergillus fumigatus (Weidner et al., 1998), Rhizomucor
pusillus (Yamazaki et al., 1999), Rhizopus oryzae (Skory, 2002) en Mortierella alpina
(Takeno et al., 2004) konden worden geïsoleerd via gedegenereerde primers. Hiervoor
werden eerst geschikte gedegenereerde primers ontwikkeld op basis van de geconserveerde
regio’s van OMPD genen of eiwitten van verwante organismen (zie Tabel 4). Eenmaal een
deel van het OMPD gen geamplificeerd is kan dit gebruikt worden als homologe probe bij de
MANU DE GROEVE
II. Literatuurstudie - 18
hybridisatie van een genomische of cDNA bank. De verder isolatie van het volledige gen
verloopt dan meestal zoals bij de heterologe hybridisatie.
Tabel 4. Isolatie van deel van een OMPD gen via gedegenereerde PCR
Organisme
Gedegenereerde primers (5’->3’)
A. fumigatus
CTTCCTYATCTTYGARGACCG
TACTGCTGMCCSAGCTTATCBCCCTT
Geconserveerde
Referentie
regio
FLIFEDRKF
Weidner et
KGDKLGQQY
al. (1998)
R. pusillus
CCCGGATCCTTTGAAGATCGYAAATTTGCTGATATYGG
CCCGGATCCAGGKGTTCTGTAYTGYTGACCCAAWCCATC
FEDRKFADIG
DGLGQQYRTP
Rhizopus
oryzae
ATTGAYATTGTKGAAGACTTYGA
CCACTCTCMACAATNACTTC
M. alpina
AARTTYGCIGAYATYGGHAAYACIGT
CKICCIACRATRATVACRTCIGCDCC
Yamazaki et
al. (1999)
Skory (2002)
KFADIGNTV
RGVIIVDAG
Takeno et al.
(2004)
Hoewel de techniek van hybridisatie van een genomische bank vaak gebruikt wordt om
nieuwe genen te kloneren is dit een omslachtige methode. De constructie van een
representatieve en volledige DNA bank is moeilijk en tijdrovend. Snellere methodes om
volledige genen kloneren zijn gebaseerd op PCR. Hierbij dient een deel van de sequentie van
het te kloneren gen gekend te zijn waarna de sequentie uitgebreid wordt via PCR. Een vaak
gebruikte techniek is inverse PCR.
In tegenstelling tot standaard PCR, waarbij een DNA segment vermenigvuldigd wordt dat
tussen twee naar elkaar gerichte primers ligt, kunnen met inverse PCR de onbekende DNAflanken van een gekende DNA sequentie geamplificeerd worden (Sambrook & Russell,
2001). Hiervoor dient eerst het (genomisch) DNA dat de gekende sequentie bevat geknipt te
worden met een restrictie-enzym (zie Figuur 3). De resulterende restrictiefragmenten worden
gecirculariseerd door intramoleculaire ligatie waarna deze gebruikt worden als template in
een PCR reactie. De onbekende sequentie wordt geamplificeerd met twee primers die
specifiek op de gekende sequentie binden en in tegengestelde richting wijzen.
Het geamplificeerde fragment is lineair en bevat één restrictieplaats voor het oorspronkelijk
gebruikte restrictie-enzym. Deze plaats wijst de verbinding aan tussen de flankerende
sequenties van de gekende sequentie. De inverse PCR techniek is één van de meer
problematische technieken voor het kloneren van genen en de kans op succes is niet
gegarandeerd.
II. Literatuurstudie - 19
MANU DE GROEVE
gekende DNA sequentie
genomisch DNA
knip met restrictie-enzym
restrictieplaatsen
circularisatie van restrictiefragmenten
primer B
primer A
PCR op gecirculariseerde fragmenten
met primer A en primer B
primer B
PCR product
primer A
Figuur 3. Schematische voorstelling van de inverse PCR techniek
(naar Sambrook & Russell, 2001)
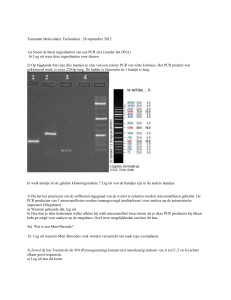
In het onderzoek waar deze scriptie deel van uitmaakt zal PCR gebaseerde genomewalking
gebruikt worden om het volledige OMPD gen van M. gramineum te kloneren. Hiervoor zal de
BD GenomeWalker Universal Kit (BD Biosciences) gebruikt worden. Met deze kit kan een
gekende DNA sequentie naar de upstream en downstream kant uitgebreid worden via PCR
met genspecifieke primers en adaptor-specifieke primers. Het principe van deze techniek
wordt meer in detail beschreven in het hoofdstuk Materialen en Methoden (III.10.2, p.51 e.v.)
en Resultaten en Bespreking (IV.1.31.3, p.77).
5.2.2. Selectie van OMPD-negatieve mutanten
OMPD negatieve mutanten kunnen geselecteerd worden aan de hand van hun resistentie aan
5-fluoro-orotinezuur (5-FOA). Dit systeem werd voor het eerst beschreven door Boeke et al.
(1984) voor de positieve selectie van OMPD negatieve gisten. Wild type gistcellen konden
niet groeien op 5-FOA bevattend medium terwijl cellen met een mutatie in het OMPD gen
(ura3) wel konden groeien. Niet alle 5-FOA-resistente cellen zijn echter OMPD negatief. In
experimenten waarbij spontane of UV-geïnduceerde 5-FOA resistente kolonies geselecteerd
werden bleek slechts 5-10% ura3-. Ook OPRT negatieve mutanten (ura5) zijn (gedeeltelijk)
resistent aan 5-FOA. ura1 (dihydroorotaat dehydrogenase), ura2 (carbamoyl-fosfaat
synthase/aspartaat carbamoyltransferase) en ura4 (dihydroorotase) mutanten zijn wel
gevoelig aan 5-FOA. Een groot voordeel van het gebruik van OMPD als selectiemerker is dat
het zowel voor positieve als negatieve selectie van mutanten kan gebruikt worden.
Op welke manier 5-FOA gistcellen doodt is nog onzeker, maar omdat OPRT- en OMPDnegatieve mutanten resistent blijken te zijn wordt vermoed dat de toxiciteit resulteert uit de
conversie van 5-FOA naar 5-fluoro-uridine-5’-monofosfaat (5-FUMP) (Boeke et al., 1984).
In Aspergillus fumigatus en A. niger kon via 19F NMR spectroscopie aangetoond worden dat
5-FUMP wordt omgezet tot 5-fluoro-uridine-5’-trifosfaat (5-FUTP) en 5-fluoro-2’deoxyuridine-5’-monofosfaat (5-FdUMP) (Chouini-Lalanne et al., 1989). Het gevormde 5-
II. Literatuurstudie - 20
MANU DE GROEVE
FUTP wordt ingebouwd in RNA, terwijl 5-FdUMP een mogelijke inhibitor is van het enzym
thymidylaat synthase (TS). In Figuur 4 wordt de conversie van 5-FOA naar 5-FUMP en de
toxische werking van de metabolieten schematisch weergegeven.
Ook bij de plant Nicotiana plumbaginifolia kon aangetoond worden dat 5-FUMP door het
celmetabolisme wordt omgezet tot 5-FdUMP dat TS inhibeert (Santoso & Thornburg, 1998).
De inhibitie van TS veroorzaakt een sterke reductie van de thymine biosynthese met als
gevolg een thymine tekort dat resulteert in celdood.
Boeke en medewerkers (1984) stelden vast dat de groei inhibitie van URA+ S. cerevisiae
cellen door 5-FOA tenminste gedeeltelijk reversibel is. Van de 40 URA+ cellen die uitgeplaat
werden op 5-FOA medium (3 dagen incubatie bij 30°C) konden 15 cellen herstellen en verder
groeien na transfer op YPD medium.
O
O
F
orotaat
fosforibosyltransferase (OPRT)
F
HN
O
HN
O
OH
NH
O
P
HO
PRPP
O
PPi
OH
O
N
O
O
OH
5-fluoro-orotinezuur
(5-FOA)
OH
OH
5-fluoro-orotidine-5'-monofosfaat
(5-FOMP)
O
F
HN
O
HO
P
O
O
orotidine-5'-monofosfaat
decarboxylase (OMPD)
N
O
CO 2
OH
OH
OH
5-fluoro-uridine-5'-monofosfaat
(5-FUMP)
5-FUTP
5-FUDP
5-FdUDP
5-FdUMP
inhibitie van
incorporatie in RNA
dUMP
dTMP
thymidylaat synthase
(TS)
Figuur 4. Metabolisme en toxische werking van 5-FOA
5-FOA blijkt een breed werkingsspectrum te hebben. Boeke en medewerkers (1984) vonden
reeds dat 5-FOA de groei inhibeert van de gisten Saccharomyces, Schizosaccharomyces en
Candida. Ook E. coli HB101 bleek gevoelig te zijn terwijl E. coli DB6507, een mutant in het
pyrF gen coderend voor OMPD, niet gevoelig is.
MANU DE GROEVE
II. Literatuurstudie - 21
Dit wees toen reeds op een gelijkaardige werking van 5-FOA bij gisten en bacteriën.
Ondertussen is de inhiberende werking van 5-FOA reeds bij vele verschillende organismen
aangetoond: de groei van onder meer bacteriën, gisten, filamenteuze schimmels, plantaardige
en dierlijke cellen wordt geïnhibeerd vanaf een bepaalde 5-FOA concentratie in het medium.
5.2.3. Creatie en complementatie van OMPD-negatieve mutanten
Verschillende OMPD negatieve mutanten van filamenteuze schimmels konden succesvol
worden geïsoleerd met het 5-FOA resistentie systeem. Daartoe worden meestal een groot
aantal fungale sporen (108) gemuteerd via ultraviolet (UV) bestraling of chemische
mutagentia en vervolgens uitgeplaat op 5-FOA bevattend selectief medium. De 5-FOA
concentraties in de gebruikte selectiemedia liggen in de grootteorde van 0.06-2.5 g/L. Verder
wordt ook nog uracil en/of uridine toegevoegd (5-20 mM) om groei toe te laten van de
auxotrofe mutanten. Een overzicht van de gebruikte mutagentia en de belangrijkste
componenten van de selectieve media is gegeven in Tabel 5.
Tabel 5. Creatie van OMPD- mutanten: gebruikte mutagentia en mediasamenstellingen
Over5-FOA
5-FOAR
Organisme
Methode
Uracil Uridine
kolonies
leving(%)
conc.
Aspergillus
UV
5-80
0.8 g/L
10 mM
11
niger
DEO*
10
A. parasiticus
MNNG*
66
2 g/L
10 mM
46
OMPDkolonies
2
Referentie
Goosen et
al. (1987)
3
Skory et al.
(1990)
Claviceps
UV
40-50
0.06 g/L
10 mM
144
9
Smit &
purpurea
Tudzynski
(1992)
Colletotrichum
Screening
0.87 g/L
5 mM
1
0
Rasmussen
graminicola
WT
et al.
(1992)
A. awamori
Transformatie
0.75 g/L
10 mM
27
16
Gouka et
al. (1995)
Trichoderma
UV
10
1 g/L
0.89
0.41
?
8
Manczinger
harzanium
mM
mM
et al.
(1995)
A. fumigatus
4-nitroquino75
1 g/L
5 mM 5 mM
100
>3
d’Enfert
line-N-oxide
(1996)
Rhizomucor
UV
1
1.2 g/L
2.2
18
1
Yamazaki
pusillus
mM
et al.
(1999)
Aureobasidium
EMS*
20
0.8 g/L
0.45
65
2
Rose et al.
pullulans
mM
(2000)
Rhizopus
MNNG
?
2.5 g/L
4.5
17
5
Skory
oryzae
mM
(2002)
Aspergillus.
UV
10-20
1.2 g/L
20 mM
200
1
Kanamasa
aculeatus
et al.
(2003)
Penicillium
UV+filtratie20
1 g/L
0.57
1
1
Fierro et al.
nalgiovense
aanrijking
mM
(2004)
Mortierella
MNNG
?
1 g/L
0.45
>6
0
Takeno et
alpina
mM
al. (2004)
* DEO = 1,2,7,8-diepoxyoctaan; MNNG = N-methyl-N’-nitro-N-nitrosoguanidine; EMS = ethyl methaan
sulfonaat
MANU DE GROEVE
II. Literatuurstudie - 22
Naast de methode van mutagenese en directe isolatie op 5-FOA bevattend medium, kunnen
OMPD negatieve mutanten ook geselecteerd worden via de filtratieaanrijkingsmethode. Deze
methode is uitermate geschikt voor de isolatie van auxotrofe mutanten van filamenteuze
schimmels. Het aanrijken van auxotrofe mutanten via filtratie van gemuteerde sporen werd
geoptimaliseerd voor Neurospora crassa door Case (1963). Bij de filtratieaanrijking worden
meestal lage mutagene dosissen gebruikt. Het principe van de filtratieaanrijking steunt op het
feit dat bij groei in minimaal medium enkel de prototrofe fungale sporen kunnen kiemen en
verder groeien. Wanneer het medium na een bepaalde incubatieperiode gefilterd wordt, zullen
de gekiemde sporen (prototrofen) op de filter achterblijven terwijl de niet-gekiemde sporen
(auxotrofen) zich in het filtraat bevinden. Het filtraat met niet-gekiemde sporen wordt
vervolgens opnieuw geïncubeerd en gefilterd. Door telkens de filtratiestap te herhalen wordt
het filtraat geleidelijk aan relatief rijker aan auxotrofe mutanten. Na enkele filtratierondes
wordt dan uitgeplaat op compleet medium waarop zowel de auxotrofe als eventueel nog
aanwezige prototrofe sporen kunnen kiemen en verder groeien. Door replica plating op
medium met en zonder de auxotrofe merker kunnen de echte auxotrofen opgespoord worden.
Deze methode bleek efficiënt voor de isolatie van auxotrofe mutanten van filamenteuze
schimmels zoals Trichoderma harzianum (Cassiolato & Soares de Melo, 1999) en Ascolobus
immersus (Lamb & Helmi, 1991). Ook temperatuurgevoelige mutanten van Neurospora
crassa konden op deze manier geïsoleerd worden (Applegate et al., 1978; Yoder, 1979).
Recent werd door Fierro et al. (2004) de isolatie van uridine auxotrofe mutanten van
Penicillium nalgiovense via filtratieaanrijking gerapporteerd. Zij gebruikten een
gemodificeerde methode van Bos & Stadler (1996). Een oplossing van 107 leefbare sporen na
UV-behandeling werd geïnoculeerd in 100 mL Czapeck minimaal medium en na incubatie in
het donker (25°C, 250 rpm, 48 h) gefiltreerd over nylon filters (20 µm poriën). Zij gebruikten
slechts één filtratieronde en de bekomen sporen in het filtraat werden uitgeplaat op compleet
medium. Na testen op uridine auxotrofie bleken van de 1800 gescreende kolonies er 6 uridine
auxotroof te zijn. Eén van deze 6 auxotrofen was 5-FOA resistent en bovendien gemuteerd in
het OMPD gen.
OMPD-negatieve mutanten kunnen zeer efficiënt worden bekomen door homologe
recombinatie van het wild type OMPD gen met een gemuteerd gen via transformatie gevolgd
door selectie op 5-FOA medium. Gouka en medewerkers (1995) creëerden op deze manier
gedefinieerde OMPD mutanten van Aspergillus awamori die werden gebruikt voor de
ontwikkeling van een site-specifiek integratie systeem. De constructie van deze gedefinieerde
mutanten verliep in twee stappen. In de eerste stap werden in vitro mutaties geïntroduceerd in
het gekloneerde OMPD gen van A. awamori. Door de vector te knippen met een restrictieenzym dat in de coderende sequentie van het OMPD gen knipt, en daarna de overhangende
eindjes in te vullen via T4 DNA polymerase gevolgd door ligatie, konden frame shift mutaties
bekomen worden. In de tweede stap voor de constructie van een OMPD mutant werden de
gemuteerde OMPD genen gebruikt om het chromosomale wild type OMPD gen te vervangen
via transformatie. De resulterende mutanten werden geselecteerd op 5-FOA medium en
II. Literatuurstudie - 23
MANU DE GROEVE
onderzocht op uridine-auxotrofie. De auxotrofen werden vervolgens verder onderzocht op het
integratiepatroon van het getransformeerde DNA. Deze methode bleek zeer efficiënt voor de
isolatie van gedefinieerde OMPD-negatieve mutanten. Gemiddeld werden 60% van de 5-FOA
resistente transformanten bekomen via gene replacement.
Het 5-FOA resistentiesysteem, dat zowel positieve als negatieve selectie toelaat, kan ook
gebruikt worden voor de ontwikkeling van nieuwe strategiën voor de manipulatie van fungale
genomen (d’Enfert, 1996). Zo werd door Alani et al. (1987) de zogenaamde “ura-blaster”
ontwikkeld voor het uitschakelen van genen bij gist. d’Enfert (1996) construeerde een
analoge pyrG-blaster voor Aspergillus fumigatus en toonde aan dat deze kan gebruikt worden
voor het uitschakelen van meerdere genen in deze schimmel. De pyrG-blaster is een
drieledige cassette die een selectiemerker bevat, in dit geval het pyrG gen van A. niger,
geflankeerd door twee identieke elementen die een directe repeat vormen en recombinatie
toelaten. In dit geval werden twee kopijen van het gen coderend voor neomycin
phosphotransferase (neo) als direct repeat gebruikt. De blaster is geflankeerd door de
sequenties van het uit te schakelen gen zodat gerichte integratie in het doelgen kan gebeuren.
Een uracil-auxotrofe stam (pyrG negatief) wordt vervolgens getransformeerd met het
construct en de prototrofe transformanten worden geselecteerd. De prototrofe stammen die de
gewenste insertie in het doelgen bezitten worden vervolgens onderworpen aan negatieve
selectie door ze over te enten op medium met 5-FOA en uracil. Enkel de stammen die het
pyrG gen verloren hebben door recombinatie tussen de directe repeats zullen op dit medium
kunnen groeien. De gewenste mutatie in het doelgen blijft behouden omdat een van de directe
repeats na recombinatie in het doelgen blijft. Een gevolg van deze strategie is dat de
resulterende stam opnieuw uracil/uridine auxotroof is en kan gebruikt worden voor een
volgende transformatieronde. Op deze manier kunnen verschillende genen of meerdere
kopijen van hetzelfde gen uitgeschakeld worden.
uit te schakelen gen
chromosoom
neo
neo
selecteerbaar merkergen (bv. pyrG)
neo
neo
+ 5-FOA
neo
neo
neo
Figuur 5. Schematische voorstelling van de pyrG-blaster techniek om meerdere genen uit te schakelen
(Brakhage & Langfelder, 2002)
MANU DE GROEVE
II. Literatuurstudie - 24
Om met zekerheid een bepaalde 5-FOA resistente mutant aan te duiden als OPRT negatief
dan wel als OMPD negatief, worden deze aan bepaalde testen onderworpen. Een eerste test
bestaat er in de mutanten over te enten op zowel minimaal medium als op uracil/uridine
bevattend medium. Niet alle 5-FOA resistente mutanten zijn immers uracil of uridine
auxotroof. Skory en medewerkers (1990) observeerden bij A. parasiticus dat 22% van de 5FOA resistente mutanten stabiele uridine auxotrofen waren. d’Enfert (1996) vond bij A.
fumigatus 79 uracil/uridine auxotrofen op 100 5-FOA resistente mutanten. Bij Claviceps
purpurea bleken 12.5% van de 5-FOA resistente kolonies pyrimidine auxotroof (Smit &
Tudzynski, 1992). De uracil/uridine auxotrofe mutanten worden vervolgens onderworpen aan
meer gespecialiseerde testen om te onderscheiden tussen OPRT- en OMPD-negatieve
mutanten.
Zoals reeds eerder vermeld zijn zowel OPRT-negatieve als OMPD-negatieve mutanten
uracil/uridine auxotroof en resistent aan 5-FOA. Deze auxotrofie en 5-FOA resistentie is het
gevolg van mutaties in het OPRT gen of het OMPD gen die leiden tot verlies van
enzymatische activiteit. Hierdoor worden UMP en het toxische 5-FUMP niet meer
geproduceerd. Door de enzymatische activiteit te meten van OPRT en/of OMPD kan men een
onderscheid maken tussen OPRT-negatieve en OMPD-negatieve mutanten. Deze
enzymatische testen zijn gebaseerd op de omzetting van orotaat tot OMP (OPRT) en de
omzetting van OMP tot UMP (OMPD). Enzymatische testen werden onder andere gebruikt
door Skory et al. (1990), Smit & Tudzynski (1992), Rasmussen et al. (1992), Skory (2002) en
Takeno et al. (2004) om een onderscheid te maken tussen OPRT en OMPD negatieve
mutanten van filamenteuze schimmels.
Rasmussen en medewerkers (1992) vonden na screening van wild type sporen van
Colletotrichum graminicola één spontane 5-FOA resistente mutant. Na genetische analyse via
seksuele kruisingen bleek dit een mutatie te zijn in één bepaalde locus, PYR1 genoemd. Via
enzymatische testen kon aangetoond worden dat het een OPRT negatieve mutant is. Takeno
et al. (2004) verkregen na MNNG mutagenese zes uracil auxotrofe mutanten die resistent
waren aan 5-FOA. Uit enzymatische testen bleek dat deze allen OPRT deficiënt waren.
Een andere mogelijkheid om een onderscheid te maken tussen OPRT- en OMPD-negatieve
mutanten is via complementatie met het homologe OMPD gen. Indien het homologe OMPD
gen nog niet geïsoleerd werd kan ook een heteroloog OMPD gen gebruikt worden. Indien de
auxotrofe en 5-FOA resistente mutant OMPD deficiënt is moet deze kunnen
gecomplementeerd worden met een werkend OMPD gen. Indien dit niet het geval is, is de
mutant waarschijnlijk OPRT negatief. In Tabel 6 wordt een overzicht gegeven van de OMPD
genen die werden gebruikt om via complementatie een onderscheid te maken tussen OPRT en
OMPD-negatieve mutanten.
MANU DE GROEVE
II. Literatuurstudie - 25
Tabel 6. Overzicht van de gebruikte OMPD genen voor complementatie van OMPD-negatieve mutanten
Organisme
OMPD gen waarmee gecomplementeerd werd
Referentie
Trichoderma harzanium
pyr4 gen van Neurospora crassa
Manczinger et al. (1995)
Aspergillus fumigatus
pyrG gen van A. niger
d’Enfert (1996)
Rhizomucor pusillus
homoloog pyr4 gen van R. pusillus
Yamazaki et al. (1999)
Aspergillus sojae
pyrG gen van A. niger
Heerikhuisen
et
al.
(2001)
Aspergillus aculeatus
pyrG gen van A. nidulans
Kanamasa et al. (2003)
Penicillium nalgiovense
pyrG gen van P. chrysogenum
Fierro et al. (2004)
Heerikhuisen et al. (2001) probeerden OMPD negatieve mutanten van Aspergillus sojae te
bekomen via verschillende mutagenese technieken en directe selectie op medium met 5-FOA
en uridine. Zij verkregen vele 5-FOA resistente kolonies maar geen enkele bleek uridineauxotroof te zijn. De experimenten werden verschillende keren herhaald maar nooit werden
onder de 5-FOA resistente kolonies uridine-auxotrofe mutanten gevonden. Pas na het
toevoegen van uracil aan het selectiemedium konden 5-FOA resistente kolonies worden
bekomen die auxotroof zijn voor uracil. Deze konden bovendien gecomplementeerd worden
met het pyrG gen van A. niger. Uit latere experimenten bleek dat de A. sojae pyrG mutanten
niet kunnen groeien op minimaal medium met enkel uridine. Dit was nog niet eerder
geobserveerd bij verwante Aspergillus soorten.
De beschikbaarheid van een OMPD negatieve stam en een transformatiesysteem gebaseerd op
de complementatie van uracil/uridine auxotrofie met het homologe OMPD gen biedt
verschillende voordelen ten opzichte van andere selectiemerkers zoals antibiotica resistentie
of het acetamidase (amdS) systeem (Fierro et al., 2004):
• De transformatie efficiëntie is veel hoger dan bij elk ander systeem
• De pyrG-blaster kan gebruikt worden om verschillende genen uit te schakelen in
opeenvolgende transformatieronden (d’Enfert, 1996)
• Gebruik makend van een mutant OMPD gen in de transformatievector kan elke
constructie als een single-copy op de OMPD locus worden geïntroduceerd via gene
targeting (Gouka et al., 1995)
• Er is veel bezorgdheid om het gebruik van heterologe genen voor de verbetering van
stammen in de voedselindustrie; het gebruik van een homologe selectiemerker zoals het
OMPD gen voorkomt dit probleem
6. HETEROLOGE EIWITPRODUCTIE DOOR SCHIMMELS
Fungi worden algemeen gebruikt als ‘celfabrieken’ voor de productie van recombinante
proteïnen, gebruik makend van sterke en gereguleerde promotoren (Larsen et al., 2004). Eén
van de meest gebruikte promotoren is de Aspergillus oryzae Taka-amylase promotor die
wordt gebruikt voor de productie van een groot aantal enzymen (Brakhage et al., 1999).
Andere veel gebruikte fungale promotoren zijn de Aspergillus gpdA, alcR en glaA promotoren
II. Literatuurstudie - 26
MANU DE GROEVE
(Punt et al., 1995; Santerre-Heriksen et al., 1999; Mathieu et al., 2000). Aspergillus
promotoren werden reeds met succes gebruikt bij andere Ascomyceten voor genetische
studies of voor de ontwikkeling van transformatiesystemen.
6.1. Structuur van fungale expressiesystemen
Heterologe genexpressiesystemen in filamenteuze schimmels bestaan uit een expressievector
die het tot expressie te brengen gen bevat geflankeerd door niet-vertaalde sequenties, en een
kloneringsgastheer die het heterologe eiwit optimaal kan synthetiseren (Radzio & Kück,
1997). Fungale expressievectoren bevatten gewoonlijk expressiecassettes die uit verschillende
componenten bestaan. Zoals weergegeven in Figuur 6 is het heterologe gen upstream
geflankeerd door DNA sequenties die verantwoordelijk zijn voor transcriptionele controle en
initiatie van translatie. Indien het genproduct gesecreteerd moet worden wordt een
signaalsequentie gefuseerd met de coderende regio van het heterologe gen. Downstream is het
heterologe gen geflankeerd door sequenties die de transcriptieterminatie controleren.
bindingsplaatsen van
specifieke transcriptiefactoren
transcriptionele controle
+ initiatie van translatie
ATG
translationeel
startcodon
signaalsequentie
voor secretie
TAA/TAG/TGA
translationeel
stopcodon
heteroloog gen
transcriptie
terminatie
Figuur 6. Schematische voorstelling van een expressiecassette voor de synthese en
secretie van heterologe eiwitten in filamenteuze schimmels
6.2. Transcriptionele controle
Efficiënte synthese van heterologe eiwitten kan bereikt worden door het heterologe gen onder
controle te plaatsen van een sterke promotor. Algemeen kunnen de fungale promotoren voor
heterologe genexpressie verdeeld worden in twee groepen (Saunders et al., 1989), namelijk
constitutieve promotoren afkomstig van huishoudgenen en induceerbare promotoren.
6.2.1. Constitutieve promotoren
De meest gebruikte constitutieve promotor is de glyceraldehyde-3-fosfaat dehydrogenase
(gpdA) promotor van A. nidulans. Deze promotor is functioneel in verschillende soorten
waaronder P. chrysogenum, A. nidulans en A. niger. Een nadeel hiervan zijn de continue,
groeigerelateerde expressieniveau’s. Een deel van het GPD gen van Myrothecium gramineum
MUCL 39210 kon reeds worden geïsoleerd met gedegenereerde primers (Jonniaux et al.,
2004). Met de primers werd een stuk van 850 bp geamplificeerd dat een groot intron (456 bp)
bevat. De afgeleide eiwit sequentie van de coderende sequentie vertoont het meest homologie
MANU DE GROEVE
II. Literatuurstudie - 27
(88%) met het GPD enzym van Neurospora crassa. De GPD promotor kan gebruikt worden
in expressiesystemen voor M. gramineum schimmel.
Het gebruik van sterke promotoren voor de expressie van heterologe genen in geschikte
gastheerorganismen is van groot belang bij biotechnologische toepassingen. De
glyceraldehyde-3-fosfaat dehydrogenase (GPD; EC 1.2.1.12) promotor is een veelbelovende
kandidaat (Kuo et al., 2004). GPD is een van de sleutelenzymen in de glycolytische en
gluconeogenese pathway. Het zorgt voor de omzetting van D-glyceraldehyde-3-fosfaat tot
1,3-bifosfoglyceraat en omgekeerd.
In vele eukaryotische micro-organismen worden GPD coderende genen constitutief en in
grote hoeveelheden tot expressie gebracht. Het oplosbare cellulaire eiwitgehalte van S.
cerevisiae en andere hogere eukaryoten zou tot 5% uit GPD bestaan (Piechaczyk et al., 1984).
Bovendien staat het mRNA van GPD in voor 2-5% van het totale poly (A)+ RNA aanwezig in
gisten (Holland & Holland, 1978). Het feit dat GPD in hoge hoeveelheden voorkomt wijst er
op dat het GPD gen gereguleerd wordt door een zeer actieve promotor (Kuo et al., 2004).
Punt et al. (1988) vergeleken de promotorregio van het gpdA gen van A. nidulans met de
promotorsequenties van andere glycolytische Aspergillus genen. Zij vonden twee regio’s van
gelijkaardige sequenties, namelijk een pgk box 600 bp upstream van de transcriptiestartplaats
(tsp) en een CT-rijke regio onmiddellijk vóór de transcriptiestartplaats. Door de
promotorregio van het gpdA gen van A. nidulans te vergelijken met de gpdA promotorregio
van A. niger vonden Punt et al. (1990) een regio met 96% homologie die de gpd box werd
genoemd. Deze gpd box bevindt zich 250 bp upstream van de major tsp in A. nidulans en op
ongeveer dezelfde afstand bij A. niger. Zowel de gpd box als de pgk box bevatten een
aanzienlijk aantal inverted repeats. Aangezien inverted repeats beschouwd worden als
regulatorische sites in promotorsequenties door binding van transcriptiefactoren en/of
regulatorische eiwitten (Johnson & McKnight, 1989) kan dit betekenen dat de pgk en gpd
boxen deel uitmaken van zo’n regulatorische site.
GPD promotoren werden reeds met succes gebruikt voor de expressie van heterologe genen in
filamenteuze fungi (Juge et al., 1998; Punt et al., 1987). Enkele voorbeelden zijn
weergegeven in Tabel 7.
Tabel 7. Toepassingen van de GPD promotor in filamenteuze schimmels voor de heterologe expressie van fungale
en andere eukaryotische genen (Radzio & Kück, 1997)
Gastheer
Promotor
Heteroloog eiwit
Opbrengst Referentie
A. awamori gpd (Aspergillus sp.)
glucoamylase (A. niger)
4.6 g/L
Finkelstein et al. (1989)
A. niger
gpdA (A. nidulans)
lysozyme (kippeneiwit)
1 mg/L
Archer et al. (1990)
MANU DE GROEVE
II. Literatuurstudie - 28
6.2.2. Induceerbare promotoren
Van de induceerbare promotoren wordt de glucoamylase (glaA) promotor van A. niger het
meest gebruikt. Andere gebruikte induceerbare promotoren zijn de alcohol dehydrogenase
(alcA) promotor van A. nidulans, de A. oryzae amylase (amy) promotor en de aldehyde
dehydrogenase (adhA) promotor van A. nidulans. Recent werd de A. oryzae thiA promotor
ontwikkeld als induceerbare promotor (Shoji et al., 2005). Het thiA gen codeert voor een
enzym dat betrokken is in de biosynthese van thiamine en is transcriptioneel gereguleerd door
thiamine. De expressieniveau’s konden gecontroleerd worden aan de hand van de
concentratie van extern thiamine. Pachlinger et al. (2005) ontwikkelden een metabolisch
onafhankelijk en sterk reguleerbaar expressiesysteem voor Aspergillus soorten. Zij maakten
gebruik van estrogeen-responsieve elementen (ERE’s) van het humane estrogeen receptor gen
zodat Aspergillus soorten gevoelig werden voor estrogene verbindingen. Op deze manier
konden de ERE’s de transcriptie reguleren van een reportergen. Induceerbare promotoren zijn
vooral nuttig wanneer het tot expressie te brengen gen onstabiel of toxisch is voor het
gastheerorganisme (Radzio & Kück, 1997). Bovendien kan door het gebruik van sterke
constitutieve promotoren het fungale secretiesysteem overbelast worden wat resulteert in
verkeerde opvouwing en glycosylatie van de geproduceerde eiwitten (Pachlinger et al., 2005).
Dit kan dan de eiwitdegradatie pathway induceren resulterend in lagere eiwitopbrengsten.
In de meeste gevallen leiden homologe promotoren tot meer efficiënte expressie dan
heterologe promotoren (Jeenes et al., 1991).
6.2.3. Regulatorische sequenties in fungale promotoren
Het is nuttig een onderscheid te maken tussen de upstream elementen die de transcriptie
startplaats (tsp) bepalen en een minimaal niveau van transcriptie teweegbrengen, en de andere
sequenties die transcriptie activatie elementen worden genoemd (Jeenes et al., 1991). Hoewel
dit een enigszins kunstmatig onderscheid is wordt de term ‘kernpromotor’ gebruikt om de
minimale sequentie aan te duiden die nodig is om transcriptie te initiëren vanaf de major tsp.
Dit resulteert echter niet altijd in even hoge mRNA niveaus als de basale niveau’s van wild
type genen.
In de meeste eukaryotische systemen blijkt de TATA-box (consensus TATAAA) essentieel
voor een functionele kernpromotor (Sawadogo & Sentenac, 1990). In de hogere eukaryoten
ligt deze sequentie bijna altijd 25-30 bp upstream van de tsp en mutaties in deze sequentie
leiden gewoonlijk tot aberrante transcriptie initiatie (Jeenes et al., 1991). Bij S. cerevisiae
komen TATA-sequenties veel minder voor. Sommige genen bevatten geen TATA-box terwijl
andere genen één of meer kopijen van dit element bevatten tot 120 bp upstream van de tsp
(Struhl, 1989). De genen van filamenteuze schimmels lijken meer op deze van gist in die zin
dat TATA-boxen zeldzaam zijn in fungale promotoren (Ballance, 1986). Hoewel AT-rijke
sequenties werden geobserveerd upstream van transcriptie initiatie plaatsen, is hun locatie
variabel en hun functionele significantie onbekend. Deletie analyse van de A. nidulans oliC
MANU DE GROEVE
II. Literatuurstudie - 29
promotor (Turner et al., 1989) toonde aan dat de TATA-box belangrijk is voor het bepalen
van het tsp maar dat deze het niveau van expressie niet beïnvloedt.
Het belangrijkste element dat de tsp bepaalt bij filamenteuze schimmels is de CT-box. Dit is
een pyrimidinerijke sequentie van 8-12 bp lang die direct upstream wordt gevonden van een
aanzienlijk aantal fungale genen (Gurr et al., 1987). Functionele analyse van de gpdA
promotor van A. nidulans toonde aan dat deletie van deze sequentie resulteert in aberrante
initiatie en suggereerde dat deze regio voldoende is voor het bepalen van de tsp (Punt et al.,
1990).
In vele hogere eukaryoten vertoont de CAAT-box kernpromotor activiteit en bevindt zich
meestal 80-90 bp upstream van het tsp (Breathnach & Chambon, 1981). Bij filamenteuze
schimmels is de CAAT box vaak afwezig, of indien toch aanwezig op variabele afstand van
de tsp (60-120 bp).
6.3. Terminatie van transcriptie
Tot op heden is slechts weinig gekend over de mechanismen van transcriptieterminatie bij
filamenteuze schimmels. De meeste expressiecassettes bevatten downstream van het
heterologe gen de 3’ onvertaalde regio van een gekend homoloog gen zodat er een efficiënte
terminatie van transcriptie is.
De meest belangrijke sequenties die geassocieerd zijn met transcriptieterminatie en
polyadenylatie van mRNA in eukaryoten zijn het YGTGTTYY en het AAUAAA motief (Jeenes
et al., 1991). Deze sequenties werden reeds gevonden in genen van filamenteuze schimmels,
maar zijn meestal aanwezig in een verkorte vorm (AUAA) ofwel ontbreken ze volledig. Mede
hierdoor is hun functionele rol onduidelijk.
Uit studies bij hogere eukaryoten is bekend dat de 3’ onvertaalde regio niet enkel de
transcriptieterminatie controleert, maar ook belangrijke functies heeft met betrekking tot
mRNA stabiliteit (Ross, 1996). Verscheidene onderzoeken toonden aan dat de terminator
sequentie bij recombinante constructen een belangrijke rol speelt bij de productie en/of
stabiliteit van mRNA. Constructie van gpdA-lacZ fusies in A. nidulans verminderde de
mRNA niveau’s tot een derde van het wild type gpdA gen (Punt et al., 1990). Dit werd
toegeschreven aan het gebruik van een heterologe terminator (A. nidulans trpC) of lacZ
sequenties. Of deze verminderde mRNA niveau’s te wijten zijn aan instabiliteit of een
verminderde transcriptie werd niet onderzocht. Via Northern blot analyse toonden Punt et al.
(1991) wel aan dat mRNA afkomstig van een recombinant glaA gen (A. niger) onder de
controle van de gpdA promotor (A. nidulans) minder stabiel was dan het wild type glaA
transcript.
MANU DE GROEVE
II. Literatuurstudie - 30
6.4. Secretie van heterologe eiwitten
De secretie van heterologe eiwitten biedt verschillende voordelen ten opzichte van
intracellulaire productie (Radzio & Kück, 1997):
• de opzuivering van het recombinante eiwit wordt vergemakkelijkt omdat het
mycelium niet hoeft opengebroken te worden
• het eiwit wordt niet blootgesteld aan intracellulaire proteasen
• het metabolisme van de schimmel wordt niet verstoord door het vreemde eiwit
Eiwitten kunnen naar het secretiesysteem getransloceerd worden door de insertie van een
secretie signaalsequentie tussen de promotor en het heterologe gen. Deze sequentie codeert
voor een signaal peptide van 13-50 aminozuren dat het eiwit naar het endoplasmatisch
reticulum brengt en vervolgens de secretiepathway binnenkomt.
Een speciale eigenschap van eiwitsecretie in filamenteuze schimmels is dat eiwitten
hoofdzakelijk aan de uiteinden van de groeiende hyfen worden gesecreteerd in het medium
(Punt et al., 1994). Het moleculair mechanisme van eiwitsecretie in schimmels is nog niet in
detail onderzocht maar omdat vele heterologe signaalsequenties functioneel zijn in schimmels
wordt aangenomen dat deze gelijkaardig is aan de mechanismen in andere organismen
(Radzio & Kuck, 1997).
Een alternatieve strategie voor de productie van heterologe eiwitten in filamenteuze
schimmels is de fusie van het heterologe gen met de complete coderende regio van een
homoloog gen dat sterk tot expressie komt. Het gesecreteerde homologe eiwit dient dan als
drager voor het heterologe eiwit wat in sommige gevallen leidt tot verbeterde opbrengsten. Zo
kon de opbrengst van kalf chymosine in A. awamori significant verbeterd worden door fusie
van het prochymosine gen met de volledige coderende sequentie van het fungale
glucoamylase, in plaats van enkel de glucoamylase leader sequentie (Ward et al., 1990). Een
gelijkaardige strategie in A. awamori waarbij Korman et al. (1990) α-amylase-prochymosine
fusies gebruikten zorgde ook voor verhoogde opbrengsten. Deze observaties leiden tot de
suggestie dat de coderende sequenties van extracellulaire enzymen informatie bevatten die de
secretie bevorderen. Het probleem van het splitsen van het heterologe eiwit en het homologe
drager eiwit wordt meestal opgelost door de insertie van een herkenningssequentie voor een
KEX2 endopeptidase (Radzio & Kück, 1997). Dit endopeptidase splitst eiwitten
endoproteolytisch tussen twee naast elkaar gelegen basische aminozuren, bij voorkeur tussen
lysine en arginine.
Ook werd aangetoond dat de sequenties van sommige recombinante eiwitten incompatibel
zijn met de secretiepathway van filamenteuze schimmels. Dit resulteert dan in geen secretie
(Saunders et al., 1989), transport naar andere celcompartimenten (Punt et al., 1991) of
proteolytische afbraak door intracellulaire proteasen (Calmels et al., 1991).
MANU DE GROEVE
II. Literatuurstudie - 31
6.5. Post-translationele modificaties
Gedurende de passage door het secretiesysteem worden eiwitten onderworpen aan
verschillende post-translationele modificaties zoals vorming van disulfide bruggen,
proteolyse en glycosylatie (Radzio & Kück, 1997). Samen met eiwit-opvouwing processen
spelen deze modificaties een belangrijke rol in het bepalen van de structuur van vele eiwitten.
De opvouwing van gesecreteerde eiwitten gebeurt in het lumen van het endoplasmatisch
reticulum (ER) en is een energievereisend proces gelijkaardig aan dit in vele andere
eukaryoten. De tertiaire structuur van vele extracellulaire eiwitten wordt gestabiliseerd door
disulfide bruggen. Het eerste fungale eiwit-disulfide isomerase werd gekloneerd uit A. niger
(Jeenes et al., 1996). Proteolytisch knippen is een ander belangrijk type post-translationele
modificatie in het ER. De meest voorkomende proteolytische modificatie is de verwijdering
van signaalsequenties door signaalpeptidasen na translocatie door de ER membraan (Dalbey
& von Heijne, 1992) en het endoproteolytische knippen door endoproteasen zoals KEX2.
De structuur en functie van gesecreteerde eiwitten kan sterk beïnvloed worden door
glycosylatie. De glycosylaties in filamenteuze schimmels zijn, voor zover geweten, zeer
gelijkaardig aan deze in andere eukaryoten, vooral S. cerevisiae (Radzio & Kück, 1997). In
tegenstelling tot S. cerevisiae waar hypermannosylatie frequent voorkomt, zijn de
glycosylatie patronen in filamenteuze schimmels meer gelijkaardig aan deze in hogere
eukaryoten (Jenkins et al., 1996). Er worden twee processen onderscheiden: N-glycosylatie
en O-glycosylatie. Gerichte modificatie van de glycosylatie kan gebruikt worden om
heterologe genexpressie te verbeteren. Zo kon door de insertie van een additionele Nglycosylatie plaats in kalf chymosine de opbrengst tot meer dan drie maal verhoogd worden
in A. awamori (Ward, 1989).
6.6. Reportergenen en expressiestudies
De eerste studies over heterologe genexpressie bij filamenteuze schimmels werden uitgevoerd
met modelgenen, waaronder de lacZ en gusA reportergenen uit E. coli. Het lacZ gen coderend
voor β-galactosidase en het gusA gen coderend voor β-glucuronidase worden frequent
gebruikt voor expressiestudies (Punt et al., 1991). Deze laten de studie toe van 5’ sequenties
die nodig zijn voor gereguleerde genexpressie (Hynes, 1996). Mini-promotors gefuseerd met
reportergenen kunnen gebruikt worden om de regulatorische effecten van ingevoegde
sequenties te bepalen (Punt et al., 1991; Punt et al., 1995).
Bacteriële reportergenen zijn een krachtig hulpmiddel bij verschillende onderzoeken en
werden reeds vaak met succes toegepast bij filamenteuze schimmels. Ook eukaryotische
reportergenen worden meer en meer gebruikt. Shoji et al. (2005) bijvoorbeeld gebruikten het
enhanced green fluorescent protein (EGFP) voor expressiestudies met de induceerbare A.
oryzae thiA promotor.
II. Literatuurstudie - 32
MANU DE GROEVE
Om de productieniveau’s van verschillende eiwitten betrouwbaar te kunnen vergelijken is het
nodig dat de productiestammen de expressievectoren in single-copy opnemen en op een
identieke chromosomale plaats. Het kopijaantal en de chromosomale omgeving kunnen
immers de genexpressie beïnvloeden (Gouka et al., 1995). Voor verschillende Aspergillus
soorten werden reeds expressie/analyse systemen ontwikkeld die de selectie
vergemakkelijken van single-copy transformanten die de expressievector op een specifieke
locus bevatten. Deze systemen zijn gebaseerd op een mutant pyrG van A. niger (Van Gorcom
& Van den Hondel, 1988) of mutante argB genen van A. nidulans (Hamer & Timberlake,
1987) als selectiemerker en de overeenkomstige pyrG of argB mutanten als recipiënten.
Gouka en medewerkers (1995) ontwikkelden voor A. awamori een plaatsspecifiek
integratiesysteem gebaseerd op mutante pyrG genen. Na transformatie van een gedefinieerde
OMPD-negatieve mutant (die een mutatie aan de 3’ kant van het pyrG gen bevat) met
vectoren die een mutatie bevatten aan de 5’ kant van pyrG, worden hoge frequenties bekomen
aan mutanten met een single-copy van de vector op de pyrG locus. Op deze manier kan een
expressievector in single-copy in het genoom geïntegreerd worden (zie Figuur 7).
CREATIE OMPD KNOCK-OUT MET 3' MUTATIE
wild type OMPD gen
COMPLEMENTATIE VAN OMPD KNOCK-OUT
3' mutant OMPD gen
5' mutant OMPD gen
Transformatie met mutant
OMPD gen dat een mutatie
bevat in de 3' regio
Transformatie met
expressievector die een
mutant OMPD gen bevat
met een 5' mutatie
expressiecassette
3' mutant OMPD gen
actief OMPD gen
inactief
5'
3'
expressiecassette
via homologe recombinatie wordt het wild type gen
vervangen door het mutante gen
RESULTERENDE MUTANT IS 5-FOA
RESISTENT EN URACIL/URIDINE AUXOTROOF
HOGE FREQUENTIE AAN SINGLE-COPY
TRANSFORMANTEN DIE ÉÉN KOPIJ VAN DE
EXPRESSIECASSETTE BEVATTEN
Figuur 7. Strategie voor de creatie van single-copy transformanten volgens Gouka et al. (1995)
6.7. Overproductie van heterologe eiwitten
Na de ontwikkeling van verschillende systemen voor de expressie van heterologe modelgenen
werd de aandacht gericht op genen die coderen voor farmaceutische en industrieel belangrijke
producten. Vooral de productie van humane eiwitten door fungale systemen is van
commercieel belang, bijvoorbeeld humaan α-interferon, interleukine-6, lactoferrine en
insuline. Een andere belangrijke toepassing is de productie van antilichaamfragmenten door
filamenteuze schimmels.
Het is moeilijk algemene conclusies te trekken wanneer bij filamenteuze schimmels
verschillende expressieniveau’s van recombinante eiwitten worden vergeleken (Jeenes et al.,
1991). De verschillen in expressieniveau’s kunnen immers het gevolg zijn van het aantal
genkopijen, de plaats van integratie van de vector in het genoom en de groeicondities, maar
MANU DE GROEVE
II. Literatuurstudie - 33
ook van het gebruikte type expressie- of secretiesignaal. Wat wel algemeen kan besloten
worden is dat bij filamenteuze schimmels de niveau’s van eiwitsecretie (zowel homologe als
heterologe fungale genen) vaak in de grootte-orde van g/L liggen (tot 30 g/L), terwijl deze bij
andere organismen eerder in de orde van mg/L liggen (Jeenes et al., 1991). De eiwitsecretie
van niet-fungale genen door filamenteuze schimmels is echter veel lager dan van fungale
genen en bereikt meestal slechts het mg/L niveau. De productie van het heterologe
kippeneiwit lysozyme in A. niger bleek vijf maal lager te zijn dat de productie van het
homologe glucoamylase hoewel dezelfde promotor werd gebruikt en evenveel kopijen
aanwezig waren in het genoom (Archer et al., 1994). De reden voor deze discrepantie is
waarschijnlijk te wijten aan post-translationele eiwitmodificatie (Radzio & Kück, 1997).
Omdat hierover slechts weinig gekend is bij filamenteuze schimmels is de optimalisatie ervan
moeilijk.
Transcriptie van recombinante genen in filamenteuze schimmels lijkt niet gelimiteerd te zijn.
Hoewel er wel problemen kunnen zijn met eiwit opbrengst worden in deze gevallen meestal
toch aanvaardbare mRNA niveau’s waargenomen (Carrez et al., 1990). Of hetzelfde kan
gezegd worden van translatie is minder duidelijk. Translatie initiatie begint bij filamenteuze
genen meestal bij de eerste ATG in de sequentie. Bijna altijd bevindt zich een purine (meestal
A) op de -3 positie (Gurr et al., 1987). Bij eukaryoten kan translatie gemoduleerd worden
door de sequenties die het startcodon omringen (Kozak, 1986). In A. nidulans werd
aangetoond dat de expressie van humaan α2-interferon door de alcA promotor verdubbeld
wordt wanneer de 8 bp direct upstream van het startcodon vervangen worden door die van de
wild type alcA promotor (Gwynne et al., 1987). Bij filamenteuze schimmels wordt vaak
codon bias waargenomen, vooral bij genen die tot hoge expressie komen (Clements &
Roberts, 1986). In vele gevallen uit deze codon bias zich in de voorkeur voor een C of G in de
derde positie. De introductie van zeldzame codons (NNA) in het am gen van N. crassa leidde
tot een verminderde eiwitopbrengst tot 35% van het wild type niveau terwijl de mRNA
niveau’s dezelfde bleven (Kinnaird et al., 1991).
Het gebruik van plaatsspecifieke integratie vectoren laat onderzoek toe van de parameters die
de expressie van recombinante genen beïnvloeden. Van Hartingsveldt et al. (1991) gebruikten
zulke vectoren in A. niger. Door gebruik te maken van glucoamylase leader peptidechymosine fusies konden zij twee belangrijke zaken observeren. Ten eerste bleek dat
afhankelijk van het gebruikte type leader peptide de mRNA niveau’s met een factor 10
konden variëren. Dit wees er op dat sequenties die coderen voor de leader peptiden van hoog
geëxpresseerde genen mee instaan voor initiatie van transcriptie of mRNA stabiliteit. Verder
werd vastgesteld dat er geen directe correlatie bestaat tussen mRNA niveau’s en de
uiteindelijke eiwitopbrengst. Een construct dat 90% minder mRNA opleverde dan een ander
leverde slechts 50% minder eiwit op. Gouka en medewerkers (1996) vergeleken bij A.
awamori de productie van een homoloog eiwit en verschillende fungale en niet-fungale
eiwitten. Zij observeerden sterke verschillen in steady-state mRNA niveau’s waarbij fungale
genen de hoogste niveau’s en niet-fungale genen veel lagere niveau’s vertoonden. In alle
MANU DE GROEVE
II. Literatuurstudie - 34
gevallen waren de eiwitniveau’s gecorreleerd met de mRNA niveau’s, behalve voor humaan
interleukine 6. Hier werden relatief hoge mRNA niveau’s geobserveerd terwijl slechts een
zeer lage eiwitopbrengst werd bekomen die waarschijnlijk te wijten is aan intracellulaire
eiwitdegradatie.
Verbeteren van genexpressie
Verschillende strategieën werden ontwikkeld om de heterologe eiwitopbrengst te verhogen
(Gouka et al., 1996):
• modificatie van het eiwit, random mutagenese en screening naar hogere
productieniveau’s
• het gebruik van protease-deficiënte kloneringsgastheren
• de introductie van een hoog aantal kopijen van de expressiecassette in het genoom
• het gebruik van sterke promotoren en secretiesignalen (zie hoger)
• het gebruik van genfusies met sterk gesecreteerde homologe eiwitten (zie hoger)
• ontwikkeling van een geschikt fermentatiemedium
Klassieke methoden zoals random mutagenese en daaropvolgende screening van de
resulterende mutanten werden reeds succesvol toegepast zoals bijvoorbeeld bij het verbeteren
van de chymosine productie in A. awamori (Dunn-Coleman et al., 1991).
Zelfs wanneer heterologe eiwitten succesvol gesecreteerd worden kan een groot deel van de
opbrengst verloren gaan door extracellulaire protease degradatie. Heterologe eiwitten zijn
gevoeliger aan proteolyse dan homologe eiwitten (Wang et al., 2005). Mattern et al. (1992)
konden aantonen dat het gebruik van protease-deficiënte mutanten van A. niger resulteerde in
hogere opbrengsten van heterologe eiwitten. Proteolytische degradatie door fungale proteasen
wordt beschouwd als een van de grootste problemen bij heterologe eiwitproductie.
Zoals reeds eerder vermeld resulteert transformatie van filamenteuze schimmels vaak in nietspecifieke integratie van het recombinante DNA in het chromosoom. Een veel voorkomende
eigenschap van dit proces is de duplicatie van het geïntegreerde DNA waardoor vele kopijen
worden geïntroduceerd in het genoom. Hoewel in het algemeen kan gesteld worden dat een
hoger aantal kopijen resulteert in een hogere eiwitopbrengst zijn deze twee niet strict
gecorreleerd (Jeenes et al., 1991). Het feit dat DNA integreert in regio’s met verschillende
transcriptie-efficiënties kan hiervoor een verklaring zijn. Om te voorkomen dat het
recombinant DNA integreert op een plaats in het chromosoom met een lage transcriptieefficiëntie kan gebruik gemaakt worden van plaatsspecifieke integratievectoren. Deze
vectoren bevatten een expressie cassette die geflankeerd is met de sequenties van een nietessentieel gen waarvan geweten is dat het tot hoge expressie komt. Via homologe
recombinatie wordt de cassette dan geïntegreerd in de transcriptioneel actieve regio. Zulke
gene replacement vectoren gebaseerd op de A. niger glaA (van Hartingsveldt et al., 1991) en
T. reesei cbh1 (Uusitalo et al., 1991) flankerende sequenties werden reeds met succes
gebruikt.
MANU DE GROEVE
II. Literatuurstudie - 35
Transformanten met vele kopijen van DNA dat sequenties bevat waar positief regulatorische
eiwitten op kunnen binden, blijken een verminderde expressie te hebben van genen die door
deze eiwitten geactiveerd worden. (Hynes, 1996). Dit fenomeen wordt toegeschreven aan
uitverdunning van gelimiteerde aantallen van de regulatorische eiwitten. Deze visie wordt
ondersteund door het feit dat meerdere kopijen van regulatorische eiwitten de uitverdunning
kunnen teniet doen. In A. nidulans kon de opbrengst van α-interferon onder controle van de
alcA promotor verhoogd worden door overproductie van de positief regulerende
transcriptiefactor ALCR dat de alcA promotor reguleert (Gwynne, 1992).
Naast bovenvermelde strategieën voor het verbeteren van de heterologe genexpressie in
filamenteuze schimmels moet ook rekening gehouden worden met fysiologische parameters
zoals bijvoorbeeld de rol van het fermentatiemedium en cultiveringscondities.
6.8. Toekomstperspectieven
Fungale expressiesystemen kunnen op verschillende niveau’s verbeterd worden. Volgens
Radzio & Kück (1997) moet bijzondere aandacht besteed worden aan volgende denkpistes:
• Een beter inzicht in de werking van fungale promotoren en de eiwitten die betrokken
zijn bij transcriptionele regulatie kan zorgen voor een verbeterde heterologe
genexpressie. De overproductie van positief regulerende transcriptiefactoren kan ook
resulteren in hogere transcriptieniveau’s.
• Het niveau van genexpressie kan afhankelijk zijn van de plaats van integratie in het
genoom. Bij planten en zoogdieren werd genexpressie waargenomen die min of meer
onafhankelijk is van de plaats van integratie wanneer het heterologe DNA geflankeerd
werd door DNA sequenties die interageren met de nucleaire matrix (Stief et al., 1989).
Op deze manier wordt de transcriptie van het heterologe DNA vergemakkelijkt en
wordt de invloed van de genomische omgeving op de expressie verminderd.
Gelijkaardige strategieën zouden ook bij filamenteuze schimmels kunnen toegepast
worden.
• Op post-translationeel niveau kan modificatie van de enzymen betrokken bij
eiwitopvouwing en glycosylatie leiden tot stammen die hogere hoeveelheden actieve
heterologe eiwitten produceren. De overproductie van chaperone-eiwitten kan
bijvoorbeeld resulteren in hogere hoeveelheden correct opgevouwde eiwitten.
Aangezien chaperonne-eiwitten ook vaak betrokken zijn bij de translocatie van
eiwitten naar het ER zou dit ook tot verbeterde eiwitsecretieniveau’s kunnen leiden.
De succesvolle toepassing en implementatie van zulke strategieën zullen een belangrijke rol
spelen in het gebruik van filamenteuze schimmels als gastheerorganismen voor de productie
van heterologe eiwitten.
MANU DE GROEVE
III. Materialen en Methoden - 36
III. MATERIALEN EN METHODEN
1. MICRO-ORGANISMEN
1.1. Fungale stammen
Myrothecium gramineum MUCL 39210 is afkomstig van de MUCL (agro)industriële
schimmel- en gistcollectie van de Belgian Co-ordinated Collection of Micro-organisms
(BCCM™/MUCL, Mycothèque de l'Université catholique de Louvain, Louvain-la-Neuve,
België).
Aspergillus nidulans FGSC A722 (pyrG89 pabaA1; fwA1 uaY9) is afkomstig van het Fungal
Genetics Stock Center (University of Kansas Medical Center, Kansas City, KS, USA). Deze
stam bevat de pyrG89 mutatie en is bijgevolg uracil en uridine auxotroof en resistent aan 5fluoro-orotinezuur (5-FOA).
De stockculturen worden bewaard in 70% glycerol in cryovials bij -20°C en -80 °C.
1.2. Bacteriële stammen
Voor het vermeerderen van plasmiden werd Escherichia coli DH5α-F’ gebruikt. Deze stam is
afkomstig uit de cultuurcollectie van het Laboratorium voor Industriële Microbiologie en
Biokatalyse (LIMAB, Gent). E. coli DH5α-F’ is geschikt voor transformatie en is
streptomycineresistent.
2. CULTUURMEDIA EN GROEICONDITIES
2.1. M. gramineum MUCL 39210 en A. nidulans FGSC A722
Om groei en sporulatie toe te laten van Myrothecium gramineum wordt deze gecultiveerd op
vast compleet groeimedium (Potato Dextrose Agar, zie Tabel 8) bij 25°C. Na 1-2 dagen zijn
de eerste kolonies zichtbaar en na 5-7 dagen is de schimmel over de volledige plaat
gesporuleerd. Sporen worden geoogst met steriele fysiologische oplossing (0.9% NaCl) voor
gebruik in verdere toepassingen of voor bewaring in cryovials.
Om mycelium te bekomen voor DNA isolatie wordt M. gramineum gecultiveerd in vloeibaar
compleet medium (Potato Dextrose Broth, zie Tabel 8) bij 25°C en 125-150 RPM.
III. Materialen en Methoden - 37
MANU DE GROEVE
Tabel 8. Samenstelling complete media voor wild type schimmelstammen
Concentratie (g/L)
Potato Dextrose Agar (PDA) Potato Dextrose broth (PD)
Component
(Oxoid/Biokar)
(Difco)
Aardappelzetmeel 4
4
Glucose
20
20
Agar
15
-
De standaard minimale media voor Myrothecium gramineum en A. nidulans FGSC A722 zijn
weergegeven in Tabel 9. A. nidulans FGSC A722 heeft zowel 10 mM uridine als uracil nodig
in het medium voor optimale groei.
Tabel 9. Samenstelling minimale media voor schimmelstammen
Concentratie (g/L)
AMM voor
Component
M. gramineum
Glucose
10
NaNO3 (Merck)
6
Uracil (10 mM, Sigma)
Uridine (10 mM, Acros)
1.52
KH2PO4
0.52
MgSO4.7H2O
KCl
0.52
Bacteriologische agar (1.5%, Oxoid)
15
Hutner’s sporenelementen
1 mL/L
AMM voor
A. nidulans FGSC A722
10
6
1.12
2.44
1.52
0.52
0.52
15
1 mL/L
De samenstelling van Hutner’s sporenelementen is weergegeven in Tabel 10.
Tabel 10. Hutner’s sporenelementen
Component
Concentratie (g/L)
22
ZnSO4.7H2O
H3BO3
11
MnCl2.4H2O
5
5
FeSO4.7H2O
CoCl2. 6H2O
1.6
1.6
CuSO4. 5H2O
1.1
(NH4)Mo7O24. 4H2O
EDTA
50
2.2. E. coli DH5α-F’
E. coli DH5α-F’ wordt gecultiveerd in vloeibaar Luria Broth (LB) medium bij 37 °C en 200
RPM. Indien nodig wordt ampicilline aan het medium toegevoegd. Na transformatie met
pGEM®-T (Promega) wordt E. coli DH5α-F’ uitgeplaat op vast LB medium met ampicilline
en X-gal. De samenstellingen van de media voor E. coli zijn weergegeven in Tabel 11.
III. Materialen en Methoden - 38
MANU DE GROEVE
Tabel 11. Samenstelling media voor E. coli
Concentratie (g/L)
Component
Luria Broth (LB)
Gistextract (Difco)
5
Bactotrypton (Difco)
10
NaCl
5
Ampicilline
X-gal
Agar (Oxoid)
12
LB + ampicilline
5
10
5
0.1
12
LB + ampicilline + X-gal
5
10
5
0.1
0.04
12
3. ISOLATIE VAN NUCLEÏNEZUREN
3.1. Isolatie van genomisch DNA
Voor de isolatie van genomisch DNA (tot 30 µg) uit de schimmelstammen werd gebruik
gemaakt van de Dneasy Plant Mini Kit (Qiagen). Indien grotere hoeveelheden DNA nodig
waren (tot 260 µg) werd gebruik gemaakt van de Dneasy Plant Maxi kit (Qiagen).
Sporen van de schimmelstam worden geënt in vloeibaar PD medium en geïncubeerd volgens
de groeicondities van de stam. Na 3-5 dagen is de schimmel voldoende gegroeid voor DNA
extractie. Als startmateriaal werd al dan niet gelyofiliseerd mycelium gebruikt.
Een cruciale stap bij de isolatie van genomisch DNA is het openbreken van de cellen. Dit
gebeurde hetzij met vloeibare stikstof en stamper in een proefbuis, hetzij door het malen van
gelyofiliseerd mycelium tot fijn poeder in een mortier. Vervolgens werd het protocol gevolgd
zoals beschreven in de kit.
3.2. Isolatie van totaal RNA
Voor de isolatie van totaal RNA uit Myrothecium gramineum werd gebruik gemaakt van de
RNeasy Plant Mini Kit (Qiagen). Hiermee kan tot 100 µg totaal RNA geïsoleerd worden uit
10-100 mg startmateriaal.
Sporen van de schimmel werden geënt in twee vloeibare 5 mL culturen (PD medium). Deze
werden gedurende twee dagen geïncubeerd bij 25°C en 125 RPM. Na de incubatie werd het
mycelium geoogst door te filteren over een 0.45 µm filter (Sartorius). Na wassen met
gedestilleerd water werd 50 mg mycelium (nat gewicht) overgebracht naar een epje. Door
middel van vloeibare stikstof en een pincet werd het mycelium in het epje fijngemalen tot
poeder waarna het protocol van de Rneasy Plant Mini Kit werd gevolgd. Er werd gebruik
gemaakt van het Rneasy Mini protocol voor de isolatie van totaal RNA uit plantencellen,
plantenweefsels en filamenteuze schimmels. Als lyse-buffer werd de RLC buffer gebruikt die
guanidine hydrochloride bevat. Deze wordt in de kit aangeraden voor filamenteuze
schimmels.
MANU DE GROEVE
III. Materialen en Methoden - 39
Omdat geen enkele RNA extractiemethode genomisch DNA contaminatie kan vermijden
werd het geïsoleerde RNA behandeld met DNAse. Hiervoor werden volgende reagentia
samengevoegd in een epje:
- 1 µL DNAse (1 unit/µL, Roche)
- 6 µL 10X DNAse buffer (100 mM Tris-Cl, pH 8.3; 500 mM KCl; 15 mM MgCl2)
- RNA staal
- DEPC-water (aanlengen tot 60 µL)
Dit reactiemengsel wordt gedurende 30 minuten geïncubeerd bij 37°C waarna het gezuiverd
wordt volgens het protocol van de BD GenomeWalker Universal Kit (zie verder).
3.3. Isolatie van plasmiden
3.3.1. Minipreps
De gewenste E. coli kolonie wordt opgegroeid in 5 mL vloeibaar LB medium met ampicilline
(37°C, 200 RPM). Wanneer de O.D.600 van de cultuur tussen 1.5 en 3.0 is wordt 1 tot 1.5 mL
van de cultuur gebruikt voor plasmiden isolatie. Voor de isolatie van plasmiden uit E. coli
werden volgende kits gebruikt: FastPlasmid Mini Kit (Eppendorf), NucleoSpin® Plasmid Kit
(BD Biosciences) en Nucleospin Plasmid Quick Pure (Macherey-Nagel).
3.3.2. Maxipreps
Voor het bekomen van grote hoeveelheden plasmiden wordt eerst 100 µL uit een cryovial
geënt in een 5 mL precultuur (LB + ampicilline). Na voldoende groei wordt de 5 mL geënt in
in 250 mL vloeibaar LB medium gesupplementeerd met ampicilline (37°C, 200 RPM).
Wanneer de O.D.600 ongeveer 1.5 is wordt de volledige cultuur gebruikt voor plasmiden
isolatie met de Hispeed Plasmid Maxi kit (Qiagen).
3.4. Bepalen nucleïnezuurconcentratie en zuiverheid
De concentratie en zuiverheid van het geïsoleerde DNA en RNA worden bepaald met een
spectrofotometer (Uvikon 922; Kontron Instruments) of via gelelektroforese (zie verder).
Om de nucleïnezuurconcentratie te bepalen met de spectrofotometer wordt de absorbantie bij
260 nm gemeten van een 200x of 1000x verdunning van het DNA of RNA staal. De stalen
wordt verdund met milli-Q water. Na correctie van de absorbantie met een blanco staal dat
enkel milli-Q water bevat wordt de DNA of RNA concentraties als volgt berekend:
DNA concentratie (ng/µL) = A260 * 50 * 200 (voor 200x verdunning)
RNA concentratie (ng/µL) = A260 * 40 * 200 (voor 200x verdunning)
MANU DE GROEVE
III. Materialen en Methoden - 40
De zuiverheid wordt bepaald door de verhouding van de absorbantie bij 260 nm tot de
absorbantie bij 280 nm. Deze waarde ligt voor een zuiver staal tussen 1.8 en 2.1 wanneer
isopropanol werd gebruikt in de opzuivering en rond 1.2 in de andere gevallen.
3.5. Concentreren van DNA
Aan het DNA staal worden 1/10 volume Na-acetaat (3M) en 2 volumes 100% ethanol
toegevoegd. Indien het DNA nog niet zichtbaar is, wordt het mengsel 10 tot 20 minuten bij 20 °C geplaatst. Vervolgens wordt er gedurende 20 minuten gecentrifugeerd bij 12.000 RPM.
Het supernatans wordt verwijderd en de pellet wordt gewassen met 500 µL 70% ethanol (die
5 à 10 minuten bij -20°C gestaan heeft). Er wordt opnieuw gecentrifugeerd gedurende 20
minuten bij 12.000 RPM. Na het verwijderen van het supernatans wordt de pellet gedroogd
aan de lucht. De pellet wordt heropgelost in milli-Q water waarna de concentratie en de
zuiverheid van het bekomen DNA bepaald worden.
3.6. Zuiveren van DNA
Voor het zuiveren van restrictie- en PCR-reactiemengsels werd gebruik gemaakt van de
Bioline DNAce Quick-Clean kit (Bioline, Gentaur).
4. GELELEKTROFORESE
4.1. Analytische gelelektroforese
Voor het scheiden en visualiseren van DNA fragmenten worden de DNA stalen geladen op
een agarosegel. Afhankelijk van de grootte van de te scheiden DNA fragmenten wordt een
hoger of lager percentage agarose gebruikt. Standaard worden 1.4% agarosegels gebruikt.
Deze worden aangemaakt door 5.6 g Agarose MP (Roche) op te lossen in 400 mL 1x TAEbuffer (40 mM Tris; 20 mM acetaat; 1 mM EDTA; Fermentas).
DNA stalen worden op gel geladen in volumes van 10 µL, bestaande uit 5 µL DNA staal, 3
µL milli-Q water en 2 µL ladingsbuffer (met glycerol om het DNA te verzwaren, TAE-buffer
om het DNA te stabiliseren en kleurstof om het zichtbaar te maken).
Standaard wordt de SmartLadder (200-10 000 bp; Eurogentec) als merker gebruikt. Met deze
merker kan zowel de grootte als de concentratie van de bekomen fragmenten bepaald worden.
Voor groottebepaling van kleine fragmenten (100-1000 bp) wordt de SmartLadderSF
(Eurogentec) gebruikt. Van de merkers wordt 5 µL op gel geladen. De grootte van de
fragmenten van beide ladders is weergegeven in Figuur 8.
MANU DE GROEVE
SmartLadder
III. Materialen en Methoden - 41
SmartLadderSF
Figuur 8. Grootte van de fragmenten van de SmartLadder en SmartLadderSF (Eurogentec)
De elektroforese wordt uitgevoerd in 1x TAE buffer bij 110 V (Bio-Rad Model 1000/500
Power supply) voor kleine gels of bij 140 V (Electroforesis Power supply EPS 500/400,
Pharmacia) voor grote gels. Na elektroforese wordt de gel gedurende 30 minuten in een
verdunde oplossing van ethidiumbromide (EtBr) gelegd. Na spoelen in gedestilleerd water
wordt de gel bekeken onder UV licht en indien gewenst gefotografeerd.
4.2. Extractie van DNA fragmenten uit agarosegel
Voor de extractie van DNA fragmenten uit gel wordt een preparatieve gelelektroforese
uitgevoerd. Daarbij worden drie lanen geladen per DNA staal. In de middelste laan wordt 40
µL staal aangebracht en in de twee buitenste 5 µL staal. De gebruikte merker is de
Smartladder (Eurogentec).
De gelelektroforese wordt uitgevoerd in 1x TAE buffer. Na gelelektroforese (140 V,
Electroforesis Power supply EPS 500/400, Pharmacia) wordt als volgt te werk gegaan:
•
•
•
•
•
het middelste van de drie laantjes wordt verwijderd en onderaan van een merkteken
voorzien zodat de stukken later weer op de juiste wijze in het gel kunnen gefit worden
de rest van de gel wordt gedurende 30 minuten in ethidiumbromide gelegd en daarna
bekeken onder UV belicht
de benodigde fragmenten worden onder UV-licht gemerkt door ze gedeeltelijk weg te
snijden uit de buitenste banen
het UV-licht wordt gedoofd en de middelste stroken gel worden teruggeplaatst
op de plaats van de insnijdingen worden de bandjes uit de middenstrook verwijderd en in
epjes overgebracht
Deze methode van laden en isoleren wordt toegepast om mutaties in de verder te gebruiken
fragmenten te vermijden. Zij komen zo niet in aanraking met mutagene stoffen, zoals
ethidiumbromide en UV-bestraling.
MANU DE GROEVE
III. Materialen en Methoden - 42
Het DNA wordt geëxtraheerd uit de weggesneden bandjes met behulp van de QIAEX ΙΙ Gel
Extraction Kit (Qiagen). Met deze kit kunnen 40 bp tot 50 kb DNA-fragmenten geëxtraheerd
worden uit 0.3-2% TAE-gels.
5. POLYMERASE CHAIN REACTION (PCR)
De PCR reactie wordt gebruikt voor het amplificeren van specifieke stukken DNA. Voor de
PCR reactie wordt gebruik gemaakt van de Progene thermocycler (Techne). Primers worden
aangekocht bij Sigma-Genosys en verdund tot een 10 pmol/µL werkoplossing. Alle
uitgevoerde PCR’s zijn hotstart PCR’s, wat concreet wil zeggen dat de PCR tubes
rechtstreeks van ijs naar de denaturatietemperatuur in het PCR-toestel worden gebracht.
5.1. Standaard Taq PCR
Standaard wordt volgend reactiemengsel samengesteld in een PCR tube (0.2 mL thin walled
PCR tubes; ABgene):
- 10-250 ng genomisch DNA; 0.1-10 ng plasmide DNA
- 5 µL Taq-polymerase buffer (Roche)
- 1 µL nucleotiden (10 mM, Sigma)
- 2 µL forward primer (10 pmol/µL, Sigma-Genosys)
- 2 µL reverse primer (10 pmol/µL, Sigma-Genosys)
- 0.2 µL Taq-polymerase (5 U/µL, Roche)
Het reactiemengsel wordt aangelengd tot 50 µL met milli-Q water.
Het cycluspatroon van de PCR-reactie wordt weergegeven in Tabel 12.
Tabel 12. Standaard PCR protocol
Cycli
Tijd
Temperatuur (°C)
1x
4 min.
94
25x
30 s
94
30 s
55
1 min. 30 s
72
1x
7 min.
72
Opmerking
denaturatie
denaturatie
annealing
elongatie
elongatie
Het resultaat van de PCR wordt gecontroleerd via agarose-gelelektroforese.
III. Materialen en Methoden - 43
MANU DE GROEVE
5.2. High Fidelity PCR Master kit (Roche)
Om te voorkomen dat de geamplificeerde DNA fragmenten fouten bevatten in hun sequentie
wordt gebruik gemaakt van de High Fidelity PCR Master kit (Roche). Het is immers bekend
dat bij de standaard PCR met Taq polymerase fouten kunnen ingebouwd worden tijdens de
elongatie. Taq polymerase bevat geen proofreading activiteit. De High Fidelity PCR Master
kit daarentegen bevat een enzymmix van thermostabiel Taq en Tgo DNA polymerase. Door
de 3’-5’ exonuclease activiteit van Tgo DNA polymerase wordt de betrouwbaarheid van de
PCR verhoogd ten opzichte van de standaard PCR.
Voor de PCR reactie zijn twee master mixen nodig. De samenstelling van de eerste master
mix (25 µL) is weergegeven in Tabel 13. Deze bevat het template DNA, de primers en steriel
water. De tweede master mix bestaat uit High Fidelity PCR Master mix (25 µL). Deze bevat
de polymerasen en een dubbel geconcentreerde reactiebuffer bestaande uit 3 mM MgCl2 en
dATP, dGTP, dCTP, dTTP elk in een concentratie van 0.4 mM.
Tabel 13. Samenstelling van Master Mix 1
Component
Volume (µL)
Eindconcentratie
Forward primer
1.5
300 nM
Reverse primer
1.5
300 nM
Template DNA
x
10-500 ng
Steriel water
aanlengen tot 25 µL
Beide master mixen worden gemengd in een PCR tube en op ijs bewaard. Het totale volume
bedraagt 50 µL. Er wordt een hotstart PCR uitgevoerd waarvan het standaard protocol
weergegeven is in Tabel 14.
Tabel 14. High Fidelity PCR protocol
Cycli
Tijd
Temperatuur (°C)
1x
2 min.
94
10x
10 s
94
30 s
50
2 min.
72
15x
15 s
94
30 s
50
2 min. +5 s inc.
72
1x
7 min.
72
5.3. Reamplificatie PCR
Reamplificatie PCR werd gebruikt voor het reamplificeren van schijnbaar mislukte PCR
mengsels. Wanneer een PCR uitgevoerd wordt met dit PCR-mengsel als template worden
dikwijls geamplificeerde fragmenten bekomen. Het standaard protocol voor reamplificatie is
weergegeven in Tabel 15.
III. Materialen en Methoden - 44
MANU DE GROEVE
Tabel 15. Reamplificatie PCR protocol
Samenstelling PCR reactie-mix
Component
Eindconcentratie
Taq DNA polymerase (5U/µL)
1.25 U
10X Taq pol. Buffer (+Mg)
1X
10mM dNTP mix
0.2 mM
Forward primer (10 pmol/µL)
0.3 µM
Reverse primer (10 pmol/µL)
0.3 µM
DNA template:
- PCR-mengsel
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
30x
1x
Tijd
1 min.
10 s
30 s
4 min.
7 min.
Volume nodig voor 50 µL
0.25 µL
5 µL
1 µL
1.5 µL
1.5 µL
0.5 µL
40.25 µL
50 µL
Temperatuur (°C)
92
92
afhankelijk van primers
72
72
5.4. Synthese van cDNA
Voor de synthese van cDNA werd gebruik gemaakt van de RevertAid™ H Minus First Strand
cDNA synthesis Kit (Fermentas). De kit bevat het RevertAid ™ H Minus M-MuLV reverse
transcriptase en werd gebruikt voor het maken van ‘eerste-streng’ cDNA uit RNA templates.
Er werd gebruik gemaakt van de oligo(dT)18 primer waardoor enkel cDNA wordt gemaakt
van mRNA templates die een 3’-poly(A) staart bevatten. De synthese van cDNA, geschikt
voor PCR amplificatie, werd uitgevoerd volgens het protocol van de fabrikant. Het bekomen
‘eerste streng’ cDNA kan rechtstreeks als template gebruikt worden voor PCR.
5.5. Kolonie-PCR
Er werd een kolonie-PCR protocol ontwikkeld om op een snelle manier getransformeerde
kolonies te kunnen controleren. In het ontwikkelde protocol kan rechtstreeks PCR uitgevoerd
worden op mycelium van een groeiende kolonie op een petriplaat. Het mycelium wordt met
een tandenstoker naar het PCR-reactiemengsel gebracht. Hierbij is het belangrijk zo weinig
mogelijk medium mee te nemen. De samenstelling van het PCR-reactiemengsel is
weergegeven in Tabel 16.
III. Materialen en Methoden - 45
MANU DE GROEVE
Tabel 16. Kolonie-PCR protocol
Samenstelling PCR reactie-mix
Component
Taq
DNA
polymerase
(5U/µL)
10X Taq pol. buffer (+Mg)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
Mycelium
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
10x
15x
1x
Eindconcentratie
2U
Volume nodig voor 50µL
0.4 µL
1X
0.2 mM
0.4 µM
0.4 µM
5 µL
1 µL
2 µL
2 µL
-
39.6 µL
50 µL
Tijd
2 min.
10 s
30 s
2 min.
15 s
30 s
2 min. +5 s inc.
7 min.
Temperatuur (°C)
94
94
45
72
94
45
72
72
5.6. Ligatie van PCR fragmenten in pGEM®-T Vector (Promega)
Voor het kloneren van PCR fragmenten worden pGEM®-T plasmiden (Promega) gebruikt.
Deze plasmiden zijn uitermate geschikt om PCR producten rechtsreeks in te ligeren omwille
van de enkelvoudige 3’-T (thymidine) overhangen. De PCR producten bevatten immers een
complementaire 3’-A (adenine) die door het Taq polymerase tijdens PCR werd ingebouwd.
pGEM®-T vectoren zijn high copy plasmiden die een ampicilline resistentie gen en de T7 en
SP6 RNA polymerase promotors bevatten die de multikloneringsplaats flankeren (zie Figuur
9). De multikloneringsplaats ligt in de coderende regio van het α-peptide (lacZ gen) van het
β-galactosidase enzym. Dit enzym hydrolyseert het chromogene X-gal (5-bromo-4-chloro-3indolyl-β-D-galactoside) tot galactose en 5-bromo-4-chloro-3-indolyl. Door de splitsing van
X-gal wordt een water-onoplosbaar blauw dichloro-dibromo-indigo precipitaat gevormd.
Wanneer het lacZ gen onderbroken wordt door het PCR fragment gaat de activiteit van het
enzym verloren. Na transformatie van E. coli DH5α-F’ kunnen de geschikte klonen
geselecteerd worden via blauw-/witkleuring op LB medium met X-gal en ampicilline (zie
Tabel 11). Alle kolonies die op de indicator plaat groeien hebben een plasmide (al dan niet
met insert) opgenomen, aangezien de gebruikte E. coli stam van nature niet resistent is aan
ampicilline. Kolonies die het plasmide zonder insert opgenomen hebben, kleuren blauw
omdat het LacZ gen niet onderbroken is en het β-galactosidase enzym dus nog functioneel is.
III. Materialen en Methoden - 46
MANU DE GROEVE
De witte kolonies daarentegen bevatten het plasmide met insert en zullen gebruikt worden om
de gewenste plasmiden uit op te zuiveren.
Figuur 9. De pGEM®-T vector (Promega)
De ligatie van PCR producten in de plasmiden gebeurt volgens de instructies van de fabrikant.
De pGEM®-T plasmiden en ligatiebuffer worden goed gevortexed, waarna in een epje
volgende componenten samengevoegd worden: 5 µL 2X Rapid Ligation Buffer, 1 µL
pGEM®-T vector (50 ng), 3 µL PCR product en 1 µL T4 DNA ligase (3 Weiss units/µL). De
reacties worden gedurende 1 uur geïncubeerd bij kamertemperatuur. Indien een maximaal
aantal transformanten nodig is worden de reacties overnacht bij 4°C geïncubeerd. Het ligase
wordt geïnactiveerd door incubatie bij 65°C gedurende 10 minuten.
6. RESTRICTIEDIGEST
Een restrictiedigest wordt uitgevoerd om grotere DNA-strengen (of plasmiden) te verknippen
met behulp van restrictie-enzymen. Een restrictiedigest kan analytisch zijn. Zij dient dan om
lengte van de gevormde fragmenten te controleren. Restrictiedigesten kunnen ook preparatief
zijn. Zij worden dan toegepast op een grotere hoeveelheid DNA en de fragmenten kunnen na
scheiding op gel opgezuiverd worden en verder gebruikt worden.
Wanneer men restricties in vitro wil uitvoeren moet men er steeds voor zorgen dat de reactie
in de juiste buffer en bij de juiste temperatuur wordt uitgevoerd. Beiden zijn enzymspecifiek.
Alle type II restrictie-enzymen vereisen Mg2+ als cofactor.
MANU DE GROEVE
III. Materialen en Methoden - 47
Analytische restrictie
Bij een analytische restrictie bestaat het reactiemengsel uit 5 µL DNA staal, 0.2 µL restrictieenzym (2 units) en 1 µL restrictiebuffer, aangelengd tot 10 µL met milli-Q water.
Preparatieve restrictie
Bij een preparatieve restrictie bestaat het reactiemengsel uit 40 µL DNA staal, 2 µL restrictieenzym (20 units) en 5 µL restrictiebuffer, aangelengd tot 50 µL met milli-Q water.
De mengsels worden geïncubeerd bij 37°C gedurende minimum 2.5 uur of overnacht.
7. LIGATIE
Om verschillende lineaire DNA fragmenten te ligeren tot circulair DNA werd volgende
procedure gevolgd:
• bereid in een epje een oplossing van lineair DNA in milli-Q of TE-buffer
• aan dit epje voeg je het volgende toe:
- 10X ligatiebuffer
- 50% polyethyleenglycol (PEG) 4000 oplossing
- milli-Q water
- T4 DNA-ligase
• dit epje vortexen en centrifugeren gedurende 3-5 seconden
• het mengsel incuberen gedurende 1 uur of overnacht bij 22°C
• ligatiereactie stoppen door het mengsel gedurende 10 minuten te incuberen bij 65°C
8. SEQUENERING VAN DNA FRAGMENTEN
De te sequeneren DNA fragmenten worden geligeerd in pGEM®-T en de bekomen plasmiden
worden getransformeerd in E. coli. Na isolatie van de plasmiden wordt via PCR gecontroleerd
of de plasmiden wel degelijk een insert bevatten. Als primers worden de T7 en SP6 primers
gebruikt. Deze amplificeren het insert indien dit aanwezig is. Het amplificatieproduct zou 149
bp groter moeten zijn dan het geïsoleerde bandje dat in het plasmide geligeerd werd. De
details van de PCR op pGEM®-T zijn weergegeven in Tabel 17.
Op basis van het resultaat van de controle PCR en concentratiebepaling via gelelektroforese
wordt beslist welke plasmiden zullen worden gesequeneerd. De sequenering gebeurt door het
Vlaams Interuniversitair Instituut voor Biotechnologie (VIB, Genetic Service Facility,
Antwerpen, België). Zij maken gebruik van de Applied Biosystems 3730 DNA Analyzer en
beschikken over de T7 en SP6 primers waarmee het insert in het pGEM®-T plasmide kan
gesequeneerd worden.
III. Materialen en Methoden - 48
MANU DE GROEVE
Tabel 17. Details van de controle PCR op geïsoleerde plasmiden
Primers
Tm (°C)
Sequentie (5’→ 3’)
TAATACGACTCACTATAGGG
T7
50.8
TATTTAGGTGACACTATAG
SP6
43.4
Lengte
20
19
Samenstelling PCR reactie-mix
Component
Taq DNA polymerase (5U/µL)
10X Taq pol. Buffer (+Mg)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
DNA template:
- plasmide DNA
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
25x
1x
Uiteindelijke
concentratie
1U
1X
0.2 mM
0.4 µM
0.4 µM
Volume nodig voor 50µL
0.2 µL
5 µL
1 µL
2 µL
2 µL
-
0.1 µL
39.7 µL
50 µL
Tijd
2 min.
30 s
30 s
2.5 min. + 5 s inc.
7 min.
Temperatuur (°C)
94
94
50
72
72
9. TRANSFORMATIE VAN E. COLI DH5α-F’
9.1. Aanmaken van competente cellen van E. coli DH5α-F’
Competente cellen van E. coli DH5α-F’ werden aangemaakt volgens de aangepaste Hanahan
methode (Hanahan et al., 1991).
Benodigde oplossingen
Stockoplossingen:
- 100 mM morpholinoëthanesulfonzuur (MES) pH 5.8 (pH met KOH)
- 500 mM RbCl
- 1 M CaCl2
- 1 M MnCl2
- 100 mM PIPES pH 6.8 (pH aanpassen met KOH)
- 15% glycerol
Deze stockoplossingen werden bewaard bij 4°C.
MANU DE GROEVE
III. Materialen en Methoden - 49
Oplossing I
Deze oplossing bevat:
10 mM MES (= 25ml stockoplossing 100 mM/250 mL)
100 mM RbCl (= 50ml stockoplossing 500 mM/250 mL)
10 mM CaCl2 (= 2.5ml stockoplossing 1 M/250 mL)
50 mM MnCl2 (= 12.5ml stockoplossing 1 M/250 mL)
(160 mL ultrapuur water)
Oplossing II
Deze oplossing bevat:
10 mM PIPES (= 25 mL stockoplossing 100mM/250 mL)
10 mM RbCl (= 5 mL stockoplossing 500mM/250 mL)
75 mM CaCl2 (= 18.75 mL stockoplossing 1M/250 mL)
15% glycerol (= 37.5 mL/250 mL)
(163.75 mL ultrapuur water)
Zowel oplossing I als oplossing II werden gesteriliseerd via filtratie.
Cellen competent maken
•
•
•
•
•
•
•
•
•
•
•
•
•
Maak een precultuur opgezet van de te transformeren stam. Groei de cellen in 5 mL Luria
Broth en ïncubeer overnacht bij 37°C aan 200 RPM.
Ent 2 x 100 mL Luria Broth met 2 mL van de precultuur.
Men laat de cellen groeien bij 37°C (200 tpm) tot de OD600 tussen 0.5 en 0.75 ligt.
Men giet ze daarna over in 8 voorgekoelde SS34-buizen (± 25 mL / buis; equilibreren!!).
Laat 5 tot 10 min. op ijs staan.
Centrifugeer: SS34-rotor, 4°C, 5030 tpm, 5 minuten.
Zet de cellen terug op ijs en giet het supernatans af.
Los de pellets op in ijskoude oplossing I (10 mL per buis, tot alles opgelost is: er mogen
geen celaggregaten meer aanwezig zijn; cellen losmaken met pipet is beter dan schudden)
Giet de inhoud van de buizen 2 per 2 bij elkaar en laat 5 tot 20 min. op ijs staan.
Centrifugeer: SS34, 4°C, 5030 tpm, 5 minuten.
Los de pellets op in ijskoude oplossing II (1 mL per buis) en giet deze 2 per 2 bij elkaar (2
x 2 mL).
Laat 5 tot 10 min. op ijs staan.
Verdeel per 100 µL in steriele buizen (ca. 44) en zet direct op CO2-ijs. Stockeer bij –
80°C .
MANU DE GROEVE
III. Materialen en Methoden - 50
9.2. Transformatieprocedure voor E. coli DH5α-F’
Competente E. coli DH5α-F’ cellen worden bewaard bij –80°C. 100 µL van deze competente
cellen wordt gedurende 15 tot 20 minuten op ijs gezet, waarna 2 µL ligatiemengsel wordt
toegevoegd. De cellen worden dan opnieuw 15 minuten op ijs gezet. Vervolgens ondergaan
de cellen een hitteschok bij 37°C gedurende 5 minuten, waarna ze opnieuw 10 minuten op ijs
gezet worden. Hierna wordt 900 µL Luria Broth aan het mengsel toegevoegd en de cellen
worden 75 minuten gegroeid bij 37°C. Van deze celsuspensie wordt vervolgens 200-300 µL
uitgeplaat op vast selectief medium (LB met ampicilline en X-gal) en overnacht geïncubeerd
bij 37°C.
10. KLONERING VAN HET OMPD GEN VAN
MYROTHECIUM GRAMINEUM MUCL 39210
10.1. Gedegenereerde PCR
Gedegenereerde PCR is zeer gelijkaardig aan de gewone standaard PCR, met die uitzondering
dat er gedegenereerde primers gebruikt worden. Gedegeneerde primers bestaan uit een mix
van enkele tot duizenden gelijkaardige primers. Deze primers worden ontworpen op basis van
geconserveerde sequenties in de eiwit- en nucleotidensequentie van een gen. In dit onderzoek
zal via gedegenereerde PCR getracht worden een deel van het homologe OMPD gen van
Myrothecium gramineum te amplificeren.
10.1.1. Ontwikkeling van gedegenereerde primers voor OMPD
Om gedegenereerde primers te kunnen ontwikkelen moeten eerst de geconserveerde
sequenties in de eiwitsequentie van OMPD gelokaliseerd worden. Hiervoor werden
alignments uitgevoerd met de OMPD eiwitsequenties van Ascomycete fungi waartoe ook
Myrothecium gramineum behoort. De alignments werden uitgevoerd met DbClustal W.
De bekomen geconserveerde aminozuursequenties werden vergeleken met deze beschreven in
de literatuur en er werd een keuze gemaakt op welke geconserveerde sequenties primers
werden ontwikkeld. Op basis van de DNA sequentie van deze geconserveerde
aminozuursequenties werd een consensus DNA sequentie opgesteld. Een deel van de
consensussequentie werd gebruikt voor het ontwerp van een gedegenereerde primer die
voldoet aan de algemene eisen van een primer (%GC, 20-21 bp). De ontworpen primers
werden besteld bij Sigma-Genosys.
10.1.2. Optimalisatie van PCR-protocol voor gedegenereerde PCR
Initieel werd de High Fidelity PCR Master kit (Roche) gebruikt. Yamazaki et al. (1999)
gebruikten ook deze kit voor gedegenereerde PCR op het OMPD gen van Rhizomucor
MANU DE GROEVE
III. Materialen en Methoden - 51
pusillus. Omdat hiermee echter geen positieve resultaten werden bekomen werd
overgeschakeld op het standaard Taq polymerase PCR protocol. Dit protocol werd
geoptimaliseerd voor gedegenereerde PCR. De optimalisatie wordt besproken onder
Resultaten en Bespreking (IV.1.2.1, p.68).
10.2. Genomewalking: BD GenomeWalker™ Universal Kit (BD
Biosciences)
10.2.1. Constructie van genomische DNA banken van Myrothecium gramineum
10.2.1.1. Controle van de kwaliteit van het genomisch DNA
Voor de constructie van de BD GenomeWalker™ DNA banken is zeer zuiver genomisch
DNA nodig van hoog moleculair gewicht. Het genomisch DNA werd geïsoleerd uit mycelium
van Myrothecium gramineum MUCL 39210 met de Dneasy Plant Maxi kit (Qiagen), zoals
eerder beschreven. Vervolgens werd de grootte en zuiverheid van het genomisch DNA
gecontroleerd zoals beschreven in de kit. De grootte werd gecontroleerd door 1 µL genomisch
DNA staal te laden op een 0.5% agarosegel met als lengtemerker de SmartLadder
(Eurogentec). Het genomisch DNA moet groter zijn dan 50 kb met zo weinig mogelijk smeer.
Om de zuiverheid te controleren wordt een controlerestrictie uitgevoerd op het genomisch
DNA met DraI als restrictie-enzym. Van het restrictiemengsel wordt 5 µL geladen op 0.5%
agarosegel. Indien een smeer te zien is, kan het DNA geknipt worden met de restrictieenzymen en is het bijgevolg zuiver genoeg voor gebruik in de GenomeWalker kit.
10.2.1.2. Restrictiedigest van genomisch DNA
Voor de constructie van de banken dient het genomisch DNA per bank geknipt te worden met
een specifiek restrictie-enzym. De restrictie werd uitgevoerd zoals beschreven in de kit. Per
bank wordt 2.5 µg genomisch DNA gebruikt in een restrictiemengsel van 100 µL. Er worden
vier verschillende restricties uitgevoerd resulterend in vier verschillende banken: DraI,
EcoRV, PvuII en StuI. Na incubatie overnacht wordt gecontroleerd of de restrictie compleet is
door 5 µL te laden op 0.5% agarosegel.
10.2.1.3. Opzuiveren van DNA uit het restrictiemengsel
Het geknipte genomische DNA werd gezuiverd uit het restrictiemengsel zoals beschreven in
de kit. Na zuivering werd via gelelektroforese gecontroleerd of er niet teveel DNA verloren
was gegaan. Er werd 1 µL geladen op een 0.5% agarosegel. Indien de helderheid van de
smeer na zuivering tweemaal minder is dan vóór de zuivering dient het DNA geconcentreerd
te worden.
MANU DE GROEVE
III. Materialen en Methoden - 52
10.2.1.4. Ligatie van de BD GenomeWalker™ Adaptors aan het genomisch DNA
De ligatiestap werd uitgevoerd zoals beschreven in de kit. De reacties werden uitgevoerd in
een PCR tube in een PCR toestel (Progene thermocycler, Techne) omdat deze een zeer
constante temperatuur aanhoudt. Na deze ligatiestap is de constructie van de banken voltooid.
10.2.2. GenomeWalking op het OMPD gen van Myrothecium gramineum
10.2.2.1. Ontwerpen van genspecifieke primers op OMPD
Eenmaal de sequentie van een deel van het OMPD gen van Myrothecium gramineum gekend
is kan dit gebruikt worden om genspecifieke primers te ontwerpen. De genspecifieke primers
werden ontworpen met het softwareprogramma Clone Manager Professional Suite 6.0
(Scientific & Educational Software) en voldoen zo veel mogelijk aan de eisen beschreven in
de kit.
In totaal werden vier verschillende primers ontwikkeld voor de genomewalking op het OMPD
gen (Tabel 18): twee voor de upstream (5’) kant van het gekende stuk en twee voor de
downstream (3’) kant. De twee primers voor elke kant bestaan uit een primer voor primary
PCR en een geneste primer voor secondary PCR.
Tabel 18. Benaming genspecifieke primers voor OMPD
Upstream (5’) kant
Downstream (3’) kant
Primary PCR
1 OMPD
3 1 OMPD
Secondary (nested) PCR
nested 1 OMPD
3 1 nested OMPD
Met deze primers kan het volledige OMPD gen gekloneerd worden via PCR-gebaseerde
genomewalking.
10.2.2.2. Procedure voor PCR gebaseerde DNA walking met de BD GenomeWalker banken
In de kit wordt aangeraden om de BD GenomeWalker PCR reacties uit te voeren met een 50X
polymerase mix geschikt voor long-distance PCR (LD PCR). Bij LD PCR wordt een
combinatie van twee thermostabiele DNA polymerasen gebruikt om de reikwijdte en
nauwkeurigheid van de PCR amplificatie te verbeteren. De elongatie zelf wordt uitgevoerd
door een eerste polymerase, terwijl een tweede polymerase zorgt voor de 3’->5’ exonuclease
activiteit die verkeerd ingebouwde nucleotiden corrigeert. Het gebruik van LD PCR in het BD
GenomeWalker protocol vergroot het bereik van mogelijke PCR producten tot 6 kb. De reden
voor deze bovenlimiet is niet duidelijk, maar is waarschijnlijk te wijten aan het verlies van het
suppressie PCR effect.
In de BD GenomeWalker Kit werd de samenstelling van het reactiemengsel en de PCR cycli
geoptimaliseerd voor gebruik met de BD Advantage™ 2 Polymerase Mix in een PE
Biosystems DNA Thermal Cycler 480. Hierbij worden geen drieledige cycli gebruikt (bv.
III. Materialen en Methoden - 53
MANU DE GROEVE
denaturatie, annealing, elongatie), maar wel tweeledige cycli (denaturatie, annealing +
elongatie).
Hier werd echter het Expand Long Template PCR System (Roche) gebruikt en als PCR
toestel de Progene thermocycler (Techne). Voor de samenstelling van het reactiemengsel en
de PCR cycli werd dan ook zoveel mogelijk het protocol gevolgd zoals beschreven in de kit
van Roche. Hoewel dit afgeraden wordt in de GenomeWalker kit, werden toch drieledige
PCR-cycli gebruikt. Het PCR protocol werd geoptimaliseerd voor gebruik met de
GenomeWalker Kit.
Primary PCR
Het geoptimaliseerde protocol voor de primary PCR is weergegeven in Tabel 19. Per PCR
reactie wordt gebruik gemaakt van de Adaptor primer 1 (AP1) en één genspecifieke primer.
Tabel 19. Primary PCR: Genome Walking naar 5’ OMPD en 3’ OMPD
Primers
Sequentie (5’→ 3’)
CGATAGGGAAACGTCTCGTCGGTAATG
1 OMPD
CCCTGCTTCTGCTTGCTGAGATGAC
3 1 OMPD
GTAATACGACTCACTATAGGGC
AP1
Samenstelling PCR reactie-mix
Component
LD polymerase mix (5U/µL)
10X PCR Buffer (met 1.75 mM MgCl2)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
DNA template:
- BD GenomeWalker bank
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
5x
25x
10x
1x
Tm (°C)
72.8
72.8
59
Lengte
27
25
22
Eindconcentratie
3.75 U
1X
0.35 mM
0.3 µM
0.3 µM
Volume nodig voor 50µL
0.75 µL
5 µL
1.75 µL
1.5 µL
1.5 µL
-
2 µL
37.5 µL
50 µL
Tijd
1 min.
10 s
30 s
4 min.
10 s
30 s
4 min.
10 s
30 s
4 min.
7 min.
Temperatuur (°C)
92
92
70
68
92
65
68
92
60
68
68
III. Materialen en Methoden - 54
MANU DE GROEVE
Secondary PCR
Het geoptimaliseerde protocol voor de secondary (nested) PCR is weergegeven in Tabel 20.
Per PCR reactie wordt gebruik gemaakt van de geneste Adaptor primer 2 (AP2) en één
genspecifieke geneste primer. Het primary PCR mengsel dat als template dient voor de
geneste PCR werd niet verdund.
Tabel 20. Secondary PCR: Genome Walking naar 5’ OMPD en 3’ OMPD
Primers
Sequentie (5’→ 3’)
GGTACTGTTTCTGGGCTGTTGAGCCAAT
nested 1 OMPD
CTGGTATCAACAGCAGCCAATCTGAC
3 1 nested OMPD
ACTATAGGGCACGCGTGGT
AP2
Samenstelling PCR reactie-mix
Component
LD polymerase mix (5U/µL)
10X PCR Buffer (met 1.75 mM MgCl2)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
DNA template:
- primary PCR-mengsel
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
5x
20x
1x
Tm (°C)
73.4
70.3
71
Lengte
28
26
19
Eindconcentratie
3.75 U
1X
0.35 mM
0.3 µM
0.3 µM
Volume nodig voor 50 µL
0.75 µL
5 µL
1.75 µL
1.5 µL
1.5 µL
-
1 µL
38.5 µL
50 µL
Tijd
1 min.
10 s
30 s
4 min.
10 s
4 min.
7 min.
Temperatuur (°C)
92
92
70
68
92
68
68
10.3. Klonering van het volledige OMPD gen
Om het volledige OMPD gen van Myrothecium gramineum MUCL 39210 te kloneren werd
dit eerst geamplificeerd via PCR. Op basis van de gesequeneerde DNA fragmenten die
bekomen werden met de BD GenomeWalker kit werden twee OMPD-genspecifieke primers
ontworpen. Deze amplificeren een fragment van 2050 bp dat een groot deel van de 5’
onvertaalde regio (5’ UTR) , de volledige coderende sequentie (CDS) en de 3’ onvertaalde
regio (3’ UTR) van het OMPD gen bevat. De gebruikte primers zijn weergegeven in Tabel
21. Er werd gebruik gemaakt van het standaard protocol voor de High Fidelity PCR Master
kit (Roche).
Het resultaat van de PCR reactie werd op agarosegel bekeken en er werd een preparatieve
agarose gelelektroforese uitgevoerd om het bekomen PCR fragment op te zuiveren. Dit
fragment werd vervolgens geligeerd in pGEM®-T en het transformatiemengsel werd
getransformeerd in E. coli DH5α-F’. De bekomen kolonies werden gecontroleerd en de
III. Materialen en Methoden - 55
MANU DE GROEVE
plasmiden van de positieve kolonies werden gesequeneerd zoals vroeger beschreven. Het
pGEM®-T plasmide met het 2.05 kb fragment van het OMPD gen van M. gramineum wordt
pOV genoemd.
Tabel 21. Gebruikte primers voor de amplificatie van het volledige OMPD gen van M. gramineum
Primers
Tm (°C)
Lengte (nt)
Sequentie (5’→ 3’)
OMPD volledig
5 O volledig
3 O transcriptiestop
CAGACCGCATGCATTCGTATGCC
ACCAAGCACCAACAC
74.1
52.5
23
15
11. TRANSFORMATIE VAN A. NIDULANS FGSC A722
Voor de protoplast-transformatie van A. nidulans FGSC A722 werd een aangepast protocol
volgens Punt & Van den hondel (1992) gebruikt (Jonniaux et al., 2004).
11.1. Oplossingen gebruikt voor de transformatie
Oplossing van CaCl2:
- 0.27 M CaCl2.2H2O
- 0.6 M NaCl
Oplossing STC:
- 1.2 M D-sorbitol
- 10 mM Tris pH 7.5
- 50 mM CaCl2.2H2O
- 35 mM NaCl
Oplossing van PEG (polyethyleenglycol):
- 60% PEG 4000
- 10 mM Tris pH 7.5
- 50 mM CaCl2.2H2O
Deze oplossingen werden allen geautoclaveerd bij 1.1 atm, 121°C gedurende 21 minuten.
11.2. Media
11.2.1. Niet-selectieve media
De vaste niet-selectieve media worden gebruikt voor het oogsten van sporen en voor het
bepalen van de leefbaarheid van de protoplasten. De media zijn gebaseerd op het PDAmedium en zijn al dan niet gestabiliseerd met 1.2 M sorbitol. De samenstelling van de vaste
niet-selectieve media is weergegeven in Tabel 22.
III. Materialen en Methoden - 56
MANU DE GROEVE
Tabel 22. Samenstelling vaste niet-selectieve media
Concentratie (g/L)
Component
PDA
PDAS
Aardappelzetmeel (Oxoid) 4
4
Glucose (Oxoid)
20
20
Sorbitol (1.2 M)
218
Agar (Oxoid)
15
15
Als vast niet-selectief medium werk ook ‘OS’ gebruikt dat dezelfde samenstelling heeft als
het minimale medium voor A. nidulans FGSC A722 (zie Tabel 9, p.37) maar gestabiliseerd is
met sorbitol (1.2 M = 218 g/L). Om mycelium te bekomen van de te transformeren schimmel
worden sporen geënt in vloeibaar niet-selectief medium (zie Tabel 23). Na incubatie worden
uit het mycelium protoplasten bereid.
Tabel 23. Samenstelling vloeibare niet-selectieve media
Concentratie (g/L)
Component
AMM
Glucose
10
NaNO3
6
Uracil (10 mM)
1.12
1.52
KH2PO4
0.52
MgSO4.7H2O
KCl
0.52
Gistextract (Difco)
5
Hutner’s sporenelementen 1 mL/L
pH
4.5
11.2.2. Selectieve media
De selectieve media bevatten geen uracil en uridine. Het zijn vaste minimale media die enkel
groei toelaten van prototrofe kolonies. De OMPD-negatieve A. nidulans FGSC A722 kan hier
dus niet op groeien. Enkel wanneer deze getransformeerd wordt met een werkend OMPD gen
kan er groei optreden op de platen. Er worden twee soorten selectief medium gebruikt. TO-S
is gestabiliseerd met 1.2 M sorbitol en wordt gebruikt voor de regeneratie van protoplasten
van A. nidulans na transformatie. Het TO- medium wordt gebruikt voor replica-plating en
controle van de prototrofe kolonies. De samenstelling van de selectieve media is weergegeven
in Tabel 24.
III. Materialen en Methoden - 57
MANU DE GROEVE
Tabel 24. Samenstelling vaste selectieve media
Component
TO-S
Glucose
10
Sorbitol (1.2 M)
218
6
NaNO3
1.52
KH2PO4
0.52
MgSO4.7H2O
KCl
0.52
Agar Noble (Difco)
15
Hutner’s sporenelementen
1 mL/L
pH
4.5
TO10
6
1.52
0.52
0.52
15
1 mL/L
4.5
11.3. Transformatieprotocol
11.3.1. Voorbereiding
•
•
•
Oogst sporen vanop een PDA plaat met 5 mL fysiologische oplossing (0.9% NaCl)
Tel de sporendensiteit met behulp van een Thoma-telkamer
Ent de sporen in 400 mL AMM in 2 liter erlenmeyers zonder baffles (zodat er ongeveer
1x109 sporen per 500 mL cultuur zijn)
• Deze culturen worden geïncubeerd: 200 RPM, 30°C, 16 uur
11.3.2. Protoplastbereiding
•
•
•
•
•
•
•
•
•
•
•
Plaats 1 fles STC (500 mL) op ijs
Zet een (steriele) trechter op een erlenmeyer; doe er een steriele Miracloth-filter in en
oogst het mycelium door de cultuur over de filter te gieten
Was met ongeveer 250 mL steriel water
Was met ongeveer 250 mL CaCl2
Neem de filter samen met een pincet en draai tot zoveel mogelijk vocht verwijderd is
Leg de filter met het mycelium open op een stukje steriele aluminiumfolie
Bepaal het gewicht van een lege, steriele falcon
Doe het mycelium met een pincet (steriel) in de falcon
Bepaal de hoeveelheid mycelium in de falcon
Vul de falcon tot 50 mL met CaCl2
Maak de enzymoplossing voor de vertering van de celwand klaar (per 5 g mycelium):
- Meng in een falcon:
- 600 mg β-D-glucanase (Interspex)
- 375 mg driselase (Interspex)
- 848 mg Yeast Lytic Enzymes (ICN)
- Vul de falcon met gedistilleerd water tot 50 mL en meng goed
- Centrifugeer: 4000 RPM, 4 minuten, 4°C
MANU DE GROEVE
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
•
III. Materialen en Methoden - 58
Giet in een 500 mL erlenmeyer het mycelium en het supernatans van de enzymoplossing
samen
Incubeer op een schudder (± 55 RPM) bij 25°C gedurende één tot anderhalf uur
Volg de protoplastvorming onder de microscoop (Olympus CH30 lichtmicroscoop)
Plaats de erlenmeyer op ijs wanneer de reactie voldoende lang is doorgegaan (als er kleine
bolletjes zichtbaar zijn naast het mycelium, maar het mycelium nog niet volledig gebroken
is)
Zet een steriele trechter op een steriele erlenmeyer en plaats er een Miracloth-filter in
Was goed met koude STC en giet daarna de erlenmeyer leeg
Zet de erlenmeyer + trechter + filter op ijs
Filtreer de protoplasten (langzaam gieten! hals niet flamberen!)
Was met ongeveer 100 mL koude STC
Verdeel over 4 steriele falcons (elk 50 mL)
Centrifugeer: 2000 RPM, 10 minuten, 4°C (toerental en duur zijn afhankelijk van de
vastheid van de protoplasten)
Verwijder het supernatans
Los de pellet van 1 falcon op in ongeveer 25 mL koude STC
Giet dit in een tweede falcon
Spoel de eerste falcon met 25ml koude STC
Giet ook dit in de tweede falcon
Doe hetzelfde met de overige twee falcons
Centrifugeer: 2000 RPM, 10 minuten, 4°C (toerental en duur zijn variabel)
Verwijder het supernatans
Los de pellets opnieuw op zoals hierboven beschreven (voeg weer twee aan twee bij
elkaar)
Centrifugeer: 2000 RPM, 10 minuten, 4°C (toerental en duur zijn variabel)
Verwijder het supernatans, maar houdt een klein beetje over om de pellet in op te lossen
(zo zijn de protoplasten ongeveer een half uur bewaarbaar op ijs)
Doe 5 µL van de protoplastoplossing in een epje en verdun met 195 µL koude STC (40x)
Bepaal de protoplastdensiteit met behulp van een Thoma-telkamer (de uiteindelijke
concentratie moet ongeveer 1x108 protoplasten per mL zijn)
Test de leefbaarheid van de protoplasten voor transformatie door een equivalent van 100
en 500 protoplasten uit te platen op PDAS en OS.
11.3.3. Transformatie en selectie
•
•
•
Controleer met afgesneden tips hoeveel µL protoplasten je hebt
Voeg 1 µL 15 mM aurintricarboxylzuur toe per 100 µL protoplasten
Voeg het DNA toe: maximum 10 µg DNA per 100 µL protoplasten in maximum 10 µL
water per 100 µL protoplasten
• Plaats 10 minuten op ijs
III. Materialen en Methoden - 59
MANU DE GROEVE
•
•
•
•
•
•
•
•
•
Zet ondertussen de oplossing PEG bij 30°C
Plaats 10 minuten bij kamertemperatuur
Voeg (per 100 µL protoplasten) toe: 250 µL PEG, 250 µL PEG en 850 µL PEG en schud
tussen elke toevoeging
Plaats 20 minuten bij kamertemperatuur
Voeg STC (niet meer koud) bij tot 50 mL in totaal
Centrifugeer: 2000 RPM, 5 minuten, 4°C
Verwijder het supernatans en hou een druppel over om de pellet in op te lossen
Test de leefbaarheid van de protoplasten na transformatie door een equivalent van 100 en
500 protoplasten uit te platen op PDAS en OS
Plaat uit op TO-S selectief medium (150 µL per plaat)
12. ULTRAVIOLET MUTATIE EN FILTRATIEAANRIJKING
Voor de constructie van een auxotrofe OMPD-negatieve mutant van M. gramineum werden
sporen van de schimmel bestraald met ultraviolet (UV) licht. De gemuteerde sporen werden
vervolgens aangerijkt aan uracil/uridine-auxotrofe mutanten via filtratieaanrijking..
12.1. Ultraviolet mutatie
Een oplossing van 2x108 sporen/mL van M. gramineum wordt 10x verdund met fysiologische
oplossing (0.9% NaCl). Van deze oplossing (2x107 sporen/mL) wordt 10 mL overgebracht
naar een steriele petriplaat.
Vervolgens wordt de UV-lamp 10-15 minuten opgewarmd en op een afstand van 30 cm
geplaatst van de petriplaat met deksel en sporenoplossing. De petriplaat wordt op een roerder
geplaatst zodat de sporen zo homogeen mogelijk verdeeld worden. De UV-behandeling dient
te gebeuren in het donker of bij (infra)rood licht om herstel van de DNA schade te
voorkomen. De UV-behandeling start van zodra het deksel van de petriplaat verwijderd
wordt. De sporen worden 6 minuten bestraald met ultraviolet licht.
Om de overleving na te gaan worden van de sporenoplossing drie stalen van 200 µL genomen
op tijdstip 0 (geen UV-belichting) en na 6 minuten UV-behandeling. Deze stalen worden bij
800 µL fysiologische oplossing gevoegd. De oplossingen worden uitgeplaat in volgende
verdunningen:
- voor tijdstip 0:
- na 6 minuten UV-behandeling:
10-2; 10-3; 10-4
10-1; 10-2; 10-3
Van elke verdunning wordt 100 µL uitgeplaat op compleet PDA-medium. Na twee dagen
incubatie bij 25°C worden de kolonies geteld en de overleving bepaald.
MANU DE GROEVE
III. Materialen en Methoden - 60
De gemuteerde sporen worden gebruikt voor een filtratieaanrijking van uracil/uridine
auxotrofe mutanten.
12.2. Filtratieaanrijking
Voor de filtratieaanrijking van auxotrofe mutanten worden de gemuteerde sporen eerst geënt
in vloeibaar minimaal medium (LMM). Na enkele filtratieronden worden de niet-gekiemde
sporen (=auxotrofen) geënt op vast minimaal medium dat de auxotrofe merker bevat (SCM).
Na replica plating op vast minimaal medium zonder de auxotrofe merker (SMM) kunnen de
auxotrofen geïdentificeerd worden. Als alternatief kunnen de sporen na de filtratieaanrijking
direct uitgeplaat worden op 5-fluoro-orotinezuur (5-FOA) bevattend medium (TO+). Kolonies
die op deze platen groeien zijn OPRT- of OMPD-negatief. De gebruikte media voor de
filtratieaanrijking zijn weergegeven in Tabel 25.
Tabel 25. Samenstelling benodigde media voor filtratieaanrijking
Concentratie (g/L)
Component
LMM
SMM
Glucose
10
10
NaNO3
6
6
Uracil
Uridine
5-fluoro-orotinezuur (FOA)
1.52
1.52
KH2PO4
0.52
0.52
MgSO4.7H2O
KCl
0.52
0.52
Bacteriologische agar (1.5%, Oxoid) 15
Hutner’s sporenelementen
1 mL/L
1 mL/L
pH
5.5
5.5
SCM
10
6
1.12
2.44
1.52
0.52
0.52
15
1 mL/L
5.5
TO+
10
6
0.224
1
1.52
0.52
0.52
15
1 mL/L
4.5
Voor de filtratieaanrijking werd gebruik gemaakt van een protocol van Bos & Stadler (1996)
dat aangepast werd voor M. gramineum.
Van de gemuteerde sporenoplossing worden 108 sporen geënt in een vloeibare 150 mL LMM
cultuur. Na incubatie (in het donker) gedurende 24 uur bij 25°C (150 RPM) wordt de cultuur
een eerste maal gefilterd over steriele Miracloth filter (Calbiochem; porie: 22-25 µm). Aan
het filtraat wordt vervolgens 50 mL vers LMM medium toegevoegd en opnieuw geïncubeerd
(in het donker) bij 25°C (150 RPM). Na 24 uur wordt opnieuw gefilterd over Miracloth, maar
wordt geen nieuw medium toegevoegd aan het filtraat. Het filtraat wordt opnieuw
geïncubeerd gedurende 24 uur waarna een laatste maal gefilterd wordt.
Het filtraat wordt vervolgens verdeeld over vier falcons en deze worden gecentrifugeerd bij
4000 RPM gedurende 5 minuten. Bij elke falcon wordt het supernatans verwijderd en wordt
de pellet heropgelost in 1 mL fysiologische oplossing. Van de heropgeloste pellet wordt
vervolgens 100 µL uitgeplaat op SCM en SMM om het aantal leefbare sporen na de
filtratieaanrijking te bepalen.
MANU DE GROEVE
III. Materialen en Methoden - 61
De sporen worden vervolgens geënt in een densiteit van ongeveer 100 sporen/plaat op SCM
en TO+ medium. Groeiende kolonies worden overgeënt naar SMM om te testen op auxotrofie.
Kolonies die niet verder groeien op het minimale SMM medium zijn auxotroof. Vervolgens
kan getest worden of de bekomen auxotrofe mutanten OMPD-negatief zijn door
complementatie met het homologe OMPD gen van M. gramineum.
IV. Resultaten en Bespreking - 62
MANU DE GROEVE
IV. RESULTATEN EN BESPREKING
Het doel van deze scriptie is het kloneren van het homologe OMPD gen van Myrothecium
gramineum MUCL39210. Dit gen kan gebruikt worden als selectiemerker in transformatieen expressiesystemen voor Myrothecium gramineum en andere filamenteuze schimmels. Om
het volledige homologe OMPD gen te kloneren werd in eerste instantie gebruik gemaakt van
gedegenereerde PCR. Het stuk van het gen dat zo werd bekomen, werd vervolgens uitgebreid
via PCR-gebaseerde genomewalking met de BD GenomeWalker™ Universal Kit (BD
Biosciences). De gevolgde strategie is schematisch weergegeven in Figuur 11.
genomisch DNA Myrothecium gramineum
gedegenereerde PCR op OMPD gen
ontwerpen van genspecifieke primers (GSP) op
gekende sequentie OMPD gen voor genomewalking
5' GSP2
5' GSP1
3' GSP2
3' GSP1
GenomeWalking
BD GenomeWalker
Universal Kit
BD GenomeWalker
Adaptor
UPSTREAM (5')
CONSTRUCTIE
GENOMWALKER BANKEN
BD GenomeWalker
Adaptor
DOWNSTREAM (3')
ontwerpen van primers voor amplificatie van
het volledige OMPD gen
High Fidelity PCR
OMPD gen
Ligatie in pGEM-T en
klonering
OMPD gen van
Myrothecium gramineum
pOV
Figuur 11. Strategie voor het kloneren van het volledige OMPD gen van Myrothecium gramineum
MANU DE GROEVE
IV. Resultaten en Bespreking - 63
De functionaliteit van het OMPD gen werd aangetoond door de complementatie van een
OMPD-negatieve Aspergillus nidulans. In wat volgt zullen de resultaten meegedeeld en
besproken worden van volgende experimenten:
• Gedegenereerde PCR op het homologe OMPD gen van Myrothecium gramineum
MUCL 39210
• Genomewalking naar de 5’ (upstream) en de 3’ (downstream) kant van het via
gedegenereerde PCR bekomen deel van het OMPD gen
• Klonering en sequenering van het volledige OMPD gen van M. gramineum
• Studie van de eiwit- en DNA sequenties van het OMPD gen van M. gramineum
• Transformatie en complementatie van de OMPD-negatieve mutant A. nidulans
FGSC A722 met het wild type OMPD gen van M. gramineum
• UV-mutatie en filtratieaanrijking van M. gramineum voor de creatie van een
OMPD-negatieve mutant
• Constructie van een OMPD knock-out vector
1. KLONERING VAN HET OMPD GEN VAN
MYROTHECIUM GRAMINEUM MUCL 39210
1.1. Ontwikkeling van gedegenereerde primers voor OMPD
Het design van de primers is van cruciaal belang bij gedegenereerde PCR. De gedegenereerde
primers voor het OMPD gen van Myrothecium gramineum werden ontworpen op basis van
geconserveerde sequenties in de aminozuur- en nucleotidensequentie van de OMPD genen
van verwante schimmels en gisten. Omdat de aminozuursequentie meer geconserveerd is dan
de nucleotidensequentie werd in eerste instantie gezocht naar de meest geconserveerde
aminozuursequenties in bekende OMPD eiwitten.
Om deze geconserveerde sequenties te vinden werd een multiple sequence alignment
uitgevoerd met de OMPD aminozuursequenties van de vijftig meest verwante schimmels en
gisten van Myrothecium gramineum. De gebruikte schimmels zijn allen Ascomyceten zoals
M. gramineum.
Een deel van het resultaat van de alignment is weergegeven in Figuur 12.
MANU DE GROEVE
IV. Resultaten en Bespreking - 64
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
1
1
1
1
1
1
1
1
1
1
------MSSKSQL--PFSTRASNHPNPLARKLFEVAEAKKSNVTVSADVTTTKELLDLADR---LGPYIA
------MSSKSQL--TYGARASKHPNPLAKRLFEIAEAKKTNVTVSADVTTTRELLDLADR---LGPYIA
------MSSKSQL--TYTARASKHPNALAKRLFEIAEAKKTNVTVSADVTTTKELLDLADR---LGPYIA
------MSSKSQL--TYSARASKHPNALVKKLFEVAEAKKTNVTVSADVTTTKELLDLADR---LGPYIA
------MSSKSQL--TYTARAQSHPNPLARKLFQVAEEKKSNVTVSADVTTTKELLDLADPSTGLGPYIA
MSTSQETQPHWSLKQSFAERVESSTHPLTSYLFRLMEVKQSNLCLSADVEHARDLLALADK---VGPSIV
MSTTQ--QPHWSLQKSFAERVESSSHPLTSYLFRLMEVKQSNLCLSDDVEHARELLALADK---IGPSIV
------MAPHPTLKSTYSTRAETVTHPLSAYLYKLMDLKASNLCLSADVANARELLHVADK---VGPSIV
------MASHPTLKTTFAARSEATTHPLTSYLLRLMDLKASNLCLSADVPTARELLYLADK---IGPSIV
------MAPHPTLKATFAARSETATHPLTAYLFKLMDLKASNLCLSADVPTARELLYLADK---IGPSIV
*
*
*
*
* *
* **
** **
** *
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
60
60
60
60
63
68
66
62
62
62
VIKTHIDILS--DFGPETING--LNALAEKHNFLIFEDRKFIDIGNTVQKQYHGGALKISEWAHIINCAV
VIKTHIDILT--DFSVDTING--LNVLAQKHNFLIFEDRKFIDIGNTVQKQYHGGALRISEWAHIINCSV
VIKTHIDILS--DFSDETIEG--LKALAQKHNFLIFEDRKFIDIGNTVQKQYHRGTLRISEWAHIINCSI
VIKTHIDILS--DFSEETITG--LKALAEKHNFLIFEDRKFIDIGNTVQKQYHGGTLRISEWAHIINCSI
VIKTHIDILS--DFSQETIDG--LNALAQKHNFLIFEDRKFIDIGNTVQKQYHNGTLRISEWAHIINCSI
VLKTHYDLITGWDYHPHTGTGAKLAALARKHGFLIFEDRKFVDIGSTVQKQYTAGTARIVEWAHITNADI
VLKTHYDLITGWDYHPHTGTGAKLAALARKHGFLIFEDRKFVDIGSTVQKQYTAGTARIVEWAHITNADI
VLKTHYDMVAGWDFTPETGTGARLAKLARKHGFLIFEDRKFGDIGNTVELQYTQGAARIIEWAHIVNVNM
VLKTHYDMVSGWDFHPDTGTGAKLASLARKHGFLIFEDRKFGDIGHTVELQYTSGSARIIDWAHIVNVNM
VLKTHYDMVSGWTSHPETGTGAQLASLARKHGFLIFEDRKFGDIGHTVELQYTGGSARIIDWAHIVNVNM
* *** *
* * * ** ** ********* *** ** ** *
* **** *
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
126
126
126
126
129
138
136
132
132
132
LPAEGIVQALAETAQA--EDFPH----------------------------------------------LPGEGIVEALAQTASA--QDFPY----------------------------------------------LPGEGIVEALAQTASA--PDFSY----------------------------------------------LPGEGIVEALAQTASA--EDFPY----------------------------------------------LPGEGIVEALAQTAQA--TDFPY----------------------------------------------HAGEAMVSAMAQAAQKWRERIPYEVKTSVSVGTPVA-DQFADEE----AE--DQVEELRKVVTRETSTTT
HAGEAMVSAMAQAAQKWRERIPYEVKTSVSVGTPVA-DQFADEE----AE--DQVDELRKIVPRENSTSK
VPGKASVTSLANAAAKWLERLPYEVKTSVTVGTPRNTDEDDDDEDDGNAGEMERSHTFGLNGNNGVPIKVPGKASVASLAQGARRWLERYPCEVKTSVTVGTP-TMDQFDDAEDAKDDEPA-TVNDNGS-NMMEKPIYA
VPGKASVASLAQGAKRWLERYPCEVKTSVTVGTP-TMDSFDDDADSRDAEPAGAVNGMGSIGVLDKPIYS
*
* *
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
147
147
147
147
150
201
199
201
199
201
--------GS---------------------------------------ERGLLILAEMTSKGSLATGEY
--------GP---------------------------------------ERGLLVLAEMTSKGSLATGEY
--------GP---------------------------------------ERGLLILAEMTSKGSLATGQY
--------GS---------------------------------------ERGLLILAEMTSKGSLATGQY
--------GS---------------------------------------ERGLLILAEMTSKGSLATGAY
-KDTDGRKSSIVSITTVTQTYEPADSPRLVKTISEDDEMVFPGIEEAPLDRGLLILAQMSSKGCLMDGKY
EKDTDGRKGSIVSITTVTQTYEPADSPRLAKTISEGDEAVFPGIEEAPLDRGLLILAQMSSKGCLMDGKY
-ESSDGRKGSIVSVTTVTQQYESAHSPRLTKTIAEEGDMLLAGLEEPPLNRGLLILAQMSSAGNFMNAEY
GRNGDGRKGSIVSITTVTQQYESAASPRLGKTIAEGDESLFPGIEEAPLNRGLLILAQMSSEGNFMTGEY
NRSGDGRKGSIVSITTVTQQYESVSSPRLTKAIAEGDESLFPGIEEAPLSRGLLILAQMSSQGNFMNKEY
**** ** * * *
*
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
170
170
170
170
173
270
269
270
269
271
TSASVDYARKYPSFVLGFVSTRALTEVSST------------VTAA-DNEDFVVFTTGVNLSSK-----TKASVDYARKYKNFVMGFVSTRALTEVQSD------------VSSASEDEDFVVFTTGVNLSSK-----TTSSVDYARKYKNFVMGFVSTRSLGEVQSE------------VSSPSDEEDFVVFTTGVNISSK-----TTSSVDYARKYKKFVMGFVSTRHLGEVQSE------------VSSPSEEEDFVVFTTGVNLSSK-----TSASVDIARKYPSFVLGFVSTRSLGEVEST------------EAPAS--EDFVVFTTGVNLSSK-----TWECVKAARKNKGFVMGYVAQQNLNGITKEALAPSYEDGESTTEEEAQADNFIHMTPGCKLPPPGEEAPQ
TWECVKAARKNKDFVMGYVAQQNLNGITKEDLAPGYEDGETSTEEEAQADNFIHMTPGCKLPPPGEEAPQ
TQACVEAAREHKDFVMGFVSQEALN-----------------SQPD---DDFIHMTPGCQLPPEHEE--TQACVEAAREHKDFVMGFISQEALN-----------------TQAD---DDFIHMTPGCQLPPEDEDQQT
TQASVEAAREHKDFVMGFISQETLN-----------------TEPD---DAFIHMTPGCQLPPEDEDQQT
*
* **
** *
*
*
* *
PYRF_CLAFU
PYRF_ASPFU
PYRF_ASPNG
PYRF_ASPOR
PYRF_PENCH
PYRF_NEUCR
PYRF_SORMA
PYRF_CEPAC
PYRF_TRIHA
PYRF_TRIRE
221
222
222
222
223
340
339
317
319
321
-----GDKLGQQYQTPQSAIG-RGADFIIAGRGIYTAPDPVEAAKQYQQQGWEAYLARVGGASQ
-----GDKLGQQYQTPASAIG-RGADFIIAGRGIYAAPDPVEAAQRYQKEGWEAYMARVCGKS-----GDKLGQQYQTPASAIG-RGADFIIAGRGIYAAPDPVQAAQQYQKEGWEAYLARVGGN------GDKLGQQYQTPESAVG-RGADFIIAGRGIYAAPDPVEAAKQYQKEGWDAYLKRVGAQ------GDKLGQQYQTPQSAVG-RGADFIISGRGIYAAADPVEAAKQYQQQGWEAYLARVGAQ------GDGLGQQYNTPDNLVNIKGTDIAIVGRGIITAADPPAEAERYRRKAWKAYQDRRERLA-----GDGLGQQYNTPDNLVNIKGTDIAIVGRGIITASDPPAEAERYRRKAWKAYQDRRERLADAELRGDGKGQQYNTPEKLIGVCGADIVIVGRGILKAGDLQHEAERYRSAAWKAYTERVR---NGKVGGDGQGQQYNTAHKIIGIAGSDIAIVGRGILKASDPVEEAERYRSAAWKAYTERLLR--NGSVGGDGQGQQYNTPHKLIGIAGSDIAIVGRGILKASDPVEEAERYRSAAWKAYTERLLR--** **** *
* * * **** * *
* *
* ** *
Figuur 12. Deel van de alignment die gebruikt werd voor het ontwikkelen van gedegenereerde primers
MANU DE GROEVE
IV. Resultaten en Bespreking - 65
Op basis van de uitgevoerde multiple sequence alignment met de OMPD eiwitsequenties
werden volgende geconserveerde domeinen gevonden:
- FLIFEDRKF-DIG-TV
- RGLLILA-M
- GD--GQQY-T
(meest aan 5’ kant gelegen)
(meest aan 3’ kant gelegen)
Deze geconserveerde sequenties werden ook in de literatuur teruggevonden (zie
Literatuurstudie, Tabel 4, p.18). De meest gebruikte sequenties voor het ontwerpen van
gedegenereerde
primers
zijn
de
FLIFEDRKF-DIG-TV
en
GD--GQQY-T
aminozuursequenties (Weidner et al., 1998; Yamazaki et al., 1999). Er werd besloten om ook
in dit onderzoek gebruik te maken van deze geconserveerde sequenties voor de ontwikkeling
van gedegenereerde primers voor de amplificatie van het OMPD gen van M. gramineum.
In een eerste stap werden de DNA sequenties van de OMPD genen van de meest verwante
schimmels opgezocht in de EMBL nucleotidensequentie database. In een volgende stap werd
dan gezocht naar de nucleotidensequenties die overeenkomen met de bovenvermelde
geconserveerde aminozuursequenties. Voor de ontwikkeling van de forward primer werden
de nucleotidensequenties van de FLIFEDRKF-DIG aminozuursequentie onder elkaar
gerangschikt, voor de reverse primer de nucleotidensequenties van de GD--GQQY-T
aminozuursequentie. De gebruikte sequenties van verwante schimmels voor de ontwikkeling
van de forward en de reverse primer zijn respectievelijk weergegeven in Tabel 26 en Tabel
27. De nucleotidensequenties zijn zo weergegeven dat onder elk aminozuur van de
geconserveerde sequentie het bijhorende codon staat. Op basis van de gerangschikte
nucleotidensequenties werd vervolgens een consensussequentie opgesteld. Omdat de
genetische code gedegenereerd is bevat deze consensussequentie op bepaalde plaatsen
gedegenereerde nucleotiden. De consensussequentie dient als uitgangspunt voor het ontwerp
van een gedegenereerde primer.
Voor het ontwerp van de primers zelf dienen enkele zaken in het oog gehouden te worden.
Onder meer het GC-gehalte (40-60%), de lengte van de primer (20-21 bp), de stabiliteit, de
smelttemperatuur en de gedegenereerdheid spelen een belangrijke rol. Hoewel er reeds
succesvol genen werden geïsoleerd met meer dan 1000 maal gedegenereerde primers is het in
het algemeen aan te raden de gedegenereerdheid zo laag mogelijk te houden. Dit kan men
doen door gebruik te maken van ‘promiscue’ nucleotiden zoals inosine (bindt met A, T, C en
G) of door een gedegenereerde base in de consensus sequentie te vervangen door de meest
voorkomende nucleotide op die plaats. Een gedegenereerde base kan ook vervangen worden
door een minder gedegenereerde base wanneer slechts enkele nucleotiden afwijken. In Tabel
26 en Tabel 27 zijn de nucleotiden waarmee geen rekening werd gehouden weergegeven op
een grijze achtergrond. Door geen rekening te houden met deze nucleotiden kon de
gedegenereerdheid van de primers laag gehouden worden.
IV. Resultaten en Bespreking - 66
MANU DE GROEVE
Tabel 26. Overzicht van de gebruikte sequenties en organismen voor het ontwerpen van een gedegenereerde
FORWARD primer voor PCR op het OMPD gen van M. gramineum
Organisme
Sordariomyceten
Acremonium chrysogenum
Epichloe typhina
Neurospora crassa
Sordaria macrospora
Trichoderma harzanium
Trichoderma reesei
Eurotiomyceten
Aspergillus awamori
Aspergillus fumigatus
Aspergillus kawachii
Aspergillus nidulans
Aspergillus niger
Aspergillus oryzae
Aspergillus oryzae
Aspergillus parasiticus
Penicllium camemberti
Penicllium chrysogenum
Penicllium nalgiovense
Auxarthron zuffianum
Coccidioides immitis
Uncinocarpus reesii
Andere
Cladosporium fulvum
Solorina crocea
Clustal consensus
Consensus sequentie
Ontworpen primers (5’->3’)
OMPD obv flifedrkf
OMPD obv flifedrkf 2
Aminozuur- en nucleotidensequentie van geconserveerd domein van OMPD
F
L
I
F
E
D
R
K
F
X
D
I
G
C
A
C
C
C
C
TTC
TTC
TTC
TTC
TTC
TTC
CTC
TTG
CTC
CTC
CTC
CTC
ATC
ATA
ATC
ATC
ATT
ATC
TTT
TTC
TTC
TTC
TTC
TTC
GAG
GAA
GAG
GAG
GAA
GAG
GAC
GAT
GAC
GAC
GAC
GAC
CGC
CGC
CGC
CGC
CGC
CGC
AAG
AAG
AAG
AAG
AAG
AAG
TTT
TTC
TTC
TTC
TTC
TTT
GGC
GCT
GTC
GTC
GGG
GGC
GAT
GAC
GAC
GAC
GAC
GAC
ATT
ATT
ATT
ATT
ATT
ATT
GG
GG
GG
GG
GG
GG
C
C
C
C
C
T
T
T
C
C
C
G
C
C
TTC
TTT
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
CTC
TTG
CTC
CTC
CTC
CTC
CTC
CAC
CTC
CTT
CTC
TTG
CTG
CTC
ATC
ATC
ATC
ATC
ATC
ATC
ATC
ATC
ATC
ATC
ATC
ATT
ATC
ATC
TTC
TTC
TTC
TTT
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTT
TTC
GAG
GAG
GAG
GAG
GAG
GAA
GAA
GAA
GAA
GAA
GAA
GAG
GAG
GAA
GAC
GAC
GAC
GAC
GAC
GAT
GAT
GAT
GAC
GAC
GAC
GAC
GAC
GAC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGC
CGG
CGG
AAA
AAA
AAA
AAG
AAA
AAG
AAG
AAG
AAA
AAA
AAA
AAG
AAA
AAG
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
TTC
ATT
ATC
ATT
ATC
ATC
ATC
ATC
AGA
ATC
ATC
ATC
GTC
GTT
GTC
GAC
GAC
GAC
GAC
GAC
GAT
GAT
GAT
GAC
GAC
GAC
GAT
GAT
GAT
ATC
ATC
ATC
ATC
ATT
ATC
ATT
ATC
ATC
ATC
ATC
ATT
ATT
ATC
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
GG
C TTT CTC
C TTT CTC
**
N TTY YTB
ATC
ATC
**
ATH
TTT
TTC
**
TTY
GAG
GAG
**
GAR
GAC
GAC
**
GAY
CGC
CGG
**
CGS
AAG
AAA
**
AAR
TTC ATC GAC ATC
TTC GTT GAT ATC
**
** **
TTY RBN GAY ATY
GG
GG
**
GG
C CTS ATC TTY GAR GAY CGC AA
C CTS ATC TTY GAR GAY CGC
Door rekening te houden met de gedegeneerdheid, het GC gehalte, de lengte en de stabiliteit
konden uiteindelijk primers ontworpen worden die zo veel mogelijk voldoen aan de eisen. In
Tabel 26 en Tabel 27 zijn de ontworpen primers weergegeven.
IV. Resultaten en Bespreking - 67
MANU DE GROEVE
Tabel 27. Overzicht van de gebruikte sequenties en organismen voor het ontwerpen van een gedegenereerde
REVERSE primer voor PCR op het OMPD gen van M. gramineum
Organisme
Sordariomyceten
Acremonium chrysogenum
Epichloe typhina
Neurospora crassa
Sordaria macrospora
Trichoderma harzanium
Eurotiomyceten
Aspergillus kawachii
Aspergillus nidulans
Aspergillus niger
Aspergillus fumigatus
Aspergillus oryzae
Aspergillus oryzae
Aspergillus parasiticus
Penicllium camemberti
Penicllium chrysogenum
Penicllium nalgiovense
Clustal consensus
Consensus sequentie
Aminozuur- en nucleotidensequentie van geconserveerd domein
G
D
X
X
G
Q
Q
Y Q/N T P/A
GGC
GGC
GGC
GGT
GGA
GAT
GAC
GAC
GAC
GAT
GGG
GGA
GGA
GGA
GGC
AAG
AAA
CTG
CTT
CAA
GGC
GGC
GGT
GGT
GGC
CAG
CAG
CAG
CAG
CAG
GGA
GGG
GGA
GGA
GGT
GGA
GGA
GGC
GGC
GGC
**
GGN
GAT
GAT
GAT
GAT
GAC
GAC
GAC
GAT
GAT
GAT
**
GAY
AAG
AAG
AAG
AAG
AAG
AAG
AAG
AAG
AAG
AAG
CTC
CTG
CTC
CTT
CTG
CTG
CTG
CTC
CTC
CTC
CAG
CAG
CAG
CAG
CAG
TAC
TAT
TAC
TAC
TAC
AAC
AAT
AAC
AAC
AAC
ACC
ACA
ACG
ACG
ACG
CCC
CCA
CCG
CCG
GCG
GA
CA
GA
GA
CA
GGT
GGG
GGT
GGA
GGA
GGA
GGA
GGT
GGT
GGT
**
RRV MWN GGN
CAG
CAG
CAG
CAG
CAG
CAG
ACA
CAG
CAG
CAG
CAG
CAG
CAG
CAA
CAG
CAG
CAA
CAG
CAG
CAG
**
MMR CAR
TAC
TAT
TAC
TAC
TAC
TAC
TAC
TAC
TAC
TAC
**
TAY
CAG
CAG
CAG
CAG
CAA
CAA
CAG
CAG
CAG
CAG
*
MAN
ACG
ACA
ACT
ACT
ACT
ACT
ACT
ACA
ACG
ACG
**
ACN
CCC
CCT
CCC
CCT
CCT
CCT
CCT
CCG
CCG
CCG
*
SCN
GC
GG
GC
GC
GA
GA
GA
CA
CA
CA
SV
Template sequentie voor primer
W GGN CAG CAG TAC MAV ACD CC
Primer=reverse complement template
GG HGT BTK GTA CTG CTG NCC W
OMPD obv gqqyxtx
GG HGT STK GTA CTG CTG HCC
OMPD obv gqqyxtx2
De theoretisch verwachte groottes van de PCR fragmenten die kunnen bekomen worden met
de ontworpen primers zijn weergegeven in Tabel 28. Hieruit blijkt dat de gedegenereerde
primers bij de Sordariomyceten een fragment van ongeveer 700-750 bp amplificeren, terwijl
dit bij alle andere schimmels een fragment van ongeveer 430 bp is. Omdat Myrothecium
gramineum tot de klasse van de Sordariomyceten behoort werd een fragment van 700 tot 750
bp verwacht .
Tabel 28. Theoretische grootte van de fragmenten na PCR met de gedegenereerde primers
Organisme
PCR fragment (bp) Organisme
PCR fragment (bp)
Eurotiomyceten
Aspergillus awamori
Aspergillus kawachii
Aspergillus nidulans
Aspergillus niger
Aspergillus oryzae
Penicllium camemberti
Penicllium chrysogenum
Penicllium nalgiovense
Andere
Cladosporium fulvum
432
432
426
432
432
429
426
429
429
Sordariomyceten
Acremonium chrysogenum
Neurospora crassa
Sordaria macrospora
Trichoderma harzianum
Trichoderma reesei
714
750
753
720
726
MANU DE GROEVE
IV. Resultaten en Bespreking - 68
1.2. Gedegenereerde PCR op het OMPD gen
1.2.1. Optimalisatie protocol voor gedegenereerde PCR op OMPD gen
Omdat in het verleden bleek dat PCR’s met de High Fidelity PCR Master kit vaker positieve
resultaten gaven en er bovendien hogere opbrengsten werden bekomen dan bij gewone PCR,
werd besloten de gedegenereerde PCR uit te voeren met deze kit. Ook Yamazaki et al. (1999)
gebruikten het standaard protocol van de Expand High Fidelity PCR kit voor gedegenereerde
PCR op een deel van het OMPD gen van Rhizomucor pusillus. Het protocol van de eerste
gedegenereerde PCR is weergegeven in Tabel 29. Als template werd genomisch DNA van
Myrothecium gramineum gebruikt dat geïsoleerd werd zoals beschreven in Materialen en
Methoden (III.3.1, p.38).
Deze eerste PCR reactie leverde echter geen PCR fragmenten op. Er werd vermoed dat dit het
gevolg was van een te hoge annealingstemperatuur (60.6°C). De smelttemperaturen (Tm) van
de gebruikte primers zijn immers respectievelijk 61.5°C en 60.6°C. De annealingstemperatuur
is best ongeveer 5°C lager dan de smelttemperatuur van de primers
(http://www.fermentas.com). Daarom werd besloten het protocol te optimaliseren. De
annealingstemperatuur werd verlaagd naar 50°C. Naast genomisch DNA van M. gramineum
werd nu ook een positieve controle meegenomen, namelijk het pDJB2-ans plasmide dat het
OMPD gen (pyr4) van Neurospora crassa bevat. Van het plasmide werd 5 µL als template
gebruikt en werd de Master Mix 1 aangelengd tot 25 µL met steriel milli-Q water.
Er werd zoals verwacht een PCR fragment van 700-800 bp bekomen bij het plasmide
(positieve controle). Bij het genomisch DNA werden echter nog steeds geen PCR fragmenten
geobserveerd. Hieruit kon besloten worden dat de oorzaak waarschijnlijk niet bij de primers
of de annealingstemperatuur moest gezocht worden, maar eerder bij het genomisch DNA zelf,
de samenstelling van de PCR-reactiemix of het PCR-programma.
In sommige gevallen kunnen door het knippen van genomisch DNA de resultaten van de PCR
sterk verbeterd worden omdat het DNA dan ‘toegankelijker’ geworden is. Dit laat efficiëntere
primerbinding en amplificatie toe. Daarom werden vier preparatieve restricties uitgevoerd op
genomisch DNA van M. gramineum met volgende restrictie-enzymen: AvaI, DraI, EcoRI en
ScaI. Dit zijn allen hexacutters die niet in het OMPD gen van N. crassa en ook niet in enkele
andere OMPD genen van Ascomyceten knippen. De samenstelling van de restrictiemengsels
is weergegeven in Tabel 30.
IV. Resultaten en Bespreking - 69
MANU DE GROEVE
Tabel 29. High Fidelity PCR protocol
Primers
Sequentie (5’→ 3’)
OMPD obv flifedrkf CCTSATCTTYGARGAYCGCAA
OMPD obv gqqyxtx GGHGTBTKGTACTGCTGNCCW
Samenstelling MASTER MIX 1
Component
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
Template:
- genomisch DNA M.
gramineum
Steriel milli-Q water
Totaal
Samenstelling MASTER MIX 2
Component
2X High Fidelity PCR Master:
(bevat polymerasen, dNTP’s
(elk 0.4 mM), 3 mM MgCl2
Eindconcentratie
300 nM
300 nM
20x
1x
Lengte
61.5
60.6
21
21
Volume
1.5 µL
1.5 µL
22 µL
25 µL
Eindconcentratie
1X
Totaal: MASTER MIX 1 + MASTER MIX 2
PCR programma (Hot Start)
Cycli
1x
10x
Tm (°C)
Tijd
2 min.
30 s
30 s
2 min.
30 s
30 s
2 min. + 5 s inc.
7 min.
Volume
25 µL
50 µL
Temperatuur (°C)
94
94
60.6
72
94
60.6
72
72
Tabel 30. Samenstelling restrictiemengsels voor preparatieve
restrictie genomisch DNA
Component
Eindhoeveelheid
Volume
DNA staal
5 µg
31 µL
Restrictie-enzym
20 U
2 µL
Restrictiebuffer
1X
5 µL
milli-Q water
12 µL
Totaal
50 µL
MANU DE GROEVE
IV. Resultaten en Bespreking - 70
Na controle op gel bleek het genomisch DNA bij de vier restricties volledig verknipt te zijn.
Vervolgens werd 22 µL (2 µg geknipt DNA) van elk restrictiemengsel gebruikt als template
voor een High Fidelity PCR reactie. Er werd hetzelfde PCR protocol gevolgd als bij de laatst
beschreven PCR (annealing bij 50°C). Na controle op gel werden echter opnieuw geen PCR
fragmenten verkregen. Een mogelijke oorzaak hiervoor is dat de restrictie-enzymen toch
knippen in het OMPD van M. gramineum of het feit dat de restrictiemengsels niet werden
gezuiverd vooraleer ze gebruikt werden in de PCR reactie.
Ondertussen werd gezocht naar meer fundamentele oorzaken voor het mislukken van de
gedegenereerde High Fidelity PCR’s. Mede aan de hand van een tekst van Michael Koelle
kunnen volgende aanbevelingen gedaan worden voor succesvolle gedegenereerde PCR:
•
•
•
•
•
•
Het is aan te raden een polymerase zonder proofreading activiteit te gebruiken.
Polymerasen met proofreading activiteit kunnen immers de gebonden gedegenereerde
primers afbreken. Daarom is het gebruik van gewone Taq polymerase te verkiezen
boven High Fidelity PCR. Dit is dus in tegenspraak met het artikel van Yamazaki et al.
(1991), waar wel High Fidelity PCR gebruikt werd om een OMPD gen te amplificeren.
De concentratie van gedegenereerde primers is best hoger dan deze van nietgedegenereerde primers in gewone PCR.
Het primer design is de belangrijkste factor die het succes van gedegenereerde PCR
bepaalt. Vooral de 3’ kant van de primer is zeer belangrijk. Deze is best zo weinig
mogelijk gedegenereerd. Daarenboven is het best aan het 3’ uiteinde een exacte match
te hebben met de template.
Hoewel er reeds genen werden geamplificeerd met primers die meer dan 1000 maal
gedegenereerd waren is het aan te raden primers te gebruiken die maximaal 100 maal
gedegenereerd zijn.
Er wordt best vertrokken van standaard PCR condities, maar met lagere
annealingstemperatuur en meer cycli (35 tot 50 cycli). Indien dit geen resultaat oplevert
kunnen de eerste cycli bij een nog lagere annealingstemperatuur uitgevoerd worden,
waarna de temperatuur stapsgewijs verhoogd wordt.
Wanneer na een eerste gedegenereerde PCR geen fragmenten zichtbaar zijn op gel, kan
het nuttig zijn een tweede (reamplificatie) PCR uit te voeren onder dezelfde condities,
maar met het eerste PCR mengsel als template.
Rekening houdend met deze aanbevelingen werd overgestapt naar gewone PCR met Taq
polymerase dat geen 3’->5’ exonuclease activiteit bezit (= geen proofreading). De
annealingstemperatuur van het standaard PCR protocol werd sterk verlaagd (tot 30°C), de
annealingstijd werd verlengd naar 1 minuut, de concentratie van de primers werd verhoogd en
er werden 10 cycli meer uitgevoerd. Bovendien werd de reverse primer in hogere concentratie
toegevoegd dan de forward primer wegens meer gedegenereerd. De details van de
uitgevoerde PCR zijn weergegeven in Tabel 31. Als template werd zowel geknipt als
ongeknipt genomisch DNA gebruikt.
IV. Resultaten en Bespreking - 71
MANU DE GROEVE
Tabel 31. Details van de gedegenereerde PCR op het OMPD gen van Myrothecium
Primers
Tm (°C)
Lengte
Sequentie (5’→ 3’)
CCTSATCTTYGARGAYCGCAA
OMPD obv flifedrkf
61.5
21
GGHGTBTKGTACTGCTGNCCW
OMPD obv gqqyxtx
60.6
21
Samenstelling PCR reactie-mix
Component
Taq DNA polymerase (5U/µL)
10X Taq pol. Buffer (+Mg)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
DNA template:
- genomisch DNA
- restrictiemengsel (AvaI, DraI,
EcoRI of ScaI)
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
10x
30x
1x
Eindconcentratie
1U
1X
0.2 mM
2 µM
6 µM
Volume
0.2 µL
5 µL
1 µL
10 µL
30 µL
750 ng
465 ng
5 µL
5 µL
-
51.2 µL
Tijd
3 min.
45 s
1 min.
2 min.
45 s
1 min.
2 min.
7 min.
Temperatuur (°C)
94
94
30
72
94
35
72
72
Met deze PCR werden opnieuw geen fragmenten bekomen. Er werd echter wel bij alle
reacties een laagmoleculaire smeer (<100 bp) geobserveerd, vermoedelijk primer-dimeren.
Dit kan te wijten zijn aan de sterk verhoogde hoeveelheid primers die voor deze reactie
gebruikt werd. De verhouding template/primer is een belangrijke factor bij PCR. Door te veel
staal te gebruiken kan de PCR onmiddellijk verzadigd zijn. Omdat volgens het Taq-protocol
de hoeveelheid genomisch DNA in gewone PCR best tussen 10 en 250 ng ligt, en er tot nog
toe met veel grotere hoeveelheden werd gewerkt, werd besloten de hoeveelheid template te
verminderen. Er werden vier verschillende PCR’s uitgevoerd met respectievelijk 37 ng, 94
ng, 187 ng en 374 ng genomisch DNA als template. Ook werd de hoeveelheid Taq
polymerase verhoogd tot 2 U. Voor de rest werd het protocol gevolgd zoals weergegeven in
Tabel 31. Er werden echter opnieuw geen fragmenten bekomen. Er waren wel nog steeds
primer-dimeren zichtbaar onderaan de gel. Bij het mengsel met 37 ng staal waren deze het
minst intens. Dit betekent dat een deel van de primers in PCR-product ingebouwd moest zijn.
IV. Resultaten en Bespreking - 72
MANU DE GROEVE
Als mogelijke oorzaak voor het feit dat er toch geen fragmenten zichtbaar waren, werd
gedacht aan de ongelijke hoeveelheid forward en reverse primer waardoor de PCR reactie uit
balans is of aan het feit dat er te weinig cycli werden uitgevoerd.
Daarom werden in een volgende PCR de concentraties van de forward en reverse primer
gelijkgesteld en verlaagd tot 1 µM, in plaats van respectievelijk 2 µM en 6 µM. Er werden
opnieuw PCR’s uitgevoerd op dezelfde hoeveelheden genomisch DNA als de vorige PCR (37
tot 374 ng), maar er werd nu ook een reamplificatie uitgevoerd op een schijnbaar mislukt
PCR staal van de vorige PCR (op 37 ng genomisch DNA). De rest van het PCR protocol werd
uitgevoerd zoals de vorige beschreven PCR. Na controle op gel waren geen PCR fragmenten
zichtbaar bij de PCR’s op genomisch DNA. Bij de reamplificatie was er wel een licht bandje
zichtbaar van ongeveer 400 bp. Ook was er een lichte smeer zichtbaar.
Daar de reamplificatie van een ‘mislukt’ PCR mengsel een positief resultaat gaf, werd
gedacht dat de PCR reactie op genomisch DNA zeer inefficiënt verliep. Het viel inderdaad op
dat er vaak primer-dimeren werden geobserveerd op gel wat wijst op het niet of onvoldoende
binden van de primers op de template. De primer-dimeren waren waarschijnlijk het gevolg
van de zeer lage annealingstemperaturen (30 tot 35°C) waardoor de primers sneller met elkaar
binden dan met de template. Indien voorgaande theorie klopt zou door het verhogen van de
annealingstemperatuur de hoeveelheid primer-dimeren moeten verminderen en de
hoeveelheid PCR product verhogen.
Om de vermoedens te bevestigen werd een PCR uitgevoerd van 40 cycli met een constante
annealingstemperatuur van 50°C. De samenstelling van het reactiemengsel is zoals bij de
vorige PCR. De details van de uitgevoerde PCR zijn weergegeven in Tabel 32. Er werd één
reactie uitgevoerd op genomisch DNA (187 ng)
Het resultaat van deze PCR is weergegeven in Figuur 13. Er werd een smeer bekomen tussen
200 en 700 bp (niet zichtbaar op foto) met een zwak bandje op 700 bp.
1
Laan 1: Gedegenereerde PCR op OMPD volgens
protocol weergegeven in Tabel 32
M
Laan M: lengtemerker (SmartLadder)
- 0.7 kb
Figuur 13. Gedegenereerde PCR op OMPD
IV. Resultaten en Bespreking - 73
MANU DE GROEVE
Tabel 32. Details van de gedegenereerde PCR op het OMPD gen van Myrothecium
Primers
Tm (°C)
Lengte
Sequentie (5’→ 3’)
CCTSATCTTYGARGAYCGCAA
OMPD obv flifedrkf
61.5
21
GGHGTBTKGTACTGCTGNCCW
OMPD obv gqqyxtx
60.6
21
Samenstelling PCR reactie-mix
Component
Taq DNA polymerase (5U/µL)
10X Taq pol. Buffer (+Mg)
10mM dNTP mix
Forward primer (10 pmol/µL)
Reverse primer (10 pmol/µL)
DNA template:
- genomisch DNA (187 ng/µL )
milli-Q water
Totaal
PCR programma (Hot Start)
Cycli
1x
40x
1x
Eindconcentratie
2U
1X
0.2 mM
1 µM
1 µM
Volume
0.4 µL
5 µL
1 µL
5 µL
5 µL
187 ng
-
1 µL
32.6 µL
50 µL
Tijd
4 min.
30 s
1 min.
2 min.
7 min.
Temperatuur (°C)
94
94
50
72
72
Omdat de bekomen PCR fragmenten een lage concentratie hadden werd besloten een
reamplificatie uit te voeren op dit PCR mengsel. Het protocol van de reamplificatie was
hetzelfde als voor de gedegenereerde PCR (zie Tabel 32), behalve dat de
annealingstemperatuur werd verhoogd tot 53°C en het aantal cycli verminderd tot 30. Dit om
de smeer te verminderen. Er werd 2 µL van het PCR mengsel gebruikt als template.
Het resultaat van de reamplificatie is weergegeven in Figuur 14 (laan 2). Er werden meer en
duidelijkere bandjes bekomen, waaronder een duidelijk van 400 bp. Om de specificiteit te
verbeteren en eventueel de concentratie van de bandjes op 700 bp en 400 bp te verhogen werd
een reamplificatie uitgevoerd op het reamplificatiemengsel. Er werd hetzelfde PCR protocol
gebruikt als bij de eerste reamplificatie, maar in de annealingsstap werd de temperatuur
verhoogd tot 55°C en de tijdsduur verkort tot 45 s. Er werden twee PCR reacties uitgevoerd
volgens dit protocol maar bij één ervan werd slechts 1.5 U Taq polymerase gebruikt om de
smeer te verminderen. Er werd 2 µL van het reamplificatiemengsel gebruikt als template. Het
resultaat van deze reamplificatie is eveneens weergegeven in Figuur 14 (laan 3 en 4).
IV. Resultaten en Bespreking - 74
MANU DE GROEVE
1
2
3
4
Laan 1: Gedegenereerde PCR op OMPD
M
Laan 2: Reamplificatie van gedegenereerde
PCR op OMPD
Laan 3: Reamplificatie van vorig
reamplificatiemengsel (laan 2) (2 U Taq)
- 0.7 kb
Laan 4: Idem laan 3 maar met 1.5 U Taq
polymerase
- 0.4 kb
- 0.2 kb
Laan M: lengtemerker (SmartLadder)
Figuur 14. Reamplificaties van de gedegenereerde PCR op OMPD
Er werden geen nieuwe fragmenten geamplificeerd, maar sommige van de eerder
waargenomen fragmenten werden duidelijker terwijl andere minder duidelijk werden. Er
werden uiteindelijk vier bandjes geobserveerd van ongeveer 200 bp, 400 bp, 640 bp en 700
bp. De fragmenten van 400 bp en 700 bp werden reeds in vorige PCR reacties geobserveerd
en kwamen bovendien overeen met de verwachte grootte.
Omdat er echter nog steeds veel primer-dimeren zichtbaar waren op gel en er relatief veel
smeer werd bekomen werden de gedegenereerde primers geoptimaliseerd. Bij de forward
primer (‘OMPD obv flifedrkf’) werden de twee adenines (A) op het einde van de 3’ kant
verwijderd zodat deze nu eindigt op een cytosine (C). Dit verbetert theoretisch de binding met
de template en het polymerase, wat resulteert in betere amplificatie. Bij de reverse primer
(‘OMPD obv gqqyxtx’) werd de W (=A of T) op het einde (3’ kant) verwijderd zodat de
primer nu eindigt op een C, wat een exacte match is en bovendien de amplificatie bevordert
door een sterkere binding. Om de degeneratie te verminderen werd een B (=C, G of T)
vervangen door een S (=G of C) en werd een N (=A, G, C, of T) vervangen door een H (=A,
C of T). Hierdoor werd de degeneratie verminderd van 144 tot 36. In Tabel 33 worden de
geoptimaliseerde gedegenereerde primers (‘nieuw’) vergeleken met de eerst gebruikte
gedegenereerde primers (‘oud’).
Tabel 33. Vergelijking eerst gebruikte gedegenereerde primers met de geoptimaliseerde primers
Richting
Naam
Sequentie (5’-> 3’)
Lengte Degeneratie Tm (°C)
CCTSATCTTYGARGAYCGCAA
Forward Oud
OMPD obv flifedrkf
21
16
61.5
Nieuw OMPD obv flifedrkf2 CCTSATCTTYGARGAYCGC
19
16
57.2
GGHGTBTKGTACTGCTGNCCW
Reverse Oud
OMPD obv gqqyxtx
21
144
60.6
Nieuw OMPD obv gqqyxtx2 GGHGTSTKGTACTGCTGHCC
20
36
59.6
Met deze geoptimaliseerde primers werd een nieuwe gedegenereerde PCR uitgevoerd op 187
ng genomisch DNA van Myrothecium gramineum. Het PCR protocol is identiek aan de reeds
eerder gelukte gedegenereerde PCR (zie Tabel 32), behalve dat als forward primer ‘OMPD
IV. Resultaten en Bespreking - 75
MANU DE GROEVE
obv flifedrkf2’ en als reverse primer ‘OMPD obv gqqyxtx2’ gebruikt werden. Het resultaat is
weergegeven in Figuur 15. Er werden drie duidelijke PCR producten bekomen, namelijk een
fragment van 900 bp en de reeds eerder bekomen fragmenten van 400 bp en 700 bp. Er
werden geen primer-dimeren of smeer waargenomen op gel. Deze waarnemingen tonen
nogmaals het belang aan van primerdesign en optimalisatie van het PCR-mengsel voor
gedegenereerde PCR.
1
M
Laan 1: Gedegenereerde PCR op OMPD met
geoptimaliseerde primers
Laan M: lengtemerker (SmartLadder)
- 0.9 kb
- 0.7 kb
- 0.4 kb
Figuur 15. Gedegenereerde PCR op OMPD met geoptimaliseerde primers
1.2.2. Sequenering van de PCR fragmenten
Het PCR reactiemengsel werd gebruikt voor een preparatieve gelelektroforese (1.4%
agarosegel, 110 V) waarna de fragmenten van 400 bp, 700 bp en 900 bp geïsoleerd werden uit
gel met de QIAEX ΙΙ Gel Extraction Kit (Qiagen). De geïsoleerde fragmenten werden
gecontroleerd op gel.
Om de fragmenten van 400 bp, 700 bp en 900 bp te sequeneren werden deze eerst geligeerd in
pGEM®-T plasmiden. De ligatie gebeurde overnacht bij 4°C om een zo hoog mogelijk aantal
transformanten te bekomen. Na het stopzetten van de ligatiereacties door incubatie bij 65°C
gedurende 10 minuten, werden de ligatiemengsels gebruikt voor de transformatie van E. coli
DH5α-F’. De transformatie werd uitgevoerd zoals eerder beschreven en er werd 300 µL van
de getransformeerde E. coli cellen uitgeplaat op LB medium met X-gal en ampicilline. De
platen werden overnacht geïncubeerd bij 37°C.
Het screenen naar de kolonies die de gewenste plasmiden (pGEM®-T + insert) opgenomen
hebben gebeurde via wit-/blauwkleuring. Alle E. coli’s die op de plaat kunnen groeien hebben
het pGEM®-T plasmide (al dan niet met insert) opgenomen, aangezien de gebruikte E. coli
DH5α-F’ stam van nature niet resistent is aan ampicilline. Kolonies die het plasmide zonder
insert (PCR fragment) opgenomen hebben, kleuren blauw omdat het LacZ gen niet
IV. Resultaten en Bespreking - 76
MANU DE GROEVE
onderbroken is en het β-galactosidase enzym dus nog functioneel is. De witte kolonies
daarentegen bevatten het plasmide met insert en werden gebruikt worden om de gewenste
plasmiden uit te isoleren. Er werden 4 witte kolonies opgepikt van pGEM®-T + 400 bp, 3 van
pGEM®-T + 700 bp en 2 van pGEM®-T + 900 bp. Deze kolonies werden overgeënt naar 5 mL
LB + ampicilline en schuddend (200 RPM) geïncubeerd bij 37°C tot de O.D. hoog genoeg
was voor plasmiden isolatie. De plasmiden werden uit de E. coli’s geïsoleerd met de
NucleoSpin® Plasmid Kit (BD Biosciences).
Om te controleren of de geïsoleerde plasmiden wel degelijk het PCR fragment bevatten werd
een analytische restrictie uitgevoerd. Er werd geknipt met NcoI en NdeI. Deze restrictieenzymen zijn single cutters in pGEM®-T en knippen het insert uit de vector. Na
gelelektroforese verwacht men dan ook een bandje ter hoogte van de grootte van het insert
(hier 400 bp, 700 bp of 900 bp) en een bandje op 3000bp (pGEM®-T).
Op basis van de restrictiedigest en de concentratie van de plasmiden werd besloten welke
plasmiden werden gesequeneerd. Er werd van elk fragment één plasmide opgestuurd naar het
Vlaams Interuniversitair Instituut voor Biotechnologie (VIB, Genetic Service Facility,
Antwerpen, België). Zij maken gebruik van de Applied Biosystems 3730 DNA Analyzer en
beschikken over de T7 en SP6 primers waarmee het insert in het pGEM®-T plasmide kan
gesequeneerd worden.
De sequeneringen van het 900 bp fragment en het 700 bp fragment waren niet gelukt. De
sequenering van het 400 bp fragment was echter wel gelukt. Omdat er gesequeneerd werd met
de T7 en SP6 primers bevatte de sequentie nog een deel van het pGEM®-T plasmide. Via
Clone Manager werden de plasmidensequenties verwijderd en werd de sequentie verder
onderzocht. Het geïsoleerde PCR fragment bleek 441 bp lang te zijn en de gedegenereerde
primers werden teruggevonden aan de uiteinden van dit fragment. De nucleotidensequentie
van dit 441 bp fragment is weergegeven in Figuur 16.
1
61
121
181
241
301
361
421
CCTGATCTTT
TGGTGGTGCC
TGATGGCGTT
TCAACGGGCC
ATACACAGAG
CTTCGTTGCC
GGACGAAGAC
GGGGCAGCAG
GAGGACCGCA
CTGCGCATCT
GTGAATGCAC
CTGCTTCTGC
CGCTGCGTCG
ACGCGAGGCC
TTTGTCGTTT
TACCACACAC
AGTTTGTGGA
CCGAGTGGGC
TGGGCCAGGT
TTGCTGAGAT
CTGTCGCACG
TCGAAGACGT
TTACTACTGG
C
TATTGGCTCA
CGACATTGTC
CATTACCGAC
GACCACGGCC
AAAGAATGGC
TGGCAACGAA
TATCAACAGC
ACAGCCCAGA
AACGTCAGTC
GAGACGTTTC
GGTAGCCTTG
ATTGCCGCTG
CCTGGGGTCG
AGCCAATCTG
AACAGTACCA
TCCTAGGGGG
CCTATCGCGG
CCGTTGAAAA
GAGTCATGGG
AGAGAGACGA
ACAATGTGCT
* De bindingsplaatsen van de gedegenereerde primers zijn onderlijnd en in vet aangeduid.
Figuur 16. Nucleotidensequentie (441 bp) van een deel van het OMPD gen van Myrothecium gramineum,
bekomen via gedegenereerde PCR
MANU DE GROEVE
IV. Resultaten en Bespreking - 77
Deze nucleotidensequentie werd vervolgens vertaald naar de aminozuursequentie in alle zes
leesramen. Eén van deze leesramen was het correcte en er werd een aminozuursequentie van
146 aminozuren verkregen. Deze sequentie is weergegeven in Figuur 17.
1 LIFEDRKFVD IGSTAQKQYH GGALRISEWA DIVNVSLLGG DGVVNALGQV ITDETFPYRG
61 QRALLLLAEM TTAGSLAVEK YTERCVAVAR KNGIAAGVMG FVATRGLEDV GNEPGVERDE
121 DEDFVVFTTG INSSQSDNVL GQQYHT
Figuur 17. Aminozuursequentie (146 AZ) van een deel van het OMPD enzym van Myrothecium gramineum.
Vervolgens werden de nucleotiden- en aminozuursequenties geblast (WU-BLAST2,
standaard parameters) tegenover respectievelijk de EMBL en de UNIPROT database. Op
deze manier kan men zien met welk gen of enzym van welke organisme het stuk OMPD het
meest verwant is. Hieruit bleek dat de nucleotidensequentie het meest similariteit vertoont
met het pyrG gen van Aspergillus oryzae (64%) en de aminozuursequentie het meest
similariteit vertoont met het OMPD enzym van Aspergillus fumigatus (58%).
Rekening houdend met bovenvermelde argumenten kon dus met grote zekerheid gesteld
worden dat via gedegenereerde PCR een 441 bp fragment van het OMPD gen van
Myrothecium gramineum werd bekomen. Oorspronkelijk werd echter een fragment van 700750 bp verwacht (zie Tabel 28, p.67). Dit wijst er op dat het OMPD gen van M. gramineum
meer gelijkt op dit van niet-Sordariomyceten. Dit is vrij verrassend aangezien M. gramineum
zelf tot de klasse van de Sordariomyceten behoort.
In een volgende stap kon de sequentie van dit 441 bp fragment uitgebreid worden naar de 5’
en de 3’ kant via genomewalking. Op deze manier werd getracht het volledige OMPD gen te
kloneren. Om het OMPD gen als selectiemerker te kunnen gebruiken moet dit immers
functioneel zijn en tot expressie kunnen komen in een OMPD-negatieve mutant.
1.3. GenomeWalking naar 5’ en 3’ kant van het OMPD gen
1.3.1. Algemeen principe
Eenmaal de sequentie van een deel van een gen gekend is kunnen de aangrenzende sequenties
gevonden worden via genomewalking. Hier werd gebruik gemaakt van de BD
GenomeWalker™ Universal Kit (BD Biosciences). Het principe is weergegeven in Figuur 18.
IV. Resultaten en Bespreking - 78
MANU DE GROEVE
Genomisch DNA
Knip verschillende stalen DNA met restrictie-enzymen
Ligeer met GenomeWalker Adaptor
BD GenomeWalker banken
DraI
Genomisch
DNA fragment
ongekend
gekend
5'N
AP1
AP2
EcoRV
PvuII
StuI
Amplificeer gewenste gen bij alle vier de banken
GSP2
GSP1
N
AP1
GSP1
Primary PCR
AP2
GSP2
Secondary (geneste) PCR
-5'
BD GenomeWalker
Adaptor
Controleer PCR producten via gelelektroforese
Kloneer PCR fragmenten
Figuur 18. Principe van de BD GenomeWalker™ Universal Kit (BD Biosciences)
In een eerste stap worden banken gemaakt van het genomisch DNA. Daarvoor wordt het
genomisch DNA geknipt met een restrictie-enzym dat blunt eindjes geeft. In de kit worden
vier verschillende restrictie-enzymen meegeleverd: DraI, EcoRV, PvuII en StuI. Hiermee
kunnen dus vier verschillende banken gemaakt worden. Na de restrictie wordt het
restrictiemengsel gezuiverd en gebruikt voor adaptorligatie. Tijdens de ligatiestap worden de
BD GenomeWalker Adaptors met gekende sequentie geligeerd aan beide uiteinden van de
restrictiefragmenten. Na deze stap zijn de banken klaar om gebruikt te worden voor de
eigenlijke genomewalking.
Er worden twee PCR’s uitgevoerd per bank. Per PCR wordt gebruik gemaakt van een adaptor
primer en een genspecifieke primer. De adaptor primers worden meegeleverd in de kit, de
genspecifieke primers moeten zelf ontworpen worden op basis van het gekende stuk gen. In
de eerste (primary) PCR reactie wordt gebruik gemaakt van de outer adaptor primer (AP1) en
een outer genspecifieke primer (GSP1). Het primary PCR mengsel wordt vervolgens gebruikt
als template voor een secondary of nested PCR. Hierbij worden de nested adaptor primer
(AP2) en een geneste genspecifieke primer gebruikt (GSP2). In het ideale geval wordt na deze
stap een duidelijk PCR product waargenomen op gel dat kan gebruikt worden voor klonering
en sequenering.
IV. Resultaten en Bespreking - 79
MANU DE GROEVE
1.3.2. De BD GenomeWalker Adaptor en het suppressie PCR effect
De BD GenomeWalker Adaptor heeft drie eigenschappen die cruciaal zijn voor een
succesvolle genomewalking:
(1) Het gebruik van een 5’-geëlongeerde adaptor die geen bindingsplaats heeft voor de AP1
primer in primary PCR. De AP1 bindingsplaats kan enkel gegenereerd worden door elongatie
van de genspecifieke primer.
(2) Door het blokkeren van het ‘blootgestelde’ 3’ einde van de adaptor met een aminegroep
wordt elongatie van het 3’ einde voorkomen (anders wordt een AP1 bindingsplaats gecreëerd)
(3) Het gebruik van een adaptor primer die korter is dan de adaptor zelf = suppressie PCR.
Zoals weergegeven in Figuur 19, voorkomt het suppressie PCR effect de amplificatie van
templates waarvan het 3’ einde toch werd geëlongeerd en er zo een AP1 bindingsplaats werd
gecreëerd. Hoewel dit eerder uitzonderlijk is, wordt dit vermoedelijk veroorzaakt door
onvolledige amine-modificatie of onvolledige adaptorligatie. Gezien het exponentiële
karakter van PCR amplificatie zou dit in afwezigheid van het suppressie effect leiden tot nietspecifieke amplificatie en onaanvaardbare achtergrond.
Uitzonderlijk kan het 3' einde van de adaptor
geëlongeerd worden waardoor een template ontstaat
met aan beide uiteinden de volledige adaptor sequentie
OF
Denaturatie bij 95°C
Suppressie PCR
Annealing bij 68°C
AP1
DNA synthese
Geen primerbinding:
hairpin structuur zorgt voor
suppressie van PCR
Figuur 19. Het suppressie PCR effect
Elke van deze eigenschappen elimineert de niet-specifieke amplificatie van DNA fragmenten.
In combinatie met touchdown PCR en geneste PCR laat dit de amplificatie toe van een
specifiek DNA fragment uit een complex mengsel. Van de drie eigenschappen is het
suppressie PCR effect het meest cruciaal.
IV. Resultaten en Bespreking - 80
MANU DE GROEVE
1.3.3. Controle kwaliteit genomisch DNA
Genomisch DNA werd geïsoleerd uit mycelium van M. gramineum met de Dneasy Plant
MAXI kit zoals eerder beschreven. De concentratie werd bepaald via de spectrofotometer.
Het geïsoleerde genomisch DNA werd bekeken op gel. Het bandje van het genomisch DNA
lag hoog (> 10 kb) en vertoonde zeer weinig smeer. Er werd een restrictie uitgevoerd op het
genomisch DNA met DraI en het resultaat werd bekeken op gel. Er was een smeer te zien
over de gehele laan wat er op wijst dat het genomisch DNA volledig verknipt was.
Vervolgens werd het restrictiemengsel gezuiverd zoals beschreven in de kit. Na controle op
gel werd nog steeds een smeer waargenomen. Er was dus weinig DNA verloren gegaan in de
zuiveringsprocedure. Tenslotte werd een PCR (standaard protocol) uitgevoerd op een gekende
sequentie van het GPD gen van M. gramineum (Jonniaux et al., 2004) met het gezuiverde
restrictiemengsel als template. Er werd zoals verwacht een fragment van 850 bp bekomen.
Omdat het geïsoleerde genomisch DNA van M. gramineum weinig smeer vertoonde, geknipt
kon worden en geschikt was als template voor PCR, kon besloten worden dat het DNA van
voldoende kwaliteit was voor gebruik met de BD GenomeWalker Universal Kit.
1.3.4. Constructie BD GenomeWalker banken
De constructie van de banken gebeurde volgens het protocol van de kit. In eerste instantie
werd enkel een DraI bank geconstrueerd. De EcoRV, PvuII en StuI bank werden later op
dezelfde wijze als de DraI bank geconstrueerd.
2.5 µg genomisch DNA van M. gramineum en humaan genomisch DNA (meegeleverd bij de
kit ter controle) werd geknipt met DraI volgens het protocol van de kit. 5 µL van elk 100 µL
restrictiemengsel werd gebruikt voor gelelektroforese (0.5% agarose, 110 V). Zoals blijkt uit
Figuur 20 werd zowel het genomisch DNA van M. gramineum als het humaan DNA volledig
verknipt. Er is immers een heldere smeer te zien in beide lanen.
1
2 M
Laan 1: DraI geknipt genomisch DNA
M. gramineum
Laan 2: DraI geknipt genomisch
humaan DNA
Laan M: lengtemerker (SmartLadder)
Figuur 20. Resultaat restrictie van genomisch DNA
MANU DE GROEVE
IV. Resultaten en Bespreking - 81
Vervolgens werden beide restrictiemengsels gezuiverd zoals beschreven in de kit. Na controle
op gel bleek dat er weinig DNA verloren was gegaan. Het gezuiverde restrictiemengsel kon
dus gebruikt worden voor adaptorligatie.
De ligatie van de BD GenomeWalker Adaptors aan de DraI-geknipte genomische DNA
fragmenten werd uitgevoerd zoals beschreven in de kit. Het ligatie-reactiemengsel werd
overnacht (16 uur) geïncubeerd bij 16°C in een PCR toestel (Techne). De ligatiereactie werd
stopgezet door 5 minuten te incuberen bij 70°C in hetzelfde PCR toestel.
1.3.5. Ontwikkeling OMPD-genspecifieke primers voor genomewalking
Op basis van de OMPD sequentie die bekomen werd via gedegenereerde PCR (zie hoger)
werden genspecifieke primers ontworpen. De genspecifieke primers werden ontworpen met
behulp van het softwareprogramma Clone Manager Professional Suite 6.0 (Scientific &
Educational Software) en voldoen zo veel mogelijk aan de eisen beschreven in de
GenomeWalker kit. In eerste instantie wordt aangeraden de outer en geneste primer niet te
laten overlappen. Daarnaast moeten de primers bij voorkeur 26-30 nucleotiden lang zijn met
een GC gehalte van 40-60%. Dit verzekert een efficiënte annealing met de template bij de
aanbevolen annealing- en elongatietemperatuur van 67°C. Verder mogen de primers geen
intramoleculaire waterstofbruggen kunnen vormen en geen homologie vertonen met de
adaptor primers. Ook wordt aangeraden geen primers te gebruiken met meer dan drie G’s en
C’s in de laatste zes posities aan de 3’ kant van de primer.
Er werden twee primers ontwikkeld om naar de upstream (5’) regio te wandelen, en twee
primers voor de downstream (3’) regio. De sequenties van de primers zijn weergegeven in
Tabel 34. De bindingsplaats van de primers op de reeds gekende OMPD sequentie is
schematisch weergegeven in Figuur 21.
Tabel 34. Gebruikte primers voor de genomewalking naar de 5’ en 3’ kant van het OMPD gen van M. gramineum
Richting
Naam
Lengte (nt) GC%
Tm (°C)
Sequentie (5’→ 3’)
CGATAGGGAAACGTCTCGTCGGTAATG
27
51
72.8
5’ OMPD 1 OMPD
GGTACTGTTTCTGGGCTGTTGAGCCAAT
nested 1 OMPD
28
50
73.4
CCCTGCTTCTGCTTGCTGAGATGAC
3’ OMPD 3 1 OMPD
25
56
72.8
CTGGTATCAACAGCAGCCAATCTGAC
3 1 nested OMPD
26
50
70.3
Adaptor
AP1
22
45
59.0
GTAATACGACTCACTATAGGGC
AP2
19
57
71.0
ACTATAGGGCACGCGTGGT
IV. Resultaten en Bespreking - 82
MANU DE GROEVE
bindingsplaats primer voor
genomewalking
nested 1 OMPD
1 OMPD 3 1 OMPD
100
200
3 1 nested OMPD
StuI 316
300
400
UPSTREAM
(5' kant)
DOWNSTREAM
(3' kant)
via gedegenereerde PCR verkregen fragment (441 bp)
van het OMPD gen van M. gramineum
Figuur 21. Bindingsplaatsen van de primers voor de genomewalking op het via gedegenereerde PCR verkregen
fragment van het OMPD gen van M. gramineum
Een overzicht van de positie van het reeds gekende 441 bp fragment in de andere OMPDenzymen en de totale lengte van deze enzymen worden gegeven in Tabel 35. Voor de DNA
sequentie werd enkel rekening gehouden met de exons en de introns.
Tabel 35. Overzicht lengte meest verwante OMPD-enzymen
Organisme
Totale lengte OMPD
Aminozuren bp (exons en introns)
Aspergillus nidulans
274
883
Aspergillus fumigatus
278
904
Aspergillus oryzae
277
898
Aspergillus niger
277
901
Cladosporium fulvum
278
897
Penicillium chrysogenum
278
882
Trichoderma harzanium
379
1139
Neurospora crassa
397
1193
Nog te wandelen
5’ AZ
3’ AZ
89
39
89
43
89
42
89
42
89
43
92
40
93
144
100
133
Uit de tabel kan afgeleid worden dat er nog een honderdtal aminozuren (300 bp) naar het 5’
einde moet gewandeld worden en 40 (eventueel 140) aminozuren naar het 3’ einde (120/420
bp). Men moet er wel rekening mee houden dat de promotor hier nog niet bijzit, net als de 3’
onvertaalde sequenties.
1.3.6. GenomeWalking naar de 5’ en 3’ kant van OMPD
DraI bank
Voor de genomewalking naar de 5’ en 3’ kant van het OMPD gen werd in eerste instantie
gebruik gemaakt van de DraI bank. Als genspecifieke primers werden “1 OMPD” en “nested
1 OMPD” gebruikt voor de upstream (5’ kant) walking, en “3 1 OMPD” en “3 1 nested
OMPD” voor de downstream (3’ kant) walking. Er werd gebruik gemaakt van het
geoptimaliseerd primary PCR protocol (Materialen en Methoden, Tabel 19, p.53). Voor de
IV. Resultaten en Bespreking - 83
MANU DE GROEVE
walking naar de 5’ kant werd een zeer helder bandje bekomen van 2000 bp en twee zwakkere
bij 4000 bp en 1650 bp. Voor de walking naar de 3’ kant werd enkel een smeer waargenomen
en een zeer zwak bandje van 700 bp.
Vervolgens werden nested PCR’s uitgevoerd op de primary PCR mengsels. Er werd gebruik
gemaakt van het geoptimaliseerde secondary PCR protocol (Materialen en Methoden, Tabel
20, p.54). Voor de 5’ kant werd zowel een 1/50ste verdunning als een onverdund staal van het
primary PCR mengsel gebruikt. Voor de 3’ kant werd onverdund primary PCR mengsel
gebruikt. Hoewel volgens het protocol van de kit steeds een 1/50ste verdunning van het
primary PCR mengsel moet gebruikt worden in de nested PCR, werd dit hier enkel gedaan bij
reacties die in de eerste PCR een zeer helder bandje opleverden. Het resultaat van de nested
PCR’s is weergegeven in Figuur 22.
1
2
3
M
- 4.0 kb
- 1.9 kb
- 1.5 kb
Laan 1: UPSTREAM (5’) - Nested PCR op 1/50ste
verdunning van primary PCR mengsel
Laan 2: UPSTREAM (5’) - Nested PCR op onverdund
primary PCR mengsel
Laan 3: DONWSTREAM (3’) - Nested PCR op onverdund
primary PCR mengsel
Laan M: lengtemerker (SmartLadder)
- 0.6 kb
Figuur 22. GenomeWalking DraI bank: nested PCR’s naar 5’ en 3’ kant van OMPD
Voor de walking naar de upstream kant van OMPD werd een zeer helder bandje van 1.9 kb
bekomen op gel. Het PCR fragment was zoals verwacht ongeveer 100 bp kleiner dan dat bij
de primary PCR. Er was zo goed als geen verschil tussen de nested PCR op onverdund en
verdund staal. Er werden opnieuw zwakkere bandjes bekomen van 4 kb en 1.5 kb.
Voor de genomewalking naar de downstream kant van OMPD werd opnieuw een smeer
waargenomen en een zeer zwak bandje op ongeveer 610 bp. Er werd een reamplificatie
uitgevoerd op dit PCR mengsel om de concentratie van het fragment te verhogen volgens het
standaard reamplificatie protocol. Dit leverde echter geen verbetering op.
Er werd besloten een preparatieve gelelektroforese uit te voeren om volgende PCR
fragmenten te isoleren uit gel:
MANU DE GROEVE
IV. Resultaten en Bespreking - 84
•
het fragment van 1.9 kb bekomen bij de upstream genomewalking (met verdund
primary PCR-mengsel als template)
• het fragment van 610 bp bekomen bij de downstream genomewalking
De geïsoleerde fragmenten van 1.9 kb (5’ OMPD) en 610 bp (3’ OMPD) werden geligeerd in
pGEM®-T, waarna E. coli getransformeerd werd met het ligatiemengsel. De E. coli’s werden
uitgeplaat op LB+X-gal+ampicilline en overnacht geïncubeerd bij 37°C. Er werden twee
witte kolonies van het 1.9 kb fragment en drie witte kolonies van het 610 bp fragment
overgeënt naar 5 mL LB+ampicilline en geïncubeerd. Van de E. coli culturen die voldoende
dens gegroeid waren werden plasmiden geïsoleerd. De plasmiden van de E. coli’s met
pGEM®-T+1.9 kb en van pGEM®-T+610 bp werden geïsoleerd met de Nucleospin QuickPure
kit. De geïsoleerde plasmiden werden gecontroleerd via PCR met de T7 en SP6 primers die
het insert in het plasmide amplificeren. Op basis hiervan werd besloten welke plasmiden
werden opgestuurd naar het VIB voor sequenering. Omdat het 1.9 kb fragment groter is dan
de sequeneringslimiet (800-1000 bp) werd zowel met de T7 als de SP6 primer gesequeneerd.
De sequenering van het 1.9 kb fragment van de upstream regio van OMPD was volledig
gelukt. Na verwijderen van de plasmide- en adaptorsequenties bleek het om een fragment van
1894 bp te gaan. Deze van het 610 bp fragment van de downstream regio was vroegtijdig
afgebroken, maar toch werd nog een 326 bp fragment bekomen (met de plasmidensequenties
reeds verwijderd). Zowel bij het 1896 bp fragment als bij het 326 bp fragment werd een
overlap met de reeds gekende sequentie van OMPD gevonden. Ook werden bij het 1894 bp
fragment de adaptorsequenties en de DraI knippplaats teruggevonden. De sequenties bleken
dus te kloppen.
Vervolgens werden de via genomewalking verkregen sequenties samengevoegd met de reeds
gekende 441 bp sequentie van OMPD. Na analyse bleek dat de totale gekende sequentie van
OMPD uitgebreid was tot 2549 bp. Deze sequentie bestond uit 1478 bp van de promotor
regio, 908 bp van startcodon (ATG) tot stopcodon, en 163 bp van de 3’ onvertaalde regio (3’
UTR). Er werd dus een groot stuk promotor en de volledige coderende sequentie van het
OMPD gen van M. gramineum gesequeneerd. De 163 bp sequentie van de 3’ UTR was echter
vrij kort en bevat bovendien geen polyA-signaal of transcriptie terminatiesignaal. Deze
signalen kunnen belangrijk zijn voor goede expressie.
Daarom werd besloten opnieuw naar de 3’ kant van OMPD te wandelen met dezelfde primers
maar met een andere bank. Er kon echter geen gebruik gemaakt worden van de StuI bank
omdat er zich een StuI knipplaats bevindt tussen de bindingsplaatsen van de primary en de
nested primer. Er was dus nog keuze uit de EcoRV of PvuII bank. Omdat er zich geen EcoRV
en PvuII restrictieplaatsen bevinden tussen de bindingsplaats van de nested primer en het
einde van de reeds gekende sequentie was de verwachte grootte van de fragmenten groter dan
360 bp na de nested PCR.
IV. Resultaten en Bespreking - 85
MANU DE GROEVE
EcoRV bank
Na de DraI bank werd de EcoRV bank gebruikt voor de walking naar het 3’ einde van
OMPD. Er werd gebruik gemaakt van hetzelfde genomewalking protocol als bij de DraI bank
en ook de genspecifieke primers voor de downstream walking waren dezelfde (‘3 1 OMPD’
voor de primary en ‘3 1 nested OMPD’ voor de nested PCR).
De primary PCR werd uitgevoerd zoals eerder beschreven en de producten werden
gecontroleerd op gel. Er werden geen PCR fragmenten waargenomen. Volgens de
handleiding van de kit betekent dit echter niet dat de PCR mislukt is. Het kan immers
gebeuren dat er bij de nested PCR nieuwe fragmenten tevoorschijn komen en andere
verdwijnen.
Er werd een nested PCR uitgevoerd op het onverdund primary reactiemengsel zoals eerder
beschreven. Het resultaat van de gelelektroforese van het nested PCR mengsel is
weergegeven in Figuur 23. Er werden twee vrij duidelijke bandjes waargenomen van
ongeveer 400 bp en 390 bp.
1
M
Laan 1: DONWSTREAM (3’) - Nested PCR op onverdund
primary PCR mengsel
Laan M: lengtemerker (SmartLadder)
- 0.4 kb
- 0.39 kb
Figuur 23. GenomeWalking EcoRV bank: nested PCR naar 3’ kant van OMPD
De verkregen fragmenten waren groter dan de verwachte minimumgrootte (> 360 bp). Omdat
het echter relatief korte fragmenten zijn - die de reeds gekende sequentie met maximaal 40
bp zouden kunnen uitbreiden - werd besloten om opnieuw te wandelen naar de 3’ kant met
een andere bank. Er werd gebruik gemaakt van de PvuII bank.
PvuII bank
Er werd opnieuw naar de 3’ kant van OMPD gewandeld volgens de eerder beschreven
protocols en met dezelfde primers, maar nu met de PvuII bank. Omdat er zich geen PvuII
restrictieplaatsen bevonden in de reeds gekende sequentie waren de verwachte groottes van de
PCR fragmenten dezelfde als bij de EcoRV bank (> 360 bp).
Er werd een primary PCR uitgevoerd met de ‘3 1 OMPD’ genspecifieke primer en de AP1
adaptor primer. Na gelelektroforese kon een duidelijk PCR fragment van ongeveer 1000 bp en
IV. Resultaten en Bespreking - 86
MANU DE GROEVE
een minder duidelijk fragment van 900 bp waargenomen worden. Het resultaat is
weergegeven in Figuur 24.
Op het onverdund primary mengsel werd vervolgens een nested PCR uitgevoerd volgens de
eerder beschreven protocols. Als primers werden de ‘3 1 nested OMPD’ genspecifieke primer
en de AP2 adaptor primer gebruikt. Het resultaat van de gelelektroforese van het nested PCR
mengsel is weergegeven in Figuur 24.
M’
1
2
M
Laan M’: kleine lengtemerker (SmartLadder SF)
Laan 1: Primary PCR op OMPD
Laan 2: Nested PCR op onverdund primary PCR mengsel
Laan M: lengtemerker (SmartLadder)
- 1.0 kb
- 0.8 kb
Figuur 24. GenomeWalking PvuII bank: primary en nested PCR naar 3’ kant van OMPD
Er werd opnieuw een duidelijk PCR fragment waargenomen op gel. Het PCR fragment was
ongeveer 800 bp groot en komt overeen met de verwachte grootte. De bindingsplaatsen van
de twee genspecifieke primers zijn immers ongeveer 200 bp van elkaar verwijderd. Het 1000
bp fragment van de primary PCR en het 800 bp fragment van de nested PCR zijn dus
waarschijnlijk afkomstig van het OMPD gen. Bij de nested PCR werd verder ook nog een
bandje op ongeveer 1000 bp waargenomen. Vermoedelijk is dit afkomstig van het primary
PCR mengsel omdat er onverdund staal werd gebruikt voor de nested PCR.
Het 800 bp fragment van de nested PCR werd gesequeneerd volgens de reeds eerder vermelde
procedures. De sequenering was gelukt en er werd een overlap gevonden tussen de gekende
sequentie en de nieuwe sequentie. In de sequentie werden zoals verwacht ook nog de eerste
drie basenparen van de PvuII knipplaats (CAG^CTG) en de adaptorsequentie gevonden. Na
verwijderen van de plasmidensequentie en aanvullen met de laatste drie basenparen van de
PvuII knipplaats (CAG^CTG) bleek het om een fragment van 730 bp te gaan. Omdat er reeds
een deel van dit fragment gekend was werd de gekende sequentie van het OMPD gen
uitgebreid met 367 bp tot 2918 bp.
IV. Resultaten en Bespreking - 87
MANU DE GROEVE
1.4. Klonering en sequenering van het volledige OMPD gen
Op basis van de gekende sequentie (2918 bp) van het OMPD gen van Myrothecium
gramineum werden primers ontworpen om het volledige OMPD gen te amplificeren en
vervolgens te kloneren.
Er werden twee forward en twee reverse primers ontworpen waarmee het OMPD gen kan
geamplificeerd worden. De sequentie en de eigenschappen van deze primers zijn
weergegeven in Tabel 36. De bindingsplaatsen van de primers op het OMPD-gen zijn
schematisch weergegeven in Figuur 25.
Tabel 36. Gebruikte primers voor de amplificatie van het volledige OMPD van Myrothecium gramineum
Primers
Tm (°C)
Lengte (nt) Positie
Sequentie (5’→ 3’)
OMPD volledig
5 O volledig
3 O transcriptiestop
OMPD in place
Oip 5’
Oip 3’
CAGACCGCATGCATTCGTATGCC
ACCAAGCACCAACAC
74.1
52.5
23
15
849-871
2884-2898
AGCCAGCTTCTATGGCTTCC
CTGCGCTCAGTGAATCAACC
64.5
65.9
20
20
595-614
2896-2915
2050 bp
5 O volledig
2321 bp
Oip 5'
DraI
EcoRV
3 O trancriptiestop
Oip 3'
EcoRV
StuI
promotor (1478 bp)
500
1000
CDS (908 bp)
1500
2000
PvuII
3' UTR (530 bp)
2500
Figuur 25. Schematisch overzicht van de bindingsplaatsen van de gebruikte primers voor de amplificatie
van het OMPD gen van M. gramineum
De forward primer ‘5 O volledig’ en de reverse primer ‘3 O transcriptiestop’ werden gebruikt
om 2050 bp van het OMPD gen te amplificeren dat zal gebruikt worden voor klonering in
pGEM®-T. Het resulterende plasmide kan gebruikt worden voor de constructie van knock-out
vectoren voor de creatie van een gedefinieerde OMPD-negatieve mutant. Zulke mutanten
kunnen dan gecomplementeerd worden met het werkende wild type OMPD gen. De forward
primer ‘Oip 5’ en de reverse primer ‘Oip 3’ amplificeren een fragment van 2321 bp van het
OMPD gen.
De PCR werd uitgevoerd met de High Fidelity PCR Master kit om de kans op fouten in de
sequentie zo klein mogelijk te houden. Er werd gebruik gemaakt van het protocol zoals
beschreven in Materialen en Methoden (III.5.2, p.54). Het resultaat van de PCR is
weergegeven in Figuur 26.
IV. Resultaten en Bespreking - 88
MANU DE GROEVE
1
2
M
Laan 1: High Fidelity PCR op OMPD “volledig”
Laan 2: High Fidelity PCR op OMPD “in place”
2321 bp2050 bp-
Laan M: lengtemerker (SmartLadder)
820 bp-
Figuur 26. Amplificatie van OMPD gen met High Fidelity PCR
De verwachte PCR fragmenten werden bekomen. Bij OMPD ‘volledig’ werd een 2050 bp
fragment bekomen en bij OMPD ‘in place’ een fragment van 2321 bp. Het fragment van 2321
bp werd gezuiverd uit het ‘in place’ PCR reactiemengsel met de Bioline DNAce Quick-Clean
kit. Het fragment van 2050 bp werd geïsoleerd uit gel na een preparatieve gelelektroforese
omdat er nog een zwak bandje zichtbaar was op ongeveer 820 bp. Zowel het 2321 bp als het
2050 bp fragment werden gekloneerd in pGEM®-T. De ligatiereacties werden uitgevoerd bij
kamertemperatuur gedurende 1 uur. Na transformatie in E. coli en plasmidenisolatie werden
de plasmiden gecontroleerd via PCR waarna ze gesequeneerd werden met de T7 en SP6
primers door het VIB. Op basis van deze sequenties en de reeds gekende sequentie van het
OMPD gen werd de definitieve sequentie van OMPD opgesteld. Deze wordt besproken in een
volgend hoofdstuk.
Het plasmide dat werd gecreëerd door het ‘OMPD volledig’ PCR-fragment van 2050 bp te
ligeren in de pGEM®-T vector wordt pOV genoemd en is 5052 bp groot. Dit plasmide bevat
het OMPD gen (632 bp van de promotor; 908 bp van startcodon tot en met stopcodon; 510 bp
3’UTR), een ampicilline-resistentiegen (β-lactamase), een onderbroken lacZ gen, een
oorsprong van replicatie, een faag f1-oorsprong en de T7 en SP6 faagpromotoren.
Het plasmide is schematisch weergegeven in Figuur 27. Het pOV plasmide werd gebruikt
voor de transformatie en complementatie van de OMPD-negatieve mutant A. nidulans FGSC
A722 en voor de creatie van een OMPD knock-out vector voor M. gramineum (zie verder).
IV. Resultaten en Bespreking - 89
MANU DE GROEVE
OMPD gen (2050 bp) van
Myrothecium gramineum
faag f1
pOV
(5052 bp)
ampR
ori
Figuur 27. Schematische voorstelling van het pOV plasmide
1.5. Studie van de DNA- en eiwitsequentie van OMPD
De volledige gekende nucleotidensequentie van het OMPD gen van Myrothecium gramineum
MUCL 39210 werd geanalyseerd. De totale lengte van deze sequentie bedraagt 2918 bp
waarvan 2050 bp gekloneerd werden. Door de sequentie te vergelijken met de OMPD genen
van andere filamenteuze schimmels kon de nucleotidensequentie opgesplitst worden in drie
grote delen: de promotorregio (1480 bp), de sequentie van startcodon tot en met stopcodon
(908 bp) en de 3’ onvertaalde regio (530 bp). De coderende sequentie codeert voor een eiwit
van 282 aminozuren.
De nucleotidensequentie van het OMPD gen en de afgeleide aminozuursequentie zijn
weergegeven in Figuur 28 (p.92). In wat volgt zullen deze sequenties in detail besproken
worden en vergeleken worden met andere OMPD sequenties.
1.5.1. Nucleotidensequentie
Het GC-gehalte (=% G+C) van de promotorregio (47.7%) en de 3’ onvertaalde regio (48.5%)
is lager dan het GC-gehalte van de sequentie van start- tot en met stopcodon (54.1%).
1.5.1.1. Promotorregio
Bij hogere eukaryoten zijn twee elementen belangrijk voor de initiatie van transcriptie: de
CAAT box die 70 tot 90 nt (nucleotiden) upstream van de transcriptiestartplaats (tsp) ligt, en
de TATA box die op 20 tot 40 nt upstream ligt (Ballance, 1986). Onderzoek bij filamenteuze
schimmels heeft aangetoond dat sommige genen TATA-achtige sequenties bevatten op de
verwachte afstand, terwijl andere enkel een AT-rijke sequentie bevatten op 30-100 bp van de
tsp. Ook sequenties die lijken op de CAAT consensus werden reeds beschreven, maar zijn
vaak afwezig bij filamenteuze schimmels.
MANU DE GROEVE
IV. Resultaten en Bespreking - 90
In de promotorregio van het OMPD gen van M. gramineum werd een CCAAT sequentie
teruggevonden op -305 nt van het startcodon. Er werden ook twee TATA-sequenties gevonden
op respectievelijk -98 nt en -62 nt van het startcodon. De significantie van deze sequenties
voor het bepalen van de transcriptiestartplaats is niet duidelijk en vereist verder onderzoek
(bv. deletiestudies). In ieder geval is de afstand tussen de CCAAT en TATA sequenties relatief
groot.
Bij genen van filamenteuze schimmels wordt de tsp vooral bepaald door een pyrimidine (CT)
rijke sequentie die zich direct upstream van de tsp bevindt (Ballance, 1986). Bij het OMPD
gen van M. gramineum werd een pyrimidinerijke regio gevonden tussen -172 en -39 nt
(100/134=74.6%) (1309-1442). Vermoedelijk ligt de transcriptiestartplaats dus net
downstream van de regio. Het is echter moeilijk te bepalen waar de CT-rijke precies stopt.
Interessant is dat de vermoedelijke TATA-boxen zich in de pyrimidinerijke regio bevinden.In
deze regio werden enkele kleinere regio’s gevonden die zeer rijk zijn aan C en T:
•
CCCTCTCCCCTTTCGTTTTCTTCTCAACATCCTTGAATTATCCTACTCCCCTCTCTCACGCCT
op -172 tot -110 (1309-1371) (52/63=82.5%)
•
CTTTTCCCACCTCTCTATACCCTCTTCAATCCCCCACTT
op -77 tot -39 (1404-1442) (33/39=84.6%)
Bovendien ligt een van de twee TATA-boxen in het midden van een zeer rijke CT-regio
(33/39=84.6%). Dit kan er op wijzen dat dit een functionele TATA-box is en dat de tsp zich
net na deze pyrimidinerijke regio bevindt op -42 tot -39 nt van het startcodon en op ongeveer
20 nt downstream van de TATA-box. De exacte tsp kan bepaald worden via primer extensie
experimenten.
De afstand tussen de tsp en het startcodon varieert bij filamenteuze schimmels tussen 25 en
400 bp (Ballance, 1986). Voor het bepalen van de start van translatie (startcodon ATG) wordt
gebruik gemaakt van de Kozak-sequentie. Dit is een consensus voor de regio rond het
startcodon. In bijna alle eukaryotische genen wordt een purine teruggevonden op positie -3
van het startcodon. In een grote meerderheid van de gevallen is dit een A. Ook bij het OMPD
gen van M. gramineum werd een A op -3 gevonden.
1.5.1.2. Sequentie van start- tot en met stopcodon
De sequentie van start- tot en met stopcodon (TGA) is 908 bp lang en bevat één intron van 59
bp. De coderende sequentie (zonder intron en stopcodon) is 846 bp lang en bestaat uit twee
exons van respectievelijk 157 bp en 689 bp.
Genen van filamenteuze schimmels bevatten meestal korte introns (48-240 bp) (Ballance,
1986). Het intron in het OMPD gen van M. gramineum voldoet aan de consensus-sequenties
IV. Resultaten en Bespreking - 91
MANU DE GROEVE
voor de 5’ splice site, het splicing signaal (lariat formatie) en de 3’ splice site. De consensussequenties zijn weergegeven in Tabel 37.
Tabel 37. Consensus-sequenties voor introns van filamenteuze schimmels
Niet-Sordariomyceten
Sordariomyceten
g|GTA(A/C/T/)GT(C/T)
g|GTAAGT
5’ splice site
(A/G)CT(A/G)A(C/A)
Splicing signaal (T/A)(G/A)CT(A/G)AC
(A/C)(C/T)AG|g
(T/A/G)(T/A/G)(T/C)AG|
3’ splice site
Referenties
Ballance (1986)
Jekosch & Kück (1999)
Voor de 5’ splice site werd volgende sequentie gevonden: g|GTAGAGT. Zowat alle introns
van filamenteuze schimmels beginnen met GT. Het intron bevat een vermoedelijk splicing
signaal (lariat formatie) met als sequentie GCTAAC op -22 nt van de 3’ splice site. In S.
macrospora en N. crassa ligt het splicing signaal op respectievelijk 12 tot 22 nt en 14 tot 30
nt van de 3’ splice site. Als 3’ splice site werd TTTAG|a gevonden. Het intron voldoet dus
aan de consensus-sequenties. Het intron heeft de typische GT...AG structuur die bij zeer
veel eukaryotische introns wordt teruggevonden. Het GC-gehalte van het intron (45.8%) is
lager dan het GC-gehalte van het eerste exon (50.3%) en het laatste exon (55.7%).
De positie en lengte van het intron werden bevestigd door middel van een twee-staps reversetranscriptase PCR (zie IV.1.5.2, p.93).
1.5.1.3. 3’ onvertaalde regio
Genen van hogere eukaryoten bevatten AATAAA polyadenylatie (polyA)-signalen, maar deze
zijn meestal afwezig of in verkorte vorm aanwezig bij filamenteuze schimmels. Er werd een
vermoedelijk AATAA polyA-signaal gevonden 208 bp downstream van het stopcodon.
De consensus-sequentie voor transcriptieterminatie in eukaryoten is YGTGTTYY (Jeenes et
al., 1991). Deze ligt logischerwijze downstream van het polyA-signaal. In de 3’ onvertaalde
regio van het OMPD gen van M. gramineum werd een gelijkaardige CGTGTT sequentie
gevonden 124 bp downstream van het polyA-signaal. Verder onderzoek is nodig om de
significantie van de gevonden sequenties te bepalen.
IV. Resultaten en Bespreking - 92
MANU DE GROEVE
1
71
141
211
281
351
421
491
561
631
701
771
841
911
981
1051
1121
1191
1261
1331
1401
1471
1
tttaaaggca
tctatgcccg
wtttactata
tcagtcaaca
ctcgtttccc
ggctctatat
tatggcgtag
tggatggtaa
ccagaatcaa
ttgtgcaatc
ttcgtagaaa
cgtatcactg
catcgtggca
tcaagaaatt
gccaagggct
gaggtttctg
tagacatgtt
tttttcacgt
ctcagcctga
ctcaacatcc
ctgcttttcc
gaaaccaagg
gagagaaagt
ggccttggtt
tacgttgatc
ggcakttttg
gaagtagacg
agctggatat
tggttaaacc
agtggttttg
ctggagagaa
acctatgagt
cagagcgttc
ccctgatggg
gaccgcatgc
tactaaagtt
gcttctggat
gatggcgctc
ccgattccca
cgaacattgc
tagtttggat
ttgaattatc
cacctctcta
ATGAGTTCAA
M S S
1541
21
GCTCGAAAGC TTTTCGCCGT TGCGGAACGC AAAAAGTCGA ACCTGATCAT ATCCGCTGAC TTGACCGATA
A R K
L F A
V A E R
K K S
N L I
I S A D
L T D
1611
44
CAAAGAGTCT CTTGGAGTGT GCTGATGgta gagtagccaa caaacccatt gcatgctcac tcgctaactt
T K S
L L E C
A D
intron
1681
53
atatgcgcct gtttagAGCT CGGGCCCTTC ATCTCAGTGT TCAAGACCCA TATCGACATC ATTCACGATT
E
L G P F
I S V
F K T
H I D I
I H D
1751
71
TTGGCGACGA GACGGTTCGG GGCCTCAAGA GTCTGGCAAG AAAGCACGAC TTCCTCATCT TCGAGGATCG
F G D
E T V R
G L K
S L A
R K H D
F L I
F E D
1821
94
CAAGTTTGTG GATATTGGCT CAACAGCCCA GAAACAGTAC CATGGTGGTG CCCTGCGCAT CTCCGAGTGG
R K F V
D I G
S T A
Q K Q Y
H G G
A L R
I S E W
1891
118
GCCGACATTG TCAACGTCAG TCTCCTAGGG GGTGATGGCG TTGTGAATGC ACTGGGCCAG GTCATTACCG
A D I
V N V
S L L G
G D G
V V N
A L G Q
V I T
1961
141
ACGAGACGTT TCCCTATCGC GGTCAACGGG CCCTGCTTCT GCTTGCTGAG ATGACCACGG CCGGTAGCCT
D E T
F P Y R
G Q R
A L L
L L A E
M T T
A G S
2031
164
TGCCGTTGAA AAATACACAG AGCGCTGCGT CGCTGTCGCA CGAAAGAATG GCATTGCCGC TGGAGTCATG
L A V E
K Y T
E R C
V A V A
R K N
G I A
A G V M
2101
188
GGCTTCGTTG CCACGCGAGG CCTCGAAGAC GTTGGCAACG AACCTGGGGT CGAGAGAGAC GAGGACGAAG
G F V
A T R
G L E D
V G N
E P G
V E R D
E D E
2171
211
ACTTTGTCGT TTTTACTACT GGTATCAACA GCAGCCAATC TGACAATGTG CTGGGACAGC AGTATCAGAC
D F V
V F T T
G I N
S S Q
S D N V
L G Q
Q Y Q
2241
234
ACCCGAGGCA GCCATACGTG GCGGCTCTGA TTTCATCATC GCCGGCAGGG GCATATATGC CTCTGAGGAT
T P E A
A I R
G G S
D F I I
A G R
G I Y
A S E D
2311
258
CGTGTTCAGG CTGCGAAGCA ATATCAAGCC GAGGGTTGGG CTGCCTATGT GGCTCGCATA GGGCCAGGGC
R V Q
A A K
Q Y Q A
E G W
A A Y
V A R I
G P G
2381
281
ATTATTGAgg ggactctaag gttcaatata ccgaaacaat attgtgtcta cttgcctggt cagttgcaga
H Y *
2451
2521
2591
2661
2731
2801
2871
gcacacgtaa
ccagggcttc
ctcgaagtcg
tcgtttgcag
cagagcagct
actcggcagt
tttctttttg
tctacttgtg
tctctgtaga
gcttaggctt
tatgcaacga
tctaggggcg
cgtcaaatct
cacgtgttgg
gacggacgga
gtaagcggtg
aagcgtaggg
tagagctgaa
ccagttgaga
tagggatatt
taatgccggc
aaaaatatga
tgcagctctc
tgttgttgga
tctgaagata
atcctatgac
attcgtatgc
tctcaccagc
tacacgaagc
ggcgttgaat
accattgttg
atattgatac
ctgcataagg
ctactcccct
taccctcttc
AATCGGGACT
K S G
agttggggtt
tcttggcctt
gatcggagct
gagccttaga
tctcgcagaa
aacagataat
tgcttggttg
agcatgccgg
ctcaggcttt
gttgatgtat
tgtwttttta
gagggaagaa
cggccagaga
cctgacaaaa
cttagttatt
tacaagccag
ttccacaatg
cgacggtgat
agtttttgac
cgagctcttc
acatgcagtg
cgtgtcaccg
ggacttccga
ctgaggaata
gctggccaag
ggttatccgc
ctctcacgcc
aatcccccac
CCCGTATCAT
L P Y H
cgactccaat
gaatttggcc
tttttcaccg
gcgagtagcg
taatatagat
caggactgac
attcactgag
tgccgtaccc
gaagtgattc
caaattcaaa
agaccaggat
ggacaaaaag
gccgctattg
agcatcttgg
attaaatgat
cttctatggc
agtcttggac
ccatctgtcc
agtttttctg
cgcttctagc
cttctacggg
gacaacccgt
cggttgtgac
gtgaatcgga
acgcgagtag
tcctgaaacc
tgcagtcaag
ttgttatgga
ATCCGAGCCA
I R A
gcatatcgtt
ggaaaggcca
gcattcgcct
agattcaacc
tcatctcgtc
tttacgcttc
cgcagctg
gattgaagtg
gatgccttct
cwtttagacg
taraaraatc
tgttaggggc
ctgaatcttt
gtatagcgat
atcaaagctc
ttcccagcag
ccccccctcc
ataagtcgca
aagaatcagt
tcagatgcgt
gttgtatgat
ggtgaaaccc
gaatagtgct
aatcgccaat
agtctgatta
ctctcccctt
aatatacgca
gaagaacgac
CAACGAATCC
T T N
tctaatcaaa
tttattaact
tctactgtag
agagatgatg
ccgagcatat
cgagccacgc
ctgcactggc
gtacgacgcc
agtacacgcg
tccctcaagt
ctgatatcga
tggttcttgt
ggatgcaagg
tttagggtag
atgcggatga
caccaggttc
tggggtagca
ggtgggtaat
agtcgtcgcg
gccaaaatct
actcgttggc
cattagtgaa
tttcggtcac
gggtatgctc
tcgttttctt
cgcacactgt
agcgaccgca
GCACCCAGTG
P H P V
ggatctcagt
gagtgacggg
cccatactgg
aagccctcta
caccacacca
tcgggaagtc
Figuur 28. Nucleotidensequentie (2918 bp) van het OMPD gen van Myrothecium gramineum
MUCL39210 en de afgeleide aminozuursequentie (282 AZ) van het OMPD enzym
IV. Resultaten en Bespreking - 93
MANU DE GROEVE
1.5.2. Bevestiging van het intron in het OMPD gen
Om de theoretische positie en lengte van het intron te bevestigen werd een PCR uitgevoerd op
cDNA van M. gramineum. Daarvoor werd eerst totaal RNA geïsoleerd uit mycelium van M.
gramineum met de Rneasy Plant Mini Kit zoals eerder beschreven. Er werd geëlueerd met 50
µL RNAse-vrij water. De concentratie van het RNA staal werd bepaald met de
spectrofotometer en bedroeg 240 ng/µL. In totaal werd dus 12 µg totaal RNA geïsoleerd. Ter
controle werd 5 µL van het RNA staal geladen op een agarose gel (1.4%). Het resultaat is
weergegeven in Figuur 29. Hieruit blijkt dat de isolatie van totaal RNA succesvol was. Naast
een laag-moleculaire smeer zijn er twee zeer heldere bandjes zichtbaar die staan voor het 18S
en 28S ribosomaal RNA. Omdat er echter ook contaminatie van genomisch DNA zichtbaar is
op gel werd het RNA-staal behandeld met DNAse zoals eerder beschreven.
Na zuivering van het totaal RNA staal werd 11 µL
gebruikt als template voor de ‘eerst streng’ cDNA
synthese met de RevertAid™ H Minus First Strand
cDNA Synthesis Kit (Fermentas). Er werd ook cDNA
gemaakt van een niet-gezuiverd RNA staal. Als primer
werd de oligo(dT)18 primer gebruikt om enkel cDNA te
maken van mRNA met een 3’-poly(A) staart. Het
gesynthetiseerde cDNA kan rechtstreeks gebruikt
worden als template bij een PCR reactie.
- genomisch DNA
(contaminatie)
- 28S ribosomaal RNA
- 18S ribosomaal RNA
Figuur 29. Geïsoleerd totaal RNA uit
M. gramineum op agarosegel
Op basis van de reeds gekende sequentie van het OMPD
gen werden twee primers ontworpen waarbij de forward
primer bindt op exon 1 en de reverse primer op exon 2. De sequenties en eigenschappen van
de ontworpen primers zijn weergegeven in Tabel 38. De bindingsplaatsen van de primers op
genomisch DNA zijn weergegeven in Figuur 30.
Tabel 38. Gebruikte primers voor de amplificatie van cDNA van het OMPD gen
Primers
Tm (°C)
Lengte (nt)
Sequentie (5’→ 3’)
ATCCGAGCCACAACGAATCC
cDNA OMPD 5’
68.3
20
AATCAGAGCCGCCACGTATG
cDNA OMPD 3’
67.2
20
31
792
cDNA OMPD 5'
exon 1
(157 bp)
genomisch DNA
intron
(59 bp)
cDNA OMPD 3'
exon 2
(689 bp)
Figuur 30. Bindingsplaatsen van de cDNA primers op genomisch DNA
Met genomisch DNA als template amplificeren deze primers een fragment van 762 bp dat het
volledige intron bevat. Omdat cDNA afkomstig van mRNA geen introns bevat kan op deze
manier de exacte positie van het intron afgeleid worden. Indien de theoretische lengte (59 bp)
IV. Resultaten en Bespreking - 94
MANU DE GROEVE
en positie van het intron klopt moeten de primers met cDNA als template een fragment van
703 bp amplificeren.
Er werd een High Fidelity PCR uitgevoerd volgens het standaard protocol met 5 µL van het
gesynthetiseerde cDNA als template. Zowel het cDNA afkomstig van het gezuiverde RNA
staal als dit van het niet-gezuiverde RNA staal werd gebruikt voor PCR. Er werden 35 cycli
(10 + 25) uitgevoerd bij een annealingstemperatuur van 50°C. Ter controle werd een zelfde
PCR uitgevoerd op genomisch DNA van M. gramineum. Het resultaat is weergegeven in
Figuur 31 (laan 1-2-3). Uit de figuur blijkt dat zowel bij de PCR op het genomisch DNA als
bij de PCR’s op de twee cDNA stalen een fragment zichtbaar is van 762 bp. Dit wijst er op
dat het gezuiverde cDNA staal nog steeds gecontamineerd was met genomisch DNA, niet
tegenstaande het cDNA gemaakt was van een RNA staal dat een DNAse behandeling had
ondergaan. De intensiteit van het 762 bp fragment is echter wel minder duidelijk dan bij het
ongezuiverde cDNA staal. Er werd van uitgegaan dat er bij het gezuiverde cDNA staal
waarschijnlijk wel een fragment van 703 bp geamplificeerd werd, maar dat de concentratie te
laag was om te kunnen observeren op gel.
Daarom werd een reamplificatie uitgevoerd op het reactiemengsel van de PCR op het
gezuiverde cDNA. De reamplificatie werd uitgevoerd volgens het standaard Taq PCR
protocol. Er werd 1 µL van het cDNA PCR mengsel gebruikt als template. Het resultaat van
de reamplificatie is weergegeven in Figuur 31 (laan 4).
1
2
3
4
PCR met cDNA primers
M
Laan 1: PCR op genomisch DNA M. gramineum
-762 bp
-703 bp
Laan 2: PCR op niet gezuiverd cDNA van M. gramineum
Laan 3: PCR op gezuiverd cDNA staal van M. gramineum
Laan 4: reamplificatie van PCR mengsel laan 3
Laan M: lengtemerker (SmartLadderSF)
Figuur 31. PCR met cDNA primers op genomisch DNA en cDNA van M. gramineum
Er konden nu twee fragmenten geobserveerd worden op gel. Een duidelijk fragment van 762
bp en een minder duidelijk fragment van 703 bp. Dit laatste fragment is vermoedelijk
afkomstig van het cDNA. Het 703 bp fragment werd uit gel gezuiverd, geligeerd in pGEM®-T
en gesequeneerd.
MANU DE GROEVE
IV. Resultaten en Bespreking - 95
Uit de sequenering bleek dat het 703 bp fragment inderdaad afkomstig is van cDNA van het
OMPD gen. De sequentie van het vermoedelijk intron werd immers niet teruggevonden in het
cDNA. Het cDNA en het genomisch DNA werden gealigneerd (Needleman-Wunsch) om de
positie en lengte van het intron te bevestigen. Het resultaat van deze alignment is
weergegeven in Bijlage (IX.1.1, p.131). Hieruit bleek dat de theoretische positie en lengte (59
bp) van het intron correct is.
Hiermee werd de theoretische positie en lengte van het intron bevestigd. Het feit dat het
cDNA van OMPD kon geamplificeerd worden is ook een bewijs dat het OMPD gen effectief
tot expressie komt in M. gramineum.
1.5.3. Aminozuursequentie en codon usage
De coderende sequentie codeert voor een eiwit bestaande uit 282 aminozuren met een
berekende moleculaire massa van 30635.44 Da. Het eiwit bevat het herkenningspatroon van
alle bekende OMPD enzymen. PROSITE geeft als herkenningspatroon voor OMPD enzymen
volgende aminozuursequentie: [LIVMFTAR]-[LIVMF]-x-D-x-K-x(2)-D-[IV][ADGP]-x-T-[CLIVMNTA]. In dit patroon is K (lysine) absoluut essentieel voor de
activiteit van het enzym. In de aminozuursequentie van het OMPD enzym van M. gramineum
wordt dit patroon onder de volgende vorm teruggevonden: IFEDRKFVDIGSTA. De bekomen
sequentie is dus wel degelijk afkomstig van een OMPD enzym [orotidine 5'-phosphate
decarboxylase (OMPdecase), EC 4.1.1.23].
In genen van filamenteuze schimmels wordt vaak codon bias gevonden (Ballance, 1986). Dit
resulteert uit het feit dat 61 codons coderen voor 20 aminzuren. Er zijn dus meerdere codons
die coderen voor een zelfde aminozuur. Codon bias treedt op wanneer er voor een bepaald
aminozuur een duidelijke voorkeur is voor een of meerdere codons. Codon bias uit zich sterk
in genen die tot hoge expressie komen. Er is dan een voorkeur voor abundante tRNA’s.
Het codongebruik in het OMPD gen van M. gramineum werd onderzocht. In Tabel 39 wordt
een frequentietabel weergegeven van de gebruikte codons in het OMPD gen.
Analyse van het codongebruik van de coderende sequentie toont aan dat er een voorkeur is
voor codons met een pyrimidine (C of T) op de derde positie waarbij C boven T verkozen
wordt. Codons eindigend op A worden blijkbaar vermeden. Dit uit zich het sterkst bij Leu en
Val maar is ook duidelijk merkbaar bij Ala, Glu, Gly, Ile en Lys. De voorkeur voor C op de
derde positie en het vermijden van A op deze positie wordt ook bij andere filamenteuze
schimmels teruggevonden (Ballance, 1986; Pöggeler, 1997; Jekosch & Kück, 1999). Verder
blijkt nog dat twee codons niet gebruikt worden, namelijk TTA (Leu) en GTA (Val). Ook bij
S. macrospora, N. crassa en Acremonium chrysogenum worden deze codons weinig gebruikt
(Pöggeler, 1997; Jekosch & Kück, 1999).
IV. Resultaten en Bespreking - 96
MANU DE GROEVE
Tabel 39. Analyse van codongebruik in het OMPD gen van M. gramineum (283 codons)
AZ
Codon
%
N
AZ
Codon
%
N
AZ
Codon
%
N
AZ
Codon
%
N
Ala
GCA
13%
4
His
CAC
43%
3
Pro
CCA
25%
2
Ser
AGC
18%
3
GCC
50%
15
CAT
57%
4
CCC
38%
3
AGT
24%
4
GCG
7%
2
ATA
21%
4
CCG
25%
2
TCA
18%
3
GCT
30%
9
ATC
53%
10
CCT
12%
1
TCC
12%
2
TGC
50%
1
ATT
26%
5
CAA
36%
4
TCG
12%
2
TGT
50%
1
AAA
31%
4
CAG
64%
7
TCT
18%
3
GAC
58%
11
AAG
69%
9
AGA
12%
2
GTA
0%
0
GAT
42%
8
CTA
5%
1
AGG
6%
1
GTC
36%
8
GAA
28%
5
CTC
33%
7
CGA
24%
4
GTG
27%
6
GAG
72%
13
CTG
33%
7
CGC
35%
6
GTT
36%
8
TTC
58%
7
CTT
19%
4
CGG
12%
2
Trp
TGG
100%
2
TTT
42%
5
TTA
0%
0
CGT
12%
2
Tyr
TAC
22%
2
GGA
11%
3
TTG
10%
2
ACA
31%
5
TAT
78%
7
GGC
46%
13
Met
ATG
100%
3
ACC
25%
4
STOP TAA
0%
0
GGG
18%
5
Asn
AAC
50%
4
ACG
31%
5
TAG
0%
0
GGT
25%
7
AAT
50%
4
ACT
13%
2
TGA
100%
1
Cys
Asp
Glu
Phe
Gly
Ile
Lys
Leu
Gln
Arg
Thr
Val
AZ=aminozuur; %=procentueel voorkomen van codon per aminozuur; N=aantal codons
Een vaak gebruikte methode om codon bias in genen te kwantificeren werd ontwikkeld door
Wright (1990). Hij introduceerde het begrip ‘effectief aantal codons’ (Nc, ‘effective number of
codons’). Deze eenheid (Nc) geeft aan in welke mate alle 61 codons gebruikt worden in een
gen. Nc benadert het getal 20 (=minimum) in genen met een sterke codon bias, terwijl in
genen zonder bias het getal 61 (=maximum) benaderd wordt. Het effectief aantal codons (Nc)
van het OMPD gen van M. gramineum werd berekend met het EMBOSS-programma CHIPS.
Er werd een waarde bekomen van Nc = 58.06. Omdat deze waarde vrij dicht tegen de
maximale waarde (61) ligt kan besloten worden dat er slechts weinig codon bias is in het
OMPD gen. Dit is niet zo verwonderlijk aangezien OMPD genen over het algemeen niet tot
hoge expressie komen.
1.5.4. Vergelijking met andere OMPD sequenties
Een krachtige methode om nieuwe sequenties te vergelijken met reeds gekende genen is het
aligneren van deze sequenties. Zowel functionele als evolutionaire informatie kan afgeleid
worden uit goed uitgevoerde alignments. De nucleotidensequentie van het OMPD gen van M.
gramineum en de afgeleide aminozuursequentie werden vergeleken met de OMPD sequenties
van andere fungi. Deze sequenties werden opgezocht in de EMBL nucleotidensequentie
database en de UniProt eiwitdatabase op het European Bioinformatics Institute (EBI). Meer
in het bijzonder werden de volgende nucleotiden- en aminozuursequenties in detail
bestudeerd:
MANU DE GROEVE
•
IV. Resultaten en Bespreking - 97
Nucleotidensequenties: Aspergillus oryzae (AB017705), Aspergillus kawachii (AB064659),
Cryptococcus humicola (AB084379), Penicillium camemberti (AB100244), Aureobasidium pullulans
(AF165169), Solorina crocea (AF206163), Cladosporium fulvum (Fulvia fulva) (AF288696),
Penicillium nalgiovense (AF510725), Blakeslea trispora (AJ534694), Aspergillus awamori
(AY530810), Rhizopus niveus (D17362), Neurospora crassa (X05993), Emericella nidulans
(Aspergillus nidulans) (M19132), Schizophyllum commune (M26019), Trichoderma harzianum
(Hypocrea lixii) (U05192), Epichloe typhina x Neotyphodium lolii (U14564), Epichloe typhina x
Neotyphodium lolii (U14565), Aspergillus niger (X06626), Penicillium chrysogenum (Penicillium
notatum) (X08037), Cephalosporium acremonium (Acremonium chrysogenum) (X15937), Phycomyces
blakesleeanus (X53601), Trichoderma reesei (Hypocrea jecorina) (X55880), Mortierella alpina
(AB109469), Rhizopus oryzae (AF497632), Sordaria macrospora (Z70291).
•
Aminozuursequenties: Aspergillus niger (P07817 Q9HGS5), Aspergillus oryzae (O13416),
Aureobasidium pullulans (Q9P8X9), Cephalosporium acremonium (Acremonium chrysogenum)
(P14017), Cladosporium fulvum (Fulvia fulva) (Q9HFV8), Emericella nidulans (Aspergillus nidulans)
(P10652), Neurospora crassa (P05035 Q7RV44), Penicillium chrysogenum (Penicillium notatum)
(P09463), Solorina crocea (Q9HF68), Sordaria macrospora (P78748), Trichoderma harzianum
(Hypocrea lixii) (Q12709), Trichoderma reesei (Hypocrea jecorina) (P21594), Aspergillus awamori
(Q5J2D0), Aspergillus fumigatus (Sartorya fumigata) (O13410), Aspergillus kawachi (Aspergillus
awamori var. kawachi) (Q96WP7), Penicillium nalgiovense (Q8J269), Penicillium camembertii
(Q7Z8L4), Epichloe typhina x Neotyphodium lolii (1) (Neotyphodium sp. (strain Lp1)) (Q12565),
Epichloe typhina x Neotyphodium lolii (2) (Neotyphodium sp. (strain Lp1)) (Q12566), Rhizopus oryzae
(Rhizopus delemar) (Q71HN5), Rhizopus niveus (P43230), Blakeslea trispora (Q6X1B7), Rhizomucor
racemosus (Mucor circinelloides f. lusitanicus) (P32431), Phycomyces blakesleeanus (P21593),
Rhizomucor pusillus (Q9Y720), Mortierella alpina (Q6F5H7), Ustilago maydis (P15188),
Schizophyllum commune (P14964), Cryptococcus humicola (Q7Z7W9).
1.5.4.1. BLAST (Basic Local Alignment Search Tool)
Een eerste methode die werd gebruikt is BLAST. Dit is een snelle methode voor het
doorzoeken van nucleotiden- en aminozuurdatabases. Het BLAST algoritme kan zowel lokale
als globale regio’s van similariteit detecteren in al dan niet gerelateerde eiwitten. Beide types
van similariteit (lokaal-globaal) kunnen belangrijke informatie verschaffen over de functie
van niet-gekarakteriseerde eiwitten.
De aminozuursequentie van het OMPD enzym van M. gramineum werd geblast ten opzichte
van de UNIPROT (Release 5.0) database. Er werd gebruik gemaakt van het WU-BLASTp
(v2.0) programma op het EBI met standaard parameters, behalve voor de scorematrix
(PAM250). Het OMPD enzym van M. gramineum bleek homologie te vertonen met de
OMPD enzymen van andere fungi. De aminozuursequentie vertoont de grootste identiteit en
similariteit met het OMPD enzym van Aspergillus oryzae (respectievelijk 60% en 79%).
Algemeen wordt de grootste identiteit en similariteit waargenomen met de OMPD enzymen
van de filamenteuze schimmels Aspergillus sp., Penicllium sp., Cladosporium fulvum en
Solorina crocea. Opmerkelijk is dat het OMPD enzym van M. gramineum slechts weinig
similariteit vertoont met de OMPD enzymen van de andere Sordariomyceten. M. gramineum
behoort nochtans tot de Sordariomyceten, een klasse binnen de Ascomyceten. De
MANU DE GROEVE
IV. Resultaten en Bespreking - 98
aminozuursequentie vertoont zelfs meer similariteit met de OMPD enzymen van gisten (6066%) dan van Sordariomyceten (47-49%).
Zowel de coderende sequentie (zonder intron) als de sequentie van start- tot stopcodon
werden geblast tegenover de EMBL database (Release 82) met het WU-BLASTn (v2.0)
programma (standaard parameters) op het EBI. De coderende sequentie (zonder intron)
vertoont zoals verwacht hetzelfde beeld als de blast met de aminozuursequentie (zie hoger).
De nucleotidensequentie vertoont homologie met de OMPD genen van andere fungi en de
hoogste identiteit werd opnieuw bekomen met het OMPD gen (pyrG) van A. oryzae (65%).
De sequentie van start- tot en met stopcodon (met intron) vertoont de hoogste identiteit met de
OMPD genen (pyrG) van C. fulvum en A. oryzae (beide 63%). Opnieuw werd slechts een lage
identiteit waargenomen met de OMPD genen (pyr4) van de Sordariomyceten.
1.5.4.2. Alignment van het OMPD enzym van M. gramineum met andere OMPD sequenties
Er werd een multiple sequence alignment uitgevoerd in het software programma Clone
Manager met de OMPD aminozuursequenties van M. gramineum en de volgende
filamenteuze schimmels: Eurotiomyceten (klasse): A. niger, A. nidulans en Penicillium
chrysogenum; Sordariomyceten (klasse): N. crassa, Sordaria macrospora, Trichoderma
harzianum en T. reesei. De alignment is weergegeven in Bijlage (IX.1.2, p.132). In deze
alignment is duidelijke de grote aminozuurinsertie te zien die typisch is voor Sordariomyceten
(N. crassa, T. reesei, T. harzianum, S. macrospora) maar niet voorkomt bij M. gramineum.
1.5.4.3. Graad van identiteit en similariteit met andere OMPD sequenties
Om een overzicht te krijgen van de graad van identiteit en similariteit van de OMPD
aminozuursequentie met andere OMPD sequenties werd de similariteit/identiteit-matrix
opgesteld. Deze matrix geeft het percentage similariteit en identiteit tussen twee bepaalde
gealigneerde sequenties. Er werd gebruik gemaakt van de OMPD aminozuursequenties van
20 Ascomycete filamenteuze schimmels, M. gramineum inbegrepen. De similariteit\identiteit
matrix werd opgesteld met het softwareprogramma MatGAT (Matrix Global Alignment Tool,
v2.03; Campanella et al., 2003) en is weergegeven in Tabel 40. Voor de alignments werd de
PAM250 scorematrix gebruikt en volgende parameters: First Gap = 12, Extending Gap = 2.
Aan de hand van de similariteit\identiteit-matrix kan besloten worden dat het OMPD eiwit
van M. gramineum de grootste similariteit en identiteit vertoont met de OMPD eiwitten van
niet-Sordariomyceten (66-79% similariteit; 42-60% identiteit), terwijl de laagste percentages
bekomen worden bij sequenties van Sordariomyceten (47-49% similariteit; 28-31%
identiteit).
IV. Resultaten en Bespreking - 99
MANU DE GROEVE
Tabel 40. Similariteit\identiteit matrix van OMPD-eiwitten van Ascomycete filamenteuze schimmels
% similariteit\identiteit*
Organisme
1
1. Aspergillus awamori
2
9 10 11 12 13 14 15 16 17 18 19
20
89 100 92 74 99 84 84 83 81 64 33 33 32 34 35 33 33 46
60
89 87 76 88 82 81 81 83 65 34 35 33 36 36 33 33 46
59
92 74 99 84 84 83 81 64 33 33 32 34 35 33 33 46
60
76 91 85 86 84 83 64 34 34 33 34 35 34 34 45
60
74 73 73 73 73 61 34 35 34 35 36 33 33 44
56
83 83 82 80 64 33 33 32 34 35 33 33 46
60
98 96 87 64 34 34 32 35 35 34 34 45
59
97 86 65 33 33 32 35 35 35 35 44
59
85 64 33 33 32 34 35 34 34 44
58
65 34 33 32 34 35 33 33 45
59
32 33 32 33 33 31 32 43
59
71 73 74 75 55 55 37
30
88 73 73 58 59 37
29
76 76 56 57 35
28
98 55 56 36
30
56 56 36
30
94 37
30
37
31
2. Aspergillus fumigatus 96
3
3. Aspergillus niger 100 97
4
4. Aspergillus oryzae 97 96 98
5
5. Aspergillus nidulans 87 86 87 87
6
6. Aspergillus kawachi 99 96 99 96 86
7
7. Penicillium camembertii 95 93 95 94 87 94
8
8. Penicillium nalgiovense 95 93 95 94 87 94 100
9. Penicillium chrysogenum 94 92 94 92 86 93 99 99
10. Cladosporium fulvum 95 95 95 95 89 94 95 95 94
11. Solorina crocea 87 88 87 87 80 86 87 87 86 88
** 12. Acremonium chrysogenum 52 52 52 51 50 52 52 52 52 51 51
13. Trichoderma harzianum 52 52 51 52 50 52 52 52 52 51 50 87
14. Trichoderma reesei 50 51 50 50 49 50 50 50 51 50 50 87 95
15. Epichloe typhina x Neotyphodium lolii(1) 51 52 51 52 50 51 51 51 52 51 53 89 89 89
16. Epichloe typhina x Neotyphodium lolii(2) 52 53 52 51 51 51 52 52 52 52 53 89 88 90 99
17. Neurospora crassa 50 50 50 50 49 50 51 51 51 51 50 76 75 75 75 76
18. Sordaria macrospora 50 50 50 50 49 50 51 51 51 51 50 77 77 75 75 76 98
19. Aureobasidium pullulans 67 68 67 66 67 67 68 68 68 69 68 52 52 50 53 53 50 50
42
20. Myrothecium gramineum 78 78 78 79 76 78 79 79 79 79 79 48 48 47 49 49 47 48 66
* Percentage identiteit is weergegeven op grijze achtergrond. ** Sordariomyceten zijn onderlijnd
1.5.4.4. Fylogenie
In de literatuur worden verschillende artikels teruggevonden waarin aangetoond wordt dat de
sequentie van OMPD kan gebruikt worden om fylogenetische verwantschappen tussen
filamenteuze schimmels aan te tonen. Radford (1993) stelde op basis van 14 fungale OMPD
aminozuursequenties een fungale fylogenie op die in grote lijnen overeenkwam met de toen
bekende taxonomie en 18S rRNA sequenties.
Hier werd besloten een fungale fylogenie op te stellen gebaseerd op de OMPD
aminozuursequenties van 30 fungi. Er werden 20 sequenties gebruikt van Ascomyceten
(inclusief M. gramineum), 3 sequenties van Basidiomyceten en 7 sequenties van
Zygomyceten. Als outgroup werd de aminozuursequentie van het OMPD enzym van
Saccharomyces cerevisiae gebruikt. De aminozuursequenties werden opgezocht in UniProt
(Release 5.0). Voor de fylogenetische analyse werd de BioEdit software (v7.0.4.1; Hall,
1999) gebruikt. Er werd besloten de fylogenetische analyse uit te voeren volgens de
Maximum Likelihood (ML) methode. Dit is de meest statistisch robuuste methode voor zulke
analyses (Whelan et al., 2001). Nara et al. (2000) gebruikten ook de ML methode voor
eiwitten om een fylogenie op te stellen op basis van OMPD. Volgende procedures werden
uitgevoerd:
MANU DE GROEVE
IV. Resultaten en Bespreking - 100
1. Eerst werden de aminozuursequenties gealigneerd met het CLUSTAL W programma
(v1.82; Higgins et al., 1994) op het EBI volgens standaard parameters.
2. De verkregen multiple sequence alignment werd vervolgens gebruikt voor Maximum
Likelihood analyse met het programma ProML (Protein Maximum Likelihood;
v3.6a2.1) in BioEdit. Als evolutief model werd het Jones-Taylor-Thornton model voor
aminozuursubstitutie gebruikt. Dit model werd ook door Nara et al. (2000) gebruikt.
3. Na de ML analyse werd een fylogenetische boom verkregen. Er werd ook een
statistische analyse van de boom verkregen met de significanties van de knopen en
takken.
De verkregen ongewortelde boom heeft een Log Likelihood van -8767.85720. De
fylogenetische boom werd bekeken in het programma TreeIllustrator (v0.5 beta; Trooskens,
2005) en is weergegeven in Figuur 33. Alle knopen en takken waren statistisch significant
(P<0.05). De verkregen boom werd geworteld met de outgroup (S. cerevisiae) in het
programma TreeView (v1.6.6). Het fylogram dat zo werd verkregen is weergegeven in Figuur
32. De outgroup is niet weergegeven. De afstanden langs de takken tussen de verschillende
bladeren (= organismen) zijn evenredig met de berekende genetische afstand tussen de
OMPD aminozuursequenties. Op deze manier kan de boom een idee geven van de evolutie
van de onderzochte organismen.
De verkregen fylogenetische bomen corresponderen goed met de taxonomie van de
filamenteuze schimmels. De fylogenie op basis van de OMPD aminozuursequenties van
filamenteuze schimmels verdeelt deze organismen in drie duidelijk te onderscheiden groepen,
namelijk de Ascomyceten, Basidiomyceten en Zygomyceten. Binnen de Ascomyceten kunnen
twee duidelijke groepen onderscheiden worden die overeenkomen met de klasse van de
Eurotiomycetes en de klasse van de Sordariomycetes. Tot de Eurotiomycetes behoren
Aspergillus sp. en Penicllium sp., terwijl Neurospora crassa en Trichoderma sp. tot de
Sordariomycetes behoren. Opmerkelijk is dat M. gramineum, die taxonomisch een
Sordariomyceet is, op basis van de hier voorgestelde fylogenie niet tot de groep van de
Sordariomyceten behoort. M. gramineum vormt daarentegen een groep met Solorina crocea
die tot de klasse van de Lecanoromycetes behoort. Deze groep bevindt zich tussen de
Eurotiomycetes en de Sordariomycetes. De lage similariteit en identiteit van het OMPD eiwit
van M. gramineum met andere Sordariomyceten werd reeds aangetoond aan de hand van de
similariteit/identiteit-matrix (zie IV.1.5.4.3, p.98).
IV. Resultaten en Bespreking - 101
MANU DE GROEVE
ASCOMYCETEN
Eurotiomycetes (klasse)
Aspergillus awamori
Aspergillus kawachi
Aspergillus niger
Aspergillus oryzae
Aspergillus nidulans
Aspergillus fumigatus
Penicillium chrysogenum
Penicillium nalgiovense
Penicillium camembertii
Cladosporium fulvum ('incertae sedis')
Solorina crocea
(Lecanoromycetes)
Myrothecium gramineum (Sordariomycetes)
Epichloe typhina x Neotyphodium lolii (1)
Acremonium chrysogenum
Trichoderma reesei
Trichoderma harzianum
Sordaria macrospora
Neurospora crassa
Aureobasidium pullulans ('incertae sedis')
Rhizopus oryzae
Sordariomycetes (Klasse)
Epichloe typhina x Neotyphodium lolii (2)
ZYGOMYCETEN
Rhizopus niveus
Blakeslea trispora
Rhizomucor racemosus
Phycomyces blakesleeanus
Rhizomucor pusillus
Mortierella alpina
Ustilago maydis
BASIDIOMYCETEN
Schizophyllum commune
Cryptococcus humicola
* klasse weergegeven tussen haakjes
Figuur 32. Fylogenie (fylogram) van filamenteuze schimmels gebaseerd op het OMPD enzym
MANU DE GROEVE
IV. Resultaten en Bespreking - 102
Ascomyceten
Basidiomyceten
Zygomyceten
Figuur 33. Ongewortelde fylogenetische boom (radiale weergave) op basis van de OMPD enzymen
van filamenteuze schimmels
1.5.4.5. Structuur van fungale OMPD-genen
De genstructuur van het OMPD gen van Myrothecium gramineum werd vergeleken met de
genstructuur van de OMPD genen van andere filamenteuze schimmels. Meer bepaald werden
de sequenties van ATG tot en met stopcodon van de filamenteuze schimmels vergeleken. Er
werden 26 OMPD genen geanalyseerd van zowel Ascomyceten, Basidiomyceten en
Zygomyceten. In Figuur 34 is deze vergelijking schematisch weergegeven. De introns zijn
weergegeven als rechthoeken, de exons als lijnen. De lengtes van de exons en introns zijn
erboven weergegeven in aantal basenparen (bp). Rechts is de lengte van het corresponderende
OMPD eiwit weergegeven in aantal aminozuren.
IV. Resultaten en Bespreking - 103
MANU DE GROEVE
ZYGOMYCETEN
Rhizopus oryzae
Rhizopus niveus
Blakeslea trispora
Phycomyces blakesleeanus
180
56
180
56
180
77
186
65
Mortierella alpina
506
58
112
64
112
67
111
111
180
58
112
265 AZ
506
265 AZ
509
266 AZ
507
173
112
267 AZ
497
262 AZ
BASIDIOMYCETEN
Schizophyllum commune
186
Cryptococcus humicola
192
50
50
48
112
539
187
112
278 AZ
506
269 AZ
ASCOMYCETEN
60
310
Aureobasidium pullulans
366
131
157
69
677
Aspergillus oryzae
157
65
677
Aspergillus kawachii
157
69
677
Aspergillus nidulans
157
59
668
157
68
677
Aspergillus awamori
Aspergillus niger
Penicillium camemberti
157
Penicillium nalgiovense
157
Penicillium chrysogenum
Cladosporium fulvum
61
157
46
157
60
157
Solorina crocea
59
132
372
270 AZ
277 AZ
277 AZ
277 AZ
274 AZ
277 AZ
674
276 AZ
674
276 AZ
680
278 AZ
680
278 AZ
686
280 AZ
692
282 AZ
ASCOMYCETEN (Klasse Sordariomycetes)
Myrothecium gramineum
157
59
Neurospora crassa
1194
397 AZ
Sordaria macrospora
1191
396 AZ
Acremonium chrysogenum
1131
1146
Trichoderma reesei
Trichoderma harzianum
1140
Epichloe typhina x
Neotyphodium lolii (1)
1104
Epichloe typhina x
Neotyphodium lolii(2)
1101
376 AZ
381 AZ
379 AZ
367 AZ
366 AZ
Figuur 34. Genstructuur van OMPD genen van filamenteuze schimmels
Uit Figuur 34 blijkt duidelijk dat de meeste Ascomyceten (behalve de Sordariomyceten) één
intron bevatten op een sterk geconserveerde positie (na 157 bp). Een uitzondering is
Aureobasidium pullulans dat twee introns bevat waarvan één relatief groot is (366 bp). De
Sordariomyceten (behalve M. gramineum) bevatten geen introns. Het OMPD gen van M.
gramineum bevat in tegenstelling tot alle andere Sordariomyceten wèl het sterk
geconserveerde intron. De Zygomyceten en Basidiomyceten bevatten twee introns die
eveneens sterk geconserveerd zijn. De afstand tussen beide introns is vrijwel invariant (111112 bp).
MANU DE GROEVE
IV. Resultaten en Bespreking - 104
De lengtes van de corresponderende eiwitten variëren tussen 262-282 AZ voor zowel
Zygomyceten, Basidiomyceten als de meeste Ascomyceten (inclusief M. gramineum). De
Sordariomyceten vormen opnieuw een uitzondering. De OMPD-eiwitten van
Sordariomyceten (behalve M. gramineum) zijn 90-130 AZ groter (366-397 AZ) dan de andere
gekende OMPD-eiwitten.
Door de sterke verschillen tussen OMPD genen van Sordariomyceten en andere filamenteuze
schimmels worden de OMPD genen van Sordariomyceten ‘pyr4’ genen genoemd, terwijl de
andere OMPD genen ‘pyrG’ genoemd worden.
1.5.4.6. Bespreking
Alle methoden die hier gebruikt werden voor het vergelijken van het OMPD gen en eiwit van
M. gramineum met andere OMPD sequenties leveren dezelfde conclusie op: hoewel M.
gramineum een Sordariomyceet is, is de OMPD sequentie duidelijk meer gelijkaardig aan
filamenteuze schimmels die niet tot de Sordariomyceten behoren. Dit is des te opmerkelijker
aangezien OMPD in de literatuur vaak vermeld wordt als evolutionaire merker. Onder meer
Kimsey & Kaiser (1992) en Radford (1993) zien het OMPD enzym als het ideale eiwit voor
moleculaire evolutionaire studies omwille van volgende redenen:
• Alle organsimen hebben dezelfde de novo pyrimidine biosynthese pathway en
moeten dus OMPD activiteit bezitten. Het OMPD eiwit is vermoedelijk een
universeel enzym;
• Bijna alle onderzochte organismen hebben slechts één OMPD gen;
• OMPD-aminozuursequenties bevatten enkele zeer geconserveerde regio’s met
absoluut geconserveerde aminozuren die bewaard zijn gebleven gedurende meer
dan een miljard jaar evolutie.
Ook Nara et al. (2000) en Traut & Temple (2000) gebruikten het OMPD enzym als
evolutionaire merker voor fylogenetisch onderzoek en om de geconserveerde structuur van
het OMPD enzym te onderzoeken. Op basis van de hier opgestelde fylogenetische boom kan
besloten worden dat het OMPD enzym inderdaad kan gebruikt worden voor de classificatie
van de meeste filamenteuze schimmels. M. gramineum werd echter foutief geclassificeerd.
Dit is te wijten aan de OMPD aminozuursequentie die te afwijkend is van die van de andere
Sordariomyceten. Een mogelijke reden hiervoor is dat M. gramineum taxonomisch
verkeerdelijk onder de Sordariomyceten wordt ondergebracht. In de Literatuurstudie (II.2,
p.4) werd reeds vermeld dat het Myrothecium genus heterogeen is en moeilijk te classificeren.
Dit wordt echter gedeeltelijk tegengesproken door het feit dat een deel van het GPD gen van
M. gramineum de hoogste similariteit en identiteit vertoont met Sordariomyceten (Jonniaux et
al., 2004).
In ieder geval zou het academisch interessant zijn om de OMPD genen te sequeneren van
andere Myrothecium soorten of zelfs andere Myrothecium gramineum stammen. Met de in
deze scriptie gebruikte primers en geoptimaliseerde protocols zou dit theoretisch niet te veel
MANU DE GROEVE
IV. Resultaten en Bespreking - 105
problemen mogen opleveren. Op basis van de verkregen OMPD sequenties zal dan
geconcludeerd kunnen worden of M. gramineum al dan niet tot het Myrothecium genus
behoort, of dat het Myrothecium genus verkeerdelijk onder de klasse van de Sordariomyceten
wordt ondergebracht.
Tot nog toe werden alle OMPD genen van Sordariomyceten meestal ‘pyr4’ genen genoemd.
OMPD genen van andere filamenteuze schimmels worden ‘pyrG’ genen genoemd. Omdat het
OMPD gen van M. gramineum een intron bevat kan overwogen worden dit gen pyrG te
noemen om zo de verwantschap aan te tonen met de andere bekende pyrG genen.
Het OMPD gen van M. gramineum is het eerste OMPD gen van een Sordariomyceet dat een
intron bevat en dat de ongeveer 100 aminozuren lange insert niet bevat die typisch is voor
Sordariomyceten.
MANU DE GROEVE
IV. Resultaten en Bespreking - 106
2. COMPLEMENTATIE VAN A. NIDULANS FGSC A722
Om aan te tonen dat het gekloneerde OMPD gen van Myrothecium gramineum codeert voor
een werkend OMPD enzym werd A. nidulans FGSC A722 getransformeerd met het pOV
plasmide. A. nidulans FGSC A722 is een gedefinieerde OMPD-negatieve mutant (bevat de
pyrG89 mutatie) die vaak in de literatuur wordt gebruikt om de werking aan te tonen van
gekloneerde OMPD genen.
De gebruikte A. nidulans stam is auxotroof en heeft uracil of uridine nodig voor groei. Door
deze stam te transformeren met een werkend OMPD gen wordt de auxotrofie opgehoffen en is
groei mogelijk op minimaal medium zonder uracil en uridine. Op deze manier kan
aangetoond worden dat het getransformeerde OMPD gen codeert voor een functioneel enzym.
Voor de transformatie van filamenteuze schimmels zijn grote hoeveelheden DNA nodig
(enkele tot tientallen µg DNA). Het pOV plasmide dat 2050 bp van het wild type OMPD gen
van Myrothecium gramineum bevat werd vermeerderd in E. coli DH5α-F’ voor een maxiprep.
De pOV plasmiden werden uit E. coli geïsoleerd met de HiSpeed Plasmid Maxi kit (Qiagen).
De DNA concentratie van de bekomen plasmidenoplossing werd bepaald met de
spectrofotometer. Er werd een concentratie van 2.06 µg DNA/µL bekomen.
Om te controleren of de vaste selectieve media (zie Tabel 24) voor A. nidulans FGSC A722
wel selectief genoeg zijn werden deze geënt met 100 µL van een 107 sporen/mL oplossing.
De gebruikte selectieve media zijn TO- en TO-S (gestabiliseerd met sorbitol). Deze minimale
media bevatten geen uridine en uracil. Na enkele dagen incubatie bij 30°C werd slechts zeer
weinig tot geen groei waargenomen op de selectieve platen (zie Figuur 35) terwijl de
schimmel wel goede groei vertoonde op niet-selectief medium mét uracil (OS). Er kon
besloten worden dat zowel de TO-S als TO- media voldoende selectief zijn.
Aspergillus nidulans FGSC A722
OS (bevat uracil)
TO-S
Figuur 35. Selectiviteit van de gebruikte media
Voor de transformatie werd A. nidulans opgegroeid in een vloeibare 400 mL cultuur. Er
werden 5.3x108 sporen geënt in 400 mL AMM medium gesupplementeerd met gistextract (5
g/L), uracil (10 mM) en uridine (10 mM). De cultuur werd geïncubeerd gedurende 16 uur bij
30°C en 125 RPM. Na filtratie werd 6.15 g mycelium bekomen. De behandeling van het
MANU DE GROEVE
IV. Resultaten en Bespreking - 107
mycelium met de celwandafbrekende enzymen werd stopgezet na 1 uur 15 min. Er werden
1.6x107 protoplasten/mL bekomen in een volume van 7.5 mL. In totaal waren dus 1.2x108
protoplasten beschikbaar voor transformatie. De protoplasten werden getransformeerd met 30
µL van de pOV plasmidenoplossing. Dit komt neer op 61.8 µg DNA.
Er werden acht selectieve TO-S platen geënt met elk 200 µL van de getransformeerde
protoplasten. Na vier dagen waren de platen volledig bedekt met honderden kleine kolonies.
Na zes dagen werden 117 kolonies overgeënt naar TO-. In de daaropvolgende dagen werden
nog meer kolonies overgeënt zodat in totaal 597 kolonies overgeënt werden. Van deze 597
overgeënte kolonies vertoonden er 352 duidelijke groei na enkele dagen. De eerste 117
kolonies werden een tweede maal overgeënt op TO- en 113 ervan (= 96.6%) groeiden verder
na enkele dagen incubatie.
Indien enkel rekening wordt gehouden met de overgeënte kolonies (597) wordt een
transformatie-efficiëntie van 9.66 transformanten per µg DNA bekomen. Gecorrigeerd naar
het aantal gebruikte protoplasten wordt dit 0.08 transformanten per µg DNA per 106
protoplasten.
Het is moeilijk transformatie-efficiënties te vergelijken tussen verschillende artikels.
Afhankelijk van de gebruikte stam en de gebruikte methode kunnen de transformatieefficiënties immers sterk variëren. Ook de aard van het gebruikte DNA kan een belangrijke
rol spelen. Er kunnen zowel integratieve als replicatieve vectoren gebruiken. Hoewel
replicatieve vectoren met een ARS sequentie meestal tot hogere transformatie-efficiënties
leiden zijn de transformanten vaak instabiel. In Tabel 41 worden de transformatie-efficiënties
van enkele filamenteuze schimmels uit de literatuur vergeleken.
In vergelijking met deze waarden is de transformatie-efficiëntie die hier werd bekomen vrij
laag. Hier kunnen enkele redenen voor gegeven worden:
• Voor het berekenen van de transformatie-efficiëntie werd hier enkel rekening
gehouden met de kolonies die na overenten verder konden groeien op selectief
medium. In de literatuur wordt echter meestal enkel het totaal aantal bekomen
kolonies gerapporteerd. Aangezien hier vele honderden kolonies per plaat werden
bekomen en er acht platen werden geënt kan besloten worden dat er in totaal enkele
duizenden transformanten werden bekomen. In dat geval is de transformatieefficiëntie veel hoger dan de eerst berekende.
• Er moet rekening gehouden worden met het feit dat hier A. nidulans werd
getransformeerd met het OMPD gen van M. gramineum, een Sordariomyceet, en dat
er 76% similariteit en 56% identiteit is tussen de OMPD eiwitten van beide
schimmels. Het zou kunnen dat A. nidulans het gen niet goed tot expressie kan
brengen omdat de promotorstructuur te verschillend is. In dat geval zou het vervangen
van de promotor van het OMPD gen van M. gramineum door een homologe promotor
van A. nidulans de transformatie-efficiëntie kunnen verhogen.
MANU DE GROEVE
IV. Resultaten en Bespreking - 108
Tabel 41. Vergelijking van de transformatie-efficiënties bij enkele filamenteuze schimmels
similariteit # transformanten
Referentie
Getransformeerde stam Oorsprong OMPD
(%)
per µg plasmide
Deze studie
Aspergillus nidulans
76
>10
Myrothecium gramineum
Goosen et al. (1987)
Aspergillus nidulans
Aspergillus niger
100
1000
A. niger
A. niger
100
8-50
Yamazaki et al. (1999) Rhizomucor pusillus
Rhizomucor pusillus
100
5
Smit & Tudzynski (1992) A. niger
A. niger
100
20-80
Gruber et al. (1990)
Trichoderma reesei
Trichoderma reesei
100
12000
Weidner et al. (1998)
A. nidulans
A. fumigatus
86
50
A. fumigatus
A. fumigatus
100
150
Ballance et al. (1983)
A. nidulans
N. crassa
49
0,33 - 0,66
Van Hartingsveldt et al. A. niger
A. niger
100
40
(1987)
A. niger
N. crassa
50
2
Gruber et al. (1990)
T. reesei
N. crassa
75
1500
T. reesei
A. niger
50
700-800
Mattern et al. (1987)
A. oryzae
A. niger
98
16
Enkele getransformeerde kolonies van A. nidulans werden gebruikt voor een kolonie-PCR om
aan te tonen dat dit geen spontane mutanten zijn maar dat ze wel degelijk het OMPD gen van
M. gramineum hebben geïntegreerd in het genoom. De kolonie-PCR’s werden uitgevoerd
zoals beschreven in Materialen en Methoden (III.5.5, p.44). Telkens werden zowel OMPDgenspecifieke primers gebruikt die het OMPD gen van M. gramineum vermenigvuldigen als
de T7 en SP6 primers om te controleren of het pOV plasmide mee geïntegreerd is in het
genoom van A. nidulans. Als positieve en negatieve controle voor het OMPD gen werd
mycelium gebruikt van respectievelijk M. gramineum en de niet-getransformeerde A.
nidulans FGSC A722. Er werd een kolonie-PCR uitgevoerd met de OMPD-specifieke ‘5 O
volledig’ en ‘3 O transcriptiestop’ primers op twee getransformeerde A. nidulans kolonies en
de positieve en negatieve controles. Met een tandenstoker werd mycelium van een petriplaat
rechtsreeks naar het PCR-reactiemengsel overgebracht. De kolonie-PCR met de OMPD
primers leverde een duidelijk fragment van 2050 bp op bij M. gramineum (positieve controle)
en een bandenpatroon bij kolonie 1. Bij kolonie 2 en de negatieve controle waren geen
fragmenten zichtbaar. Ook werd bij geen enkele PCR op plasmide een fragment
geobserveerd. In Figuur 36 is enkel de kolonie-PCR op M. gramineum weergegeven als
bewijs dat het kolonie-PCR protocol efficiënt werkt voor deze schimmel.
IV. Resultaten en Bespreking - 109
MANU DE GROEVE
M
1
2
KOLONIE-PCR
Laan M: grote lengte merker (SmartLadder)
-2050 bp
Laan 1: Positieve controle (M. gramineum) (OMPD primers)
Laan 2: M. gramineum (plasmide primers)
Figuur 36. Kolonie-PCR op Myrothecium gramineum (OMPD en plasmide primers)
Omdat er enkel bij M. gramineum een duidelijk fragment werd bekomen en niet bij de
getransformeerde kolonies werd vermoed dat het kolonie-PCR protocol niet efficiënt is voor
de A. nidulans kolonies. Daarom werd een reamplificatie uitgevoerd op alle kolonie-PCR’s
volgens het standaard reamplificatieprotocol. Het resultaat is weergegeven in Figuur 37.
M 1
2 3 4
5
6 7 8
KOLONIE PCR (reamplificatie)
Laan M: grote lengtemerker (SmartLadder)
Laan 1: Positieve controle (M. gramineum) (OMPD primers)
Laan 2: M. gramineum (plasmide primers)
Laan 3: Getransformeerde kolonie 1 (OMPD primers)
Laan 4: Getransformeerde kolonie 1 (plasmide primers)
Laan 5: Getransformeerde kolonie 2 (OMPD primers)
Laan 6: Getransformeerde kolonie 2 (plasmide primers)
Laan 7: Negatieve controle (A. nidulans FGSC A722) (OMPD primers)
Laan 8: A. nidulans FGSC A722 (plasmide primers)
Figuur 37. Reamplificatie van kolonie-PCR’s op getransformeerde kolonies van A. nidulans
Bij de kolonie-PCR op M. gramineum (positieve controle) werd zoals verwacht een fragment
van 2050 bp geobserveerd. Bij kolonie 2 werd nu ook een bandenpatroon geobserveerd
identiek aan dit van kolonie 1. Hoewel het bandenpatroon bij de kolonies niet meteen kan
verklaard worden kan er toch besloten worden dat deze minstens een deel van het OMPD gen
van M. gramineum bevatten aangezien de negatieve controle van A. nidulans FGSC A722
geen bandenpatroon opleverde. Bij de PCR’s op de plasmiden zijn nergens fragmenten
zichtbaar wat er op wijst dat geen plasmide is geïntegreerd. Het zou echter ook kunnen dat de
plasmide primers (SP6 en T7) niet geschikt zijn voor het hier gebruikte kolonie-PCR protocol
waardoor geen fragmenten vermenigvuldigd kunnen worden.
Uit bovenstaande gegevens kan besloten worden dat de OMPD-negatieve A. nidulans FGSC
A722 gecomplementeerd kan worden met het OMPD gen van M. gramineum. Hiermee werd
MANU DE GROEVE
IV. Resultaten en Bespreking - 110
aangetoond dat het gekloneerde OMPD gen in het pOV plasmide codeert voor een functioneel
OMPD enzym. Voor het eerst werd een gen van Myrothecium gramineum heteroloog tot
expressie gebracht in een andere filamenteuze schimmel.
In het vorige hoofdstuk werd reeds gewezen op het feit dat hoewel M. gramineum een
Sordariomyceet is, de coderende sequentie van het OMPD gen meer homoloog is aan dit van
niet-Sordariomyceten zoals Aspergillus soorten. Of dit een voordeel dan wel een nadeel is om
het OMPD gen van Myrothecium gramineum als selectiemerker te gebruiken in andere
filamenteuze schimmels is discussieerbaar. Enerzijds zou men kunnen stellen dat het gen van
M. gramineum efficiënt kan gebruikt worden in zowel Sordariomyceten als nietSordariomyceten. Belangrijke argumenten die deze stelling ondersteunen zijn de volgende:
• Omdat M. gramineum een Sordariomyceet is kan verondersteld worden dat de
promotor van het OMPD gen ook efficiënt kan functioneren in andere
Sordariomyceten. Het feit dat de coderende sequentie van het OMPD gen slechts
weinig similariteit en identiteit vertoont is misschien minder belangrijk omdat het
OMPD enzym efficiënt lijkt te functioneren in M. gramineum. Voorwaarde is wel dat
het intron herkend wordt in andere Sordariomyceten.
• Omdat de coderende sequentie van het OMPD gen van M. gramineum de meeste
similariteit en identiteit vertoont met OMPD genen van niet-Sordariomyceten
functioneert het gen bij deze organismen waarschijnlijk beter dan de OMPD genen
van andere Sordariomyceten.
Anderzijds zou natuurlijk ook gesteld kunnen worden dat het OMPD gen van M. gramineum
nergens tot goede expressie kan komen door de duale structuur van het gen: de coderende
sequentie die het meest homoloog is aan niet-Sordariomyceten staat onder controle van de
promotor van een Sordariomyceet.
Verder onderzoek zal moeten aantonen of het OMPD gen van M. gramineum beter werkt dan
andere OMPD genen van Sordariomyceten. Door A. nidulans FGSC A722 te transformeren in
een parallel experiment met zowel het OMPD gen van M. gramineum als dit van Neurospora
crassa zullen de transformatie-efficiënties betrouwbaar kunnen vergeleken worden.
MANU DE GROEVE
IV. Resultaten en Bespreking - 111
3. CREATIE OMPD-KNOCKOUT VAN M. GRAMINEUM
Om het OMPD gen als selectiemerker te kunnen gebruiken in Myrothecium gramineum moet
eerst een OMPD-negatieve mutant van deze schimmel gecreëerd worden. Dergelijke
mutanten zijn uracil- en/of uridine-auxotroof. De OMPD-negatieve mutanten kunnen dan
gecomplementeerd worden met een werkend OMPD gen waardoor ze opnieuw prototroof
worden.
Er werd getracht OMPD-negatieve mutanten van M. gramineum te creëren via UV-mutatie en
daaropvolgende filtratie-aanrijking. Ook werd een knock-out vector ontwikkeld om het
OMPD gen van M. gramineum uit te schakelen via homologe recombinatie.
3.1. Ultraviolet (UV) mutatie en filtratieaanrijking
De isolatie van auxotrofe mutanten van filamenteuze schimmels via klassieke mutagenese en
filtratieaanrijking werd reeds vaak toegepast in de literatuur. Hier zal de methode gebruikt
worden voor de isolatie van OMPD-negatieve mutanten die uracil en/of uridine auxotroof
zijn. In de literatuur worden OMPD-negatieve mutanten echter meestal geïsoleerd door
directe selectie op 5-FOA medium waarna de 5-FOA resistente kolonies getest worden op
auxotrofie. Vaak wordt hierbij slechts een laag percentage auxotrofe OMPD-mutanten
bekomen. Het voordeel van de filtratieaanrijking in vergelijking met directe selectie op 5FOA medium is dat er een verhoogd aantal auxotrofen zijn onder de 5-FOA resistente
kolonies.
Het principe van de filtratieaanrijking steunt op het feit dat wanneer gemuteerde sporen
worden geënt in vloeibaar minimaal medium zonder uracil en uridine, de auxotrofen niet
kunnen groeien. Door na een bepaalde periode de cultuur te filteren zullen de gegroeide
prototrofen achterblijven op de filter, terwijl de ongekiemde sporen zich in het filtraat
bevinden. Het filtraat kan dan opnieuw geïncubeerd worden waarna opnieuw gefilterd wordt.
Na elke filtratieronde wordt het filtraat rijker aan auxotrofe mutanten.
10 mL van een sporenoplossing van 107 sporen/mL van M. gramineum werd bestraald met
UV licht gedurende 6 minuten. De overleving werd bepaald door stalen van 200 µL te nemen
vóór de behandeling en na 6 minuten UV-behandeling. De stalen werden in verschillende
verdunningen (10-1 tot 10-4) uitgeplaat op compleet PDA medium (in tweevoud) en na twee
dagen werd de overleving bepaald door het aantal kolonies per plaat te tellen. Het percentage
overleving wordt berekend door per verdunning het aantal getelde kolonies bij de behandelde
stalen te delen door het aantal kolonies bij de niet-behandelde stalen. De resultaten zijn
weergegeven in Tabel 42.
IV. Resultaten en Bespreking - 112
MANU DE GROEVE
Tabel 42. Overleving na UV-mutatie: getelde kolonies
Verdunning
Behandeling
10-4
10-3
10-2
10-1
16
11
488
0 minuten
20
18
451
Gemiddelde
18
15
470
6 minuten
Gemiddelde
Overleving
-
8
7
8
52%
208
215
212
45%
1020
960
990
Hieruit blijkt dat de overleving na de UV-mutatie 45-52% bedroeg. Dit is een ideaal
afstervingspercentage indien de gemuteerde sporen worden gebruikt voor een
filtratieaanrijking (Bos & Stadler, 1996).
De filtratieaanrijking werd uitgevoerd zoals eerder beschreven. De vloeibare cultuur werd in
totaal drie maal gefilterd over Miracloth waarna het filtraat verdeeld werd over vier falcons.
De sporen in de falcons werden afgecentrifugeerd en heropgelost in 1 mL fysiologische
oplossing. Van één falcon werd twee keer 100 µL uitgeplaat op SCM (= minimaal medium
met uracil en uridine) en SMM (minimaal medium zonder uracil en uridine). Door stalen van
het filtraat uit te platen op SCM kan het aantal leefbare sporen (prototrofen + auxotrofen) na
de aanrijking bepaald worden. Van de stalen die uitgeplaat werden op SMM zullen enkel de
prototrofen kunnen groeien. Door het aantal kolonies op SCM en SMM te vergelijken kan een
schatting gemaakt worden van het aantal uracil/uridine-auxotrofen.
Er werd een duidelijk verschil waargenomen in het aantal kolonies op SCM en SMM
medium. De platen waren echter niet telbaar omdat er meer dan 500 kolonies gegroeid waren.
Het zichtbaar verschil tussen de SCM en SMM platen wijst er in ieder geval op dat er
effectief een aanrijking aan uracil/uridine-auxotrofe mutanten gebeurd was. Er waren echter
nog steeds veel prototrofe kolonies aanwezig. De sporen in de falcon werden 10x en 100x
verdund en uitgeplaat op selectief medium (TO+) dat 5-FOA en uracil bevat. Dit selectief
medium laat theoretisch enkel groei toe van uracil-auxotrofe kolonies die OPRT- en/of
OMPD-negatief zijn. Er werden zes TO+ platen geënt met volgende stalen:
- drie platen werden geënt met 100 µL van de 10x verdunning
- drie platen werden geënt met 100 µL van een 100x verdunning
Er werden ook vier SMM (= zonder uracil/uridine) platen geënt met dezelfde stalen:
- twee platen werden geënt met 100 µL van de 10x verdunning
- twee platen werden geënt met 100 µL van een 100x verdunning
Na vijf dagen werden de kolonies geteld op de TO+ en SMM platen. Enkel de platen van de
van de 100x verdunning waren telbaar. De resultaten zijn weergegeven in Tabel 43.
MANU DE GROEVE
IV. Resultaten en Bespreking - 113
Tabel 43. Aantal getelde sporen op TO+ en SMM medium
Aantal getelde kolonies
TO+
SMM
Plaat 1
165
176
Plaat 2
192
229
Plaat 3
252
Gemiddelde
203
203
Op de vijfde dag na het uitplaten werden 20 kolonies van de TO+ platen met een tandenstoker
overgeënt naar minimaal medium (SMM) om te onderzoeken of dit inderdaad auxotrofe
mutanten zijn. Op de zesde dag na het uitplaten konden de eerste gesporuleerde kolonies
geobserveerd worden op de TO+ platen. Van deze kolonies werden er 13 overgeënt naar
SMM. Na incubatie gedurende vijf dagen bleken echter alle overgeënte kolonies verder te
groeien op SMM. Aangezien SMM medium geen uracil en uridine bevat betekent dit dat geen
enkele van de overgeënte kolonies auxotroof was.
Tien dagen na het uitplaten waren op de TO+ platen praktisch alle kolonies gesporuleerd. Een
aantal kolonies vertoonde echter geen sporulatie. Er werden 22 van deze kolonies overgeënt
naar SCM en SMM om te testen op auxotrofie. Na vijf dagen vertoonden alle kolonies groei
op het minimale medium. Eén kolonie groeide echter duidelijk veel beter op SCM dan op het
minimale SMM medium. Deze kolonie werd opnieuw overgeënt op zowel SMM als SCM.
Tot vijf dagen na het uitplaten werd even goede groei op SMM als SCM medium vastgesteld.
Hierna viel de groei op het minimale SMM medium stil, terwijl de kolonie op SCM verder
bleef groeien. Na 14 dagen bestreek de kolonie op SCM reeds een oppervlakte van ongeveer
20 cm² (diameter = 5 cm), terwijl de kolonie op SMM slechts 1.8 cm² (diameter 1.5 cm)
bestreek. Omdat er toch nog groei optreedt op SMM is dit echter geen 100% auxotrofe
mutant. Deze kan dus niet gebruikt worden voor transformatie met het wild type OMPD gen
aangezien de niet-getransformeerde kolonie kan groeien op medium zonder uracil en uridine.
Er werd dus niet in geslaagd auxotrofe OMPD-negatieve mutanten te bekomen via UVmutatie en daaropvolgende filtratieaanrijking. Op de 5-FOA platen bleken nog vele prototrofe
kolonies te groeien. Ook in de literatuur worden slechts lage frequenties auxotrofe OMPDnegatieve mutanten gevonden op 5-FOA medium (zie Literatuurstudie, Tabel 5, p.21). Rose et
al. (2000) vonden slechts 6.2% uracil-auxotrofen onder de 5-FOA resistente kolonies bij
Aureobasidium pullulans. Gelijkaardige resultaten werden bekomen door Smit & Tudzynski
(1992) bij Claviceps purpurea (12.5%). Een mogelijke reden voor het feit dat hier geen
geschikte mutanten werden gevonden is dus dat er onvoldoende kolonies gecontroleerd
werden op auxotrofie. Niet tegenstaande er een filtratie-aanrijking werd uitgevoerd zijn er
nog steeds veel prototrofen aanwezig in het filtraat wat er op wijst dat het protocol nog niet
voldoende geoptimaliseerd is voor M. gramineum. Mogelijk zijn er nog meer filtratierondes
nodig om alle prototrofen te verwijderen. Hierbij moet er wel op gelet worden dat de
eventueel aanwezige auxotrofe mutanten niet afsterven.
MANU DE GROEVE
IV. Resultaten en Bespreking - 114
3.2. Constructie van een OMPD knock-out vector
3.2.1. Principe
Door transformatie van een schimmel met een mutant OMPD gen kunnen met een hoge
efficiëntie mutanten gecreëerd worden die OMPD-negatief zijn. In dit onderzoek is het echter
de bedoeling een mutant te creëren die geschikt is voor expressiestudies. Bij zulke studies is
het belangrijk dat de expressiecassette op een gedefinieerde plaats in het genoom integreert in
single-copy, en liefst aan een hoge frequentie. Gouka et al. (1995) ontwikkelde een strategie
gebaseerd op mutante pyrG genen om een hoge frequentie aan single-copy transformanten te
bekomen. Hierbij dienen twee knock-out vectoren geconstrueerd te worden: een vector met
een mutatie in het OMPD gen aan het 5’ einde en een vector met een mutatie in het OMPD
gen aan het 3’ einde. Wanneer men knock-outs maakt met de ene soort vector (bijvoorbeeld
met de mutatie aan het 3’ einde) en die bij latere experimenten complementeert met de andere
soort vector (bijvoorbeeld met de mutatie aan het 5’ einde), dan wordt het gen hersteld. Met
een dergelijk systeem wordt de kans op integratie van 1 kopij van de vector op die specifieke
plaats verhoogd. Gouka et al. (1995) verkregen op deze manier tot 35% single-copy
transformanten bij A. awamori. Deze strategie zal ook gevolgd worden voor de constructie
van een OMPD-negatieve mutant van M. gramineum die kan gebruikt worden voor
expressiestudies.
In deze scriptie werd enkel een knock-out vector met een 3’ mutatie in het OMPD gen
gecreëerd. De knock-out vector werd geconstrueerd op basis van het gekloneerde OMPD gen
van M. gramineum in het pOV plasmide.
3.2.2. Strategie
Voor het creëren van de 3’ mutatie werd gezocht naar sterk geconserveerde stukken, omdat de
kans op inactivatie van het OMPD enzym dan groter is. Gouka et al. (1995) creëerde voor de
3’ strategie een mutatie 0.6 kb downstream van het startcodon die een frameshift in het
OMPD gen veroorzaakt.
Er werd gezocht naar een gelijkaardige mutatie voor het OMPD gen van Myrothecium
gramineum. Door het pOV plasmide te knippen met Eco47III, SpeI en StuI en de fragmenten
van 4202 bp en 783 bp te ligeren wordt zowel een frameshift als een deletie van 67 bp
gecreëerd aan de 3’ kant van het OMPD gen. De afstand tot het startcodon bedraagt 0.5 kb.
De gevolgde strategie is schematisch weergegeven in Figuur 38.
IV. Resultaten en Bespreking - 115
MANU DE GROEVE
OMPD M. gramineum
(2050 bp)
pOV
Eco 47III (1203)
StuI (1270)
(5052 bp)
CREATIE VAN 67 bp DELETIE +
FRAMESHIFT MUTATIE
- knippen met Eco47III, SpeI en StuI
resulteert in drie fragmenten: 4202 bp,
783 bp en 67 bp
Mutant OMPD
M. gramineum
(1983 bp)
pOV3'mut
- ligatie van 4202 bp en 738 bp
fragmenten resulteert in deletie van 67 bp
en frameshift mutatie
SpeI (2055)
67 bp deletie
(4985 bp)
SpeI (1988)
Figuur 38. Gevolgde strategie voor de creatie van een OMPD knock-out vector met een mutatie aan de 3’ kant
3.2.3. Constructie van een OMPD knock-out vector met een mutatie aan het 3’ einde
Het pOV plasmide dat 2050 bp van het wild type OMPD gen van M. gramineum bevat werd
eerst geknipt (2 µg) met Eco47III (AGC^GCT). Dit leverde een lineair fragment van 5052 bp
op met stompe uiteinden. Het restrictiefragment werd vervolgens opgezuiverd met de DNAce
Quick-Clean (Bioline, Gentaur) en geknipt met SpeI (A^CTAGT). Op deze manier werden
twee fragmenten bekomen van 4202 bp en 850 bp. Het restrictiemengsel werd opgezuiverd en
geknipt met StuI (AGG^CCT). Dit restrictie-enzym knipt in het 850 bp fragment. In Figuur 39
is het resultaat van de restrictie weergegeven op een 2% agarosegel. In totaal werden drie
fragmenten bekomen van volgende groottes: 4202 bp, 783 bp en 67 bp (=deletie). Het 67 bp
fragment is niet zichtbaar.
M
1
2
- 4202 bp
Laan M: kleine lengtemerker (SmartLadder SF)
Laan 1: pOV plasmide geknipt met Eco47III, SpeI
Laan 2: pOV plasmide geknipt met Eco47III, SpeI en StuI
-850 bp
-783 bp
Figuur 39. pOV plasmide geknipt met Eco47III, SpeI en StuI
De fragmenten van 4202 bp en 783 bp werden opgezuiverd na een preparatieve
gelelektroforese. Beide fragmenten bevatten elk één stomp uiteinde en één overhang en
kunnen dus direct geligeerd worden in de juiste oriëntatie. Na ligatie van beide fragmenten
werd E. coli DH5α−F’ getransformeerd met het ligatiemengsel. De plasmiden werden
geïsoleerd uit E. coli en gecontroleerd via een controlerestrictie. Voor de controlerestrictie
werden de knock-out plasmiden en het oorspronkelijke pOV plasmide geknipt met NsiI en
Eco47III. Theoretisch moeten dan voor de knock-out vector fragmenten van 467 bp, 2565 bp,
IV. Resultaten en Bespreking - 116
MANU DE GROEVE
en 2953 bp bekomen worden, en voor het pOV plasmide 440 bp, 467 bp, 1192 bp en 2953 bp.
Het resultaat van de controlerestrictie is weergegeven in Figuur 40.
1
2
3
4
5
M
Laan 1-5: Knock-out plasmiden geknipt met Eco47III en
NsiI
7
- 3420 bp
- 2953 bp
Laan M: lengtemerker (SmartLadder)
- 2032 bp
- 1565 bp
- 1192 bp
Laan 7: pOV plasmide geknipt met Eco47III en NsiI
Figuur 40. Knock-out vector en pOV plasmide geknipt met Eco47III en NsiI
Zoals uit de figuur blijkt was de controlerestrictie in de lanen 1-4 gelukt. Er werden
fragmenten bekomen van de verwachte groottes. Er waren ook fragmenten zichtbaar met
groottes die kunnen verklaard worden door onvolledige restrictie: bijvoorbeeld 3420 bp (2953
+ 467) en 2032 bp (1565 + 467 bp). De fragmenten van 467 bp en 440 bp zijn niet zichtbaar
op de foto.
De gecreëerde OMPD knock-out vector met een mutatie in het 3’ einde van het OMPD gen
wordt pOV3’mut genoemd en is 4985 bp groot. De mutatie bestaat uit een deletie van 67 bp
en een frameshift-mutatie. Het pOV3’mut plasmide kan gebruikt worden voor de creatie van
een OMPD-negatieve mutant van M. gramineum via transformatie. De gecreëerde mutant zal
gebruikt kunnen worden voor expressiestudies.
V. Besluit - 117
MANU DE GROEVE
V. BESLUIT
Door een combinatie van gedegenereerde PCR en PCR-gebaseerde genomewalking werd er
in geslaagd 2918 bp van het OMPD gen van Myrothecium gramineum MUCL 39210 te
sequeneren. Voor het eerst werd de promotor, de volledige coderende sequentie en de 3’
onvertaalde regio van een gen van deze schimmel gesequeneerd en gekarakteriseerd. Via
reverse transcriptase PCR kon de aanwezigheid van een intron in het OMPD gen aangetoond
worden. Hiermee werd ook het bewijs geleverd dat het OMPD gen tot expressie komt in M.
gramineum.
Het OMPD gen van de Sordariomyceet M. gramineum en de afgeleide aminozuursequentie
vertoont homologie met de OMPD genen en eiwitten van andere filamenteuze schimmels (tot
79% similariteit). Verrassend genoeg bleek de genstructuur en het resulterende eiwit van
OMPD meer gelijkaardig te zijn aan de OMPD sequenties van niet-Sordariomyceten. Het
OMPD gen van M. gramineum bevat namelijk een intron wat nooit eerder werd waargenomen
bij OMPD genen van Sordariomyceten. Ook is het OMPD enzym van M. gramineum
ongeveer 100 aminozuren korter dan alle andere bekende OMPD enzymen van
Sordariomyceten. Fylogenetische analyse op basis van de OMPD aminozuursequenties van
filamenteuze schimmels classificeert M. gramineum niet onder de Sordariomyceten, wat in
tegenspraak is met de huidige taxonomie.
Een fragment van 2050 bp van het OMPD gen van M. gramineum werd geligeerd in pGEM®T. Het resulterende pOV plasmide werd gekloneerd in E. coli. De functionaliteit en werking
van het OMPD gen in het pOV plasmide werd aangetoond door de complementatie van
Aspergillus nidulans FGSC A722, een uracil/uridine auxotrofe OMPD-negatieve mutant.
Voor het eerst werd een gen van Myrothecium gramineum heteroloog tot expressie gebracht
in een filamenteuze schimmel. Hiermee werd ook bewezen dat het OMPD gen van M.
gramineum kan gebruikt worden als selectiemerker in filamenteuze schimmels.
Er werd nog niet in geslaagd een OMPD-negatieve mutant van M. gramineum te creëren. De
UV-mutatie en filtratieaanrijking bleken nog niet voldoende geoptimaliseerd voor M.
gramineum. Voor de creatie van een mutant via transformatie werd een OMPD knock-out
vector geconstrueerd die een mutatie bevat aan de 3’ kant van het OMPD gen van M.
gramineum.
Er werd een kolonie-PCR protocol voor M. gramineum ontwikkeld voor de amplificatie van
fragmenten tot meer dan 2 kb. Het kolonie-PCR protocol bleek ook te werken voor
transformanten van A. nidulans, zij het minder efficiënt. Het kolonie-PCR protocol zal
gebruikt kunnen worden als snelle methode voor de controle van transformanten van M.
MANU DE GROEVE
V. Besluit - 118
gramineum. Meer bepaald zal het integratiepatroon van het getransformeerde DNA kunnen
bepaald worden en zullen single-copy mutanten gedetecteerd kunnen worden.
Het kloneren van het homologe OMPD gen van M. gramineum heeft de deur geopend voor
verder genetisch onderzoek bij deze filamenteuze schimmel. Het OMPD gen zal in de eerste
plaats gebruikt kunnen worden voor de constructie van knock-out vectoren om zo een
gedefinieerde OMPD-negatieve mutant te creëren. Het OMPD gen kan ook gebruikt worden
voor het bekomen van hoge frequenties aan single-copy transformanten die nuttig zijn bij
expressiestudies.
Uiteindelijk zal het OMPD gen van M. gramineum gebruikt kunnen worden als
selectiemerker in transformatie- en expressiesystemen voor deze schimmel en andere
filamenteuze schimmels. Myrothecium gramineum zal dan als expressiegastheer gebruikt
kunnen worden voor de overproductie van industrieel interessante enzymen die hun
toepassing vinden in allerhande industrieën zoals bijvoorbeeld de voedingsindustrie en
farmaceutische industrie.
VI. Samenvatting - 119
MANU DE GROEVE
VI. SAMENVATTING
KLONERING EN GENEXPRESSIE IN DE SCHIMMEL
MYROTHECIUM GRAMINEUM
In hun natuurlijke omgeving zijn filamenteuze schimmels in staat een grote variëteit aan
enzymen te produceren in grote hoeveelheden. Dit maakt hen aantrekkelijk als
expressiegastheren voor de productie van recombinante eiwitten. Tot op heden werden
hiervoor slechts een beperkt aantal filamenteuze schimmels onderzocht en bovendien zijn
vele expressiegastheren beschermd via patenten.
In een preliminair onderzoek werd een screening uitgevoerd bij 3000 verschillende fungale
soorten naar nieuwe fungale transformatie- en expressiegastheren (Jonniaux et al., 2004).
Myrothecium gramineum MUCL 39210 vertoonde alle gewenste karakteristieken voor een
performante kloneringsgastheer. Er werd een transformatieprotocol voor deze schimmel
ontwikkeld en geoptimaliseerd met een antibioticaresistentie gen als selectiemerker. Omdat
het gebruik van antiobioticaresistentie-genen in de voedingsindustrie vermeden wordt, werd
gezocht naar andere merkers. Het orotidine-monofosfaat decarboxylase (OMPD) gen werd
reeds vaak gebruikt als selectiemerker bij fungi en zal ook voor M. gramineum gebruikt
worden.
In deze scriptie wordt de klonering en sequenering van het volledige OMPD gen van de
filamenteuze schimmel Myrothecium gramineum MUCL 39210 gerapporteerd. Dit is het
eerste volledige gen dat gekloneerd werd in deze Ascomycete schimmel. De OMPD sequentie
werd geanalyseerd en vertoont homologie met andere OMPD sequenties. Het OMPD enzym
van M. gramineum vertoont verrassend genoeg slechts weinig similariteit met andere
Sordariomyceten, een klasse waartoe ook het Myrothecium genus behoort. Het OMPD gen
van M. gramineum is het eerste OMPD gen van een Sordariomyceet dat een intron bevat en
de grote aminozuurinsertie die voorkomt bij andere Sordariomyceten, niet bevat.
De OMPD-negatieve Aspergillus nidulans FGSC A722 kon gecomplementeerd worden met
het OMPD gen van M. gramineum. Dit betekent dat het OMPD gen codeert voor een
functioneel enzym en bijgevolg kan gebruikt worden als selectiemerker. Voor het eerst werd
een gen van M. gramineum heteroloog tot expressie gebracht.
Er werd niet in geslaagd een OMPD-negatieve mutant van M. gramineum te creëren via
ultraviolet-mutatie en daaropvolgende filtratieaanrijking. Voor de creatie van een mutant via
transformatie werd een OMPD knock-out vector geconstrueerd die een mutatie bevat aan de
3’ kant van het OMPD gen van M. gramineum. Tot slot werd een kolonie-PCR protocol
ontwikkeld voor M. gramineum voor de snelle identificatie van de gewenste transformanten.
VII. Summary - 120
MANU DE GROEVE
VII. SUMMARY
CLONING AND GENE EXPRESSION IN THE
FILAMENTOUS FUNGUS MYROTHECIUM GRAMINEUM
In their natural environment filamentous fungi secrete a wide range of enzymes in high
amounts. This makes them attractive as expression hosts for large scale recombinant protein
production. To date, only a limited number of filamentous fungi has been explored for this
purpose and most of the expression hosts are covered by patent applications.
In a preliminary study, a screening procedure for a new fungal cloning and expression host
involving 3000 fungal species and strains was performed (Jonniaux et al., 2004).
Myrothecium gramineum MUCL 39210 seemed to present all the characteristics needed for a
performing cloning host. A transformation system for this fungus was developed and
optimised using an antibiotic resistance selection marker. For applications in the food
industry the use of antibiotic resistance genes is avoided and therefore other selection markers
were searched for. The orotidine monophosphate decarboxylase (OMPD) gene is widely used
in fungi as a selection marker and will also be used for a M. gramineum transformation
system.
In this study, the cloning and sequencing of the complete orotidine monophosphate
decarboxylase (OMPD) gene of the filamentous fungus Myrothecium gramineum MUCL
39210 is reported. This is the first full-length gene cloned in this fungus. Analysis of the
OMPD sequence revealed strong homology to other fungal OMPD sequences. Surprisingly,
the OMPD enzyme of M. gramineum shows low levels of similarity with other
Sordariomycetes. The OMPD gene of M. gramineum is the first OMPD gene of a
Sordariomycete harbouring an intron and lacking the large amino acid insert seen with other
Sordariomycetes.
The OMPD gene of M. gramineum was able to complement the OMPD-negative Aspergillus
nidulans FGSC A722, meaning the OMPD gene codes for a functional enzyme and
subsequently can be used as a selection marker in filamentous fungi. This is the first report of
the heterologous expression of a gene from M. gramineum.
For the creation of an OMPD-negative M. gramineum strain, a method involving ultraviolet
light mutagenesis and subsequent filtration enrichment was unsuccessful. For the creation of a
mutant strain by transformation, an OMPD knock-out vector carrying a mutation near the 3’
region of the OMPD gene of M. gramineum was created. Finally, a colony-PCR protocol for
M. gramineum was developed for the fast detection of the desirable transformants.
MANU DE GROEVE
VIII. Literatuurlijst - 121
VIII. LITERATUURLIJST
AGNAN, J., KORCH, C. & SELITRENNIKOFF, C. (1997). Cloning heterologous genes: problems and
approaches. Fungal Genetics and Biology, 21, 292-301.
AHRAZEM, O., GÓMEZ-MIRANDA, B., PRIETO, A., BERNABÉ, M. & LEAL, J.A. (2000). Heterogeneity
of the genus Myrothecium as revealed by cell wall polysaccharides. Archives of Microbiology, 173, 296-302.
ALANI, E., CAO, L. & KLECKNER, N. (1987). A method for gene disruption that allows repeated use of
URA3 selection in the construction of multiply disrupted yeast strains. Genetics, 116, 541-545.
ALEKSENKO, A. & IVANOVA, L. (1998). In vivo linearization and autonomous replication of plasmids
containing human telomeric DNA in Aspergillus nidulans. Molecular and General Genetics, 260, 159-164.
APPLEGATE, P.J., NELSON, R.E. & METZENBERG, R.L. (1978). Mutant enrichment by filtration
concentration: a variation for the selection of temperature-conditional heterocaryons. Neurospora Newsletter,
25, 17.
ARCHER, D.B., JEENES, D.J., MACKENZIE, D. A., BRIGHTWELL, M., LAMBERT, N., LOWE, G.,
RADFORD, S.E. & DOBSON, C.M. (1990). Hen egg white lysozyme expressed in and secreted from
Aspergillus niger is correctly processed and folded. Bio/Technology, 8, 741-745.
ARCHER, D.B. (1994). Enzyme production by recombinant Aspergillus. In: Murooka, Y. & Imanaka, T. (eds.)
Recombinant microbes for industrial and agricultural applications. New York, Marcel Dekker, 373-393.
ASCH, D.K. & KINSEY, J.A. (1990). Relationship of vector insert size to homologous integration during
transformation of Neurospora crassa with the cloned am (gdh) gene. Molecular and General Genetics, 221, 3743.
BAHLER, J., WU, J.Q., LONGTINE, M.S., SHAH, N.G., MCKENZIE, A., STEEVER, A.B., WACH, A.,
PHILIPPSEN, P. & PRINGLE, J.R. (1998). Heterologous modules for efficient and versatile PCR-based gene
targeting in Schizosaccharomyces pombe. Yeast, 14, 943-951.
BALLANCE, D.J., BUXTON, F.P. & TURNER, G. (1983). Transformation of Aspergillus nidulans by the
orotidine-5’-phosphate decarboxylase gene of Neurospora crassa. Biochemical and Biophysical Research
Communications, 112, 284-289.
BALLANCE, D.J. (1986). Sequences important for gene expression in filamentous fungi. Yeast, 2, 229-236.
BEJ, A.K. & PERLIN, M.G. (1989). A high efficiency transformation system for the basidiomycete Ustilago
violacea employing hygromycin resistance and lithium-acetate treatment. Gene, 80, 171-176.
BENITO, E.P., DÍAZ-MÍNGUEZ, J.M., ITURRIAGA, E.A., CAMPUZANO, V. & ESCLAVA, A.P. (1992).
Cloning and sequence analysis of the Mucor circinelloides pyrG gene encoding orotidine-5’-monophosphate
decarboxylase: use of pyrG for homologous transformation. Gene, 116, 59-67.
BENNETT, J.W. (1998). Mycotechnology: the role of fungi in biotechnology. Journal of Biotechnology, 66,
101–107.
BINNINGER, D.M., SKRZYNIA, C., PUKKILA, P.J. & CASSELTON, L.A. (1987). DNA-mediated
transformation of the basidiomycete Coprinus cinereus. EMBO Journal, 6, 835-840.
BOEKE, J.D., LACROUTE, F. & FINK, G.R. (1984). A positive selection for mutants lacking orotidine-5’phosphate decarboxylase activity in yeast: 5-fluoro-orotic acid resistance. Molecular and General Genetics, 197,
345–346.
MANU DE GROEVE
VIII. Literatuurlijst - 122
BOLDT, R. & ZRENNER, R. (2003). Purine and pyrimidine biosynthesis in higher plants. Physiologia
Plantarum, 117, 297-304.
BOS, C.J. & STADLER, D. (1996). Mutation. In: Bos, C.J. (ed.) Fungal Genetics: principles and practice.
Marcel Dekker, New York, p. 31-35.
BRAKHAGE, A.A., ANDRIANOPOULOS, A., KATO, M., STEIDL, S., DAVIS, M.A., TSUKAGOSHI, N. &
HYNES, M.J. (1999). HAP-Like CCAAT-Binding Complexes in Filamentous Fungi: Implications for
Biotechnology. Fungal Genetics and Biology, 27, 243-252.
BRAKHAGE, A.A. & LANGFELDER, K. (2002). Menacing mold: the molecular biology of Aspergillus
fumigatus. Annual Review of Microbiology, 56, 433-455.
BREATHNACH, R. & CHAMBON, P. (1981). Organization and expression of eukaryotic split genes coding
for proteins. Annual Review of Biochemistry, 50, 349-383.
BRUCHEZ, J.J.P., EBERLE, J. & RUSSO, V.E.A. (1993a). Regulatory sequences involved in the translation of
Neurospora crassa mRNA: Kozak sequences and stop codons. Fungal Genetics Newsletter, 40, 85-88.
BRUCHEZ, J.J.P., EBERLE, J. & RUSSO, V.E.A. (1993b). Regulatory sequences in the transcription of
Neurospora crassa genes: CAAT box, TATA box, introns, poly(A) tail formation sequences. Fungal Genetics
Newsletter, 40, 89-96.
BUXTON, F.P. & RADFORD, A. (1983). Cloning of the structural gene for orotidine 5'-phosphate carboxylase
of Neurospora crassa by expression in Escherichia coli. Molecular and General Genetics, 190, 403-405.
BUXTON, F.P., GWYNNE, D.I. & DAVIES, R.W. (1985). Transformation of Aspergillus niger using the argB
gene of Aspergillus nidulans. Gene 37, 207-214.
BUXTON, F.P., GWYNNE, D.I. & DAVIES, R.W. (1989). Cloning of a new bidirectionally selectable marker
for Aspergillus strains. Gene, 84, 329-334.
CALMELS, T.P.G., MARTIN, F., DURAND, H. & TIRABY, G. (1991). Proteolytic events in the processing of
secreted proteins in fungi. Journal of Biotechnology, 17, 51-66.
CAMPANELLA, J.J., BITINCKA, L. & SMALLEY, J. (2003). MatGAT: an application that generates
similarity/identity matrices using protein or DNA sequences. BMC Bioinformatics, 4, 29.
CARREZ, D., JANSSENS, W., DEGRAVE, P., VAN DEN HONDEL, C.A.M.J.J., KINGHORN, J.R., FIERS,
W. & CONTRERAS, R. (1990). Heterologous gene expression by filamentous fungi: secretion of human
interleukin-6 by Aspergillus nidulans. Gene, 94, 147-154.
CASE, M. (1963). Procedure for filtration-concentration experiments. Neurospora Newsletter, 3, 7-8.
CASE, M.E., SCHWEIZER, M., KUSHNER, S.R. & GILES, N.H. (1979). Efficient transformation of
Neurospora crassa by utilizing hybrid plasmid DNA. Proceedings of the National Academy of Sciences of the
United States of America, 76, 5259-5263.
CASSIOLATA, A.M.R. & SOARES DE MELO, I. (1999). Filtration enrichment method for isolation of
auxotrophic mutants of Trichoderma harzianum rifai. Revista de Microbiologia, 30, 43-46.
CASTLEBURY, L.A., ROSSMAN, A.Y., SUNG, G.H., HYTEN, A.S. & SPATAFORA, J.W. (2004).
Multigene phylogeny reveals new lineage for Stachybotrys chartarum, the indoor air fungus. Mycological
Research, 108, 864–872.
CHAKRABORTY, B.N., PATTERSON, N.A. & KAPOOR, M. (1991). An electroporation-based system for
high-efficiency transformation of germinated conidia of filamentous fungi. Canadian Journal of Microbiology,
37, 858-863.
MANU DE GROEVE
VIII. Literatuurlijst - 123
CHOUINI-LALANNE, N., MALET-MARTINO, M. C., MARTINO, R. & MICHEL, G. (1989). Study of the
metabolism of flucytosine in Aspergillus species by 19F nuclear magnetic resonance spectroscopy. Antimicrobial
Agents and Chemotherapy, 33, 1939-1945.
CLEMENTS, J.M. & ROBERTS, C.F. (1986). Transcription and processing signals in the 3-phosphoglycerate
kinase (pgk) gene from Aspergillus nidulans. Gene, 44, 97-105.
COLLETT, M.A., BRADSHAW, R.E. & SCOTT, D.B. (1995). A mutualistic fungal symbiont of perennial
ryegrass contains two different pyr4 genes, both expressing orotidine-5'-monophosphate decarboxylase. Gene,
158, 31-39.
CONESA, A., PUNT, P.J., VAN LUIJK, N. & VAN DEN HONDEL, C.A.M.J.J. (2001). The secretion pathway
in filamentous fungi: a biotechnological view. Fungal Genetics and Biology, 33, 155–171.
DABOUSSI, M.J., DJEBALLI, A., GERLINGER, C., BLAISEAU, P.L., BOUVIER, I., CASSAN, M.,
LEBRUN, M.H., PARISOT, D. & BRYGOO, Y. (1989). Transformation of seven species of filamentous fungi
using the nitrate reductase gene of Aspergillus nidulans. Current Genetics, 15, 453-456.
DALBEY, R.E. & VON HEIJNE, G. (1992). Signal peptidase in prokaryotes and eukaryotes - a new protease
family. Trends in Biochemistry, 17, 474-478.
DE GROOT, M.J.A., BUNDOCK, P., HOOYKAAS, P.J.J. & BEIJERSBERGEN, A.G.M. (1998).
Agrobacterium tumefaciens-mediated transformation of filamentous fungi. Nature Biotechnology, 16, 839-842.
D'ENFERT, C. (1996). Selection of multiple disruption events in Aspergillus fumigatus using the orotidine-5'decarboxylase gene, pyrG, as a unique transformation marker. Current Genetics, 30, 76-82.
DHAWALE, S.S., PAIETTA, J.V. & MARZLUF, G.A. (1984). A new rapid and efficient transformation
procedure for Neurospora crassa. Current Genetics, 8, 77-79.
DÍAZ-MÍNGUEZ, J.M., ITURRIAGA, E.A., BENITO, E.P., CORROCHANO, L.M. & ESLAVA, A.P. (1990).
Isolation and molecular analysis of the orotidine-5'-phosphate decarboxylase gene (pyrG) of Phycomyces
blakesleeanus. Molecular and General Genetics, 224, 269-278.
DOMSCH, K.H., GAMS, W. & ANDERSON, T.H. (1980). Compendium of soil fungi. Volume 1. London,
Academic Press.
DUNN-COLEMAN, N.S., BLOEBAUM, P., BARKA, M., BODIE, E., ROBINSON, N., ARMSTRONG, G.,
WARD, M., PRZETAK, M., CARTER, G.L., LAMSA, M. & HIENSOHN, H. (1991). Commercial levels of
chymosin production by Aspergillus. Molecular and General Genetics, 230, 288-294.
DURAND, H., CLANET, M., & TIRABY, G. (1988). Genetic improvement of Trichoderma reesei for large
scale cellulase production. Enzyme and Microbial Technology, 10, 341-345.
FARR, D.F., ROSSMAN, A.Y., PALM, M.E. & MCCRAY, E.B. (2004). Fungal Databases. Systematic Botany
and Mycology Laboratory, ARS, USDA. http://nt.ars-grin.gov/fungaldatabases/.
FIERRO, F., LAICHA, F., GARCÍA-RICO, R.O. & MARTÍN, J.F. (2004). High efficiency transformation of
Penicillium nalgiovense with integrative and autonomously replicating plasmids. International Journal of Food
Microbiology, 90, 237-248.
FINCHAM, J.R.S. (1989). Transformation in fungi. Microbiological Reviews, 53, 148-170.
FINKELSTEIN, D.B., RAMBOSEK, J., CRAWFORD, M.S., SOLIDAY, C.L., MCADA, P.C. & LEACH, J.
(1989). Protein secretion in Aspergillus niger. In: Hershberger, C.L., Queener, S.W. & Hegeman, G. (eds.)
Genetics and molecular biology of industrial microorganisms. Washington, American Society of Microbiology,
295-300.
GEMS, D., JOHNSTONE, I.L. & CLUTTERBUCK, A.J. (1991). An autonomously replicating plasmid
transforms Aspergillus nidulans at high frequency. Gene, 98, 61-67.
MANU DE GROEVE
VIII. Literatuurlijst - 124
GOMES-BARCELLOS, F., PELEGRINELLI-FUNGARO, M.H., FURLANETO, M.C., LEJEUNE, B.,
PIZZIRANI-KLEINER, A.A. & AZEVEDO, J.L. (1998). Genetic analysis of Aspergillus nidulans unstable
transformants obtained by the biolistic process. Canadian Journal of Microbiology, 44, 1137-1141.
GOMI, K., IIMURA, Y. & HARA, S. (1987). Integrative transformation of Aspergillus oryzae with a plasmid
containing the Aspergillus nidulans argB gene. Agricultural and Biological Chemistry, 51, 2549-2555.
GOOSEN, T., BLOEMHEUVEL, G., GYSLER, C., DE BIE, D.A., VAN DEN BROEK, H.W.J. & SWART, K.
(1987). Transformation of Aspergillus niger using the homologous orotidine-5’-phosphate-decarboxylase gene.
Current Genetics, 11, 499-503.
GOUKA, R.J., GERK, C., HOOYKAAS, P.J.J., BUNDOCK, P., MUSTERS, W., VERRIPS, C.T. & DE
GROOT, M.J.A. (1999). Transformation of Aspergillus awamori by Agrobacterium tumefaciens-mediated
homologous recombination. Nature Biotechnology, 17, 598-601.
GOUKA, R.J., HESSING, J.G.M., STAM, H., MUSTERS, W. & VAN DEN HONDEL C.A.M.J.J. (1995). A
novel strategy for the isolation of defined pyrG mutants and the development of a site-specific integration
system for Aspergillus awamori. Current Genetics, 27, 536-540.
GOUKA, R.J., PUNT, P.J., HESSING, J.G.M. & VAN DEN HONDEL, C.A.M.J.J. (1996). Analysis of
heterologous protein production in defined recombinant Aspergillus awamori strains. Applied and
Environmental Microbiology, 62, 1951-1957.
GOUKA, R.J., PUNT, P.J. & VAN DEN HONDEL, C.A.M.J.J. (1997). Efficient production of secreted
proteins by Aspergillus: progress, limitations and prospects. Applied Microbiology and Biotechnology, 47, 1-11.
GRIFFIN, D.H. (1996). Fungal physiology. Second edition. New York, Wiley, 472 p.
GRUBER, F., VISSER, J., KUBICEK, C.P. & DEGRAAFF, L.H. (1990). Cloning of the Trichoderma reesei
pyrG gene and its use as a homologous marker for a high-frequency transformation system. Current Genetics,
18, 447-451.
GURR, S.J., UNKLES, S.E. & KINGHORN, J.R. (1987). The structure and organisation of nuclear genes of
filamentous fungi. In: Kinghorn, J.R. (ed.) Gene Structure in eukaryotes. Oxford, IRL Press, 93-139.
GWYNNE, D.I. (1992). Foreign proteins. In: Kinghorn, J.B. & Turner, G. (eds.) Applied molecular genetics of
filamentous fungi. Glasgow, Blackie Academic and Professional, 132-151.
GWYNNE, D.I., BUXTON, F.P., WILLIAMS, S.A., GARVEN, S. & DAVIES, R.W. (1987). Genetically
engineered secretion of active human interferon and a bacterial endoglucanase from Aspergillus nidulans. BioTechnology, 5, 713-719.
HALL, T.A. (1999). BioEdit: a user-friendly biological sequence alignment editor and analysis program for
Windows 95/98/NT. Nucleic Acids Symposium Series, 41, 95-98.
HAMER, J.E. & TIMBERLAKE, W.E. (1987). Functional organization of the Aspergillus nidulans trpC
promoter. Molecular and Cellular Biology, 7, 2352-2359.
HANAHAN, D., JESSEE, J. & BLOOM, F.R. (1991). Plasmid transformation of Escherichia coli and other
bacteria. Methods in Enzymology, 204,63-113.
HAZELL, B.W., TE'O, V.S., BRADNER, J.R., BERGQUIST, P.L. & NEVALAINEN, K.M. (2000). Rapid
transformation of high cellulaseproducing mutant strains of Trichoderma reesei by microprojectile
bombardment. Letters in Applied Microbiology, 30, 282-286.
HEERIKHUISEN, M., VAN DEN HONDEL, C.A.M.J.J., PUNT, P., VAN BIEZEN, N., ALBERS, A. &
VOGEL, K. (2001). Novel means of transformation of fungi and their use for heterologous protein production.
World Intellectual Property Organisation (WIPO) Patent WO 01/09352.
MANU DE GROEVE
VIII. Literatuurlijst - 125
HIGGINS, D., THOMPSON, J., GIBSON, T., THOMPSON, J.D., HIGGINS, D.G. & GIBSON, T.J. (1994).
CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence
weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 22, 4673-4680.
HOLLAND, M.J. & HOLLAND, J.P. (1978). Isolation and identification of yeast messenger ribonucleic acids
coding for enolase, glyceraldehyde-3-phosphate dehydrogenase, and phosphoglycerate kinase. Biochemistry, 17,
4900-4907.
HYNES, J. (1996). Genetic transformation of filamentous fungi. Journal of Genetics, 75, 297-311.
JARAI, G. (1997). Heterologous gene expression in filamentous fungi. In: Anke, T. (ed.) Fungal biotechnology.
London, Chapmann and Hall, 251-264.
JEENES, D.J., MACKENZIE, D.A., ROBERTS, I.N. & ARCHER, D.B. (1991). Heterologous protein
production by filamentous fungi. Biotechnology and Genetic Engineering Reviews, 9, 327-367.
JEENES, D., NGIAM, C. & ARCHER, D.B. (1996). Isolation and characterisation of PDI-family genes from
Aspergillus niger. Fungal Genetics Newsletter, 43B, 23.
JEKOSCH, K. & KÜCK, U. (1999). Codon bias in the ß-lactam producer Acremonium chrysogenum. Fungal
Genetics Newsletter, 46, 11-13.
JENKINS, N., PAREKH, R.B. & JAMES, D.C. (1996). Getting the glycosylation right: implications for the
biotechnology industry. Nature Biotechnology, 14, 975-981.
JOHNSON, P.F. & MCKNIGHT, S.L. (1989). Eukaryotic transcriptional regulatory proteins. Annual Review of
Biochemistry, 58, 799-839.
JONNIAUX, J.-L., VALEPYN, E., CORBISIER, A.-M. & DAUVRIN, T. (2004). Myrothecium sp.
transformation and expression system. World Intellectual Property Organisation (WIPO) Patent WO
2004/020611.
JUGE, N., SVENSSON, B. & WILLIAMSON, G. (1998). Secretion, purification, and characterisation of barley
alpha-amylase produced by heterologous gene expression in Aspergillus niger. Applied Microbiology and
Biotechnology, 49, 385-392.
KIMSEY, H.H. & KAISER D. (1992). The orotidine-5’-monophosphate decarboxylase gene of Myxococcus
xanthus. Comparison to the OMP decarboxylase gene family. The Journal of Biological Chemistry, 267, 819824.
KORMAN, D.R., BAYLISS, F.T., BARNETT, C.C., CARMONA, C.L., KODAMA, K.H., ROYER, T.J.,
THOMPSON, S.A., WARD, M., WILSON, L.J. & BERKA, R.M. (1990). Cloning, characterization, and
expression of 2 alpha-amylase genes from Aspergillus niger var awamori. Current Genetics, 17, 203-212.
KINNAIRD, J.H., BURNS, P.A. & FINCHAM, J.R. (1991). An apparent rare-codon effect on the rate of
translation of a Neurospora gene. Journal of Molecular Biology, 221, 733-736.
KOZAK, M. (1986). Point mutations define a sequence flanking the AUG initiator codon that modulates
translation by eukaryotic ribosomes. Cell, 44, 283-292.
KUO, C.-Y., CHOU, S.-Y. & HUANG, C.-T. (2004). Cloning of glyceraldehyde-3-phosphate dehydrogenase
gene and use of the gpd promoter for transformation in Flammulina velutipes. Applied Microbiology and
Biotechnology, 65, 593-599.
KWON-CHUNG, K.J., GOLDMAN, W.E., KLEIN, B. & SZANISZLO, P.J. (1998). Fate of transforming DNA
in pathogenic fungi. Medical Mycology, 36, 38-44.
LAMB, B.C. & HELMI, S. (1991). Filtration-enrichment methods for selecting auxotrophs and other mutants in
Ascobolus immersus and similar filamentous fungi. Fungal Genetics Newsletter, 38, 75-77.
MANU DE GROEVE
VIII. Literatuurlijst - 126
LARSEN, G.G., APPEL, K.F., WOLFF, A.-M., NIELSEN, J. & ARNAU, J. (2004). Characterisation of the
Mucor circinelloides regulated promoter gpd1P. Current Genetics, 45, 225-234.
LONG, D.M., SMIDANSKY, E.D., ARCHER, A.J. & STROBEL, G.A. (1998). In vivo addition of telomeric
repeats to foreign DNA generates extrachromosomal DNAs in the taxol-producing fungus Pestalotiopsis
microspora. Fungal Genetics and Biology, 24, 335-344.
MANCZINGER, L., ANTAL, Z. & FERENCZY, L. (1995). Isolation of uracil auxotrophic mutants of
Trichoderma harzianum and their transformation with heterologous vectors. FEMS Microbiology Letters, 130,
59-62.
MATHIEU, M., FILLINGER, S. & FELENBOK, B. (2000). In vivo studies of upstream regulatory cis-acting
elements of the alcR gene encoding the transactivator of the ethanol regulon in Aspergillus nidulans. Molecular
Microbiology, 36, 123-131.
MATTERN, I.E., UNKLES, S., KINGHORN, J.R., POUWELS, P.H. & VAN DEN HONDEL, C.A.M.J.J.
(1987). Transformation of Aspergillus oryzae using the A. niger pyrG gene. Molecular and General Genetics,
210, 460-461.
MATTERN, I.E., VAN NOORT, J.M., VAN DEN BERG, P., ARCHER, D.B., ROBERT, I.N. & VAN DEN
HONDEL, C.A.M.J.J. (1992). Isolation and characterization of mutants of Aspergillus niger which are deficient
in extracellular proteases. Molecular and General Genetics, 234, 332-337.
MAY, G.S. (1992). Fungal technology. In: Kinghorn, J.R. & Turner, G. (eds.) Applied molecular genetics of
filamentous fungi. Glasgow, Blackie Academic and Professional, 1-27.
MEYER, V., MUELLER, D., STROWIG, T. & STAHL, U. (2003). Comparison of different transformation
methods for Aspergillus giganteus. Current Genetics, 43, 371-377.
MICHIELSE, C.B., ARENTSHORST, M., RAM, A.F.J. & VAN DEN HONDEL, C.A.M.J.J. (2005).
Agrobacterium-mediated transformation leads to improved gene replacement efficiency in Aspergillus awamori.
Fungal Genetics and Biology, 42, 9-19.
MISHRA, N.C. & TATUM, E.L. (1973). Non-mendelian inheritance of DNA-induced inositol independence in
Neurospora. Proceedings of the National Academy of Sciences of the United States of America, 70, 3875-3879.
MORTIMER, P.H., CAMPELL, J., DI MENNA, M.E. & WHITE, E.P. (1971). Experimental
myrotheciotoxicosis and poisoning in ruminants by verrucarin A and roridin A. Research in Veterinary Science,
12, 508–515.
NARA, T., HSHIMOTO, T. & AOKI, T. (2000). Evolutionary implications of the mosaic pyrimidinebiosynthetic pathway in eukaryotes. Gene, 257, 209-222.
NICOT, J. & OLIVRY, C. (1961). Contribution a l’étude du genre Myrothecium Tode. 1. Les éspèces a spores
striées. Revue générale de Botanique, 68, 672-685.
OZEKI, K., KYOYA, F., HIZUME, K., KANDA, A., HAMACHI, M. & NUNOKAWA, Y. (1994).
Transformation of intact Aspergillus niger by electroporation. Bioscience, Biotechnology and Biochemistry, 58,
2224-2227.
PACHLINGER, R., MITTERBAUER, R., ADAM, G. & STRAUSS, J. (2005). Metabolically independent and
accurately adjustable Aspergillus sp. expression system. Applied and Environmental Microbiology, 71, 672–678
PAREKH, S., VINCI, V.A. & STROBEL, R.J. (2000). Improvement of microbial strains and fermentation
processes. Applied Microbiology and Biotechnology, 54, 287-301.
PENTILLÄ, M.E., ANDRÉ, L., LEHTOVAARA, P., BAILEY, M., TEERI, T.T. & KNOWLES, J.K.C. (1988).
Efficient secretion of two fungal cellobiohydrolases by Saccharomyces cerevisiae. Gene, 63, 103-112.
MANU DE GROEVE
VIII. Literatuurlijst - 127
PIECHACZYK, M., BLANCHARD, J.M., MARTY, L., DANI, C., PANABIERES, F., EL SABOUTY, S.,
FORT, P. & JEANTEUR, P. (1984). Post-transcriptional regulation of glyceraldehyde-3-phosphatedehydrogenase gene expression in rat tissues. Nucleic Acids Research, 12, 6951-6963.
PÖGGELER, S. (1997). Sequence characteristics within nuclear genes from Sordaria macrospora. Fungal
Genetics Newsletter, 44, 41-44.
POWELL, W.A. & KISTLER, H.C. (1990). In vivo rearrangement of foreign dna by Fusarium oxysporum
produces linear self-replicating plasmids. Journal of Bacteriology, 172, 3163-3171.
PUNT, P.J., OLIVER, R.P., DINGEMANSE, M.A., POUWELS, P.H. & VAN DEN HONDEL, C.A.M.J.J.
(1987). Transformation of Aspergillus based on the hygromycin B resistance marker from Escherichia coli.
Gene, 56, 117-124.
PUNT, P.J., DINGEMANSE, M.A., JACOBSMEIJSING, B.J.M., POUWELS, P.H. & VAN DEN HONDEL,
C.A.M.J.J. (1988). Isolation and characterization of the glyceraldehyde-3-phosphate dehydrogenase gene of
Aspergillus nidulans. Gene, 69, 49-57.
PUNT, P.J., DINGEMANSE, M.A., KUYVENHOVEN, A., SOEDE, R.D.M., POUWELS, P.H. & VAN DEN
HONDEL, C.A.M.J.J. (1990). Functional elements in the promoter region of the Aspergillus nidulans gpdA
gene encoding glyceraldehyde-3-phosphate dehydrogenase. Gene, 93, 101-109.
PUNT, P.J., GREAVES, P.A., KUYVENHOVEN, A., VAN DEUTEKOM, J.C., KINGHORN, J.R.,
POUWELS, P.H. & VAN DEN HONDEL, C.A.M.J.J. (1991). A twin-reporter vector for simultaneous analysis
of expression signals of divergently transcribed, contiguous genes in filamentous fungi. Gene, 104, 119-122.
PUNT, P.J. & VAN DEN HONDEL, C.A.M.J.J. (1992). Transformation of filamentous fungi based on
hygromycin B and phleomycin resistance markers. Methods in Enzymology, 216, 447-457.
PUNT, P.J., VELDHUISEN, G. & VAN DEN HONDEL, C.A.M.J.J. (1994). Protein targeting and secretion in
filamentous fungi. Antonie van Leuwenhoek, 65, 211-216.
PUNT, P.J., KUYVENHOVEN, A. & VAN DEN HONDEL C.A.M.J.J. (1995). A minipromoter lacZ gene
fusion for the analysis of fungal transcription control sequences. Gene, 158, 119-123.
PUNT, P.J., VAN BIEZEN, N., CONESA, N., ALBERS, A., MANGNUS, J. & VAN DEN HONDEL,
C.A.M.J.J. (2002). Filamentous fungi as cell factories for heterologous protein production. Trends in
Biotechnology, 20, 200-206.
QUILES-ROSILLO, M.D., RUIZ-VÁZQUEZ, R.M., TORRES-MARTÍNEZ, S. & GARRE, V. (2003).
Cloning, characterization and heterologous expression of the Blakeslea trispora gene encoding orotidine-5’monophosphate decarboxylase. FEMS Microbiology Letters, 222, 229-236.
RADFORD, A. (1993). A fungal phylogeny based upon orotidine 5’-monophosphate decarboxylase. Journal of
Molecular Evolution, 36, 389-395.
RADFORD, A. & DIX, N.I.M. (1988). Comparison of the orotidine 5'-monophosphate decarboxylase sequences
of 8 species. Genome, 30, 501-505.
RADZIO, R. & KÜCK, U. (1997). Synthesis of biotechnologically relevant heterologous proteins in filamentous
fungi. Process Biochemistry, 32, 529-539.
RIACH, M.B.R. & KINGHORN, J.R. (1996). Genetic transformation and vector developments in filamentous
fungi. In: Bos, C.J (ed.) Fungal Genetics: principles and practice. New York, Marcel Dekker, 209-233.
RODRÍGUEZ, L., CHÁVEZ, F.P., GONZÁLEZ, M.E., BASABE, L. & RIVERO, T. (1998). Isolation and
sequence analysis of the orotidine-5’-phosphate decarboxylase gene (URA3) of Candida utilis. Comparison with
the OMP decarboxylase gene family. Yeast, 14, 1399-1406.
ROSS, J. (1996). Control of messenger RNA stability in higher eukaryotes. Trends in Genetics, 12, 171-175.
MANU DE GROEVE
VIII. Literatuurlijst - 128
ROSSIER, C., PUGIN, A. & TURIAN, G. (1985). Genetic analysis of transformation in a microconidiating
strain of Neurospora crassa. Current Genetics, 10, 313-320.
RUIZ-DÍEZ, B. (2002). Strategies for the transformation of filamentous fungi: a review. Journal of Applied
Microbiology, 92, 189-195.
RUIZ-DÍEZ, B. & MARTÍNEZ-SUÁREZ, J.V. (1999). Electrotransformation of the human pathogenic fungus
Scedosporium prolificans mediated by repetitive rDNA sequences. FEMS Immunology and Medical
Microbiology, 25, 275-282.
SAMBROOK, J. & RUSSELL, D.W. (2001). Molecular cloning: a laboratory manual. Third edition. New York,
Cold Spring Harbor Laboratory Press, 999 p.
SÁNCHEZ, O. & AGUIRRE, J. (1996). Efficient transformation of Aspergillus nidulans by electroporation of
germinated conidia. Fungal Genetics Newsletter, 43, 48-51.
SANTERRE-HENRIKSEN, A.L., EVEN, S., MULLER, C., PUNT, P.J., VAN DEN HONDEL, C.A.M.J.J. &
NIELSEN, J. (1999). Study of the glucoamylase promoter in Aspergillus niger using green fluorescent protein.
Microbiology, 145, 729-734.
SANTOSO, D. & THORNBURG, R. (1998). Uridine 5’-monophosphate synthase is transcriptionally regulated
by pyrimidine levels in Nicotiana plumbaginifolia. Plant Physiology, 116, 815-821.
SAUNDERS, G., PICKNETT, T.M., TUITE, M.F. & WARD, M. (1989). Heterologous gene expression in
filamentous fungi. Trends in Biotechnology, 7, 283-287.
SAWADOGO, M. & SENTENAC, A. (1990). RNA polymerase-b (II) and general transcription factors. Annual
Review of Biochemistry, 59, 711-754.
SCHAEFER, D.G. (2001). Gene targeting in Physcomitrella patens. Current Opinion in Plant Biology, 4, 143150.
SHOJI, J.Y., MARUYAMA, J.I., ARIOKA, M., KITAMOTO, K. (2005). Development of Aspergillus oryzae
thiA promoter as a tool for molecular biological studies. FEMS Microbiology Letters, 244, 41-46.
SKORY, C.D., HORNG, J.S., PESTKA, J.J. & LINZ, J.E. (1990). Transformation of Aspergillus parasiticus
with a homologous gene (pyrG) involved in pyrimidine biosynthesis. Applied and Environmental Microbiology,
56, 3315-3320.
SKORY, C.D. (2002). Homologous recombination and double-strand break repair in the transformation of
Rhizopus oryzae. Molecular Genetics and Genomics, 268, 397-406.
SMIT, R. & TUDZYNSKI, P. (1992). Efficient transformation of Claviceps purpurea using pyrimidine
auxotrophic mutants: cloning of the OMP decarboxylase gene. Molecular and General Genetics, 234, 297-305.
STIEF, A., WINTER, D.M., STRÄTLING, W.H. & SIPPEL, A.E. (1989). A nuclear DNA attachment element
mediates elevated and position-independent activity. Nature, 341, 343-345.
STRUHL, K. (1989). Molecular mechanisms of transcriptional regulation in yeast. Annual Review of
Biochemistry, 58, 1051-1077.
TAKENO, S., SAKURADANI, E., MURATA, S., INOHARA-OCHIAI, M., KAWASHIMA, H., ASHIKARI,
T. & SHIMIZU, S. (2004). Cloning and sequencing of the ura3 and ura5 genes, and isolation and
characterisation of uracil auxotrophs of the fungus Mortierella alpina 1S-4. Bioscience, Biotechnology and
Biochemistry, 68, 277-285.
TILBURN, J., SCAZZOCCHIO, C., TAYLOR, G.G., ZABICKY-ZISSIMAN, J.H., LOCKINGTON, R.A. &
DAVIES, R.W. (1983). Transformation by integration in Aspergillus nidulans. Gene, 26, 205-221.
TIMBERLAKE, W.E. & MARSHALL, M.A. (1989). Genetic engineering of filamentous fungi. Science, 24,
1313-1317.
MANU DE GROEVE
VIII. Literatuurlijst - 129
TRAUT, T.W. & TEMPLE, B.R.S. (2000). The chemistry of the reaction determines the invariant amino acids
during the evolution and divergence of orotidine 5’-monophosphate decarboxylase. The Journal of Biological
Chemistry, 275, 28675-28681.
TROOSKENS, G. (2005). Fylogenetische visualisatie, congruentie en analyse van geconserveerde residu’s.
Scriptie, Faculteit Bio-ingenieurswetenschappen, Universiteit Gent.
TSUKUDA, T., CARLETON, S., FOTHERINGHAM, S. & HOLLOMAN W.K. (1988). Isolation and
characterization of an autonomously replicating sequence from Ustilago maydis. Molecular and Cellular
Biology, 8, 3703-3709.
TUDZYNSKI, P. & TUDZYNSKI, B. (1997). Fungal genetics: novel techniques and regulatory circuits. In:
Anke, T. (ed.) Fungal biotechnology. London, Chapmann and Hall, 229-249.
TULLOCH, M. (1972). The genus Myrothecium Tode ex FR. Mycological Papers, 130, 1-44.
TURNER, G., BROWN, J., KERREY-WILLIAMS, S., BAILEY, A.M., WARD, M., PUNT, P.J. & VAN DEN
HONDEL, C.A.M.J.J. (1989). Analysis of the oliC promoter of Aspergillus nidulans . Foundation for
Biotechnological and Industrial Fermentation Research, 6, 101-109.
VANDAMME, E.J. (2002). Cursus Algemene Microbiologie. Gent, Faculteit Bio-ingenieurswetenschappen,
Universiteit Gent.
VAN GORCOM, R.F. & VAN DEN HONDEL, C.A..M.J.J. (1988). Expression analysis vectors for Aspergillus
niger. Nucleic Acids Research, 16, 9052.
VAN HARTINGSVELDT, W., MATTERN, I.E., VAN ZEIJL, C.M., POUWELS, P.H. & VAN DEN
HONDEL, C.A.M.J.J. (1987). Development of a homologous transformation system for Aspergillus niger based
on the pyrG gene. Molecular and General Genetics, 206, 71-75.
VAN HARTINGSVELDT, W., VAN ZEIJL, C.M.J., VEENSTRA, A.E., VAN DEN BERG, J.A., POUWELS,
P.H., VAN GORCOM, R.F.M. & VAN DEN HONDEL, C.A.M.J.J. (1991). Heterologous gene expression in
Aspergillus: analysis of chymosin production in single-copy transformants of A. niger. In: Heslot, H., Bavies, J.,
Florent, J., Bobichon, L., Durand, G. & Penasse, L. (eds.) Proceedings of the 6th international symposium on the
genetics of industrial microorganisms, Société Francaise de Microbiologie, Strasbourg, 107-116.
WACH, A., BRACHAT, A., POHLMANN, R., & PHILIPPSEN, P. (1994). New heterologous modules for
classical or PCR-based gene disruptions in Saccharomyces cerevisiae. Yeast, 10, 1793-1808.
WANG, H., KIM, S. & BREUIL, S. (1999). Transformation of sapstaining fungi with hygromycin B resistance
plasmids pAN7-1 and pCB1004. Mycological Research, 103, 77-80.
WANG, L., RIDGWAY, D., GU, T. & MOO-YOUNG, M. (2005). Bioprocessing strategies to improve
heterologous protein production in filamentous fungal fermentations. Biotechnology Advances, 23, 115-129.
WARD, M. (1989). Production of calf chymosin by Aspergillus awamori. In: Hershberger, C.L., Queener, S.W.
& Hegeman, G. (eds.) Genetics and molecular biology of industrial microorganisms. Washington, American
Society of Microbiology, 288-294.
WARD, M., WILSON, L.J., KODAMA, K.H., REY, M.W. & BERKA, R.M. (1990). Improved production of
chymosin in Aspergillus by expression as a glucoamylase-chymosin fusion. Bio-Technology, 8, 435-440.
WEIDNER, G., D’ENFERT, C., KOCH, A., MOL, P.C. & BRAKHAGE, A.A. (1998). Development of a
homologous transformation system for the human pathogenic fungus Aspergillus fumigatus based on the pyrG
gene encoding orotidine 5’-monophosphate decarboxylase. Current Genetics, 33, 378-385.
WERNARS, K., GOOSEN, T., WENNEKES, B.M.J., SWART, K., VANDENHONDEL, C.A.M.J.J. &
VANDENBROEK, H.W.J. (1987). Cotransformation of Aspergillus nidulans - a tool for replacing fungal genes.
Molecular & General Genetics, 209, 71-77.
MANU DE GROEVE
VIII. Literatuurlijst - 130
WERTEN, M.W., VAN DEN BOSCH, T.J., WIND, R.D., MOOIBROEK, H. & DE WOLF, F.A. (1999). Highyield secretion of recombinant gelatins by Pichia pastoris. Yeast, 15, 1087-1096.
WHELAN, S., PIETRO, L. & GOLDMAN, N. (2001). Molecular phylogenetics: state-of-the-art methods for
looking into the past. Trends in Genetics, 17, 262-272.
WRIGHT, F. (1990). The 'effective number of codons' used in a gene. Gene, 87, 23-29.
WYSS, M., PASAMONTES, L., FRIEDLEIN, A., REMY, R., TESSIER, M., KRONENBERGER, A.,
MIDDENDORF, A., LEHMANN, M., SCHNOEBELEN, L., ROTHLISBERGER, U., KUSZNIR, E., WAHL,
G., MULLER, F., LAHM, H. W., VOGEL, K. & VAN LOON, A. P. (1999). Biophysical characterization of
fungal phytases (myo-inositol hexakisphosphate phosphohydrolases): Molecular size, glycosylation pattern, and
engineering of proteolytic resistance. Applied and Environmental Microbiology, 65, 359-366.
YAMAZAKI, H., OHNISHI, Y., TAKEUCHI, K., MORI, N., SHIRAISHI, N., SAKATA, Y., SUZUKI, H. &
HORINOUCHI, S. (1999). Genetic transformation of a Rhizomucor pusillus mutant defective in asparaginelinked glycosylation: production of a milk-clotting enzyme in a less-glycosylated form. Applied Microbiology
and Biotechnology, 52, 401-409.
YODER, O.C (1979). Experience with the Applegate-Nelson-Metzenberg method of mutant enrichment in high
sorbose medium. Neurospora Newsletter, 26, 23.
ZHU, J., OGER, P.M., SCHRAMMEIJER, B., HOOYKAAS, P.J.J., FARRAND, S.K. & WINANS, S.C.
(2000). The bases of crown gall tumorigenesis. Journal of Bacteriology, 182, 3885-3895.
Bijlage - 131
MANU DE GROEVE
IX. BIJLAGE
1. ALIGNMENTS
1.1. Alignment cDNA en genomisch DNA van het OMPD gen
Alignment uitgevoerd in Clone Manager 6.0
cDNA
Genomisch DNA
ATCCGAGCCACAACGAATCCGCACCCAGTGGCTCGAAAGCTTTTCGCCGTTGCGGAACGC
ATCCGAGCCACAACGAATCCGCACCCAGTGGCTCGAAAGCTTTTCGCCGTTGCGGAACGC
************************************************************
cDNA
Genomisch DNA
AAAAAGTCGAACCTGATCATATCCGCTGACTTGACCGATACAAAGAGTCTCTTGGAGTGT
AAAAAGTCGAACCTGATCATATCCGCTGATTTGACCGATACAAAGAGTCTCTTGGAGTGT
***************************** ******************************
cDNA
Genomisch DNA
GCTGATG----------------------------------------------------GCTGATGGTAGAGTAGCCAACAAACCCATTGCATGCTCACTCGCTAACTTATATGCGCCT
*******
cDNA
Genomisch DNA
------AGCTCGGGCCCTTCATCTCAGTGTTCAAGACCCATATCGACATCATTCACGATT
GTTTAGAGCTCGGGCCCTTCATCTCAGTGTTCAAGACCCATATCGACATCATTCACGATT
******************************************************
cDNA
Genomisch DNA
TTGGCGACGAGACGGTTCGGGGCCTCAAGAGTCTGGCAAGAAAGCACGACTTCCTCATCT
TTGGCGACGAGACGGTTCGGGGCCTCAAGAGTCTGGCAAGAAAGCACGACTTCCTCATCT
************************************************************
cDNA
Genomisch DNA
TCGAGGATCGCAAGTTTGTGGATATTGGCTCAACAGCCCAGAAACAGTACCATGGTGGTG
TCGAGGATCGCAAGTTTGTGGATATTGGCTCAACAGCCCAGAAACAGTACCATGGTGGTG
************************************************************
cDNA
Genomisch DNA
CCCTGCGCATCTCCGAGTGGGCCGACATTGTCAACGTCAGTCTCCTAGGGGGTGATGGCG
CCCTGCGCATCTCCGAGTGGGCCGACATTGTCAACGTCAGTCTCCTAGGGGGTGATGGCG
************************************************************
cDNA
Genomisch DNA
TTGTGAATGCACTGGGCCAGGTCATTACCGACGAGACGTTTCCCTATCGCGGTCAACGGG
TTGTGAATGCACTGGGCCAGGTCATTACCGACGAGACGTTTCCCTATCGCGGTCAACGGG
************************************************************
cDNA
Genomisch DNA
CCCTGCTTCTGCTTKCTGAGATGACCACGGCCGGTAGCCTTGCCGTTGAAAAATACACAG
CCCTGCTTCTGCTTGCTGAGATGACCACGGCCGGTAGCCTTGCCGTTGAAAAATACACAG
************** *********************************************
cDNA
Genomisch DNA
AGCGCTGCGTCGCTGTCGCACGAAAGAATGGCATTGCCGCTGGAGTCATGGGCTTCGTTG
AGCGCTGCGTCGCTGTCGCACGAAAGAATGGCATTGCCGCTGGAGTCATGGGCTTCGTTG
************************************************************
cDNA
Genomisch DNA
CCACGCGAGGCCTCGAAGACGTTGGCAACGAACCTGGGGTCGAGAGAGACGAGGACGAAG
CCACGCGAGGCCTCGAAGACGTTGGCAACGAACCTGGGGTCGAGAGAGACGAGGACGAAG
************************************************************
cDNA
Genomisch DNA
ACTTTGTCGTTTTTACTACTGGTATCAACAGCAGCCAATCTGACAATGTGCTGGGACAGA
ACTTTGTCGTTTTTACTACTGGTATCAACAGCAGCCAATCTGACAATGTGCTGGGACAGA
************************************************************
cDNA
Genomisch DNA
AGTATCAGACACCCGAGGCAGCCATACGTGGCGGCTCTGATT
AGTATCAGACACCCGAGGCAGCCATACGTGGCGGCTCTGATT
******************************************
MANU DE GROEVE
Bijlage - 132
1.2. Alignment OMPD enzym M. gramineum met OMPD
enzymen van andere filamenteuze schimmels
Alignment uitgevoerd in Clone Manager 6.0 (Multi-Way, PAM250 scorematrix)
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
1
1
1
1
1
1
1
1
------MSSKSGL--PYHIRATTNPHPVARKLFAVAERKKSNLIISADLTDTKSLLECADE---LGPFIS
------MSSKSHL--PYAIRATNHPNPLTSKLFSIAEEKKTNVTVSADVTTSAELLDLADR---LGPYIA
------MSSKSQL--TYTARASKHPNALAKRLFEIAEAKKTNVTVSADVTTTKELLDLADR---LGPYIA
------MSSKSQL--TYTARAQSHPNPLARKLFQVAEEKKSNVTVSADVTTTKELLDLADPSTGLGPYIA
MSTSQETQPHWSLKQSFAERVESSTHPLTSYLFRLMEVKQSNLCLSADVEHARDLLALADK---VGPSIV
MSTTQ--QPHWSLQKSFAERVESSSHPLTSYLFRLMEVKQSNLCLSDDVEHARELLALADK---IGPSIV
------MASHPTLKTTFAARSEATTHPLTSYLLRLMDLKASNLCLSADVPTARELLYLADK---IGPSIV
------MAPHPTLKATFAARSETATHPLTAYLFKLMDLKASNLCLSADVPTARELLYLADK---IGPSIV
m
L
R
pl
Lf
e K sN
SaDv
eLL lAD
GP I
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
60
60
60
63
68
66
62
62
VFKTHIDIIH--DFGDETVRG--LKSLARKHDFLIFEDRKFVDIGSTAQKQYHGGALRISEWADIVNVSL
VLKTHIDILT--DLTPSTLSS--LQSLATKHNFLIFEDRKFIDIGNTVQKQYHGGALRISEWAHIINCAI
VIKTHIDILS--DFSDETIEG--LKALAQKHNFLIFEDRKFIDIGNTVQKQYHRGTLRISEWAHIINCSI
VIKTHIDILS--DFSQETIDG--LNALAQKHNFLIFEDRKFIDIGNTVQKQYHNGTLRISEWAHIINCSI
VLKTHYDLITGWDYHPHTGTGAKLAALARKHGFLIFEDRKFVDIGSTVQKQYTAGTARIVEWAHITNADI
VLKTHYDLITGWDYHPHTGTGAKLAALARKHGFLIFEDRKFVDIGSTVQKQYTAGTARIVEWAHITNADI
VLKTHYDMVSGWDFHPDTGTGAKLASLARKHGFLIFEDRKFGDIGHTVELQYTSGSARIIDWAHIVNVNM
VLKTHYDMVSGWTSHPETGTGAQLASLARKHGFLIFEDRKFGDIGHTVELQYTGGSARIIDWAHIVNVNM
V KTH D
d
T g L LA KH FLIFEDRKF DIG TvqkQY G RI eWAhI N
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
126
126
126
129
138
136
132
132
LGGDGVVNALGQ---------------------------------------------------------LPGEGIVEALAQTTK--------------------SPDFK---------------------------DAN
LPGEGIVEALAQTAS--------------------APDFS---------------------------YGP
LPGEGIVEALAQTAQ--------------------ATDFP---------------------------YGS
HAGEAMVSAMAQAAQKWRERIPYEVKTSVSVGTPVADQFADE------EAEDQVEELRKVVTRETSTTTHAGEAMVSAMAQAAQKWRERIPYEVKTSVSVGTPVADQFADE------EAEDQVDELRKIVPRENSTSKE
VPGKASVASLAQGARRWLERYPCEVKTSVTVGTPTMDQFDDAEDAKDDEPA-TVNDNGS-NMMEKPIYAG
VPGKASVASLAQGAKRWLERYPCEVKTSVTVGTPTMDSFDDDADSRDAEPAGAVNGMGSIGVLDKPIYSN
G
V alaQ a
f
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
138
149
149
152
201
200
200
202
-------------------------------VITDETFPYRG------QRALLLLAEMTTAGSLAVEKYT
------------------------------------------------QRGLLILAEMTSKGSLATGEYT
------------------------------------------------ERGLLILAEMTSKGSLATGQYT
------------------------------------------------ERGLLILAEMTSKGSLATGAYT
KDTDGRKSSIVSITTVTQTYEPADSPRLVKTISEDDEMVFPGIEEAPLDRGLLILAQMSSKGCLMDGKYT
KDTDGRKGSIVSITTVTQTYEPADSPRLAKTISEGDEAVFPGIEEAPLDRGLLILAQMSSKGCLMDGKYT
RNGDGRKGSIVSITTVTQQYESAASPRLGKTIAEGDESLFPGIEEAPLNRGLLILAQMSSEGNFMTGEYT
RSGDGRKGSIVSITTVTQQYESVSSPRLTKAIAEGDESLFPGIEEAPLSRGLLILAQMSSQGNFMNKEYT
RgLLiLA M s G l g YT
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
171
171
171
174
271
270
270
272
ERCVAVARKNGIAAGVMGFVATRGLEDVGNEPGV-----------ERDEDEDFVVFTTGINSSQS----ARSVEYARK--YKGFVMGFVSTRALSEVLPEQ------------KEES--EDFVVFTTGVNLSDK----TSSVDYARK--YKNFVMGFVSTRSLGEVQSEV------------SSPSDEEDFVVFTTGVNISSK----SASVDIARK--YPSFVLGFVSTRSLGEVES--------------TEAPASEDFVVFTTGVNLSSK----WECVKAARK--NKGFVMGYVAQQNLNGITKEALAPSYEDGESTTEEEAQADNFIHMTPGCKLPPPGEEAP
WECVKAARK--NKDFVMGYVAQQNLNGITKEDLAPGYEDGETSTEEEAQADNFIHMTPGCKLPPPGEEAP
QACVEAARE--HKDFVMGFISQEALN--------------------TQADDDFIHMTPGCQLPPEDEDQQ
QASVEAARE--HKDFVMGFISQETLN--------------------TEPDDAFIHMTPGCQLPPEDEDQQ
V ARk
k fVmGfv
L
F
T G l
M.
A.
A.
P.
N.
S.
T.
T.
gramineum
nidulans
niger
chrysogenum
crassa
macrospora
harzianum
reesei
225
220
222
223
339
338
318
320
------DNVLGQQYQTPEAAI-RGGSDFIIAGRGIYASEDRVQAAKQYQAEGWAAYVARIGPGHY
------GDKLGQQYQTPGSAVGR-GADFIIAGRGIYKADDPVEAVQRYREEGWKAYEKRVGL--------GDKLGQQYQTPASAIGR-GADFIIAGRGIYAAPDPVQAAQQYQKEGWEAYLARVGGN-------GDKLGQQYQTPQSAVGR-GADFIISGRGIYAAADPVEAAKQYQQQGWEAYLARVGAQ-Q-----GDGLGQQYNTPDNLVNIKGTDIAIVGRGIITAADPPAEAERYRRKAWKAYQDRRERLAQ-----GDGLGQQYNTPDNLVNIKGTDIAIVGRGIITASDPPAEAERYRRKAWKAYQDRRERLATNGKVGGDGQGQQYNTAHKIIGIAGSDIAIVGRGILKASDPVEEAERYRSAAWKAYTERLLR--TNGSVGGDGQGQQYNTPHKLIGIAGSDIAIVGRGILKASDPVEEAERYRSAAWKAYTERLLR--gd lGQQY Tp
G D I GRGI a Dpv a Y
W AY R
Bijlage - 133
MANU DE GROEVE
2. DE GENETISCHE CODE, GEDEGENEREERDE
NUCLEOTIDEN EN AFKORTINGEN VAN AMINOZUREN
DE GENETISCHE CODE
1ste positie
(5’ einde)
2de positie
3de positie
(3’ einde)
↓
U
C
A
G
U
Phe
Phe
Leu
Leu
Ser
Ser
Ser
Ser
Tyr
Tyr
STOP
STOP
Cys
Cys
STOP
Trp
U
C
A
G
C
Leu
Leu
Leu
Leu
Pro
Pro
Pro
Pro
His
His
Gln
Gln
Arg
Arg
Arg
Arg
U
C
A
G
A
Ile
Ile
Ile
Met
Thr
Thr
Thr
Thr
Asn
Asn
Lys
Lys
Ser
Ser
Arg
Arg
U
C
A
G
G
Val
Val
Val
Val
Ala
Ala
Ala
Ala
Asp
Asp
Glu
Glu
Gly
Gly
Gly
Gly
U
C
A
G
↓
Bijlage - 134
MANU DE GROEVE
Tabel 44. Niet-gedegenereerde en gedegenereerde nucleotiden
Nucleotide
Benaming
Complement
Adenosine
A
T
Cytidine
C
G
Guanine
G
C
Thymidine
T
A
Uridine (RNA)
U
A
puRine (G of A)
R
Y
pYrimidine
(C
of
T)
Y
R
Weak (A of T)
W
W
Strong (G of C)
S
S
aMino (A of C)
M
K
Keto (G of T)
K
M
niet T (A, C of G)
V
B
niet A (C, G of T)
B
V
niet G (A, C of T)
H
D
niet C (A, G of T)
D
H
A, G, C of T
N
N
Tabel 45. Aminozuren en gebruikte afkortingen
Aminozuur
Alanine
3 letter code
1 letter code
Ala
A
Arginine
Arg
R
Asparagine
Asn
N
Asparaginezuur
Asp
D
Cysteine
Cys
C
Glutamine
Gln
Q
Glutaminezuur
Glu
E
Glycine
Gly
G
Histidine
His
H
Isoleucine
Ile
I
Leu
L
Lysine
Lys
K
Methionine
Met
M
Fenylalanine
Phe
F
Proline
Pro
P
Serine
Ser
S
Threonine
Thr
T
Tryptofaan
Trp
W
Tyrosine
Tyr
Y
Valine
Val
V
Leucine