cDNA analyse van DNA repair
genen in hoogrisico
borstkankerfamilies
Grunewald Anneke
Verhandeling ingediend tot
het verkrijgen van de graad van
Master in de Biomedische Wetenschappen
Promotor: Prof. Kathleen Claes
Begeleider: Dr. Kim De Leeneer
Vakgroep: Pediatrie en genetica
Academiejaar 2012-2013
Voorwoord
Het laatste jaar Gent, het thesisjaar. Een jaar waarbij leuke, grappige momenten zich
afwisselden met stress en deadlines. Zonder de hulp van bepaalde personen was het niet
mogelijk geweest om deze thesis te schrijven, daarom zou ik graag de mensen willen
bedanken die dit mogelijk maakten.
In eerste instantie zou ik graag mijn promotor Kathleen en begeleider Kim willen bedanken
voor de kans die ze me boden om deel uit te maken van dit onderzoek. Ondanks hun drukke
agenda vonden ze steeds de tijd om mij bij te staan met raad en daad. Ik mocht steeds
‘binnenspringen’ in jullie bureau. Dankzij jullie kennis, ervaring, toewijding en goede
ondersteuning was dit een bijzonder leerrijke ervaring.
Ik zou graag ook alle laboranten, die ik vaak genoeg heb lastig gevallen, hartelijk bedanken.
Ondanks jullie eigen werk, mochten we steeds bij jullie aankloppen, waarna jullie ons met
plezier hielpen.
Ook zou ik graag Lode, Elise, Jolien, Morgane, Jasper, Mattias en Liza willen bedanken voor
de (te) leuke momenten die we samen gedeeld hebben. Het was steeds gezellig in onze ‘kast’,
deels door plaatsgebrek, maar we hebben niets dan goede momenten beleefd. Jullie zorgden
voor de nodige afleiding en koffiepauzes en niet te vergeten de welgemeende steun.
Via deze weg zou ik ook graag mijn ouders en zus bedanken, die me in alle keuzes 100%
gesteund hebben. Ondanks dat ik dit jaar minder tijd kon doorbrengen met jullie, hoop ik dat
jullie trots zijn op dit werk. Mijn diploma zou ik nooit behaald hebben moesten jullie er niet
geweest zijn!
Ten laatste, maar zeker niet de minst belangrijke, zou ik graag mijn vriend Surcouf bedanken.
Hij heeft deze 5 jaar, en voornamelijk dit laatste jaar, van dichtbij meegemaakt. Dit betrof
zowel alle goede en leuke momenten als de mindere. Ook al heb ik je het niet altijd even
gemakkelijk gemaakt, je bleef me in alles steunen. Dankjewel hiervoor!
Dankjewel !!
Afkortingen
53BP1
p-53 bindend eiwit
AT
ataxia-telangiectasia
ATM
ataxia-telangiectasia mutated
AZ
aminozuur
BAP1
BRCA1-associated protein 1
BARD1
BRCA1-associated RING domain 1
BRCA1
breast cancer susceptibility gene 1
BRCA2
breast cancer susceptibility gene 2
BRCT
BRCA1 carboxyl terminus
BRIP1
BRCA1 interacting protein C-terminal helicase
cDNA
complementair DNA
CHEK2
checkpoint kinase 2
CMGG
centrum medische genetic Gent
DNA
deoxyribonucleic acid
DSB
dubbelstrengbreuken
ddNTP
dideoxyribonucleotides trifosfaat
dNTP
deoxyribonucleotides trifosfaat
DMSO
ditmethylsulfoxide
dsDNA
dubbelstrengig DNA
DTT
dithiothreitol
EBV
Epstein-Barr virus
EF3
elongatiefactor 3
ENIGMA
evidence-based network for the interpretation of germline mutant alleles
EXO-AP
exonuclease I en antartic phosphatase
FA
Fanconi anemia
FA-D1
Fanconi anemia subgroup D1
FANCJ
Fanconi anemia complementation group J
FAT
FRAP, ATM, TRAPP
FHA
fork head-associated
gDNA
genomisch DNA
GITC
guanidine isothiocyanaat
HEAT
Hungtingtin, EF3, PP2A, TOR1
LFS
Li-Fraumeni syndroom
lncRNA
long non-coding RNA
miRNA
microRNA
ncRNA
non-coding RNA
NLS
nucleair lokalisatie signal
NMD
nonsense mediated decay
M-MULV
moloney murine leukemia virus
PALB2
partner and localizer of BRCA2
PCR
polymerase-kettingsreactie
PP2A
phosphatase 2A
Rcf
relatieve centrifugale kracht
RING
really interesting new gene
RNA
ribonucleïnezuur
RNAsin
ribonuclease inhibitor
RR
relatief risico
RT
reverse transcriptase
SBS
substraatbinding
SCD
SQ/TQ cluster domein
SNP
single nucleotide polymorfismes
ssDNA
enkelstrengig DNA
Tm
smelttemperatuur
TSG
tumorsupressorgenen
UV
unclassified variants
UZG
universitair ziekenhuis Gent
Inhoudstabel
1. Samenvatting ........................................................................................................................ 1
2. Inleiding................................................................................................................................. 2
2.1. Kanker......................................................................................................................................................... 2
2.2. Borstkanker ................................................................................................................................................ 4
2.2.1. Algemeen .............................................................................................................................................. 4
2.2.2. Risicofactoren ...................................................................................................................................... 4
2.3. Genetische voorbeschiktheid ..................................................................................................................... 5
2.3.1. Inleiding ............................................................................................................................................... 5
2.3.2. Hoog penetrante borstkankergenen .................................................................................................. 6
2.3.2.1. BRCA1 en BRCA2......................................................................................................................... 6
2.3.2.2. Andere hoog penetrante genen .................................................................................................. 10
2.3.3. Matig penetrante borstkankergenen ............................................................................................... 11
2.3.3.1. ATM ............................................................................................................................................. 11
2.3.3.2. CHEK2 ......................................................................................................................................... 13
2.3.3.3. BRIP1 .......................................................................................................................................... 15
2.3.3.4. PALB2 ......................................................................................................................................... 16
2.3.4. Laag penetrante varianten ............................................................................................................... 18
2.4. Doel- en vraagstelling ............................................................................................................................... 18
2.4.1. Model van borstkankersusceptibiliteit ............................................................................................ 18
2.4.2. Probleemstelling en doelstelling ....................................................................................................... 19
2.4.2.1. cDNA analyse .............................................................................................................................. 19
2.4.2.2. Enigma project ........................................................................................................................... 20
3. Materialen en methoden .................................................................................................... 21
3.1. Gebruikte methoden ................................................................................................................................ 21
3.2. Selectie patiënten ...................................................................................................................................... 21
3.3. RNA extractie ........................................................................................................................................... 22
3.4. cDNA synthese .......................................................................................................................................... 23
3.5. RT-PCR..................................................................................................................................................... 24
3.5.1. Primerdesign ...................................................................................................................................... 25
3.6. Optimalisatie ............................................................................................................................................. 26
3.7. Caliper LabChip GX ................................................................................................................................ 27
3.8. Klonering .................................................................................................................................................. 27
3.9. Sanger sequenering .................................................................................................................................. 28
3.9.1. Principe .............................................................................................................................................. 28
4. Resultaten ............................................................................................................................ 30
4.1. Enigma Project ......................................................................................................................................... 30
4.2. Vergelijking cDNA synthese kits............................................................................................................. 32
4.3. cDNA analyse van BRCA1, BRCA2, ATM, BRIP1, PALB2 en CHEK2 ............................................... 33
4.3.1. Primerdesign en optimalisatie .......................................................................................................... 33
4.3.2. RNA 1300042 ..................................................................................................................................... 34
4.3.3. PCR + sequenering overige patiënten ............................................................................................. 36
4.4. Analyse van 4 varianten op cDNA niveau .............................................................................................. 37
5. Discussie .............................................................................................................................. 40
5.1. Enigma ...................................................................................................................................................... 40
5.1.1. BRCA1 c.671-2A>G ........................................................................................................................... 40
5.1.2. Vergelijking Takara Ex Taq en Kapa2G Robust ........................................................................... 41
5.1.3. Vergelijking 3 cDNA synthese kits ................................................................................................... 42
5.2. cDNA analyse van BRCA1, BRCA2, ATM, BRIP1, PALB2 en CHEK2 ............................................... 43
5.2.1. Primeroptimalisatie ........................................................................................................................... 43
5.2.2. RNA 1300042 ..................................................................................................................................... 44
5.2.3. Overige patiënten .............................................................................................................................. 44
5.3. Analyse van 4 varianten op cDNA niveau .............................................................................................. 45
6. Referenties ........................................................................................................................... 48
1. SAMENVATTING
Het doel van deze studie is om met behulp van cDNA analyse na te gaan of germinale
mutaties aanwezig zijn BRCA1/2, ATM, BRIP1, PALB2 of CHEK2 in families waarin tot nu
toe geen moleculaire verklaring werd gevonden voor de familiale clustering van borstkanker.
Voor deze studie werden 25 patiënten uit 24 niet-verwante families geselecteerd die voldeden
aan de inclusiecriteria. De beschikbare EBV cellijnen werden opgekweekt voor deze
patiënten, waarbij één deel behandeld werd met puromycine. Vervolgens werd hieruit totaal
RNA
geëxtraheerd
en
cDNA
gesynthetiseerd.
Gelijktijdig
werden
PCR
en
sequeneringsprimers ontwikkeld en geoptimaliseerd voor bovenvermelde genen.
Bij de 25 patiënten waarop cDNA analyse werd uitgevoerd konden 10 varianten
geïdentificeerd worden in 3 verschillende genen (BRCA1, BRCA2 en ATM). Deze varianten
worden frequent aangetroffen op gDNA niveau en betreffen waarschijnlijk neutrale sequentievarianten.
In een tweede deelproject van deze masterproef werd van vier BRCA2 varianten die
geïdentificeerd werden op gDNA niveau geëvalueerd wat hun effect op splicing is. Slechts
één variant, BRCA2 c.517C>G, leidt tot aberrante splicing en creëerde een aberrant transcript
waarbij exon 7 gedeleteerd was. Uit bijkomend onderzoek bleek dat deze variant niet meer in
staat was om het full length transcript te genereren en bijgevolg beschouwd kan worden als
een pathogene mutatie geassocieerd met een sterk verhoogd risico op borstkanker.
In een laatste luik van deze masterproef werd deelgenomen aan een kwaliteitscontroleproject
van de splicing werkgroep van het internationale ENIGMA consortium. Hierbij werd de
BRCA1 c.671-2A>G variant, waarvoor uit vroeger onderzoek gebleken is dat deze leidt tot
een complex patroon van aberrante transcripten, onderzocht op cDNA niveau door
verschillende deelnemende labo’s. Na klonering en sequenering van de gekloneerde
fragmenten konden 5 (r.671-4096del; r.594-4096del; r.671-3897del; r.671-3881del; r.7874096del) verschillende fragmenten opgepikt worden, terwijl er bij de controle maar 2 (r.6714096del; r.787-4096del) werden geïdentificeerd. Bij dit onderzoek kon men ook besluiten dat
zowel de positie van de primers, het gebruikte DNA polymerase en cDNA synthese kit,
alsook klonering een rol kunnen spelen in welke transcripten worden opgepikt.
1
2. INLEIDING
2.1. KANKER
Kanker is de verzamelnaam voor honderden verschillende soorten ziekten met een
gemeenschappelijk kenmerk, namelijk ongecontroleerde celgroei. Normale cellen zullen
enkel delen wanneer dit effectief nodig is, bijvoorbeeld bij celhernieuwing of beschadiging
van cellen en zullen vervolgens differentiëren waarbij ze hun specifieke vorm verkrijgen.
Deze beide processen, celproliferatie en –differentiatie, worden gereguleerd door
verschillende factoren die, ofwel celdeling promoten of afremmen. Men kan dus stellen dat
kanker ontstaat als gevolg van genetische veranderingen waarbij de celproliferatie verstoord is
doordat de celdeling gepromoot wordt en/of celdood geïnhibeerd wordt (1).
Deze genetische veranderingen komen voor in genen die de celdeling controleren (2). Deze
genen kunnen opgesplitst worden in twee brede klassen: de tumorsupressorgenen (TSG) en de
proto-oncogenen. Oncogenen zijn de veranderde vorm van proto-oncogenen en coderen voor
eiwitten die de celproliferatie stimuleren. Wanneer hier gain-of–functions mutaties in
voorkomen spreken we van oncogenen, deze eiwitten zullen een verhoogde activiteit hebben
of zullen in hoeveelheid zijn toegenomen, waardoor er dus een teveel aan cellen zal
geproduceerd worden (3). Dit in tegenstelling tot TSG, deze genen coderen eiwitten die de
groei van de cellen zullen remmen. Ze spelen een rol in stabilisatie van het genoom (zoals
deoxyribonucleic acid (DNA) herstel), induceren van apoptose en reguleren van de celcyclus
(3). In tegenstelling tot de proto-oncogenen dienen deze genen geïnactiveerd te worden voor
de transformatie tot maligne cellen (2).
Het Knudson two-hit model stelt dat kanker ontstaat door accumulatie van mutaties in het
DNA en dat inactivatie van beide allelen van een TSG nodig is om het gedrag van cellen te
kunnen veranderen. Bij erfelijke kankers is hiervan één mutatie overgeërfd in het DNA,
waarna een tweede hit snel kan leiden tot het ontstaan van kanker. In sporadische kankers
dienen twee hits in het TSG verzameld te worden, wat de latere leeftijd van het ontstaan van
deze kankers verklaard.
2
Figuur 1: Knudson 2 hit hypothese.
Normale cellen hebben 2 niet beschadigde chromosomen, 1 afkomstig van de moeder en 1 afkomstig van de
vader. Deze chromosomen bevatten duizenden genen. Mensen met een hereditaire susceptibiliteit tot kanker
hebben een beschadigd gen overgeërfd op één van hun chromosomen. Dus hun eerste ‘hit’ of mutatie verkrijgen
ze bij de conceptie. Andere personen kunnen hun eerste ‘hit’ verkrijgen op een later stadium, voor of na de
geboorte. In elk geval, als een cel schade verkrijgt op hetzelfde gen op het tweede chromosoom, dan kan deze cel
uitgroeien tot een kankercel. (http://www/fccc.edu.html)
TSG kunnen vervolgens nog eens onderverdeeld worden in gatekeepers, caretakers en
landscapers (4). Gatekeeper genen zullen de celproliferatie direct reguleren via het promoten
van celdood of het inhiberen van de celgroei en inactivatie van deze genen zal aanleiding
geven tot initiatie van tumorvorming (4). Dit in contrast tot caretaker genen waarbij
inactivatie niet direct verantwoordelijk is voor tumorontwikkeling, maar zal leiden tot
verhoogde accumulatie van genomische mutaties (4). Deze accumulatie van mutaties kan dan
op zijn beurt leiden tot inactivatie van de gatekeepers of activatie van de oncogenen (5). De
derde groep van TSG, de landscaper genen, coderen voor eiwitten die de micro-omgeving
waarin de cellen groeien zullen controleren. Wanneer deze genen mutaties verwerven kunnen
deze dus bijdragen tot de groei van cellen door de stromale omgeving te beïnvloeden,
waardoor er een milieu ontstaat dat de overleving van kankercellen ter goede komt (6).
Kanker is niet het gevolg van één enkele mutatie in een gen. Het effect van de activatie van
een proto-oncogen zal niet onmiddellijk leiden tot de ontwikkeling van kanker, doordat het
effect kan weerlegd worden door de TSG (door deze cellen te dwingen tot apoptose) en vice
versa. Kanker zal dus ontstaan door accumulatie van verschillende mutaties in verschillende
genen, waarbij met elke mutatie de kans op nieuwe mutaties zal verhogen.
3
2.2. BORSTKANKER
2.2.1. ALGEMEEN
Borstkanker is de meest voorkomende vorm van kanker bij vrouwen. Ongeveer één op tien
vrouwen zal borstkanker ontwikkelen gedurende haar leven, maar de incidentie varieert
tussen verschillende populaties (7, 8). Jaarlijks krijgen ongeveer 9700 vrouwen de diagnose
van borstkanker in België, dit aantal stelt 35.3% voor van alle kankers die worden vastgesteld
bij vrouwen. Bij Belgische mannen is de incidentie van borstkanker veel lager (0.3%, 85
gevallen/jaar) (zie tabel 1) (http://www.kankerregister.org).
Tabel 1: Borstkanker: incidentie en mortaliteit per geslacht en regio, 2008.
2.2.2. RISICOFACTOREN
Borstkanker is een complexe, multifactoriële aandoening, waarbij een sterke interactie
verondersteld wordt tussen genetische en omgevingsfactoren. Leeftijd, geslacht, levenswijze
en de familiale voorgeschiedenis zijn belangrijke risicofactoren voor het ontwikkelen van
borstkanker (7, 8).
Hoewel mannen ook borstkanker ontwikkelen is het risico bij vrouwen ongeveer 100 maal
hoger. Het verhoogd risico dat men verkrijgt door enkel en alleen vrouw te zijn is te wijten
aan het feit dat vrouwen meer borstweefsel hebben in vergelijking met mannen en dat ze
gedurende hun leven continu worden blootgesteld aan groeistimulerende hormonen
(oestrogeen en progesteron). Zo zijn ook verschillende gynaecologische gebeurtenissen,
waarbij een verlengde of verhoogde blootstelling van oestrogeen plaatsvindt, gelinkt met een
verhoogd risico op borstkanker: late menopauze, geen of weinig bevallingen of op latere
leeftijd en vroege menarche. Daarentegen zou langdurige borstvoeding een beschermende
factor zijn.
4
Een ander belangrijk risicofactor is de leeftijd, borstkanker wordt vaak opgesplitst in lateonset en early-onset. Uit verscheidene studies is gebleken dat de incidentie op borstkanker
verhoogd met toenemende leeftijd. Dit suggereert dat langdurige accumulatie van genetische
afwijkingen noodzakelijk is voor zowel de initiatie als de progressie van (borst)kanker.
Daarnaast worden oudere personen gedurende een langere periode blootgesteld aan mogelijke
risicoverhogende factoren.
Veranderingen in de levenswijze van een persoon kunnen het risico op het ontwikkelen van
borstkanker verlagen zoals een dieet met een laag vetgehalte, fysieke activiteit, stoppen met
roken en het beperken van de alcoholinname.
Het hebben van een familiegeschiedenis met clustering van borstkanker is één van de sterkste
risicofactoren. Hierbij stelt men vast dat het individueel risico stijgt met een stijgend aantal
verwanten met borstkanker en de dalende leeftijd waarbij het werd vastgesteld (7, 8).
2.3. GENETISCHE VOORBESCHIKTHEID
2.3.1. INLEIDING
Ongeveer 5-10% van alle borstkankerpatiënten worden geacht te wijten te zijn aan erfelijke
factoren (7-10). Erfelijke borstkanker werd drie decaden geleden voor het eerst beschreven,
het komt doorgaans samen voor met een sterk belaste familiegeschiedenis en manifesteert
zich vaak bilateraal of op jongere leeftijd (9, 11). Uit deze vaststelling blijkt dat genetische
factoren belangrijke determinanten zijn voor het bepalen van het borstkankerrisico (9, 11).
Figuur 2: Indeling in hoog, intermediair en laag
penetrante borstkankersusceptibiliteitsgenen.
Genotype-fenotype correlatie van heterozygote en
homozygote/compound heterozygote mutatiedragers voor borstkankerpredipositiegenen en
laagpenetrante borstkankersusceptibiliteit SNPs.
Genen zijn alfabetisch geordend in elke groep
(10).
5
Genen geassocieerd met een verhoogd risico op borstkanker worden ingedeeld volgens hun
penetrantie, we onderscheiden hoog, matig en laag penetrante genen (zie bovenstaande figuur
2) (10).
2.3.2. HOOG PENETRANTE BORSTKANKERGENEN
Hoog penetrante genen worden gekarakteriseerd door multipele, individueel zeldzame, lossof-functions mutaties die geassocieerd zijn met een hoog risico op de ontwikkeling van
borstkanker (relatief risico (RR) >10) (7, 12).
2.3.2.1. BRCA1 EN BRCA2
A)
BRCA1
In 1990 werd breast cancer susceptibility gene 1 (BRCA1) voor het eerst gelinkt aan de 17q21
chromosomale arm en werd vervolgens in 1994 door middel van positionele clonering
geïdentificeerd (13). BRCA1 is een groot gen met 22 coderende exonen (in totaal 24 exonen),
waarbij exon 11 ongeveer 50% van de coderende sequentie beslaat (7, 13, 14). Het is een
tumorsupressorgen waarbij het ene allel vaak mutaties draagt, waarbij er verlies of reductie
van de genfunctie optreedt, en waarbij het overige allel verloren is gegaan in het tumorweefsel
(15). De evidentie van een tumorsupressorgen wordt verder ondersteund door volgende
observaties: bij inhibitie van BRCA1 expressie zag men groeiacceleratie van zowel de
normale als kwaadaardige epitheliale borstcellen. Terwijl na transfectie van het wild-type
BRCA1 in borstkankercellijnen inhibitie van groei werd geobserveerd. Hierbij werd
vastgesteld dat de waargenomen groei-inhibitie tumorspecifiek is, het kan de groei van andere
tumortypes niet inhiberen (15).
BRCA1 codeert voor een 1863 aminozuur (AZ) eiwit, waarvan het grootste deel geen
similariteit vertoont met andere gekende eiwitten (16-19). Uitzonderingen hierop zijn aan de
N-terminus een sterk geconserveerd really interesting new gene (RING) domein (exon 2-5) en
in de C-terminus 2 BRCA1 carboxyl terminus (BRCT) repeats (16). RING domeinen zijn
zinc-bindende domeinen bestaande uit geconserveerde Cys3-His-Cys4 patronen die eiwit-eiwit
en eiwit-DNA interacties mediëren (17). Deze domeinen zijn meestal betrokken bij de
ubiquitinilatie pathway, waardoor het BRCA1 RING domein zou kunnen functioneren als een
ubiquitine-ligase, om zo eiwitten te merken voor degradatie via proteosomen (18). Het RING
vinger domein is ook de regio waar heterodimerisatie plaatsvindt van BRCA1 en BRCA16
associated RING domein 1 (BARD1) en binding met BRCA1-associated protein (BAP1), een
de-ubiquitinatie enzyme (18). De BRCT module daarentegen vertoont grote similariteit met
de C-terminale regio van een p-53 bindend eiwit (53BP1) en RAD9, die betrokken zijn in
DNA herstel (19). ( Zie figuur 3)
Figuur 3: BRCA1 en BRCA2 functionele domeinen (20).
B)
BRCA2
Een jaar na de ontdekking van BRCA1 werd een tweede borstkankersusceptibiliteitgen, breast
cancer susceptibility gen 2 (BRCA2), geïdentificeerd eveneens dmv positionele klonering
(21). Dit gen is gelegen op chromosoom 13q12 (7, 14, 21) en wordt net als BRCA1
beschouwd als een tumorsupressorgen. Dit gen bestaat uit 27 exonen, waarvan 26 coderend
zijn (exon 1 niet) en overspant ongeveer een 84 kb regio van het genomische DNA (gDNA)
(22). Net zoals bij BRCA1 beslaat het centrale exon 11 meer dan 50% van de coderende
sequentie (22).
BRCA2 codeert voor een groot eiwit bestaande uit 3418 AZ, waarbij geen sequentie
similariteit gevonden werd met andere gekende eiwitten (16, 22). In de regio van het eiwit dat
gecodeerd wordt door exon 11 werd een BRC motief bestaande uit 8 kopijen van 30-80
aminozuren geïdentificeerd dat bindt met RAD51 (20, 22). Daarnaast bevat dit eiwit ook nog
een DNA-bindend domein dat kan binden met enkelstrenging (ssDNA) en dubbelstrengig
DNA (dsDNA) (20). Dit domein is opgebouwd uit 5 componenten: een 190 AZ α-helicaal
domein, 3 oligonucleotide bindende-domeinen en een tower domein (20). De C-terminus
bevat een nucleair lokalisatie signaal (NLS) en op AZ 3191 een cycline-afhankelijke kinase
fosforylatieplaats dat ook kan binden met RAD51 (20). PALB2-binding gebeurt aan de Nterminus via AZ 21-39 (20). (Zie figuur 3)
7
C)
F UNCTIE
BRCA1 en BRCA2 worden geëxpresseerd in een brede waaier van weefsels, waarbij ze een
gelijkaardig ruimtelijk en temporeel expressiepatroon vertonen (16). Beide genen coderen
voor multifunctionele eiwitten, die een belangrijke rol vervullen in het onderhouden van de
genomische
stabiliteit.
Ze
vervullen
deze
functie
door
het
herstel
van
DNA
dubbelstrengbreuken (DSB) te vergemakkelijken en via hun betrokkenheid in homologe
recombinatie (7, 14, 18, 20). Dit laatste is een vitaal DNA herstelproces dat gebruik maakt
van het niet-beschadigde zusterchromatide om DSB te herstellen (20). Deze vorm van
beschadiging van het DNA wordt beschouwd als de meest gevaarlijke vorm van DNA schade,
doordat de integriteit van beide strengen van het DNA simultaan aangetast is (20).
BRCA1 is betrokken bij verscheidene processen zoals DNA herstel door homologe
recombinatie en nucleotide excisie, checkpointcontrole en in de regulatie van de celcyclus
(20, 23). In het herstelproces van DSB functioneert BRCA1 als een signaalgeleider, net als
CHEK2, en zal helpen bij de rekrutering van RAD51 via interactie met BRCA2 en PALB2
(20). Daarnaast is BRCA1 ook direct betrokken bij het herstel van DSB dmv van
wisselwerking met andere eiwitten. De activatie van verschillende (G1/S, S-fase en G2/M)
checkpoints wordt gereguleerd door onder andere het BRCA1-BARD1 complex (20). (Zie
figuur 4)
Net zoals BRCA1 vervult BRCA2 een belangrijke rol in herstel, via homologe recombinatie,
door controle uit te oefenen op de RAD51 filament formatie (7, 8, 20). Daarnaast ondersteunt
BRCA2 de verplaatsing van het RAD51 recombinase naar de plaats van DSB (23). BRCA2
zal zorgen voor stabilisatie van de RAD51 multimeren, zodanig dat homoloog herstel kan
plaatsvinden. Dit proces is niet enkel essentieel voor homologe recombinatie, maar het is ook
verantwoordelijk voor de tumorsupressieve functie (20). Een tweede functie van BRCA2 is
weggelegd in het beschermen van de replicatievork en voorkomt replicatievork stalling (14).
8
Figuur 4: Model van dubbelstrengbreuk herstel mechanisme en de Fanconi Anemia pathway en de link tussen
de twee processen (23).
D)
G EASSOCIEERD R ISICO EN MUTATIESPECTRUM
Pathogene mutaties in deze genen komen relatief zelden voor (gecombineerde frequentie van
0.4% in de algemene populatie (7)) en zijn gerelateerd met een 10- tot 20-voudig verhoogd
risico op borstkanker (7, 14). Ze verhogen de kans op het ontwikkelen van borstkanker tijdens
het leven met 60-85% (BRCA1) en 40-85% (BRCA2) waarbij men een sterk verhoogd RR ziet
op jongere leeftijd (<35 jaar) (7). Pathogene mutaties in deze genen zijn ook verantwoordelijk
voor een verhoogd risico op ovariumkanker met een levensduur risico van 40-60% voor
BRCA1 en tot 30% voor BRCA2 (7). Alhoewel er geen bruikbaar genotype-fenotype correlatie
blijkt te bestaan, heeft men vastgesteld dat BRCA1 mutaties in het 5’ deel van het gen en
mutaties gelegen in de ovarium cluster regio, gelegen in exon 11 en geflankeerd door
nucleotiden 3035-6629, van BRCA2 geassocieerd zijn met een hoger risico op ovariumkanker
(7).
9
Mutaties in deze genen worden doorheen de volledig coderende sequentie aangetroffen,
hierbij maken mutaties die leiden tot een vervroegd stopcodon (frameshift en nonsense
mutaties) het grootste deel uit (7, 14). Missense mutaties zijn verantwoordelijk voor ongeveer
2% van de pathogene mutaties in BRCA1 en deze mutaties komen ook frequent voor in
BRCA2 (7). Het probleem bij deze mutaties is dat ze vaak moeilijk te interpreteren en te
onderscheiden zijn van neutrale sequentie varianten (7). De frequentie van voorkomen van
deze mutaties is verschillend van populatie tot populatie en zowel in BRCA1 als in BRCA2
komen sommige mutaties, founder mutaties genaamd, frequenter voor in bepaalde etnische
groepen: bijvoorbeeld c.68_69delAG en c.5266dupC in BRCA1 (1.2% van de populatie) en
c.5946delT in BRCA2 in de Ashkenazi Joodse populatie (7, 14, 23). Ondertussen werden
reeds honderden verschillende mutaties in de BRCA genen geïdentificeerd.
Biallelische mutaties werden tot nu toe nog niet aangetroffen in BRCA1, waarschijnlijk
doordat deze lethaal zouden zijn voor het embryo (24). Wel werd reeds aangetoond dat
biallelische BRCA2 mutaties een zeldzame subgroep van Fanconi anemia (FA-D1)
veroorzaken (24).
2.3.2.2. A NDERE HOOG PENETRANTE GENEN
Andere
hoog
penetrante
genen
werden
geïdentificeerd
als
deel
van
erfelijke
kankersyndromen, waarbij ziekte veroorzaakt wordt door mutaties in één enkel allel
(dominante aandoening) (25). Mutaties in TP53, PTEN en STK11 worden in tegenstelling tot
BRCA1 en BRCA2, in minder dan 1% van de familiale clustering van borstkanker
aangetroffen (8). Zo veroorzaken germinale mutaties in het TP53 gen het Li-Fraumeni
syndroom, mutaties in het PTEN gen het Cowden syndroom en mutaties in STK11 het PeutzJegher syndroom. Al deze vermelde syndromen zijn geassocieerd met een verhoogd risico op
borstkanker (10, 25). (Zie tabel 2)
Tabel 2: Borstkanker-geassocieerde kanker predisponerende syndromen (20).
10
Aangezien verscheidene genetische linkage studies faalden om andere hoog penetrante genen
te identificeren werd het duidelijk dat de overblijvende clustering van borstkanker niet
verklaard zal kunnen worden door overerving van varianten in additionele hoog penetrante
borstkankergenen met dezelfde prevalentie als BRCA1/2 (7, 8). Alhoewel gemeenschappelijke
omgeving en levensstijl factoren een deel van de familiale clustering van borstkanker zouden
kunnen verklaren, blijkt uit tweelingenstudies dat een significant deel van het overblijvende
familiaal borstkankerrisico te wijten is aan genetische factoren. Zo stelde men vast bij
eeneiige tweelingen, waarvan 1 van deze gediagnosticeerd was met borstkanker, dat het risico
voor de niet-aangetaste tweeling hoger is dan in het geval van twee-eiige tweelingen (7).
Er wordt verondersteld dat de overblijvende clustering van borstkanker het best verklaard kan
worden door uit te gaan van een polygenisch model, waarbij verscheidene matig en laag
penetrante allelen samen kunnen leiden tot een verhoogd borstkankerrisico, alhoewel dit het
bestaan van andere zeldzame maar hoog penetrante allelen niet volledig uitsluit (7).
2.3.3. MATIG PENETRANTE BORSTKANKERGENEN
Matig of intermediair penetrante borstkankersusceptibiliteitsgenen, ATM, CHEK2, BRIP1 en
PALB2 zijn gekenmerkt door zeldzame, loss-of-functions mutaties die zorgen voor een
bescheiden toename in het borstkankerrisico (RR 2-4) (7, 12). Deze matig penetrante
borstkankergenen werden ontdekt door hun gemeenschappelijk voorkomen in de DNA
herstelpathway, waarin BRCA1 en BRCA2 ook een rol spelen (7, 8).
2.3.3.1. ATM
In 1976 werd er voor het eerst een link gelegd tussen borstkanker en het AtaxiaTelangiectasia mutated (ATM) gen, bijna 2 decaden later werd dit bevestigd (8, 23). Ataxiatelangiectasia (AT) is een zeldzame autosomale recessieve ziekte met predispositie voor
kanker, waarbij het ATM gen mutaties draagt (23). Deze ziekte is gekarakteriseerd door
progressieve
neuronale
degradatie,
immunologische
deficiënties,
gevoeligheid
aan
ioniserende straling en een verhoogd risico op ontwikkeling van kanker (26). Het merendeel
van de AT patiënten zijn compound heterozygoot of homozygoot (beide allelen zijn
aangetast) voor mutaties in het ATM gen (8, 27). De link tussen ATM en borstkanker werd
gelegd toen men vaststelde dat er meer borstkankerpatiënten voorkwamen dan verwacht bij
familieleden van AT patiënten en dit voornamelijk bij de obligaat heterozygoten (één van de
allelen draagt een mutatie) (26).
11
Het ATM gen werd gelokaliseerd op chromosoom 11q via genetische linkage analyse in 1988
en geïdentificeerd door positionele clonering in 1995 (27). Dit gen bestaat uit 66 exonen,
waarvan 64 coderen voor een 3056 AZ eiwit (27). ATM behoort tot de phosphatidylinositol
3-kinase-gerelateerde kinases (PIKK), deze worden gekarakteriseerd door een domein dat
gelijkaardig is aan deze in phosphatidylinositide 3-kinases (PI3K) en zijn actieve
serine/threonine kinasen (26, 27).
ATM bevat aan zijn C-terminus een FAT domein (FRAP, ATM, TRAPP) met een sterk
geconserveerde staart, ook wel gekend als het FATC domein (26, 27). Het ATM FATC
domein functioneert als een bindingssite voor het acetyltransferase TIP60, waarbij het lysine
op positie 3016 van ATM geacetyleerd
wordt. Deze acetylering is belangrijk voor
de activatie van dit eiwit (27, 28). Verder
bevat
ATM
verscheidene
elongatiefactor
aan
HEAT
3
zijn
N-terminus
(Hungtingtin,
(EF3),
protein
phosphatase 2A (PP2A) en TOR1)
domeinen, die interacties met andere
Figuur 5: Schematische weergave van ATM (28).
eiwitten kan beïnvloeden, en een regio
essentieel voor substraatbinding (SBS) (26-28).
ATM codeert voor een serine/threonine kinase en heeft een centrale rol in het herstel van DSB
(20, 27). Het zorgt voor herkenning van beschadigd DNA, rekrutering van DNA herstelgenen,
het doorgeven van signalen naar de checkpoints van de celcyclus, transcriptionele regulatie en
activatie van gecontroleerde celdood (20, 27). In normale omstandigheden is ATM aanwezig
als inerte di- of multimeren, maar bij detectie van DSB zal het dissociëren in actieve
monomeren, waarbij het autofosforylatie zal ondergaan van het Serine op positie 1918 (27).
Op de plaats van DNA schade zal het zorgen voor de initiatie van een fosforylatiecascade,
waarbij verschillende eiwitten downstream geactiveerd worden, zoals BRCA1, CHEK2 en
p53 en deze zullen op hun beurt zorgen voor een stop in de celcyclus, DNA herstel of
gecontroleerde celdood (26). ATM en CHEK2, dat voor zijn activatie afhankelijk is van
ATM, zullen na ioniserende straling zorgen voor stabilisatie van p53 door het plaatsen van
een fosfaatgroep op het serine op positie 15 (20, 26). Zoals eerder vermeld wordt BRCA1 ook
gereguleerd door ATM via fosforylatie op verschillende plaatsen van het eiwit (Ser-1387,
Ser-1423, Ser-1457, Ser-1524) (26). (Zie figuur 5)
12
Verschillende mutaties zijn reeds geïdentificeerd in ATM die geassocieerd zijn met een RR
van ongeveer 2 (8). Niet enkel truncerende varianten, maar ook missense varianten,
voornamelijk gelegen in het 3’deel van de coderende regio’s (kinase, FATC-domein) zijn
gerelateerd met een verhoogd risico op borstkanker (8, 29). Er werd gesuggereerd dat
sommige missense varianten in het ATM gen zouden kunnen aanleiding geven tot dominant
negatieven, waardoor ze een hoger risico op borstkanker in heterozygote vorm kunnen
veroorzaken, terwijl ze een mildere vorm van AT vertonen in homozygote vorm (30).
Hieropvolgend werd een missense mutatie in ATM (c.7271T>G, p.Val2424Gly)
geïdentificeerd door Chenevix et al. dat geassocieerd is met een 9 tot 13-voudig verhoogd
risico op borstkanker en zich als een dominant negatieve zou gedragen (26, 30).
2.3.3.2. CHEK2
Het checkpoint kinase 2 (CHEK2) gen werd in 2002 als eerste matig penetrante
borstkankergen herkend (8, 23). Identificatie van dit gen gebeurde via linkage analyse, in een
groot BRCA1/2 mutatie negatieve familie, dat verwees naar de chromosomale regio 22q. Deze
regio werd later geïdentificeerd als CHEK2 (8). Initieel werd CHEK2 gezien als een LiFraumeni syndroom (LFS) gen, maar na identificatie van de 1100delC mutatie bleek dat de
populatiefrequentie van deze mutatie te hoog was om een gen te zijn van dit sterk penetrant en
zeer zeldzaam syndroom (8).
Het CHEK2 gen overspant ongeveer 50 kb van het genomisch DNA en is opgebouwd uit 14
exonen. Het CHK2 eiwit vertoont drie karakteristieke domeinen: het N-terminale SQ/TQ
cluster domein, het fork head-associated (FHA) domein en een katalytisch kinase domein.
(31). Het N-terminale SQ/TQ cluster domein (AZ 20-75) is een regulatorisch domein
bestaande uit 7 serine of threonine residu’s gevolgd door glutamine. Het wordt ook
beschouwd als een mogelijke fosforylatie plaats dat de voorkeur geniet van het ATM eiwit
(31, 32). Het tweede domein, FHA domein (AZ 112-175), is betrokken bij de binding van
gefosforyleerde eiwitten, gedeeltelijk via herkenning van fosfothreonine residu’s. Deze regio
zorgt voor dynamische eiwit-fosfoeiwit interacties met CHK2 gedurende activatie en
signalering bij DNA schade (31, 32). Het derde domein is het katalytisch kinase domein (AZ
225-490), die bijna de volledige C-terminus overspant en deze vertoont structurele homologie
met andere serine/threonine kinases (31, 32). (zie figuur 6)
13
Figuur 6: Schematische weergave van CHK2.
Het CHK2 eiwit bevat 543 aminozuren en heeft drie verschillende functionele domeinen; SQ/TQ cluster domein
(SCD); fork-head-associated (FHA) domein en kinase domein met een T-loop. (32)
ATM zal in respons op ioniserende straling en andere genotoxische stresstoestanden, die DSB
voortbrengen, CHK2 activeren op de plaats van DNA schade. De activatie van CHK2 gebeurt
door fosforylatie van verschillende SQ/TQ domeinen, voornamelijk het threonine 68.
Hierdoor vindt homodimerisatie plaats via het FHA domein, wat op zijn beurt zal leiden tot
trans-autofosforylatie van CHK2 op threonine 383 en 387 en op serine 516. Deze threonines
zijn gelegen in de autoinhibitorische loop van het eiwit en het serine 516 in het kinase domein
wat zal leiden tot volledige activatie van het eiwit (33). Het geactiveerde CHK2 eiwit zal zich
verplaatsen doorheen het nucleoplasma om zo het signaal van het beschadigde DNA te
verspreiden en zijn downstream substraten te activeren (20, 33). De reeds gekende substraten
zijn: eiwitten betrokken in de celcyclus controle (CDC25 familie eiwitten, PLK3 kinase en
E2F1 TF) en bij DNA herstel (via onderandere fosforylatie van serine 988 van BRCA1) en bij
de regulatie van celdood (p53 en PML1) (7, 14, 23, 31, 33).
Het CHEK2 gen is een tumorsupressor gen waarbij germinale mutaties geassocieerd zijn met
borstkanker in verschillende populaties (33). Het RR voor de germinale mutatie, CHEK2
1100delC, bij homozygote en heterozygote dragers wordt geschat op respectievelijk 101.34
en 4.04 in patiënten die geen BRCA mutatie dragen (34). Deze mutatie is gelegen in exon 10
in het kinase domein en is aanwezig in 0.2-1% van de Europese bevolking en in ongeveer
4.2% van de borstkankerfamilies, alhoewel de mutatiefrequentie varieert tussen verschillende
populaties (7, 33).
Door de ontdekking van deze mutatie in CHEK2, werd dit gen beschouwd als een borstkanker
susceptibiliteitsgen, wat geleid heeft tot verscheidene mutatiescreeningen van het volledige
gen (7). Uit deze analyses werden nog 3 additionele varianten in dit gen geïdentificeerd, 2
truncerende mutaties: IVS2+1G>A, del5395 en de missense variatie: I157T geassocieerd met
borstkanker in Oost-Europa (35). Hoewel er weinig geweten is over de impact van missense
mutaties op de functie van het eiwit, bleek uit de eerste bevindingen dat substituties in het
FHA domein en het kinase domein meestal zullen leiden tot verlies van de functie/activiteit
van het CHK2 eiwit en dus meestal van pathogene aard zouden zijn (35). Net zoals bij de
14
1100delC blijkt dat deze varianten ook leiden tot een matig verhoogd risico op borstkanker,
waarbij de mutatiefrequentie varieert naargelang de populatie (8).
Uit verscheidene studies is gebleken dat men een lagere mutatiefrequentie terugvindt van
CHEK2 in familieleden van BRCA1/2 mutatie positieve stambomen in vergelijking met
familieleden afkomstig van BRCA1/2 mutatie negatieve stambomen (12). De verklaring
hiervoor zou functionele redundantie kunnen zijn, waarbij de aanwezigheid van een mutatie in
1 gen (in dit geval BRCA1 of BRCA2) geen of weinig additioneel risico ondervindt door de
aanwezigheid van een mutatie in een tweede gen (CHEK2) (12). In tegenstelling tot BRCA1
en BRCA2 mutaties bleek dat mutaties in CHEK2 niet leiden tot een verhoogd risico voor de
ontwikkeling van ovariumkanker (31).
2.3.3.3. BRIP1
Het BRCA1 interacting protein C-terminal helicase 1 (BRIP1) wordt ook wel BACH1 of
FANCJ (voor Fanconi anemia complementation Group J) genoemd (8, 23, 36, 37). Fanconi
anemia (FA) is een zeldzame (prevalentie van ongeveer 1 op 350000 geboortes) genetische
aandoening, gekarakteriseerd door multipele congenitale afwijkingen, extreme gevoeligheid
aan agentia die DNA interstrand crosslinks induceren, predispositie voor kanker en
beenmergfalen (8, 23, 36). Het is een genetisch heterogene aandoening onderverdeeld in 13
subtypes (FANCA-FANCN) (8, 23, 36, 37). Biallelische inactivatie van BRIP1 werd
aangetoond en beschreven in patiënten met Fanconi Anemia behorende tot de subtype J,
vandaar dat BRIP1 ook wel FANCJ wordt genoemd (36, 38). In 2002 werden BRCA2
mutaties geassocieerd met FA subtype D1 wanneer deze overgeërfd werden in homozygote of
compound heterozygote toestand (8). Deze link tussen FA en borstkanker heeft geleid tot
mutatie analyse van gekende FA genen als mogelijke borstkanker susceptibiliteitsgenen (8).
BRIP1 is gelegen op chromosoom 17q23, naast de BRCA1 locus, en bestaat uit 20 exonen die
meer dan 180 kb van het genomisch DNA overspannen (36, 38). Het codeert voor een 1249
AZ eiwit, dat zal binden met de BRCT domeinen in BRCA1, hiervoor ligt in de C-terminus
een fosfo-SXXF motief (AZ 990-993) (36, 38). Deze binding is afhankelijk van de celcyclus
gereguleerde fosforylatie van BRIP1 op het serine op positie 990 (36, 38). Naast het BRCA1bindend domein (AZ 979-1006) vertoont BRIP1 aan zijn N-terminale zijde sequentie
homologie met de katalytische en nucleotide-bindende domeinen van gekende leden van de
DEAH helicase familie, wat het helicase domein (AZ 1-888) genoemd wordt (36, 38). Later
werd vervolgens aangetoond dat BRIP1 een ATP-afhankelijke 5’-3’ DNA helicase is. In deze
15
N-terminale regio ligt ook een voorspeld NLS, alsook een ijzer-zwavel (Fe-S) cluster domein
die essentieel zijn voor de enzymactiviteit (36). (Zie figuur 7)
Figuur 7: Structurele weergave van het humane FANCJ/BRIP1 (39).
BRIP1 zal, via binding met BRCA1, co-lokaliseren naar de plaats van DNA schade, waar het
zal bijdragen tot DNA herstel (38). De BRIP1-BRCA1 interactie is nodig voor tijdelijk herstel
van DNA DSB en voor DNA schade-geïnduceerde checkpoint controle gedurende de G2-M
fase van de celcyclus (38).
BRIP1 werd verondersteld, net als de andere genen, een tumorsupressor gen te zijn, waarbij
verlies van het wildtype allel en retentie van het mutante allel teruggevonden wordt in
tumorcellen. Later werd dit ook bevestigd (36). In verschillende studies werd een beperkt
aantal pathogene mutaties in BRIP1 aangetroffen, deze bestonden uit heterozygote
truncerende en missense mutaties, die frequenter voorkwamen in borstkankerpatiënten dan in
gezonde controles (8). Het merendeel van deze mutaties zijn gelegen dichtbij/in het helicase
domein en sommige van deze mutaties waren reeds geïdentificeerd in homozygote of
compound heterozygote toestand in FA-J patiënten (8, 36). Men schat dat deze mutaties
aanleiding zouden geven tot een tweevoudig verhoogd risico op de ontwikkeling van
borstkanker, met een hoger risico voor vrouwen jonger dan 50 jaar (8).
2.3.3.4. PALB2
Partner and localizer of BRCA2 (PALB2) werd voor het eerst klinisch gekarakteriseerd in
patiënten met FA subtype N en wordt hierdoor ook wel FANCN genoemd (40, 41). FA-N is
geassocieerd met biallelische mutaties in PALB2, maar na de ontdekking van de link tussen
FA en borstkanker, werd ook PALB2 onderzocht als een mogelijk borstkankersusceptibiliteitsgen (40).
PALB2 is gelegen op chromosoom 16p12.1 waar het een regio van 38.2 kb beslaat (25, 42).
Het gen bestaat uit 13 exonen, waarvan exon 4 en exon 5 veel groter zijn dan de andere
exonen, en codeert voor een 1186 AZ eiwit (25, 42). Dit eiwit bevat aan zijn C-terminus een
zeven-bladig β-propeller WD40-domeinen (AZ 836-1186), deze regio’s zijn vaak betrokken
16
in eiwit-eiwit interacties en uit onderzoek is gebleken dat het kan binden met het N-terminale
deel van het BRCA2 eiwit (AZ 21-39) (42, 43). Deze binding is belangrijk voor de lokalisatie
van BRCA2 in de nucleaire chromatine structuren. Aan de andere zijde, de N-terminus,
bevindt zich een coiled-coil domein (AZ 9-44) dat zal interageren met een regio dat enkele
residues voor de BRCT-domeinen gelegen is van het BRCA1 eiwit (AZ 1393-1424) (42, 43).
Daarnaast kan PALB2 ook binden met RAD51 via AZ 1-200 en 836-1186 (Zie figuur 8) (42,
43).
Figuur 8: PALB2 exonische structuur en interacties met andere eiwitten
(36).
Via BRCA1 zal er accumulatie gebeuren van PALB2 en BRCA2 in regio’s van de cel waar
DNA schade werd gesignaleerd, waarbij deze drie eiwitten samen zullen lokaliseren naar deze
omgeving. Hierbij verzorgt PALB2 de fysieke en functionele link tussen BRCA1 en BRCA2
die nodig is in het antwoord op DNA schade. Hierbij is RAD51 eveneens betrokken (44, 45).
Heterozygote mutaties in PALB2 werden voor het eerst gedetecteerd in families met
hereditaire borstkanker in 2007 (46) en zijn geassocieerd met een 2 tot 6-voudige verhoging
van het risico op het ontwikkelen van borstkanker afhankelijk van de variant (8, 40).
Pathogene PALB2 mutaties worden teruggevonden over de gehele coderende sequenties en
het overgrote deel hiervan zijn truncerende frameshift en stop codon mutaties (8, 42). In Finse
en Frans Canadese families werden founder mutaties geïdentificeerd, waarbij de steeds
terugkerende Finse c.1592delT mutatie geassocieerd is met een cumulatief risico van 40% bij
een leeftijd van 70 jaar (42). Dit risico is vergelijkbaar met het risico gerelateerd aan BRCA2
mutaties (45%) (42, 47). Ook werd een nog andere mutatie geïdentificeerd (c.3113G>A), die
nog een groter cumulatief risico opleverde (42). PALB2 mutaties worden ook teruggevonden
17
in ongeveer 1% van de populatie van mannelijke borstkankerpatiënten en werd in 2009
geïdentificeerd in families met pancreas kanker (42).
2.3.4. LAAG PENETRANTE VARIANTEN
De nog onverklaarde fractie van familiale clustering van borstkanker zou, voor een deel,
kunnen verklaard worden door de interacties van een combinatie van individuele varianten,
die geassocieerd zijn met een licht verhoogd risico op borstkanker (RR 1.08-1.26) (12, 25).
Deze laag penetrante varianten werden geïdentificeerd dmv genoomwijde associatie studies in
heel grote studiepopulaties (12). Het doel van deze studies was om polymorfismes te
identificeren die elk maar een klein individueel verhoogd risico gaven op borstkanker.
Aangezien dat deze polymorfismes heel frequent voorkomen in de populatie, kunnen ze een
significante impact hebben op het borstkankerrisico (10). De lijst van deze laag penetrante
varianten blijft aangroeien.
2.4. DOEL- EN VRAAGSTELLING
2.4.1. MODEL VAN BORSTKANKERSUSCEPTIBILITEIT
BRCA1 en BRCA2 mutaties, alhoewel ze zeldzaam zijn,verklaren ongeveer 15-20% van de
familiale clustering van borstkanker (8). Als verklaring voor de overblijvende familiale
clustering van borstkanker zijn er 2 (mutueel niet-exclusieve) hypotheses. Enerzijds gaat men
ervan uit dat de ontbrekende familiale clustering te wijten zou kunnen zijn aan het cumulatief
effect van frequente allelen geassocieerd met laag tot matig verhoogd risico op borstkanker
(polygenisch model). Anderzijds zouden verschillende zeer zeldzame mutaties in
hoogpenetrante genen een verklaring voor de familiale vormen van borstkanker kunnen zijn.
Evidentie voor beide hypothesen is reeds gevonden : zie figuur 9 en bovenstaande tekst.
Figuur 9: Genetische architectuur van borstkanker ((48, 49); aangepast door Kim De Leeneer).
18
Het ontrafelen van de pathway van het DNA herstel mechanisme heeft bijgedragen tot het
identificeren van ‘nieuwe’ borstkankersusceptibiliteitsgenen, alsook tot het ontwikkelen van
nieuwe benaderingen voor targeted therapie (38). Hierdoor heeft men een aantal genen
kunnen identificeren waarbij mutaties geassocieerd zijn met een matig tot sterk verhoogd
risico op de ontwikkeling van borstkanker (ATM, BRIP1, PALB2, CHEK2,…).
Hoewel er de laatste decaden veel vooruitgang is geboekt omtrent het definiëren van de
genetische susceptibiliteit van borstkanker, blijft er nog veel te onderzoeken.
2.4.2. PROBLEEMSTELLING EN DOELSTELLING
2.4.2.1. C DNA ANALYSE
Sinds de start van het mutatie onderzoek in 1997 in het Centrum voor Medische Genetica
Gent (CMGG) werden ongeveer 2000 borstkankerpatiënten uit 1700 niet-verwante families
onderzocht. Deze families voldoen aan volgende inclusiecriteria: families met 3
eerstegraadsverwanten
met
borstkanker
en/of
ovariumkanker
of
minimum
2
eerstegraadsverwanten met borstkanker en/of ovariumkanker vóór een gemiddelde leeftijd
van 50 jaar. De inclusiecriteria die gehanteerd worden voor de selectie van sporadische
patiënten: diagnose van borst- of ovariumkanker voor de leeftijd van 38 jaar of diagnose van
bilaterale of multipele ipsilaterale borstkanker, waarbij alle tumoren werden vastgesteld vóór
een gemiddelde leeftijd van 50 jaar of diagnose van gelijktijdige borst en ovariumkanker,
waarbij beide kankers vastgesteld werden vóór een gemiddelde leeftijd van 50 jaar. In minder
dan 15% van de families die voldoen aan bovenstaande criteria werd een mutatie gevonden in
BRCA1&2. Daarnaast werd volledig mutatie onderzoek uitgevoerd van ATM en PALB2 in
250 niet-verwante families. Er werd in ongeveer 4% van de onderzochte patiënten een mutatie
aangetroffen in het ATM gen en in ongeveer 1% in PALB2. In een standaard mutatie
onderzoek wordt enkel de volledige coderen regio en splice sites onderzocht op gDNA
niveau. Hierdoor kunnen diep-intronische mutaties en mutaties in regulatorische sequenties
die leiden tot aberrante splicing niet gedetecteerd worden.
Het doel van deze studie is om in de families waarin tot nu toe geen moleculaire verklaring
werd gevonden voor de familiale clustering van borstkanker, na te gaan met behulp van
cDNA analyse of germinale mutaties aanwezig zijn in BRCA1/2, PALB2, CHEK2, ATM of
BRIP1. Het voordeel van een studie op cDNA niveau is dat het aantal te onderzoeken
amplicons kleiner is. Bovendien laat dit toe om in deze genen naast de truncerende en
missense mutaties, ook mutaties die buiten de coderende regio gelegen (diep-intronische) te
19
detecteren. Andere voordelen van werken op cDNA niveau is het kunnen aantonen van
bijvoorbeeld inserties van Alu repeats die leiden tot aberrante splicing. Dit mechanisme is
reeds beschreven in het BRCA2 gen en in NF1 (50-52).
Hiervoor zullen stalen van patiënten geselecteerd worden, waarin een erfelijke predispositie
vermoed wordt, op basis van een familiale voorgeschiedenis van borstkanker en/of jonge
leeftijd bij diagnose (=premenopausaal) en waarin mutaties in BRCA1/2 werden uitgesloten
met behulp van gDNA screening.
Detectie van pathogene mutaties in deze genen laat toe om predictief onderzoek aan te bieden
aan familieleden, waardoor aan personen met een germinale mutatie een intensievere followup kan aangeboden worden vanaf jongere leeftijd. Dit laat toe om tumoren te detecteren in
een vroeg stadium- met als gevolg grotere overlevingskansen. Dit onderzoekt kadert in de
onderzoekslijn naar erfelijke borstkanker die loopt in het CMGG.
Een ander deel van dit project zal bestaan uit de evaluatie van het effect op cDNA niveau van
4 potentiële splice varianten in BRCA2. Een effect zoals exon skipping of alternatieve splicing
zal na deze studie kunnen bevestigd of uitgesloten worden. Dit draagt bij tot de interpretatie
van de klinische betekenis van deze varianten zodat adequate counseling van families waarin
deze varianten segregeren mogelijk wordt. De te bestuderen varianten zijn: BRCA2 c.517G>C
(1ste base exon 7) (RNA 1300081); BRCA2 c.681+111C>G (RNA 1300073); BRCA2 c.76186G>T (RNA 1200405) en BRCA2 c.3326C>T (RNA 1300007).
2.4.2.2. E NIGMA PROJECT
De Evidence-based Network for the Interpretation of Germline Mutant Alleles (ENIGMA) is
een international consortium opgericht in 2009 met de bedoeling om zowel data, methoden als
bronnen uit te wisselen om de klinische significantie van ongeclassificeerde varianten (UV) te
achterhalen.
In deze masterproef is er meegewerkt aan een project van de splicing groep, namelijk project
1 dat bestaat uit kwaliteitscontrole. Hiervoor werden er vanuit Australië 20 EBV cellijnen
opgestuurd: 11 controles en 9 cellijnen die een bepaalde variant dragen. In de eerste fase van
dit project wordt er nagegaan met zelf ontworpen primers welke transcripten opgepikt worden
bij patiënten met de BRCA1 c.671-2A>G. Het doel is een vergelijking van de bekomen
resultaten door de verschillende deelnemende groepen.
20
3. MATERIALEN EN METHODEN
3.1. GEBRUIKTE METHODEN
Figuur 10: Flowchart van dit onderzoek.
In eerste instantie dient de selectie te gebeuren van de patiënten die in dit onderzoek voldeden aan de
inclusiecriteria. Hiervan zullen EBV cellijnen worden opgekweekt waaruit vervolgens RNA zal geëxtraheerd
worden en in een volgende stap zal hieruit cDNA gesynthetiseerd worden. Gelijktijdig met dit proces zullen
primers ontwikkeld worden voor PCR en sequenering en deze zullen ook geoptimaliseerd worden. Op dit cDNA
zal sequenering gebeuren voor de 6 geselecteerde genen die vervolgens geanalyseerd zullen worden.
3.2. SELECTIE PATIËNTEN
De patiëntenstalen, die opgenomen werden in dit onderzoek zijn afkomstig van vrouwen die
in het Universitair Ziekenhuis van Gent (UZG), CMGG geconsulteerd zijn op basis van een
vermoeden van een erfelijke vorm van borstkanker. Deze personen werden nog aan extra
inclusiecriteria onderworpen: patiënten uit families met 3 eerste of tweedegraadsverwanten
met borstkanker of 2 eerste of tweedegraadsverwanten met borstkanker voor een gemiddelde
leeftijd van 50 jaar. Bij deze vrouwen werd eerder de coderende regio van BRCA1/2
onderzocht op gDNA niveau, maar er werd geen mutatie gevonden. In totaal zijn 25 vrouwen
uit 24 niet-verwante families geselecteerd voor dit onderzoek. Een overzicht van de
geselecteerde patiënten (RNA nummer, gDNA nummer en familienummer) is terug te vinden
in bijlage 1.
Figuur 11: Schematische weergave inclusie patiënten in onderzoek.
21
3.3. RNA EXTRACTIE
Om complementair DNA (cDNA) te synthetiseren dient men in eerste instantie
ribonucleïnezuur (RNA) te extraheren. Totaal RNA zal geïsoleerd worden uit Epstein-Barr
virus (EBV) cellijnen, die beschikbaar zijn van de bovenvermelde patiënten met behulp van
de RNeasy kit (Qiagen). EBV cellijnen zijn culturen van lymfocyten, die geïnfecteerd worden
met het Epstein-Barr virus, zodanig dat men een geïmmortaliseerde cellijn verkrijgt en dus
een onuitputbare bron van RNA vormen.
Vooraleer er gestart wordt met de RNA extractie zal elke cellijn worden opgesplitst in 2
delen; op één van de aliquots gebeurt een behandeling met puromycine. Puromycine is een
translatie inhibitor en door het toevoegen van deze stof aan de celcultuur zal de mogelijke
degradatie door de nonsense mediated mRNA decay (NMD) gedeeltelijk verhinderd worden
(53, 54). NMD is een kwaliteitscontrole mechanisme van de cel, dat zorgt voor de specifieke
eliminatie van mRNA’s die premature stopcodons bevattten en beschermt ons tegen de
synthese van abnormale eiwitten (55, 56). Door het toevoegen van deze stof wordt het
mogelijk om transcripten waarin mutaties voorkomen te detecteren.
Na de incubatie kan overgegaan worden tot extractie van totaal RNA. De cellijn wordt
overgepipetteerd in een 15ml falcon en afgedraaid (5 minuten, 300 relatieve centrifugale
kracht (rcf)). Het supernatans wordt afgegoten en de overblijvende pellet van lymfocyten
wordt losgemaakt. De cellen worden gelyseerd en gehomogeniseerd door toevoeging van een
RLT-mix (RLT-buffer + β-mercapto). De RLT-buffer bevat guanidine isothiocyanaat (GITC),
wat zorgt voor denaturatie, zodanig dat de RNases snel geïnactiveerd worden en men intact
RNA kan isoleren. Tijdens de denaturatie zal de celwand en de plasmamembranen van cellen
en organellen worden afgebroken zodanig dat het RNA vrijgesteld wordt. In een volgende
stap wordt (70%) ethanol toegevoegd om optimale bindingscondities te verzekeren en dit
mengels wordt vervolgens op de RNeasy mini kolom aangebracht. Deze kolom bevat een
speciaal ontwikkeld silica-gel membraan waaraan het (totaal) RNA kan binden en ervoor
zorgt dat alle overige contaminanten kunnen worden weggewassen. De wasstappen worden
uitgevoerd met de RPE en RW1 buffers. Elutie van het RNA gebeurt door toevoeging van
RNase vrij water (zie flowchart). In een volgende fase zal de concentratie van het
geëxtraheerde RNA bepaald worden, alsook de zuiverheid met behulp van de DropSense96
(Trinean).
22
Figuur 12: Flowchart van RNA extractie met de RNeasy kit (Qiagen) (http://www.qiagen.com).
Het protocol met de gebruikte hoeveelheden kan teruggevonden worden in bijlage 2.
3.4. CDNA SYNTHESE
Voor de synthese van cDNA zullen we gebruik maken van de Superscript II cDNA synthesis
kit van BioRad. Hierbij zal first-strand cDNA gesynthetiseerd worden met behulp van
random primers, vertrekkende van het totaal RNA geëxtraheerd in de vorige stap. Het gebruik
van deze random hexameren als primers is de minst specifieke methode en hierbij zullen alle
RNAs in de populatie gebruikt worden als template bij de cDNA synthese. Er is gekozen voor
superscript, aangezien er gewerkt wordt met longe-range PCR fragmenten, en deze kit in staat
is om reverse transcriptase (RT)-producten te genereren tot 12 kb lang.
Als eerste stap zal het totaal RNA (2µg, aanvullen met sigma water tot 10 µl) samengevoegd
worden met de random primers en deoxyribonucleotide trifosfaten (dNTP’s). Deze dNTP’s
zullen gebruikt worden bij de opbouw van de nieuwe gesynthetiseerde cDNA streng. Deze
mix wordt vervolgens 5 minuten verhit op 65°C om denaturatie van het dubbelstrengig RNA
te verkrijgen, waarna het vervolgens gekoeld wordt op ijs. In een volgende stap wordt een
tweede mix samengesteld die zal toegevoegd worden aan de initiële mix. Deze bestaat uit: 5x
23
RT buffer (250 mM Tris pH 8.3, 375 mM KCl, 15 mM MgCl2), 1M Dithiothreitol (DTT) en
een ribonuclease inhibitor (RNAsin (Promega)). DTT is een reducerend agents dat helpt om
verbindingen, zoals zwavelverbinding, te verbreken, waardoor het RT enzym zijn functie
makkelijker kan uitoefenen. Het RNAsin zal tegelijkertijd eiwitten die het RNA afbreken
(RNases) inhiberen, zodanig dat de kwaliteit van het RNA niet zal worden aangetast. Om
optimale aanhechting van de random hexameren aan het template RNA te verkrijgen zal dit
mengels gedurende 2 minuten op 25°C gebracht worden en na toevoeging van het RT enzym
nog eens gedurende 10 minuten.
SuperScript® II RT is een DNA polymerase dat de complementaire DNA streng zal
synthetiseren vertrekkende van enkelstrengig RNA, die verkregen is uit de RNA extractie.
Aangezien dit enzym een optimale activiteit heeft bij 42°C, zal het gedurende 50 minuten
geïncubeerd worden op deze temperatuur, zodanig dat extensie kan plaatsvinden en dus de
complementaire cDNA strengen gesynthetiseerd kunnen worden.
In deze masterproef werden ook twee andere cDNA synthese kits vergeleken met de
hierboven besproken kit: GoScript (Promega) en M-MuLV Reverse Transcriptase (RNase H-)
(BioLabs). Deze twee andere kits hebben een gelijkaardig werkingsmechanisme en een
gelijkaardig protocol, maar het gebruikte RT enzym is verschillend.
De gedetailleerde protocols van de 3 cDNA synthesis kits zijn terug te vinden in bijlage 3.
3.5. RT-PCR
De polymerase-kettingsreactie (PCR) is de gouden standaard om vanuit minimale
hoeveelheden (c)DNA specifieke gedeeltes (amplicons) te amplificeren, zodanig dat er
genoeg is om hierop analyses uit te voeren.
Dit principe bestaat uit verschillende cycli van 3 opeenvolgende stappen: denaturatie,
aanhechting en extensie, waarbij elke stap zijn eigen specifieke temperatuur heeft. Tijdens de
eerste stap gebeurt de denaturatie waarbij het dubbelstrengig cDNA uit elkaar zal gaan tot
enkelstrengig cDNA onder invloed van een hoge temperatuur (92°C -97°C). Vervolgens zal
er in de tweede fase aanhechting gebeuren van de primers (forward en reverse) op ssDNA
(55°C-65°C). Wanneer de primers gebonden zijn op hun complementair gedeelte van het
cDNA kan gestart worden met de 3de fase, de extensie. De synthese van de nieuwe
complementaire streng zal gebeuren door toevoeging van dNTP’s, magnesiumchloride
(MgCL2) en een hittestabiel DNA polymerase. Deze laatste is een enzym met een 5’->3’
polymerase activiteit, die vanuit de uiteinden van de primers complementaire dNTP’s zal
24
inbouwen, terwijl de doelwitsequentie gelezen wordt van 3’->5’. Hierdoor ontstaat een streng
die complementair is aan de sequentie gelegen tussen de twee primers. Deze nieuw
gesynthetiseerde strengen kunnen dan in een volgende cyclus dienen als template, waardoor
er exponentiële amplificatie gebeurd van de initiële hoeveelheid DNA. In de meeste PCRreactie zal men gebruik maken van 35 opeenvolgende cycli.
In dit onderzoek zal men als DNA polymerase gebruik maken van het Takara Ex Taq DNA
polymerase. Dit enzym heeft een hoge sensitiviteit vanwege de proofreading capaciteit dat
het bevat en is beter geschikt voor amplificaties van langere amplicons.
3.5.1. PRIMERDESIGN
Primers zijn korte sequenties van opeenvolgende nucleotiden (enkelstrengig), die
complementair zijn aan een specifieke doelwitsequentie. Deze zijn zodanig ontwikkeld dat ze
met behulp van PCR de volledige coderende regio van de verschillende genen amplificeren.
Het doel is om overlappende PCR fragmenten te generen van ongeveer 2 kb in lengte. Ook
dienen er sequeneringsprimers ontwikkeld te worden die ongeveer om de 400-500 bp gelegen
zijn in het PCR fragment en die eveneens elkaar overlappen. Voor de PCR fragmenten dient
een forward en reverse primer ontwikkeld te worden. Voor de sequenering zullen we in dit
onderzoek enkel gebruik maken van forward primers.
Voor de ontwikkeling van de primers gaat men uit van de referentiesequentie die men kan
afhalen van Ensembl of UCSC, men kiest hier best voor de cDNA sequentie. Vervolgens
zullen met behulp van de Primer3Plus en Oligocalc software de primers ontwikkeld worden.
Voorwaarden waaraan de primers dienen te voldoen :
Amplicongrootte: PCR primers rond de 2000 basenparen (bp),
sequeneringsprimers rond de 400-500 bp.
Het ideale GC-gehalte is 50%.
De optimale grootte van de primers ligt tussen de 18 tot 22 bp.
De smelttemperatuur (Tm), dit is het punt waarbij de twee DNA strengen uit
elkaar gaan (denaturatie), bedraagt in optimale omstandigheden 60°C. Hierbij
mogen de Tm van de forward en reverse primers niet te ver uit elkaar liggen.
In de primers zijn er best geen single nucleotide polymorfismes (SNP) of
repeats gelegen.
25
In een volgende stap van het primerdesign zal de specificiteit van de naar voorgedragen
primers worden nagegaan. Via Bisearch, primer-BLAST tool van NCBI en de UCSC in-silico
PCR programma kan men nagaan of de primerset nog andere PCR fragmenten zal
amplificeren, dan deze die men wenst. Indien uit één van deze tools blijkt dat de voorgestelde
primerset niet specifiek is dient het volledige proces van primerontwikkeling voor dit paar
opnieuw doorlopen te worden tot men primers ontwikkeld die specifiek zijn.
3.6. OPTIMALISATIE
Na het ontwikkelen van de primers dient nog overgegaan te worden tot optimalisatie van de
PCR reactie. De PCR primers zullen onderworpen worden aan verschillende PCR
programma’s (variabele aanhechtingstemperatuur en extensietijd) en condities om zo tot
specifieke fragmenten te komen. Er zal gebruik worden gemaakt van twee verschillende PCR
programma’s, waarbij het eerste PCR programma een kortere extensietijd heeft dan het
tweede. Het volledig PCR programma is uitgeschreven in figuur 13. Vervolgens zal ook in de
aanhechtingstemperatuur gevarieerd worden van 58°C tot 64°C, dit meestal in stappen van
2°C.
PCR
Programma 1
PCR
Programma 2
94°C
98°C
°C
72°C
72°C
5 min
10 s
30 s
1 min
7 min
94°C
94°C
°C
72°C
94°C
4 min
30 sec
30 sec
3 min
30 sec
x20
15°C
10 min
°C
30 sec
x25 increase
10s
x35
72°C
72°C
elke
stap
3 min
10 min
Figuur 13: De 2 PCR programma’s gebruikt bij optimalisatie van de PCR.
De gekleurde vakjes stellen de aanhechtingstemperatuur voor die kan variëren (58°C -65°C). PCR programma 1
heeft een kortere (1 min) extensietijd in vergelijking met PCR programma 2 (3 min).
Indien dit nog niet tot een bevredigend resultaat leidt zal overgegaan worden tot een gradiënt
PCR, waarbij elk laantje van de PCR een specifieke aanhechtingstemperatuur verkrijgt en dit
in een bereik van 50°C tot 70°C (dit kan men zelf kiezen). Gelijktijdig zullen we hierbij ook
de condities van de PCR reactie aanpassen door toevoeging van dimethylsulfoxide (DMSO).
Deze stof zorgt ervoor dat de Tm verlaagd wordt, alsook de aspecifieke binding van de
primers aan de (c)DNA template, en vergemakkelijkt de amplificatie van GC-rijke zones.
De lengte van het gegenereerde PCR product zal gecontroleerd met fragment electroforese
(Caliper LabChip GX).
26
In een volgende stap dienen de sequeneringsprimers geoptimaliseerd te worden. Hierbij wordt
gestart vanaf het PCR product en zal nagegaan worden of de ontwikkelde primers aanleiding
geven tot interpreteerbare sequenties na Sanger sequenering. Deze sequenties zullen
geanalyseerd worden met Seqscape (Applied Biosystems).
3.7. CALIPER LABCHIP GX
Er werd gebruik gemaakt van Caliper LabChip om DNA- en/of RNA-fragmenten
elektroforetisch te scheiden en vervolgens te visualiseren. Dit toestel vervangt de agarose gel
electroforese methode om na te gaan of het PCR product het correcte fragment bevat en of de
non template controle niet gecontamineerd is. Hiervoor wordt 17 µl H2O en 3µl van het PCR
product gepipetteerd op een 384-well plaat. Vervolgens zal er 150 nanoliter worden
opgezogen per well. De verschillende fragmenten uit een well worden elektroforetisch
gescheiden en vervolgens gedetecteerd via laser-geïnduceerde fluorescentie. De grootte en
concentratie van een specifieke band kunnen bepaald worden door het toevoegen van een
ladder en interne merkers.
De resultaten worden voorgesteld aan de hand van een virtuele gel, grafiek en samenvattende
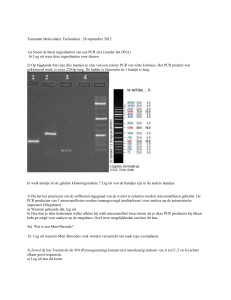
tabellen. Een printscreen wordt voorgesteld in figuur 14.
Figuur 14 : Weergave van de data van de LabChip.
3.8. KLONERING
Klonering zal gebruikt worden om de verschillende verkregen transcripten bij de PCR te
kunnen identificeren, aangezien er 1 transcript per kloon verkregen wordt. Er werd een Topo
TA cloning (Invitrogen) uitgevoerd in One shot TOP10F’ chemische competente E. Coli. Als
27
selectie werd een ampicilline en Xgal (blauw/wit) screening uitgevoerd. Bij de klonering
werd gebruik gemaakt van Taq polymerase geamplificeerde PCR producten. Vervolgens
werden de bekomen witte klonen overgeënt in een mastermix en geamplificeerd in een PCR
reactie. (De klonering werd uitgevoerd door Kim De Leeneer en zal dus niet verder in detail
besproken worden).
3.9. SANGER SEQUENERING
Via sequenering zal de volgorde van de nucleotiden in het DNA worden blootgelegd, waarna
men op zoek kan gaan naar eventuele veranderingen die aanwezig zijn.
3.9.1. PRINCIPE
Eerst dient er een opzuivering te gebeuren van de PCR producten. Dit betreft een
enzymatische opzuivering door toevoeging van Exo-AP (antartic phosphatase en exonuclease
I). Het exonuclease I enzym zal ssDNA degraderen en zal dus de overblijvende primers van
de PCR reactie afbreken, terzelfdertijd zal het antartic phosphatase de 5’ fosfaat groepen van
nucleotiden verwijderen, waardoor de dNTP’s van de PCR reacties inert worden. Vervolgens
wordt deze Exo-AP mix toegevoegd aan het PCR product dat opgezuiverd dient te worden.
De gebruikte enzymen voor de opzuivering dienen geïnactiveerd te worden, zodanig dat deze
de sequentiereactie niet verstoren.
Na opzuivering kan worden overgegaan tot de sequeneringsreactie. Hiervoor wordt een
sequeneringsreactie mix samengesteld uit: ABI-buffer (sequencing buffer (5X)), de gewenste
primer, RR-mix (ready reaction mix). Deze sequeneringsreactie is gebaseerd op de dideoxy
methode, ook wel Sanger methode genoemd. In
de reactiemix zitten naast zowel de 4 deoxynucleotidefosfaten
fluorochroom
ook
gelabelde
in
ondermaat
4
gemodificeerde
nucleotiden: dideoxyNTP’s (ddNTP’s). Tijdens
de synthese van het DNA zal vanaf de primer,
aan de 3’OH kant, nucleotide na nucleotide
Figuur 15: Links: deoxyribonucleoside trifosfaat;
Rechts:
dideoxyribonucleoside
trifosfaat
(http://core.iddrc.org)
worden aangebouwd, die complementair zijn aan de nucleotides van de DNA-streng. De
synthese van het DNA zal doorgaan zolang een dNTP wordt ingebouwd en zal telkens
stoppen wanneer een ddNTP wordt ingebouwd, dit doordat er geen vrij 3’OH meer is die
nodig is voor de verdere synthese. Zo verkrijgt men DNA fragmenten van alle mogelijke
lengtes die allemaal eindigen op een ddNTP. De sequenering zal plaatsvinden tijdens een
28
aantal cycli van hogere temperaturen, die nodig zijn voor de denaturatie en lagere
temperaturen, zodanig dat de primer kan hechten en de synthese kan gebeuren.
Vooraleer de bekomen fragmenten geanalyseerd kunnen
worden, dient er eerst nog opzuivering te gebeuren van deze
sequentiereactie,
dit
dmv
magnetische
beads.
Na
toevoeging van de magnetische beads aan het opgezuiverd
sequentiemengsel, zullen de DNA fragmenten binden aan
deze beads. Na binding kan opzuivering gebeuren door
toevoeging van 85% ethanol en het plaatsen van de
sequentieproducten op een magnetische plaat. Om de DNA
fragmenten van de beads te extraheren wordt water
toegevoegd, waarna de DNA fragmenten zullen loskomen
van de beads. Door dit opnieuw op een magnetische plaat te
plaatsen kunnen deze opgezuiverde DNA fragmenten
overgeplaatst worden in een ander epje of plaat. Een
schematisch overzicht van de verschillende opvolgende
Figuur 16: Schematische voorstelling
van
de
Sanger
methode.
http://www.genomebc.ca
stappen zijn neergeschreven in bijlage 4.
Voor de uitlezing van de sequenties zullen de sequentieproducten geladen worden op de
sequencer van Applied Biosystem (ABI 3730 of ABI 3130). De toegepaste techniek is
capillaire elektroforese, waarbij de DNA-fragmenten gescheiden worden op basis van hun
lengte door een spanning aan te leggen over de capillairen. Er bevindt zich een laserdetector
op het einde van de kolom die het fluorescente signaal. De resultaten worden geanalyseerd
met behulp van SeqScape (Applied Biosystems). De SeqScape 2.5 software (Applied
Biosystems) maakt het mogelijk om sequenties van controles of patiënten te vergelijken met
zelfgekozen referentiesequenties, deze kunnen zelf aangemaakt worden.
29
4. RESULTATEN
4.1. ENIGMA PROJECT
De ontwikkelde primers voor de BRCA1 c.671-2A>G variant zijn gelegen in exon 9 (forward)
en exon 13 (reverse). Deze primers zullen een full length product amplificeren van 3721 bp
(wild-type fragment). De sequentie van de gebruikte primers, alsook hun startplaats zijn
opgelijst in tabel 2.
Tabel 2: Overzicht van het primerpaar gebruikt bij de amplificatie van het fragment van exon 9 tot exon 13.
c.671-
Sequentie
exon
Forward
GAAGATACCGTTAATAAGGCAAC
exon 9
c.552
23
Reverse
GGGAGCCAGCCTTCTAACA
exon 13
c.4282
19
2A>G
Startplaats lengte primer (bp)
Bisearch
full lenght product : 3721 bp
Deze primers gaven een optimaal resultaat bij gebruik van de Takara Ex Taq polymerase bij
een aanhechtingstemperatuur van 64°C en bij het gebruik van PCR programma 2 (zie figuur
13). Bij de optimalisatie werd ook gebruik gemaakt van de Kapa2G Robust mix om te
vergelijken welke PCR condities het beste resultaat opleverden. De samenstelling van de
Takara ex Taq mastermix en Kapa2G Robust mix zijn weergegeven in tabel 3.
Tabel 3: Samenstelling mastermix Takara Ex Taq en Kapa2G Robust.
Na vergelijking van de resultaten bekomen met Takara Ex Taq en Kapa2G Robust kon men
vaststellen dat de PCR bij het gebruik van de Takara mix veel efficiënter doorging met
specifiekere transcripten als gevolg. Niet alleen ging de PCR minder efficiënt door bij gebruik
van de Kapa2G Robust mix, maar een aantal fragmenten, voornamelijk de grotere en de
alternatieve transcripten die bij de patiënt voorkwamen, werden niet geamplificeerd. Deze
verschillen zijn visueel voorgesteld in figuur 17.
30
Figuur 17: Vergelijking Kapa2G Robust en Takara Ex Taq, vergelijking geamplificeerde fragmenten van de
patiënt met variant BRCA1 c.671-2A>G en een controle.
Takara Ex Taq levert na het uitvoeren van een PCR veel duidelijkere banden dan bij het gebruik van de Kapa 2G
Robust mix, alsook mist men een aantal fragmenten (bij de patiënt en de grotere fragmenten). Opgepikte
fragmenten bij de patiënt (in bp): 246; 318; 447; 507; 689; 873; 3888; bij de controle (in bp): 448bp ; 495; 691;
888; 3894.
Na optimalisatie van de primers werd de PCR uitgevoerd op 3 controles en op de patiënt,
waaruit vastgesteld kon worden dat de patiënt 7 transcripten (grootte in bp: 246; 318; 447;
507; 689; 873; 3888) vertoonde en de controle 5 transcripten (grootte in bp: 448 ; 495; 691;
888; 3894). De groottes van de fragmenten werden bepaald aan de hand van de Labchip en
deze kunnen afwijken van de werkelijke waarde. Aangezien zowel bij de patiënt als bij de
controle meerdere alternatieve transcripten werden vastgesteld werd een klonering uitgevoerd,
zodanig dat er maar 1 transcript per kloon verkregen werd.
Klonering gebeurde op het PCR product afkomstig van de patiënt (RNA 1200398) en van één
controle (RNA 1200363). Dit leverde slechts 24 kolonies op van de patiënt en 27 kolonies van
de controle. Vervolgens werden deze kolonies overgeënt in de mastermix voor de PCR
reactie, en werd deze opnieuw uitgevoerd om het specifiek transcript van de kloon te
amplificeren. De visualisatie van de PCR reactie van de gekloneerde producten kan
teruggevonden worden in bijlage 5. Na visualisatie konden op basis van de grootte van de
fragmenten 5 verschillende transcripten (305, 422, 859, 975, 1276 (bp)) vastgesteld worden
bij de controle, hierbij kwam in het merendeel van de klonen het transcript met een grootte
van ongeveer 422 bp voor. 6 verschillende transcripten konden onderscheiden worden op de
labchip resultaten bij de patiënt (140, 228, 305, 422, 504, 520, 2379 in bp). Hierbij viel op dat
voornamelijk de kleinere fragmenten (228, 305 (bp)) geamplificeerd werden na PCR van de
gekloneerde producten.
31
Sequenering werd uitgevoerd op alle PCR producten verkregen uit de klonering, hiervoor
werd gebruik gemaakt van de forward en reverse primer van de PCR reactie. Na analyse van
de sequenties werden er twee verschillende transcripten geïdentificeerd bij de controle en vijf
transcripten bij de patiënt, deze zijn weergegeven in tabel 4.
Tabel 4: Overzicht van de opgepikte transcripten bij patiënt (BRCA1 c.671-2A>G) en controle.
5 verschillende transcripten werden opgepikt bij de patiënt: r.671-4096del; r.594-4096del; r.671-3897del; r.6713881del; r.787-4096del, terwijl er slechts twee verschillende transcripten werden geïdentificeerd bij de controle:
c.671-4096del; r.787-4096del.
controle
patiënt
RNA niveau
grootte fragment
deletie exon 11
1 kloon
4 klonen
r.671-4096del
295 bp
deletie exon 10 en 11
/
7 klonen
r.594-4096del
218 bp
deletie 5' deel exon 11
/
2 klonen
r.671-3897del
494 bp
deletie 5' deel exon 11
/
3 klonen
r.671-3881del
510 bp
deletie 3' deel exon 11
10 klonen
1 kloon
r.787-4096del
411 bp
Hierbij dient ook vermeld te worden dat de sequenering van sommige PCR producten geen
interpreteerbaar resultaat opleverde, waardoor er waarschijnlijk nog een aantal transcripten
ontbreken, waaronder het wild-type fragment (3721 bp).
In bijlage 6 wordt een overzicht van welke kloon welk transcript opleverde en van welke
klonen hun bekomen sequentie niet interpreteerbaar was.
4.2. VERGELIJKING C DNA SYNTHESE KITS
Er werd cDNA gesynthetiseerd met drie verschillende kits (M-MULV RNase
H-
; Superscript
II, GoScript) van drie verschillende controles en van de patiënt met BRCA1 c.671-2A>G
(RNA 1200398). Deze werden vervolgens gebruikt in eenzelfde PCR reactie met
bovenvermelde primers en condities.
Hierbij stelde men vast dat SuperScript II, in vergelijking met de andere kits, veel beter in
staat is om de grotere PCR producten te amplificeren. Deze werden bijna niet (GoScript) of
veel minder sterk (M-MULV) geamplificeerd, terwijl er sterke banden aanwezig waren bij
gebruik van SuperScript II. De kleinere fragmenten werden na PCR geamplificeerd bij
gebruik van de drie kits, maar ook hier met verschillende efficiëntie. De kleinere transcripten
gaven een specifieker resultaat bij gebruik van de SuperScript kit in vergelijking met de twee
andere kits, waarbij de gegenereerde banden moeilijker te onderscheiden waren van elkaar.
Het resultaat van deze vergelijking is samengevat in onderstaande figuur (figuur 18).
32
Figuur 18: Vergelijking drie cDNA kits.
4.3. CDNA ANALYSE VAN BRCA1, BRCA2, ATM, BRIP1, PALB2 EN CHEK2
4.3.1. PRIMERDESIGN EN OPTIMALISATIE
Primers voor BRCA1, BRCA2, en PALB2 waren reeds ontwikkeld voor de start van deze
masterproef, alsook de optimale PCR condities voor BRCA1 en BRCA2 waren reeds gekend.
Voor alle andere genen (BRIP1, CHEK2, ATM) werden de primers zelf ontwikkeld (zie
bijlage 7 voor een overzicht van de ontwikkelde primers).
Een aantal amplicons gaven na een eerste optimalisatie geen specifiek resultaat. Deze primers
werden getest bij een aanhechtingstemperatuur van 62°C en 65°C bij gebruik van PCR
programma 1 (zie figuur 13) waarna vervolgens een gradiënt (50°C-70°C) werd uitgevoerd bij
gebruik van hetzelfde PCR programma. In deze gradiënt PCR werd er gebruik gemaakt van
twee verschillende mastermixen: met 5% DMSO en zonder. De samenstelling van de
mastermix met en zonder DMSO kan nagelezen worden in bijlage 8. Aangezien deze gradiënt
nog steeds geen specifiek resultaat gaf werd een nieuwe primerset ontwikkeld, die na
verschillende optimalisatiestappen het gewenste resultaat gaf. De chronologische volgorde
van de optimalisatiestappen kan nagekeken worden in bijlage 8.
Voor CHEK2 was het onmogelijk om 2 nieuwe primersets te ontwikkelen die specifiek
genoeg waren, deze leverden namelijk bij de in silico predictie software veel alternatieve
fragmenten op, daarom werd overgegaan tot de ontwikkeling van 3 primersets voor het
CHEK2 gen. (Volledige optimalisatieprocedure van CHEK2 is ingesloten in bijlage 8.)
33
Alle andere primersets van de verschillende genen gaven na een aantal optimalisatiestappen
een specifiek resultaat. Een overzicht van de PCR condities voor de verschillende
ontwikkelde primerset zijn weergegeven in de onderstaande tabel.
Tabel 5: Overzicht PCR condities per ontwikkeld primerpaar.
Gen
BRCA1
BRCA2
ATM
PALB2
BRIP1
CHEK2
PCR fragment
PCR1
PCR2
PCR3
PCR1
PCR2
PCR3
PCR1
PCR2
PCR3
PCR4
PCR5
PCR6
PCR1
PCR2
PCR1
PCR2
PCR3
PCR1
PCR2
PCR3
DMSO
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
/
5%
/
PCR programma
1
1
1
1
1
1
2
2
2
2
2
2
2
2
2
2
2
2
2
2
Aanhechtingstemperatuur
58 °C
58 °C
58 °C
58 °C
58 °C
58 °C
64°C
58 °C
58 °C
58 °C
58 °C
58 °C
60 °C
64 °C
60 °C
60°C
64 °C
68 °C
65 °C
65°C
In een volgende stap werden de sequeneringprimers ontwikkeld voor de verschillende genen
en geoptimaliseerd. Na optimalisatie bleek dat bepaalde primers (PALB2 PCR1F; BRIP1
PCR1F; ATM 3FB; ATM 5FC) aanleiding gaven tot sequenties die niet interpreteerbaar waren
of waarbij de sequeneringsreactie faalde. Hiervoor werden nieuwe ontwikkeld. Een overzicht
van de ontwikkelde sequeneringsprimers kunnnen teruggevonden worden in bijlage 7.
4.3.2. RNA 1300042
Na het uitvoeren van de PCR reactie op cDNA van een patiënt (RNA 1300042) zag men dat
deze alternatieve fragmenten opleverde bij verschillende genen, zoals kon vastgesteld worden
in figuur 19.
34
Figuur 19: Alternatieve fragmenten patiënt RNA 1300042.
De eerste figuur is het resultaat van PCR met primerset ATM PCR1, de tweede met PALB2 PCR1 en de derde
met BRIP1 PCR2. Op alle drie de figuren vertoont 1300042 (2de resultaat op elke figuur) andere fragmenten dan
de andere stalen die geamplificeerd werden.
Deze PCRs werden gesequeneerd. Na analyse van de sequentie van een aantal fragmenten
(BRCA1 fragment 1 en ATM fragment 2-5) van de patiënt met RNA 1300042 werd een groot
aantal homozygote veranderingen geïdentificeerd. Een voorbeeld van deze homozygote
varianten in BRCA1 en ATM zijn weergegeven in figuur 20. Hierdoor werd er beslist om exon
7 (grootst aantal homozygote veranderingen) van BRCA1 en exon 14 van ATM op gDNA
niveau, geëxtraheerd uit bloed, te sequeneren en na te gaan als deze veranderingen ook hierin
aanwezig waren.
Figuur 20: Homozygote varianten in patiënt (RNA 1300042).
De bovenste sequenties zijn afkomstig van een gezonde controle, de onderste van de patiënt met RNA 1300042.
De linkse sequenties zijn afkomstig van het BRCA1 gen, de rechtse van het ATM gen. Als men de controle
vergelijkt met de patiënt dan ziet men 3 homozygote varianten in de sequentie afkomstig van het BRCA,1 5 bij
de sequentie afkomstig van het ATM.
Op gDNA niveau vertoonde deze patiënt geen alternatieve fragmenten na de PCR reactie en
werden geen homozygote veranderingen vastgesteld na analyse van de sequenties. De
vergelijking tussen de sequentie bekomen voor varianten in ATM op cDNA en gDNA niveau
is weergegeven in figuur 21.
35
Figuur 21: Vergelijking sequenties afkomstig van
cDNA en gDNA (ATM).
Beide sequenties zijn afkomstig van RNA 1300042
patiënt. De bovenste sequentie is afkomstig van
gDNA, de onderste van cDNA. De homozygote
varianten die geïdentificeerd werden in het cDNA,
zijn niet aanwezig in het gDNA.
4.3.3. PCR + SEQUENERING OVERIGE PATIËNTEN
De PCRs voor de verschillende fragmenten werden voor alle patiënten uitgevoerd, behalve
voor CHEK2 en BRIP1 PCR3. Deze primersets konden niet tijdig geoptimaliseerd worden. Er
werd slechts één afwijkend resultaat (exclusief patiënt met RNA 1300042) geïdentificeerd, dit
voor het tweede PCR fragment van PALB2 (PALB2 PCR2). Deze werd vervolgens nog een
tweede maal uitgevoerd met vers bereid cDNA, waarbij het verwachtte resultaat werd
vastgesteld. Het resultaat van de eerst uitgevoerde PCR en de tweede PCR voor patiënt met
RNA 1300042 zijn weergegeven in figuur 22.
Figuur 22: Resultaat PALB2 PCR2 patiënt RNA 1300044.
Het rode kader duidt de patiënt met RNA 1300044 aan. Op de linkse figuur ziet men een donkere band die
duidelijk lager gelegen is in vergelijking met de andere patiënten. De rechtse figuur is het resultaat bij een
tweede uitvoering van de PALB2 PCR2 amplificatie. Bij vergelijking van de twee bekomen resultaten kan men
vaststellen dat op de linkse figuur veel meer alternatieve fragmenten (lichter) aanwezig zijn dan bij de tweede
uitgevoerde PCR.
Na sequenering van de bekomen fragmenten van de verschillende patiënten konden 10
varianten in 3 verschillende genen (BRCA1, BRCA2, ATM) geïdentificeerd worden. Overzicht
van de geïdentificeerde varianten per patiënt in onderstaande tabel.
36
Tabel 6: Overzicht geïdentificeerde varianten per patiënt.
Patiënt
gen
variant
1200304
1200308
1200347
1200349
1200353
1300040
ATM
ATM
BRCA2
BRCA2
BRCA1
BRCA2
ATM
ATM
BRCA2
BRCA2
BRCA2
ATM
ATM
BRCA2
BRCA2
BRCA1
BRCA2
BRCA2
BRCA2
ATM
c.5557G>A
c.2572T>C
c.7242A>G
c.7242A>G
c.4837A>G
c.7242A>G
c.4578C>T
c.5557G>A
c.1365A>G
c.1818G>A
c.7242A>G
c.2805G>C
c.5557G>A
c.7242A>G
c.7242A>G
c.4837A>G
c.1114A>C
c.865A>C
c.1365A>G
c.5557G>A
1300044
1300050
1200047
1200049
1200068
1200071
eiwitniveau
p.Asp1853Asn
p.Phe858Leu
p. Ser2414Ser
p. Ser2414Ser
p.Ser1613Gly
p. Ser2414Ser
p.Pro1526Pro
p.Asp1853Asn
p.Ser455Ser
p.Pro606Pro
p. Ser2414Ser
p.Thr935Thr
p.Asp1853Asn
p. Ser2414Ser
p. Ser2414Ser
p.Ser1613Gly
p.Asn372His
p.Asn289His
p.Ser455Ser
p.Asp1853Asn
status
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
heterozygoot
homozygoot
heterozygoot
heterozygoot
homozygoot
homozygoot
homozygoot
homozygoot
heterozygoot
RS
RS 1801516
RS 1800056
RS 1799955
RS 1799955
RS 1799966
RS 1799955
RS 18000889
RS 1801516
RS 1801439
RS 76844014
RS 1799955
RS 55934812
RS 1801516
RS 1799955
RS 1799955
RS 1799966
RS 144848
RS 766173
RS 1801439
RS 1801516
Soort variant
missense variant
missense variant
synonieme variant
synonieme variant
missense variant
missense variant
synonieme variant
missense variant
synonieme variant
synonieme variant
synonieme variant
synonieme variant
missense variant
synonieme variant
synonieme variant
missense variant
missense variant
missense variant
synonieme variant
missense variant
4.4. ANALYSE VAN 4 VARIANTEN OP C DNA NIVEAU
Bij de analyse van de BRCA1/2 genen in patiënten met een vermoedelijke erfelijke
predispositie voor borstkanker worden in 5% van de patiënten varianten aangetroffen waarvan
de klinische betekenis niet duidelijk is. Een aantal hiervan liggen in regio’s die belangrijk zijn
voor correcte splicing. Door middel van cDNA onderzoek kan nagegaan worden wat het
effect van deze varianten is op cDNA niveau om op die manier adequate counseling te kunnen
doen van de families waarin deze varianten worden aangetroffen.
Vooraleer over te gaan tot PCR gevolgd door sequenering werd gebruik gemaakt van in silico
predictie softwares, via Alamut. Dit kan kunnen een eerste aanwijzing van het mogelijk effect
op splicing veroorzaakt door een bepaalde variant.
Voor BRCA2 c.517G>C en de BRCA2 c.7618-6G>T werd slechts een heel klein verschil
voorspeld in de sterkte van de acceptor splice site. De predictie voor de BRCA2 c.3326C>T
variant is de aanwezigheid van mogelijks een nieuwe donor splice site op de positie van de
variant. Alamut voorspelde voor de BRCA2 c.681+111C>G een nieuwe (SpliceSiteFinder-like
37
en NNSPLICE) of de versterking van een reeds bestaande (MaxEntScan en Human Splicing
Finder) acceptor splice site, de eerst volgende mogelijke donor splice site is gelegen op
positie c.681+390. Resultaten van de predicties door Alamut kunnen nagekeken worden in
bijlage 9.
Voor de varianten BRCA2 c.517G>C en BRCA2 c.681+111C>G werd een PCR uitgevoerd
met de primers van BRCA2 PCR1. Deze primerset overspant het gebied waarin de varianten
gelegen zijn. Voor de variant BRCA2 c.7618-6G>T werd gebruik gemaakt van de primerset
BRCA2 PCR2. Een primerset voor de vierde variant, BRCA2 c.3326C>T, werd niet
ontwikkeld in deze masterproef, aangezien exon 11 (het grootste exon) in deze masterproef
niet overspannen werd. Om deze variant te analyseren werd gebruik gemaakt van primers die
exon 10-13 zullen overspannen en reeds aanwezig waren in het labo.
Na analyse van de gegenereerde fragmenten bleek dat de BRCA2 c.517G>C variant een
transcript amplificeerde dat ongeveer 90 bp korter was dan deze van de gezonde controles. Bij
de drie andere varianten werden fragmenten geamplificeerd die dezelfde grootte hadden als de
gegenereerde controle fragmenten. Resultaten van deze PCR zijn weergegeven in figuur 23 en
24.
Figuur 23: PCR resultaat c.517G>C,
c.681+111C>G en c.7618-6G>T.
Resultaat labchip van de PCR
uitgevoerd
op
cDNA;
het
geamplificeerde fragment van de
variant c.517G>C is ongeveer 90 bp
korter dan de controles. De andere
varianten c.681+111C>G en c.76186G>T geven na PCR fragmenten met
dezelfde grootte als de controles.
Figuur 24: Resultaat labchip PCR van
patiënt met c.3326 C>T.
De
variant
leverde
na
PCR
amplificatie
dezelfde
grootte
fragmenten als deze van de controles.
38
Vervolgens werden deze PCR producten gesequeneerd met dezelfde sequeneringsprimers als
gebruikt bij de cDNA analyse. Voor de c.3326 C>T variant werden primers gebruikt die de
exon-exon overgangen zullen overspannen en twee primers die gebruikt zullen worden om de
sequenties gelegen in de nabijheid van de variant te analyseren. Een schematische
voorstelling wordt gegeven in figuur 25.
Figuur 25: Overzicht ligging sequeneringsprimers c.3326 C>T.
De figuur stelt de exonen 10-13 voor, waarbij de pijlen de positie en richting van de sequeneringsprimers
weergeven en het rode kruis de variant. De bovenste pijlen duiden de primers aan die gebruikt zullen worden bij
de analyse van de exon-exon boundaries/overgangen, de onderste pijltjes stellen de primers voor die gebruikt
zullen worden bij de analyse van de variant.
Er werd geen aberrante splicing geïnduceerd tengevolge van de aanwezigheid van de BRCA2
c.3326C>T, c.681+111C>G en c.7618-6G>T varianten. Bij de c.517G>C variant kon men na
sequenering een frameshift vaststellen, beginnende na de laatste base van exon 6. Deze
frameshift werd veroorzaakt door deletie van het volledige zevende exon en wordt aangeduid
in figuur 26.
Figuur 26: Sequentie variant c.517G>C.
Net na de laatste base van exon 6 (c.516) ziet men een frameshift (overlappende sequenties), deze werd
veroorzaakt door de volledige deletie van exon 7, terwijl ook het wild-type fragment werd aangetroffen.
Vervolgens werd nog een tweede PCR uitgevoerd om na te gaan of het full length fragment
ook de variant C bevatte op positie 517 of enkel de G base. De PCR werd uitgevoerd met
dezelfde forward primer en een reverse primer gelegen in exon 7 (deze was reeds aanwezig in
het labo). Bij de sequenering werd gebruik van dezelfde primers als bij de eerste uitgevoerde
PCR.
39
Men stelde vast dat enkel het wild-type nucleotide, G, aanwezig was in het full length
fragment op positie c.517, zoals weergeven in figuur 27.
Figuur 27: Resultaat sequenering full length product.
Het oranje kader geeft de positie BRCA2 c.517 aan, hierbij stelt men
vast dat enkel de G base aanwezig is, de base C van de variant komt
in het full length fragment niet voor.
5. DISCUSSIE
5.1. ENIGMA
5.1.1. BRCA1 C.671-2A>G
De BRCA1 c.671-2A>G variant is gelegen in intron 10, twee basenparen voor de start van
exon 11. Deze basen spelen een belangrijke rol bij het splicing mechanisme, waarbij de
intronen (niet-coderende delen) weggeknipt worden, zodanig dat enkel het coderende deel
(exonen) van het DNA overblijft. Veranderingen in de basen, die instaan voor de herkenning
door het spliceosoom, kunnen leiden tot alternatieve splicing met intron retentie ((deel van
het) intron wordt weerhouden) of exon skipping ((deel van het) exon wordt verwijderd) als
gevolg. De BRCA1 c.671-2A>G variant was reeds gekend verantwoordelijk te zijn voor
alternatieve splicing en zowel de procedure als de opgepikte transcripten zullen vergeleken
worden tussen de deelnemende labo’s.
Bij vergelijking van het Labchip resultaat verkregen na PCR met dit van de gekloneerde
producten kon men vaststellen dat een aantal fragmenten, waaronder het wild-type fragment,
niet geïdentificeerd werd bij de gekloneerde producten. Het wild-type fragment werd
waarschijnlijk door de lage prevalentie niet ingebouwd in de vectoren gebruikt bij klonering
of door preferentiële inbouw van kleinere fragmenten. Na sequenering van de gekloonde PCR
producten werden 5 verschillende transcripten geïdentificeerd bij de patiënt met de variant en
slechts 2 verschillende transcripten bij de gezonde controle. De 2 alternatieve fragmenten:
r.671_4096del (deletie exon 11) en r.591_4096del (deletie exon 10 en 11) kunnen beiden
verklaard worden door het wegvallen van de acceptor splice site (zie bijlage 10 voorspelling
Alamut). Bij zowel het r.671_3897del en r.671_3881del transcript verwacht men de
aanwezigheid van een acceptor splice site, terwijl bij het r.787_4096del fragment de
aanwezigheid van een donor splice site verwacht wordt. (Deze werden voorspeld op deze
40
posities door Alamut en deze resultaten zijn samengevat in bijlage 10.) Alhoewel deze
acceptor splice sites zwakkere scores hebben (vnl op positie 3881) kunnen deze toch
geactiveerd worden bij het wegvallen van de sterkere acceptor splice site op positie 671.
In BRCA1 werden reeds verscheidene alternatieve splicing transcripten vastgesteld,
waaronder bepaalde natuurlijk voorkomende isovormen (Δ9/10, Δ9/10/11 en Δ9/10/11q) (57).
Aangezien de ontwikkelde forward primer gelegen was in exon 9, was het onmogelijk om
deze te amplificeren en te identificeren. Dit geldt eveneens voor mogelijke onbekende variantgeassocieerde alternatieve transcripten, waarin (een deel van) exon 9 eruit geknipt zou zijn.
In een volgende stap zou via Western blots kunnen nagegaan worden of deze geïdentificeerde
transcripten kunnen vertaald worden naar eiwitten. Een eerste indicatie kan verkregen worden
door na te gaan of deze transcripten afgebroken worden via nonsense mediated decay (NMD).
Out of frame transcripten leiden tot een vervroegd stopcodon en hebben meer kans om NMD
te ondergaan. De transcripten r.671_4096del en r.591_4096del zijn in frame deleties en
kunnen mogelijks leiden tot translatie naar aberrante proteïnen. Er werd reeds gesuggereerd in
muizen dat getrunceerde eiwitten via een dominant negatief effect kunnen leiden tot verlies
van de tumorsupressor functie van BRCA1 (58).
We slaagden er niet in om voor alle gekloneerde producten een interpreteerbaar resultaat te
bekomen; voornamelijk bij de grotere fragmenten. Hiervoor werd tot nu toe nog geen
verklaring gevonden, sequenering met de aangrenzende sequenties van de gebruikte vector
zou hiervoor een oplossing kunnen bieden.
5.1.2. VERGELIJKING T AKARA E X TAQ EN KAPA2G ROBUST
Verschillende Taq polymerases worden aangeboden door verscheidene bedrijven. De keuze
van deze twee DNA polymerases berustte op het feit dat deze veelvuldig gebruikt worden in
het labo en hier reeds goede resultaten mee bekomen waren. Takara Ex Taq wordt gebruikt
voor de amplificatie van longe-range PCR producten, terwijl de Kapa2G robust een brede
waaier aankan van verschillende soorten (GC- en AT-rijke) templates met het gebruik van
eenzelfde kort PCR programma. Hoewel de Kapa2G Robust kit niet specifiek bedoeld is voor
longe-range PCR producten werden reeds bevredigende resultaten bekomen bij de
amplificatie van 6kb fragmenten.
Bij de vergelijking van deze twee types Taq polymerases ging men na of het volledig
spectrum van fragmenten geamplificeerd werd en de sterkte. Men zag hierbij dat de
41
amplificatie bij het gebruik van Takara Ex Taq veel efficiënter doorging en dat alle mogelijke
fragmenten aanwezig waren, iets wat men niet vaststelde bij het gebruik van Kapa2G Robust,
deze kon maar een beperkt aantal fragmenten (licht) amplificeren. Hierbij werd een duidelijk
verschil vastgesteld bij het wild-type fragment (3721 bp). Deze was sterk geamplificeerd
(duidelijke banden) bij het gebruik van Takara Ex Taq terwijl deze afwezig was bij Kapa2G
Robust. Voor dit experiment bleek het gebruik van Takara Ex Taq de beste optie.
Studies waarin verschillende DNA polymerases vergeleken werden zijn eerder zeldzaam, dit
komt waarschijnlijk doordat ieder labo werkt met een bepaalde DNA polymerases waarmee
goede resultaten bekomen werden en waarmee ze vertrouwd geraakt zijn. In het artikel van
Purzycka et al (59) werden 7 verschillende DNA polymerases vergeleken. Ook hier kon een
verschil in efficiëntie vastgesteld worden bij vergelijking van de amplificatie tussen de
verschillende DNA polymerases. De vaststelling die uit deze resultaten voortvloeit is dat de
keuze van DNA polymerase een effect kan hebben op de efficiëntie waarmee amplificatie van
bepaalde templates doorgaat. Bijkomend onderzoek dringt zich op, zodanig dat de gebruikers
van deze DNA polymerases een weloverwogen keuze kunnen maken die het best aanleunt bij
hun onderzoek.
5.1.3. VERGELIJKING 3 CDNA SYNTHESE KITS
Aangezien de PCR een spectrum van verschillende transcripten opleverde werd een
vergelijkende studie opgezet van verschillende cDNA synthese kits. In het CMGG wordt
gebruik gemaakt van SuperScript II voor longe-range RT-PCR en hiervoor dus grotere cDNA
fragmenten nodig zijn als template. De twee andere kits werden ons aangeboden door de
desbetreffende bedrijven. Bij de drie kits wordt als reverse transcriptase enzym gebruik
gemaakt van een genetisch gemodificeerd Moloney Murine Leukemia Virus (M-MULV) en
alle drie de kits zijn zodanig ontwikkeld om grote fragmenten cDNA te synthetiseren, die
nadien gebruikt kunnen worden in een longe-range PCR. cDNA synthese gebeurde conform
de protocols die bij de kits geleverd werden en om de variabelen zo constant mogelijk te
houden werd gebruik gemaakt van dezelfde drie controles en hetzelfde patiënt RNA.
Na PCR amplificatie werd een duidelijk verschil in efficiëntie waargenomen tussen de drie
gebruikte cDNA synthese kits, waarbij vastgesteld werd dat SuperScript II de meest
aangewezen kit is voor het uitvoeren van (deze) longe-range RT-PCR. Dit resultaat komt
overeen met twee andere studies waarin verschillende RT kits uitgetest en vergeleken werden
(60, 61). Hoewel deze studies gebruik maakten van andere cDNA synthese kit ondersteunen
42
deze beide studies de hypothese dat er een verschil in efficiënt van de RT reactie bestaat
tussen de verschillende cDNA synthese kits. In de studie van Stahlberg et al (60) werd
waargenomen dat deze variatie genafhankelijk is, maar bijkomend onderzoek is nodig om
deze hypothese te bevestigen.
Hierbij dient vermeld te worden dat het gebruikte template RNA bij SuperScript II 2 µg en bij
de M-MULV en GoScript 1 µg bedroeg. Maar dit biedt geen verklaring waarom de grotere
fragmenten (GoScript) of bepaalde alternatieve fragmenten (M-MULV) niet geamplificeerd
werden.
Samenvattend kan men dus stellen dat zowel de positie van de primers, het gebruikte DNA
polymerase en cDNA synthesis kit, alsook klonering een invloed kunnen hebben in welke
fragmenten preferentieel geamplificeerd en vervolgens geïdentificeerd zullen worden. Het is
belangrijk om met deze factoren rekening te houden vooraleer men start met het onderzoek,
zodanig dat men er zich van bewust is dat de mogelijkheid bestaat dat sommige transcripten
niet zullen worden opgepikt.
In deze kwaliteitscontrole georganiseerd door ENIGMA waaraan meer dan 20 labs
deelnamen, werd heel wat variatie vastgesteld bij de analyse op cDNA niveau van een BRCA1
variant in de acceptor site van exon 11. Het doel van deze internationale studie was om
richtlijnen te formuleren voor cDNA analyse wat zou kunnen bijdragen tot uniformiteit van
de resultaten in verschillende labs. Dit is belangrijk om clinici en patiënten correcte
informatie te bezorgen omtrent het risico op borstkanker in de familie.
5.2. CDNA ANALYSE VAN BRCA1, BRCA2, ATM, BRIP1, PALB2 EN CHEK2
5.2.1. PRIMEROPTIMALISATIE
Alle ontworpen primers voor de verschillende genen werden geoptimaliseerd. Het merendeel
van de primers (behalve de primers van BRCA1 en BRCA2) gaven een specifiekere
amplificatie bij gebruik van programma 2, waarbij een extensietijd gebruikt werd van 3
minuten ten opzichte van 1 minuut die gebruikt werd bij programma 1. Hiervoor is een
logische verklaring: aangezien de primers als doel hebben om een groot (>1000 bp) amplicon
te amplificeren zal deze meer tijd nodig hebben om gesynthetiseerd te worden (extensie).
43
5.2.2. RNA 1300042
De patiënt met RNA 1300042 viel op door de vele alternatieve fragmenten die geamplificeerd
werden bij de PCR van verschillende genen. Normaal verwacht men dat er alternatieve
fragmenten vastgesteld worden bij een bepaalde PCR van een bepaald gen. Deze vaststelling
leidde tot het vermoeden dat er waarschijnlijk degradatie van de EBV cellijn had
plaatsgevonden, waarschijnlijk door de ouderdom (15 jaar) van deze. Na sequenering van
deze fragmenten werd dit vermoeden versterkt door de vele homozygote veranderingen die
eveneens in verschillende genen werden geïdentificeerd.
Om deze veronderstelling te ondersteunen werd overgegaan tot sequenering van exon 14 van
het ATM gen en exon 7 van het BRCA1 gen op gDNA niveau. Deze fragmenten bevatten een
groot aantal homozygote veranderingen op cDNA niveau. De gegenereerde fragmenten na
PCR vertoonden geen afwijkend beeld in vergelijking met controles, alsook in de sequenties
bekomen na sequenering van het gDNA, konden geen homozygote veranderingen
geïdentificeerd worden. Deze resultaten bevestigden het vermoeden dat de EBV cellijn van
patiënt met RNA 1300042 gedegradeerd is, en deze zal niet meer opgenomen worden in
toekomstige analyses.
5.2.3. OVERIGE PATIËNTEN
Het hoofddoel van deze studie was om in 25 geselecteerde patiënten mogelijke (diepintronische) varianten te identificeren die aanleiding gaven tot alternatieve splicing.
Tezelfdertijd konden de coderende regio’s van PALB2, CHEK2, ATM en BRIP1 op
puntmutaties gescreend worden (BRCA1/2 werden hierop reeds gescreend). Meerdere
pathogene mutaties die aanleiding gaven tot alternatieve splicing (intronisch en exonisch)
werden reeds geïdentificeerd in deze genen (62-65). Deze mutaties komen veel zeldzamer
voor dan exonische puntmutaties, maar dit zou voor een deel verklaard kunnen worden
doordat deze in de meeste studies over het hoofd worden gezien.
Enkel bij de patiënt met RNA 1300044 werd een alternatief bandje op de Labchip vastgesteld
na amplificatie met de PALB2 PCR2 primers. Deze PCR werd een tweede maal uitgevoerd
voor deze patiënt en hierbij was dit alternatief fragment niet aanwezig. Na sequenering van
deze fragmenten kon men geen veranderingen identificeren. Een mogelijke verklaring
hiervoor zou kunnen zijn dat het cDNA gebruikt in de eerste PCR reactie mogelijks
gedegradeerd zou zijn, anderzijds zou het een artefact kunnen zijn van de Labchip. Wetende
dat bij de tweede uitgevoerde PCR reactie gebruik werd gemaakt van vers bereid cDNA en bij
44
vergelijking van de bekomen alternatieve fragmenten bij alle patiënten meegenomen in deze
PCR reactie (lichtere banden op de Labchip) werd het vermoeden versterkt dat dit sterk
geamplificeerd kleiner fragment mogelijks het gevolg was van degradatie van het gebruikte
cDNA.
In de geanalyseerde sequenties werden 10 varianten geïdentificeerd, hiervan zijn er 5
synonieme en 5 missense varianten. Deze varianten werden reeds frequent geïdentificeerd
waardoor dat deze varianten waarschijnlijk sequentie varianten zijn zonder (gekend)
pathogeen effect.
De mogelijkheid bestaat dat mogelijke varianten aanwezig in deze patiënten niet werden
gedetecteerd. Enerzijds zou dit kunnen komen doordat deze over het hoofd werden gezien
door de aanwezige achtergrond, anderzijds zouden deze kunnen liggen in de fragmenten die
nog dienen gesequeneerd te worden of in deze die faalden tijdens de sequeneringsreactie. Het
falen van deze sequeneringsreacties zou mogelijks kunnen komen doordat de interne primers,
gebruikt bij de sequenering, gelegen zouden zijn in een gedeleteerde deel, maar deze kans is
relatief klein aangezien men in eerste instantie een verschil zou merken bij de lengte van het
geamplificeerde fragment en vanwege het gebruik van overlappende sequeneringsprimers.
Aangezien de zeldzaamheid van mutaties die aanleiding geven tot alternatieve splicing en de
frequentie waarmee mutaties in de coderende regio voorkomen in families met clustering van
borstkanker, ATM, BRIP1, PALB2, CHEK2 mutaties verklaren ongeveer 4-8% van de
familiale clustering van borstkanker, is het niet verwonderlijk dat er geen mutaties
geïdentificeerd werden in dit onderzoek gezien de grootte van de gescreende
patiëntenpopulatie (25 patiënten).
5.3. ANALYSE VAN 4 VARIANTEN OP C DNA NIVEAU
In een ander deel van deze masterproef werd het mogelijke effect op splicing van vier
varianten nagegaan op cDNA niveau. Deze varianten werden geïdentificeerd op gDNA
niveau en zijn gelegen in exon-intron grenzen (c.517G>C), intronisch (c.681+111C>G;
c.7618-6G>T) of in het exon (c.3326C>T) van het BRCA2 gen.
Na analyse van de Labchip resultaten van de PCR reactie kon men bij de patiënt die drager
was van de c.517G>C variant een aberrant fragment vaststellen dat kleiner was dan het full
length transcript. Dit kortere fragment was afwezig bij controles. Dit wees op de mogelijkheid
dat er aberrante splicing had plaatsgevonden tengevolge van deze variant. Na sequenering van
45
het geamplificeerde fragment van de patiënt met c.517G>C kon men een transcript, naast het
wild-type fragment, identificeren met een deletie van exon 7. Deze deletie betrof een in-frame
deletie, waarbij het oorspronkelijk open leesraam nog steeds aanwezig was, van positie r.517r.631. Via een tweede PCR, waarbij enkel het wild-type fragment werd geamplificeerd, stelde
men vast dat enkel de G base aanwezig was op positie c.517. Dit leidde tot de
veronderstelling dat deze variant niet meer in staat is om het full length transcript te
produceren en dus zeer waarschijnlijk een pathogeen effect heeft door verlies van het wildtype product.
De bekomen resultaten van de c.517G>C variant zijn vergelijkbaar als deze beschreven in
Gaildrat et al (66), hoewel hier onderzoek verricht werd naar de c.517G>T variant. Deze
varianten zijn gelegen op dezelfde positie in het BRCA2 gen, enkel het mutant allel is
verschillend. Bij deze mutant werd eveneens skipping van exon 7 gedetecteerd, terwijl in
controles dit alternatief fragment niet geïdentificeerd kon worden. Dit leidde tot een
gelijkaardige hypothese: daling van het functioneel BRCA2 eiwit kan een pathogeen effect tot
gevolg hebben, doordat het zijn functie in DSB herstel en de bescherming die dit eiwit biedt
tegen tumorontwikkeling niet meer zou kunnen vervullen. Om deze hypothese te bevestigen
dient men te onderzoeken welke hoeveelheid BRCA2 eiwit nodig is voor het correct
uitvoeren van zijn functie en indien, alhoewel er skipping van exon 7 is plaatsgevonden, dit
alternatief fragment toch geen functioneel eiwit kan produceren.
Een mogelijke volgende stap is om een allelic imbalance studie uit te voeren voor de
c.517G>C variant. Indien het fragment, die de exon 7 deletie draagt, niet geamplificeerd
wordt bij gebruik van RNA, geëxtraheerd uit cellen die niet behandeld werden met
puromycine, zal het aberrante fragment waarschijnlijk afgebroken geweest zijn door het NMD
mechanisme en niet functioneel geweest zijn.
De sequenering van de andere varianten vertoonden allen een normaal beeld, en hierin kon
geen aberrante splicing vastgesteld worden. De hypothese die uit deze resultaten volgde was
dat de varianten c.681+111C>G, c.7618-6G>T en c.3326C>T waarschijnlijk neutrale
sequentie veranderingen zijn zonder pathogeen effect. Deze resultaten konden niet vergeleken
worden met andere studies, aangezien het effect van deze varianten op splicing nog niet werd
beschreven. Dit zou mogelijks het bekomen resultaat kunnen ondersteunen, aangezien er bij
deze varianten geen effect op splicing kon worden vastgesteld en negatieve resultaten minder
frequent beschreven worden in de literatuur.
46
In silico predictie softwares kunnen een nuttige tool zijn om het mogelijke effect van
varianten op splicing te voorspellen, maar zoals uit de resultaten van dit onderzoek blijkt,
dient men hier héél voorzichtig mee om te gaan. Daar waar de in silico predictie softwares een
effect voorspelden, kon na sequenering geen alternatieve splicing gedetecteerd worden. Bij de
c.517 G>C variant werd slecht een heel kleine verzwakking voorspeld van de acceptor splice
site, maar men stelde vast dat deze variant toch leidde tot aberrante splicing. Een correcte
voorspelling van mogelijke splicing werd gesteld voor de c.7618-6G>T variant. Om met
zekerheid het effect van varianten op splicing te bepalen een cDNA analyse nodig.
5.4. Toekomstperspectieven
Deze cDNA analyse zou nog voortgezet kunnen worden naar CHEK2 en BRIP1 PCR3. Deze
sequeneringreacties dienen nog uitgevoerd te worden, alsook ontbreken er nog bepaalde
sequenties van andere genen voor alle patiënten. Zodanig dat mutaties in deze genen kunnen
worden uitgesloten voor deze patiënten.
In een volgende stap kunnen de geïdentificeerde heterozygote SNP’s als basis dienen voor
allelic imbalance studies. In deze studie zal er onderzocht worden of er een verschil in
expressie van een gen heeft plaatsgevonden. Deze studie zal uitgevoerd worden op cDNA
afkomstig uit cellen die niet werden behandeld met puromycine. De expressie van een
bepaald allel kan opgereguleerd of neergereguleerd worden via verschillend mechanismes.
Indien er allelic imbalance gedetecteerd wordt dient er verder onderzoek te gebeuren van de
niet-coderende regio’s (UTR, promotor/enhancer regio).
De expressie van bepaalde genen kan gewijzigd worden onder invloed van bijvoorbeeld noncoding RNAs (ncRNAs). NcRNAs kunnen ingedeeld worden in twee groepen op basis van
hun grootte in long-non-coding RNA (lncRNA) (>200 nucleotiden) en small non-coding
RNA (sncRNA) waartoe de microRNAs (miRNA) behoren. Deze laatste kunnen binden op de
3’untranslated region (UTR) van het mRNA van een gen met wijzigingen (meestal daling)
van het mRNA niveau tot gevolg (via blokkering van translatie, degradatie of gene silencing)
(67). Zo kunnen mutaties in de 3’UTR van een gen leiden tot het ontstaan van een nieuwe
bindingsplaats voor een bepaald miRNA, waarna zijn expressie onderdruk zal worden, of een
bestaande bindingsplaats verstoren met upregulatie van de expressie. In tegenstelling tot de
reeds veelvuldig bestudeerde miRNAs, zijn de lncRNA een recent opkomend onderzoek.
lncRNA kunnen genexpressie wijzigen via regulatie van de transcriptie in cis (bv binding op
een transcriptiefactor (TF) bindingsplaats) of in trans (bv betrokkenheid bij transport van TF),
47
via epigenetische veranderingen of via post-transcriptionele en post-translationele regulatie
(67, 68). Zo kunnen mutaties in de promotorregio of UTR bindingsplaatsen van een TF
verstoren met als gevolg een wijziging in de genexpressie.
In dit onderzoek werd slechts gebruik gemaakt van een patiëntenpopulatie bestaande uit 25
personen. In een volgende fase zou dit onderzoek kunnen worden toegepast op een grotere
patiëntenpopulatie om zo de kans te vergroten om alternatieve splicing of mutaties in deze
genen te identificeren. Er bestaat natuurlijk ook de kans dat de clustering van borstkanker in
deze families mogelijks te wijten zou kunnen zijn aan mutaties in andere (nog te identificeren)
hoog penetrante borstkankerpredispositiegenen of via het cumulatief effect van frequent
voorkomende laag penetrante allelen. In dit geval is exoom sequenering een mogelijke optie.
6. REFERENTIES
1.
Hanahan D, Weinberg RA. The hallmarks of cancer. Cell. 2000;100(1):57-70. Epub 2000/01/27.
2.
Levitt NC, Hickson ID. Caretaker tumour suppressor genes that defend genome integrity. Trends in
molecular medicine. 2002;8(4):179-86. Epub 2002/04/03.
3.
Hinds PW, Weinberg RA. Tumor suppressor genes. Current opinion in genetics & development.
1994;4(1):135-41. Epub 1994/02/01.
4.
Kinzler KW, Vogelstein B. Cancer-susceptibility genes. Gatekeepers and caretakers. Nature.
1997;386(6627):761, 3. Epub 1997/04/24.
5.
Oliveira AM, Ross JS, Fletcher JA. Tumor suppressor genes in breast cancer: the gatekeepers and the
caretakers. American journal of clinical pathology. 2005;124 Suppl:S16-28. Epub 2006/02/14.
6.
Macleod K. Tumor suppressor genes. Current opinion in genetics & development. 2000;10(1):81-93.
7.
Lalloo F, Evans DG. Familial Breast Cancer. Clin Genet. 2012;82(2):105-14.
8.
Hollestelle A, Wasielewski M, Martens JW, Schutte M. Discovering moderate-risk breast cancer
susceptibility genes. Current opinion in genetics & development. 2010;20(3):268-76. Epub 2010/03/30.
9.
Foulkes WD. Inherited susceptibility to common cancers. The New England journal of medicine.
2008;359(20):2143-53. Epub 2008/11/14.
10.
Ripperger T, Gadzicki D, Meindl A, Schlegelberger B. Breast cancer susceptibility: current knowledge
and implications for genetic counselling. Eur J Hum Genet. 2009;17(6):722-31.
11.
Wong MW, Nordfors C, Mossman D, Pecenpetelovska G, Avery-Kiejda KA, Talseth-Palmer B, et al.
BRIP1, PALB2, and RAD51C mutation analysis reveals their relative importance as genetic susceptibility factors
for breast cancer. Breast cancer research and treatment. 2011;127(3):853-9.
12.
Turnbull C, Seal S, Renwick A, Warren-Perry M, Hughes D, Elliott A, et al. Gene-gene interactions in
breast cancer susceptibility. Human molecular genetics. 2012;21(4):958-62. Epub 2011/11/11.
13.
Miki Y, Swensen J, Shattuck-Eidens D, Futreal PA, Harshman K, Tavtigian S, et al. A strong candidate for
the breast and ovarian cancer susceptibility gene BRCA1. Science. 1994;266(5182):66-71. Epub 1994/10/07.
14.
Turnbull C, Rahman N. Genetic predisposition to breast cancer: past, present, and future. Annual
review of genomics and human genetics. 2008;9:321-45. Epub 2008/06/12.
15.
Callebaut I, Mornon JP. From BRCA1 to RAP1: A widespread BRCT module closely associated with DNA
repair. FEBS letters. 1997;400(1):25-30.
16.
Rahman N, Stratton MR. The genetics of breast cancer susceptibility. Annual review of genetics.
1998;32:95-121. Epub 1999/02/03.
17.
Thakur S, Zhang HB, Peng Y, Le H, Carroll B, Ward T, et al. Localization of BRCA1 and a splice variant
identifies the nuclear localization signal. Mol Cell Biol. 1997;17(1):444-52.
18.
Welcsh PL, Owens KN, King MC. Insights into the functions of BRCA1 and BRCA2. Trends in Genetics.
2000;16(2):69-74.
19.
Koonin EV, Altschul SF, Bork P. BRCA1 protein products ... Functional motifs. Nature genetics.
1996;13(3):266-8. Epub 1996/07/01.
48
20.
Roy R, Chun J, Powell SN. BRCA1 and BRCA2: different roles in a common pathway of genome
protection. Nature reviews Cancer. 2012;12(1):68-78. Epub 2011/12/24.
21.
Wooster R, Bignell G, Lancaster J, Swift S, Seal S, Mangion J, et al. Identification of the breast cancer
susceptibility gene BRCA2. Nature. 1995;378(6559):789-92. Epub 1995/12/21.
22.
Raicevic-Maravic L, Radulovic S. Breast cancer susceptibility gene 2 -BRCA2.Archive of Oncology.
2001;9(2):3.
23.
Shuen AY, Foulkes WD. Inherited Mutations in Breast Cancer Genes-Risk and Response. J Mammary
Gland Biol. 2011;16(1):3-15.
24.
Stratton MR, Rahman N. The emerging landscape of breast cancer susceptibility. Nature genetics.
2008;40(1):17-22. Epub 2007/12/29.
25.
Mavaddat N, Antoniou AC, Easton DF, Garcia-Closas M. Genetic susceptibility to breast cancer.
Molecular oncology. 2010;4(3):174-91. Epub 2010/06/15.
26.
Chenevix-Trench G, Spurdle AB, Gatei M, Kelly H, Marsh A, Chen X, et al. Dominant negative ATM
mutations in breast cancer families. Journal of the National Cancer Institute. 2002;94(3):205-15.
27.
Ahmed M, Rahman N. ATM and breast cancer susceptibility. Oncogene. 2006;25(43):5906-11. Epub
2006/09/26.
28.
Lavin MF. Ataxia-telangiectasia: from a rare disorder to a paradigm for cell signalling and cancer.
Nature reviews Molecular cell biology. 2008;9(10):759-69. Epub 2008/09/25.
29.
Tavtigian SV, Oefner PJ, Babikyan D, Hartmann A, Healey S, Le Calvez-Kelm F, et al. Rare, evolutionarily
unlikely missense substitutions in ATM confer increased risk of breast cancer. American journal of human
genetics. 2009;85(4):427-46. Epub 2009/09/29.
30.
Goldgar DE, Healey S, Dowty JG, Da Silva L, Chen X, Spurdle AB, et al. Rare variants in the ATM gene
and risk of breast cancer. Breast cancer research : BCR. 2011;13(4):R73. Epub 2011/07/27.
31.
Nevanlinna H, Bartek J. The CHEK2 gene and inherited breast cancer susceptibility. Oncogene.
2006;25(43):5912-9. Epub 2006/09/26.
32.
Berge EO, Staalesen V, Straume AH, Lillehaug JR, Lonning PE. Chk2 splice variants express a dominantnegative effect on the wild-type Chk2 kinase activity. Biochimica et biophysica acta. 2010;1803(3):386-95.
33.
Desrichard A, Bidet Y, Uhrhammer N, Bignon YJ. CHEK2 contribution to hereditary breast cancer in
non-BRCA families. Breast cancer research : BCR. 2011;13(6):R119. Epub 2011/11/26.
34.
Adank MA, Jonker MA, Kluijt I, Van Mil SE, Oldenburg R, Mooi WJ, et al. Risk of breast cancer in
women with a CHEK2 mutation with and without familie history of breast cancer. Journal of Clinical oncology.
2011;29(28):8.
35.
Cybulski C, Wokolorczyk D, Jakubowska A, Huzarski T, Byrski T, Gronwald J, et al. Risk of breast cancer
in women with a CHEK2 mutation with and without a family history of breast cancer. Journal of clinical
oncology : official journal of the American Society of Clinical Oncology. 2011;29(28):3747-52. Epub 2011/08/31.
36.
Cantor SB, Guillemette S. Hereditary breast cancer and the BRCA1-associated FANCJ/BACH1/BRIP1.
Future Oncol. 2011;7(2):253-61. Epub 2011/02/25.
37.
Vahteristo P, Yliannala K, Tamminen A, Eerola H, Blomqvist C, Nevanlinna H. BACH1 Ser919Pro variant
and breast cancer risk. BMC cancer. 2006;6:19. Epub 2006/01/25.
38.
De Nicolo A, Tancredi M, Lombardi G, Flemma CC, Barbuti S, Di Cristofano C, et al. A novel breast
cancer-associated BRIP1 (FANCJ/BACH1) germ-line mutation impairs protein stability and function. Clinical
cancer research : an official journal of the American Association for Cancer Research. 2008;14(14):4672-80.
39.
Ali AM, Singh TR, Meetei AR. FANCM-FAAP24 and FANCJ: FA proteins that metabolize DNA. Mutat ResFund Mol M. 2009;668(1-2):20-6.
40.
Casadei S, Norquist BM, Walsh T, Stray S, Mandell JB, Lee MK, et al. Contribution of Inherited
Mutations in the BRCA2-Interacting Protein PALB2 to Familial Breast Cancer. Cancer research. 2011;71(6):22229.
41.
Tischkowitz M, Xia B, Sabbaghian N, Reis-Filho JS, Hamel N, Li G, et al. Analysis of PALB2/FANCNassociated breast cancer families. Proc Natl Acad Sci U S A. 2007;104(16):6788-93. Epub 2007/04/11.
42.
Poumpouridou N, Kroupis C. Hereditary breast cancer: beyond BRCA genetic analysis; PALB2 emerges.
Clin Chem Lab Med. 2012;50(3):423-34.
43.
Oliver AW, Swift S, Lord CJ, Ashworth A, Pearl LH. Structural basis for recruitment of BRCA2 by PALB2.
EMBO reports. 2009;10(9):990-6.
44.
Zhang F, Ma JL, Wu JX, Ye L, Cai H, Xia B, et al. PALB2 Links BRCA1 and BRCA2 in the DNA-Damage
Response. Curr Biol. 2009;19(6):524-9.
45.
Zhang F, Fan Q, Ren KQ, Andreassen PR. PALB2 Functionally Connects the Breast Cancer Susceptibility
Proteins BRCA1 and BRCA2. Molecular Cancer Research. 2009;7(7):1110-8.
49
46.
Rahman N, Seal S, Thompson D, Kelly P, Renwick A, Elliott A, et al. PALB2, which encodes a BRCA2interacting protein, is a breast cancer susceptibility gene. Nature genetics. 2007;39(2):165-7. Epub 2007/01/04.
47.
Erkko H, Dowty JG, Nikkila J, Syrjakoski K, Mannermaa A, Pylkas K, et al. Penetrance analysis of the
PALB2 c.1592delT founder mutation. Clinical cancer research : an official journal of the American Association
for Cancer Research. 2008;14(14):4667-71. Epub 2008/07/17.
48.
McClellan J, King MC. Genetic Heterogeneity in Human Disease. Cell. 2010;141(2):210-7.
49.
Zhang B, Beeghly-Fadiel A, Long JR, Zheng W. Genetic variants associated with breast-cancer risk:
comprehensive research synopsis, meta-analysis, and epidemiological evidence. Lancet Oncology.
2011;12(5):477-88.
50.
Wimmer K, Callens T, Wernstedt A, Messiaen L. The NF1 gene contains hotspots for L1 endonucleasedependent de novo insertion. PLoS genetics. 2011;7(11):e1002371. Epub 2011/11/30.
51.
Teugels E, De Brakeleer S, Goelen G, Lissens W, Sermijn E, De Greve J. De novo Alu element insertions
targeted to a sequence common to the BRCA1 and BRCA2 genes. Human mutation. 2005;26(3):284.
52.
Teugels E, De Greve J. About the c.156_157insAlu BRCA2 breast cancer predisposing mutation. Breast
cancer research and treatment. 2009;116(3):621-2. Epub 2008/09/23.
53.
Chen X, Truong TT, Weaver J, Bove BA, Cattie K, Armstrong BA, et al. Intronic alterations in BRCA1 and
BRCA2: effect on mRNA splicing fidelity and expression. Human mutation. 2006;27(5):427-35.
54.
Chen X, Weaver J, Bove BA, Vanderveer LA, Weil SC, Miron A, et al. Allelic imbalance in BRCA1 and
BRCA2 gene expression is associated with an increased breast cancer risk. Human molecular genetics.
2008;17(9):1336-48. Epub 2008/01/22.
55.
Holbrook JA, Neu-Yilik G, Hentze MW, Kulozik AE. Nonsense-mediated decay approaches the clinic.
Nature genetics. 2004;36(8):801-8.
56.
Chang YF, Imam JS, Wilkinson ME. The nonsense-mediated decay RNA surveillance pathway. Annu Rev
Biochem. 2007;76:51-74.
57.
Lixia M, Zhijian C, Chao S, Chaojiang G, Congyi Z. Alternative splicing of breast cancer associated gene
BRCA1 from breast cancer cell line. Journal of biochemistry and molecular biology. 2007;40(1):15-21.
58.
Sylvain V, Lafarge S, Bignon YJ. Dominant-negative activity of a Brca1 truncation mutant: effects on
proliferation, tumorigenicity in vivo, and chemosensitivity in a mouse ovarian cancer cell line. International
journal of oncology. 2002;20(4):845-53. Epub 2002/03/15.
59.
Purzycka J, I. O, I. S, W. P, J J. Efficiency comparison of seven different Taq polymerases used in
hemogenetics. International Congress series 1288. 2006:3.
60.
Stahlberg A, Kubista M, Pfaffl M. Comparison of reverse transcriptases in gene expression analysis.
Clinical chemistry. 2004;50(9):1678-80.
61.
Franca A, Freitas AI, Henriques AF, Cerca N. Optimizing a qPCR Gene Expression Quantification Assay
for S. epidermidis Biofilms: A Comparison between Commercial Kits and a Customized Protocol. PloS one.
2012;7(5).
62.
Coutinho G, Xie J, Du L, Brusco A, Krainer AR, Gatti RA. Functional significance of a deep intronic
mutation in the ATM gene and evidence for an alternative exon 28a. Human mutation. 2005;25(2):118-24.
63.
Baralle D, Baralle M. Splicing in action: assessing disease causing sequence changes. Journal of medical
genetics. 2005;42(10):737-48. Epub 2005/10/04.
64.
Rutter JL, Smith AM, Davila MR, Sigurdson AJ, Giusti RM, Pineda MA, et al. Mutational analysis of the
BRCA1-interacting genes ZNF350/ZBRK1 and BRIP1/BACH1 among BRCA1 and BRCA2-negative probands from
breast-ovarian cancer families and among early-onset breast cancer cases and reference individuals. Human
mutation. 2003;22(2):121-8. Epub 2003/07/23.
65.
Sanz DJ, Acedo A, Infante M, Duran M, Perez-Cabornero L, Esteban-Cardenosa E, et al. A high
proportion of DNA variants of BRCA1 and BRCA2 is associated with aberrant splicing in breast/ovarian cancer
patients. Clinical cancer research : an official journal of the American Association for Cancer Research.
2010;16(6):1957-67. Epub 2010/03/11.
66.
Gaildrat P, Krieger S, Di Giacomo D, Abdat J, Revillion F, Caputo S, et al. Multiple sequence variants of
BRCA2 exon 7 alter splicing regulation. Journal of medical genetics. 2012;49(10):609-17. Epub 2012/09/11.
67.
Mattick JS, Makunin IV. Non-coding RNA. Human molecular genetics. 2006;15 Spec No 1:R17-29.
68.
Tufarelli C, Stanley JA, Garrick D, Sharpe JA, Ayyub H, Wood WG, et al. Transcription of antisense RNA
leading to gene silencing and methylation as a novel cause of human genetic disease. Nature genetics.
2003;34(2):157-65. Epub 2003/05/06.
50
BIJLAGE 1: Patiëntentabel
1
EBV
nummer
E10-21
RNA nummer
(P+)
1200300
DNA
nummer
D1014173
BR-32-0698
Manchester
score
19
2
172
1200302
5192
BR-32-0005
20
3
531
1200304
6286
BR-32-0007
24
4
718
1200306
D0811251
BR-32-0076
20
5
727
1200308
M9903340
BR-32-0095
18
6
730
1200310
M0000107
BR-32-0093
24
7
755
1200312
M0000666
BR-32-0071
26
8
675
1200327
11023
BR-32-0039
24
9
593
1200347
M9900890
BR-32-0066
32
10
517
1200349
11285
BR-32-0041
16
11
758
1200351
M0000709
BR-32-0102
32
12
445
1200353
10600
BR-32-0020
40
13
420
1200355
10259
BR-32-0027
48
14
673
1300002
10712
BR-32-0029
26
15
529
1300040
11503
BR-32-0050
/
16
485
1300042
11100
BR-32-0010
/
17
767
1300044
M0000821
BR-32-0109
12
18
743
1300046
M0000341
BR-32-0099
12
19
571
1300048
M9900570
BR-32-0063
18
20
383
1300050
9787
BR-32-0024
24
21
191
1300052
05198A
BR-32-0004
17
22
G1100019
1200047
/
/
33
23
G1100020
1200049
/
/
36
24
G1100021
1200068
/
/
47
25
220
1200071
6729
BR-32-0007
14
Familienummer
BIJLAGE 2: PROTOCOL: RNA extractie (RNeasy kit)
Volume bepalen van de T75 cultuurflessen, waarin de EBV cellijn werd opgekweekt
Cellijn splitsen in twee T25 cultuurflessen
Aan één T25 wordt puromycine toegevoegd
=> 200 µg puromycine/ ml milieu op te lossen in 200 µl RPMI per staal
(1 volume meer rekenen)
4-6 uur incuberen (37°C, 5% CO2)
Alles uit de T25 cultuurflessen overpipetteren in 15ml falcons (voorzien van RNA
nummer en naam van de patiënt)
5 minuten centrifugeren bij 300 rcf
Supernatans afgieten
Flickn’ the tube (pellet losmaken)
RLT-mix maken : 600 µl RLT-buffer per staal + 10 µl β-mercapto per ml RLT-buffer
600 µl RLT-mix in falcon brengen
30 “ vortexen
600 µl 70% ethanol in falcon brengen (dit goed mengen door op en neer te pipetteren)
Breng 700 µl per falcon over op de RNeasy mini spin kolommen (roze)
15” centrifugeren op volle kracht
Filtraat weggieten
De overblijvende rest van in de falcons wordt overgebracht op de kolom
15” centrifugeren op volle kracht
Filtraat weggieten
700 µl wasbuffer (RW1) op kolom aanbrengen
15” centrifugeren op volle kracht
Filtraat weggieten en collection tube wegsmijten
De mini spin kolom wordt vervolgens in een nieuwe collection tube geplaatst
500 µl RPE op kolom aanbrengen (Bij eerste gebruik: ethanol toevoegen)
15” centrifugeren op volle kracht
Filtraat weggieten
500 µl RPE op kolom
2 minuten afdraaien op volle kracht
Filtraat weggieten en collection tube wegsmijten
De kolom wordt vervolgens in een 1.5ml RNase-vrij epje geplaatst
50 µl RNase-vrij water centraal pipetteren op de kolom
1 minuut laten staan en vervolgens 1 minuut afdraaien op volle snelheid
Kolom wegsmijten en vervolgens onmiddellijk het RNA op ijs plaatsen
Vervolgens kan men een RNA-meting uitvoeren om de kwaliteit en concentratie te
achterhalen
BIJLAGE 3: Protocol van de 3 verschillende cDNA synthese kit’s
PROTOCOL: cDNA synthese SuperScript II
Voeg 2 µg RNA in een epje (200 µl)
Aanvullen met HPLC H2O tot 10 µl
Voeg 2 µl per epje toe van mix 1
mix 1
per reactie
random primers (50ng/µl)
1 µl
dNTP's (10mM)
1 µl
Incubeer gedurende 5 minuten bij 65°C
Onmiddellijk afkoelen op ijs en centrifugeren
Voeg 7 µl per epje toe van mix 2
mix 2
per reactie
5x First strand buffer
4 µl
0,1M DTT
2 µl
RNAsin (40U/ µl)
1 µl
Incubeer gedurende 2 minuten bij 25°C
Voeg 1 µl superscript II RT toe per epje
Plaats in PCR-machine met volgend programma:
25 °C
42 °C
70 °C
4 °C
10 min
50 min
15 min
Oneindig
PROTOCOL: cDNA synthese M-MULV RT (RNase H-)
Voeg 1 µg RNA in een epje (200 µl)
Aanvullen met HPLC H2O tot 9 µl
Voeg 3 µl per epje toe van mix 1
mix 1
per reactie
random primers (50ng/µl)
2 µl
dNTP's (10mM)
1 µl
Incubeer gedurende 5 minuten bij 65°C
Onmiddellijk afkoelen op ijs en centrifugeren
Voeg 8 µl per epje toe van mix 2
mix 2
5x reaction buffer
0,1M DTT
RNAsin (40U/µl)
M-MULV RT (200U/µl)
per reactie
4 µl
2 µl
1 µl
1 µl
Plaats in PCR-machine bij volgend programma:
25 °C
42 °C
80 °C
4 °C
10 min
50 min
5 min
Oneindig
PROTOCOL: cDNA synthese GoScript
Voeg 1 µg RNA in een epje (200 µl)
Aanvullen met HPLC H2O tot 4 µl
Voeg 1 µl random primers (500 ng/µl) toe
Incubeer gedurende 5 minuten bij 70°C
Onmiddellijk afkoelen op ijs en centrifugeren
Voeg 15 µl per epje toe van mix 1
mix 1
GoScript 5x Reaction Buffer
MgCl2 (25mM)
RNAsin (40U/µl)
Go Script RT
dNTP's (0,5mM)
sigma H2O
per reactie
4 µl
2,4 µl
0,5 µl
1 µl
1 µl
6,1 µl
Plaats in PCR-machine bij volgend programma:
25 °C
42 °C
70 °C
4 °C
5 min
60 min
15 min
Oneindig
BIJLAGE 4: PROTOCOL: Sanger Sequering
1. Opzuivering
Voeg 1 µl toe van mix 1
Handelsnaam
New England
BioLabs
New England
BioLabs
VWR
Mix 1
Exonuclease I
Antarctic phosphatase
H2O
Voeg hieraan 5 µl PCR product toe
Plaats in PCR machine bij volgend programma:
15 min
20 min
µl per reactie
0,05
0,20
0,75
37°C
80°C
Na opzuivering toevoegen van 20 µl H2O om te verdunnen
2. Sequencing reactie
Voeg 8,5 µl toe van mix 2 (in nieuw epje)
Mix 2
RR-mix
ABI buffer
primer (1/50 verd. van 50µM)
H2O
Handelsnaam
Applied Biosystems
eigen bereiding
Life technologies
µl per reactie
0,50
2,00
3,00
VWR
3,00
Voeg 1,5 µl toe van het verdund opgezuiverd PCR product
Plaats in PCR machine bij volgend programma:
5 min
10 sec
5 sec
4 min
10 sec
95 °C
95 °C
55 °C
60 °C
15 °C
25 cycli
3. Precipitatie met beads
Resuspendeer de magnetische beads, maak een homogene oplossing
Voeg 10 µl beads toe bij elk sequentieproduct
Voeg 45 µl 85% EtOH en mix door 7 maal op en neer te pipetteren
Plaats de sequentieproducten gedurende 3 minuten op een magnetische plaat
Verwijder het supernatans
Voeg 100 µl 85% EtOH toe en incubeer gedurende 30 seconden op een magnetische
plaat
Verwijder opnieuw het supernatans
Laat de sequentieproducten gedurende 10 minuten drogen (op kamertemperatuur of
37°C )
Voeg 40 µl H2O toe en wacht gedurende 3 minuten
Mix het mengels door op en neer te pippeteren
Plaats het op een magnetische plaat en wacht tenminste 30 seconden
Breng 30 µl zuiver sequentieproduct over in een vers epje of plaat
BIJLAGE 5: Resultaat Labchip gekloneerde producten
Controles
Patiënten
Figuur: Resultaat labchip: PCR van gekloneerde producten.
Bijlage 6: Overzicht sequenering van de gekloonde PCR producten
Patiënten
1: c.787_4096del (del 3’deel van exon 11)
2: c.671_4096del (del exon 11)
3: c.594_4096del (del exon 10 +11)
4: c.594_4096del (del exon 10 +11)
5: c.671_3897 del (del 5’deel van exon 11)
6: c.594-4096del (del exon 10 +11)
7: c.671_4096del (del exon 11)
8: mislukt
9: mislukt
10: sequentie uit MFSD6
11: c.594-4096del (del exon 10 +11)
12: c.671_3897 del (del 5’deel van exon 11)
13: c.671_3881del (del 5’deel van exon 11)
14: mislukt
15: mislukt
16: c.594-4096del (del exon 10 +11)
17: mislukt
18: c.671-4096del (del exon 11)
19: c.671-4096del (del exon 11)
20: c.671_3881del (del 5’deel van exon 11)
21: c.671_3881del (del 5’deel van exon 11)
22: c.594-4096del (del exon 10 +11)
23: c.594-4096del (del exon 10 +11)
24: sequenering mislukt
Controles
1: c.787_4096del (del 3’deel van exon 11)
2: c.787_4096del (del 3’deel van exon 11)
3: mislukt
4: mislukt
5: mislukt
6: mislukt
7: c.787_4096del (del 3’deel van exon 11)
8: c.787_4096del (del 3’deel van exon 11)
9: mislukt
10: mislukt
11: sequentie afkomstig van KANSL1-001
12: mislukt
13: c.671-4096del (del exon 11)
14: mislukt
15: mislukt
16: c.787_4096del (del 3’deel van exon 11)
17: mislukt
18: c.787_4096del (del 3’deel van exon 11)
19: mislukt
20: c.787_4096del (del 3’deel van exon 11)
21: mislukt
22: mislukt
23: c.787_4096del (del 3’deel van exon 11)
24: mislukt
25: mislukt
26: c.787_4096del (del 3’deel van exon 11)
27: c.787_4096del (del 3’deel van exon 11)
BIJLAGE 7: Overzicht PCR en sequeneringsprimers
Gen
Referentiesequentie
BRCA1
NM_007294.3
BRCA2
NM_000059.3
ATM
NM_000051.3
PALB2
NM_024675.3
BRIP1
NM_032043.2
CHEK2
NM_001005735.1
Gebruikte primers BRCA1
PCR
Sequentie
Startplaats
lengte primer (bp)
Tm (salt) (°C)
GC %
bisearch
PCR1F
CCCTCTGCTCTGGGTAAAG
82
19
59,5
58
9 producten :
PCR1R
GAGTAATAAACTGCTGTTCTCATG
995
24
60,3
38
(5x) 914; 1003; 836; 1030; 225
PCR2F
CTGAAAGCCAGGGAGTTGG
4102
19
59,5
58
6 producten :
PCR2R
GTCAATTCTGGCTTCTCCCTG
5038
21
61,2
52
(4x) 937; 1003; 1000
PCR3F
CATACCATCTTCAACCTCTGC
4895
21
59,5
48
6 producten
PCR3R
GGACTGAAGAGTGAGAGGAG
5807
20
60,5
55
(5x) 913; 839
Sequenering
Sequentie
Startplaats
lengte primer (bp)
Tm (salt) (°C)
GC %
lengte
PCR1F
1FA seq
CCCTCTGCTCTGGGTAAAG
GATTTAGTCAACTTGTTGAAGAGC
82
352
19
24
59,5
60,3
58
38
270
643
PCR2F
2FA seq
CTGAAAGCCAGGGAGTTGG
GCTGACAAGTTTGAGGTGT
4102
4539
19
19
59,5
55
58
47
437
356
PCR3F
CATACCATCTTCAACCTCTGC
4895
21
59,5
48
BRCA2
PCR
Sequentie
Startplaats
lengte primer (bp)
Tm (salt) (°C)
GC %
bisearch
PCR1F
PCR1R
GCGGTTTTGTCAGCTTACTC
CCCAAAAGAGCTAGTTAAGGAC
143
2225
20
22
58,4
60,1
50
45
2082
PCR2F
PCR2R
CGAAAATGAGGAAATGGTTTTGTC
GGCCTCCACATATTTTGCTGC
7004
8784
24
21
60,3
61,2
38
52
1801
PCR3F
PCR3R
GGAGCAGAACTGGTGGGC
CCTTTACAAGACTTTGAAGTCATC
8496
9997
18
24
60,8
60,3
67
38
1502
Sequenering
Sequentie
startplaats lengte primer (bp)
Tm (salt) (°C)
GC %
lengte (bp)
PCR1F
1FA seq
1FB seq
1FC seq
1FD seq
GCGGTTTTGTCAGCTTACTC
GGAAACCATCTTATAATCAGC
GGAGTGAGGTGGATCCTG
GTGGAACCAAATGATACTGATCC
CTTCTCCAGTGGCTTCTTCA
143
433
786
1305
1705
20
21
18
23
20
58,4
55,4
58,4
60,9
58,4
50
38
61
43
50
290
353
519
400
520
PCR2F
2FA seq
2FB seq
CGAAAATGAGGAAATGGTTTTGTC
CAGGCAGACCAACCAAAGTC
CACACTGAAGATTATTTTGGTAAGG
7004
7429
7920
24
20
25
60,3
60,5
60,9
38
55
36
425
491
576
PCR3F
3FA seq
3FB seq
GGAGCAGAACTGGTGGGC
CCTTGAATAATCACAGGCAAATG
CCTTAATGAGGACATTATTAAGCC
8496
9016
9557
18
23
24
60,8
59,2
60,3
67
39
38
520
541
ATM
PCR
Sequentie
startplaats
lengte
Tm (primer3plus) (°C)
Tm (°C) (salt)
GC (%)
Bisearch
PCR product
PCR1F
PCR1R
CCTGCTGCCCAGATATGACT
CTGTTGGGGTAGAAGCTGAG
132
1527
20 bp
21 bp
60,2
59,5
60,5
60,5
55
55
1 product
1396 bp
PCR2F
PCR2R
GATCACCTTCAGAAGTCACAG
ATCCCTTGTGTTCTCAGAGTC
1381
3207
21 bp
21 bp
/
/
59,5
59,5
48
48
3 producten
1848
PCR3F
PCR3R
GGAGCTTCCTGGAGAAGAG
ATCCTTACAGTAAGTCACGGC
3045
4722
19 bp
21 bp
/
/
59,5
59,5
58
48
2 producten
1678
PCR4F
PCR4R
GATCCTGCTCCTAATCCACC
AAGTGAGCAGCACAAGACTGA
4306
6044
20 bp
21 bp
58,5
58,5
59,5
59,5
55
48
3
1739
PCR5F
PCR5R
GGGATTTTTCACCAGCTGTCT
CATCATCTTGGTCCCCATTCT
5790
7785
21 bp
21 bp
60,5
61,1
59,5
59,5
48
48
4
1996
PCR6F
GAAAGAGGATCGTAAACGCTTC
7553
22 bp
59,4
60,1
45
3
2001
PCR6R
GCAGAGATGTTCCTTAAGACCA
9554
22 bp
60,1
60,1
45
Sequenering
Sequentie
startplaats
PCR1F
CCTGCTGCCCAGATATGACT
132
1FA seq
CTC CAG GCA GAA AAA GAT GCA
453
1FB seq
CAT GCT GTT ACC AAA GGA TGC T
763
1FC seq
GTC ATA TAG GAA GTA GAG GAA AG
1160
Lengte
20
21
22
23
Tm (°C) (salt)
60,5
59,5
60,1
59,2
GC %
55
48
45
39
lengte
321 bp
310 bp
397 bp
221 bp
PCR2F
2FA seq
2FB seq
2FC seq
GATCACCTTCAGAAGTCACAG
GGAAGTTATTTACTGGGTCAGC
GGTATAGAAAAGCACCAGTCC
GATTGCATCTGGCTTTTTCCTG
1381
1760
2197
2580
21
22
21
22
59,5
60,1
59,5
60,1
48
45
48
45
379 bp
437 bp
383 bp
465 bp
PCR3F
3FA seq
3FB seq
GGAGCTTCCTGGAGAAGAG
GAGAACCCTGAAACTTTGGATG
CCATTGCTAATCAGATTCAAGAG
3045
3604
3998
19
22
23
59,5
60,1
59.2
58
45
39
559 bp
394 bp
308 bp
PCR4F
4FA seq
4FB seq
4FC seq
GATCCTGCTCCTAATCCACC
TGT GTA TGA GCA GGT GGA GG
GTT GCA GTT ATC CAA GAT GGC
GACTGGACATAGTTTCTGGGA
4306
4770
5152
5421
20
20
21
21
59,5
60,5
59,5
59,5
55
55
48
48
464 bp
382 bp
269 bp
369 bp
PCR5F
5FA seq
5FB seq
5FC seq
GGGATTTTTCACCAGCTGTCT
CAATGTGGGGCAAAGCCCTA
TCTGTGTATTCGCTCTATCCC
GCCCTGAGTATTCTCAAGCAA
5790
6308
6682
7111
21
20
21
21
59,5
60,5
59,5
59.5
48
55
48
48
518 bp
374 bp
429 bp
442 bp
PCR6F
6FA
6FB
6FC
6FD
6FE
GAAAGAGGATCGTAAACGCTTC
ACTAAACCAGAGGTAGCCAGA
CCAGCAGACCAGCCAATTAC
GAACTGTCCCCATTGGTGAAT
GGCAAAATCCTTCCTACTCCT
GTGCTCAGTGTTGGTGGACA
7553
7906
8130
8504
8878
9262
22
21
20
21
21
20
60,1
59,5
60,5
59,5
59,5
60,5
45
48
55
48
48
55
353 bp
224 bp
374 bp
374 bp
384 bp
PALB2
PCR
Sequentie
startplaats lengte primer (bp)
PCR1F
PCR1R
GACGGCTGCTCTTTTCGTTC
CTCAAAGGGCTCCACTGGTT
121
2070
PCR2F
PCR2R
CGATTGTTAACAGGTCCAAGGAAGAAG
GTCTGGACATAAACAAGCAATCATTT
1763
3837
Sequenering
Sequentie
SEQ1F
TTTTCGTTCTGTCGCCTGCC
132
1FA seq
GCCTGAGTCCTTTAACCCTGG
1FB seq
Tm (salt) (°C)
GC %
bisearch
20
20
60,5
60,5
55
55
2 producten
1950; 1933
27
26
66,6
61,7
44
35
2 producten
2075
startplaats lengte primer (bp)
Tm (salt) (°C)
GC %
lengte
20
60,5
55
341
462
21
63,2
57
417
CACCAGGGCGACTACAGTTCC
879
21
65,3
62
405
1FC seq
CCTAAGAGTCTTAGCCTGGAAGCA
1284
24
65,2
50
479
PCR2F
CGATTGTTAACAGGTCCAAGGAAGAAG
1763
27
66,6
44
425
2FAseq
CCAGGAAAATCACATCCCAAAAGGC
2188
25
65,8
48
431
2FB seq
GCTCTGTAGAAACACATGCCAGG
2619
23
64,6
52
441
2FC seq
GCTTGGCCTGACAAAGAG
3060
18
56,3
56
317
2FD seq
GTCATCCCTGTGCCAAAGAGAGTG
3377
24
66,9
54
BRIP1
PCR
PCR1F
PCR1R
Sequentie
GGGCTCCGCTTTATTTGCTC
CACAGCTCGTAGGGGTTCAT
Startplaats
145
1591
lengte
20 bp
20 bp
Tm (primer3plus) (°C)
63,2
60,1
Tm (°C) (salt)
60,5
60,5
GC (%)
55
55
Bisearch
1 product
PCR product
1477 bp
PCR2F
PCR2R
CACAGCCCGAGAACTAATACA
TAGATGACTTGCTGCTTCCAG
1327
3055
21 bp
21 bp
57,9
58,3
59,5
59,5
48
48
2 producten
1729 bp
PCR3F
PCR3R
GAGCTCTCCTGGTAGCAGTT
AAGGTTGCAGTGAGCCCAGA
2529
4250
20 bp
20 bp
/
/
60.5
60.5
55
55
1 product
1722
Sequenering
SEQ1F
1FA SEQ
1FB SEQ
Sequentie
GCTCTCAGAAGTCGGTTTCC
CCAAGCACACCACCTTCTGA
ACAGGGAAATCCAAGATACCC
startplaats
161
599
980
Lengte
20 bp
20 bp
21 bp
Tm (°C) (salt)
60,5
60,5
59,5
GC%
55
55
48
lengte (bp)
438
371
347
PCR2F
2FA SEQ
2FB SEQ
2FC SEQ
CACAGCCCGAGAACTAATACA
TCACCACTGCTACTTTTCCCA
GTTCACGACAGAAAACTGCAG
TGGCTCTCTACTGGTTTATGG
1327
1713
2004
2390
21 bp
21 bp
21 bp
21 bp
59,5
59,5
59,5
59,5
48
48
48
48
386
291
386
382
PCR3F
3FA SEQ
3FB SEQ
3FC SEQ
ACAGAAATGATTGGGGAGCTC
CAGATCCACAAGCCCAACTTT
CCCTGGATCCAGACATTGAAT
CAGAGAAGTGAAAGCTGAGGA
2772
3226
3561
3814
21 bp
21 bp
21 bp
21 bp
59,5
59,5
59,5
59,5
48
48
48
48
454
335
253
CHEK2
PCR
Sequentie
startplaats lengte primer (bp) Tm (primer3plus) (°C) Tm (salt) (°C)
GC %
bisearch
PCR1F
PCR1R
AGGTTTAGCGCCACTCTGCT
GTGCCTCACACCTCTTATCC
7
499
20
20
/
/
60,5
60,5
55
55
1 Product: 493 bp
PCR2F
PCR2R
CCAATCTTGAGACAGAGTCTG
ACTACCAACAACTTCTTGACAAG
377
1601
21
23
/
/
59,5
59,2
48
39
1 Product: 1225 bp
PCR3F
PCR3R
GAGCATAGGACTCAAGTGTCA
AGTTCACAACACAGCAGCACA
1510
1840
21
21
/
/
59,5
59,5
48
48
3 verschillende producten: 351 bp
pseudogenen: 350 en 389
BIJLAGE 8: Overzicht optimalisatieprocedure PALB2 PCR1; BRIP1 PCR3; CHEK2
Gebruikte mastermixen
Takara Ex Taq:
ExBuffer (10x)
dNTP's (2,5mM elk dNTP)
H20 (HPLC)
Primer F (50 µM)
Primer R (50 µM)
Ex Taq (5U/µl)
cDNA
Totaalvolume
Handelsnaam
Takara
Takara
VWR
Life Technologies
Life Technologies
Takara
per reactie
(µl)
1,5
1
11,21
0,1
0,1
0,09
1
15
Takara Ex Taq + 5% DMSO:
ExBuffer (10x)
dNTP's (2.5 mM elk dNTP)
H20 (HPLC)
Primer F (50 µM)
Primer R (50 µM)
DMSO
Ex Taq (5U/µl)
cDNA
totaalvolume
PCR programma 1 en 2 zie figuur 13.
Handelsnaam
Takara
Takara
VWR
Life Technologies
Life Technologies
Takara
per reactie
(µl)
1,5
1
10,46
0,1
0,1
0,75
0,09
1
15
PALB2 PCR1
Bestaande primers (1):
PALB2
Sequentie
startplaats
Bisearch
PCR1F
PCR1R
GGGTGCGATCCCGGGCT
GCATTCCATCCCTATGAAATGGAGCC
78
1992
2 producten
1898, 1915
PCR Programma 1: aanhechtingstemperatuur 62°C en 65°C
Bij dit programma en deze aanhechtingstemperaturen zien we geen geamplificeerd
PCR product
PCR programma 1: gradiënt PCR 50°C-70°C
Bij de aanhechtingstemperatuur van 50°C-70°C zien we geen banden
PCR programma1: gradiënt PCR 50°C-70°C met Takara Ex Taq + 5% DMSO
Ook bij toevoeging van 5% DMSO aan de mastermix zien we geen banden. Hier werd
er overgegaan tot de ontwikkeling van nieuwe PALB2 PCR1 primers.
Nieuwe PALB2 PCR1 primers (2):
PALB2
Sequentie
startplaats
bisearch
PCR1F
PCR1R
GACGGCTGCTCTTTTCGTTC
CTCAAAGGGCTCCACTGGTT
121
2070
2 producten
1950; 1933
PCR programma 1: aanhechtingstemperatuur 58°C
De fragmenten die in deze PCR werden geamplificeerd zijn van de verkeerde
groottes.
PCR programma 2: aanhechtingstemperatuur 58°C, 60°C en 62°C
Labchip resultaten respectievelijk bij 58°C, 60°C en 62°C. De PCR reactie bij
60°C werd weerhouden, alhoewel deze niet specifiek is, is het PCR product van de
gewenste grootte sterker aanwezig dan de aspecifieke fragmenten. Er werd geen
rekening gehouden met controle 2939 aangezien dit staal tekenen van degradatie
vertoond.
CHEK2
Primerparen (1):
CHEK2
Sequentie
startplaats
bisearch
PCR1F
PCR1R
AGGTTTAGCGCCACTCTGCT
GCAGTGGTTCATCAAAGCAATATT
6
593
6 producten
(5x) 458; (1x) 587
PCR2F
PCR2R
CCTGCTGTCATCTGATCCTC
AACACAGCAGCACACACAGC
436
1833
1 product
1398 bp
Programma 2: aanhechtingstemperatuur 58°C
Beide PCR reacties gaan aspecifiek door, waarbij het fragment de gewenste lengte
niet aanwezig is.
PCR programma 2: aanhechtingstemperatuur 60°C
Idem als bij 58°C.
PCR programma 2: aanhechtingstemperatuur 62°C en 64°C
Beide PCR’s geven bij de verschillende aanhechtingstemperaturen aspecifieke
fragmenten. Ook hierbij is het fragment met de correcte grootte niet aanwezig.
PCR programma 2: gradiënt 45°C-69°C
PCR 1 geeft nog steeds een aspecifieke reactie bij deze verschillende
temperaturen, maar omdat sommige fragmenten in de buurt liggen qua grootte van
het gewenste fragment werden sommige fragmenten gesequeneerd om na te gaan
als deze het gewenste fragment opleverden. Dit was niet het geval.
Bij PCR 2 komen geen van de fragmenten in de buurt van het gewenste product.
Er werd besloten om nieuwe primers voor CHEK2 te ontwikkelen.
Primerparen (2):
CHEK2
Sequentie
startplaats
Bisearch
PCR1F
PCR1R
AGGTTTAGCGCCACTCTGCT
GTGCCTCACACCTCTTATCC
7
499
1 product
493 bp
PCR2F
PCR2R
CCAATCTTGAGACAGAGTCTG
ACTACCAACAACTTCTTGACAAG
377
1601
1 product 1225 bp
1225 bp
PCR3F
PCR3R
GAGCATAGGACTCAAGTGTCA
AGTTCACAACACAGCAGCACA
1510
1840
3 producten
351 bp + pseudogenen : 350 en 389
PCR programma 2: aanhechtingstemperatuur 58°C en 60°C
Alle drie de PCR’s verlopen aspecifiek, waarbij de gewenste groottes van
fragmenten niet aanwezig zijn.
PCR programma 2: aanhechtingstemperatuur 62°C en 64°C
Idem als bij aanhechtingstemperatuur 58°C en 60°C. Hierbij valt wel op dat PCR
duidelijk minder goed doorgaat.
PCR programma 1: gradiënt 50°C-70°C
De amplificatie voor alle drie de fragmenten is slechts heel licht doorgegaan.
Hoewel er fragmenten van de correcte grootte werden geamplificeerd zullen we
deze gradiënt PCR eerst uitvoeren met gebruik van PCR programma 2.
Programma 2: gradiënt 45°C-69°C
CHEK2 PCR1 geeft een specifiek fragment van de correcte grootte in laan 12,
deze laan komt overeen met een aanhechtingstemperatuur van 68°C. Dit
programma en de aanhechtingstemperatuur van 68°C zal weerhouden worden voor
de amplificatie van dit fragment
Bij de uitgevoerde gradiënt PCR voor CHEK2 PCR2 levert slechts enkel
aspecifieke amplificatie op. In een volgende fase zal deze gradiënt uitgevoerd
worden met een mastermix waaraan 5% DMSO aan toegevoegd werd.
CHEK2 PCR3 geeft na PCR een specifiek fragment in laan 11, hoewel hierbij nog
een ander fragment geamplificeerd wordt (lichter bandje), is het gewenste
fragment sterker geamplificeerd. Programma 2 met een aanhechtingstemperatuur
van 65°C zal gebruikt worden bij de amplificatie van dit fragment.
PCR programma 2: gradiënt PCR 45-69°C; mastermix met 5% DMSO
Specifieke amplificatie van CHEK2 PCR2 stelt men vast in laan 9 bij een
aanhechtingstemperatuur van 65°C. Voor amplificatie van dit fragment zal PCR
programma 2 gebruikt worden met een aanhechtingstemperatuur van 65°C. Aan de
mastermix zal voor amplificatie van dit fragment 5% DMSO toegevoegd worden.
BIJLAGE 9: Overzicht predicties Alamut
Resultaat Alamut predictie op splicing voor c.517G>C (links) en c.7618-6G>T (rechts).
Alamut voorspeld een lichte daling in de sterkte van de acceptor splice site voor de c.517G>C, terwijl de
acceptor splice site versterkt zou worden door de aanwezigheid van de c.7618-6G>T variant.
In silico predictie voor c.3326C>T variant.
Een nieuwe donor splice site wordt voorspeld voor de c.3326C>T variant (aangeduid door de blauwe balken).
Predictie voor de c.681+111C>G variant.
SpliceSiteFinder-Like en NNSPLICE voorspellen een nieuwe acceptor splice site op de positie van de variant,
terwijl MaxEntScan en Human Splicing Finder een lichte versterking geven aan de mogelijk aanwezige acceptor
splice site (Figuur rechts). De eerstvolgende donor splice site wordt voorspeld op positie c.681+390 (figuur
rechts)
BIJLAGE 10: Alamut donor- en acceptor splice predicties
Predictie c.671-2A>G.
Verscheidene in silico predictie tools voorspellen het verlies van de acceptor splice site op positie c.671.
Acceptor splice site gelegen op positie c.594.
Acceptor splice site aanwezig op positie c.3881 (zwak!) en c.3897.
Voorspelde donor splice site na positie 787.