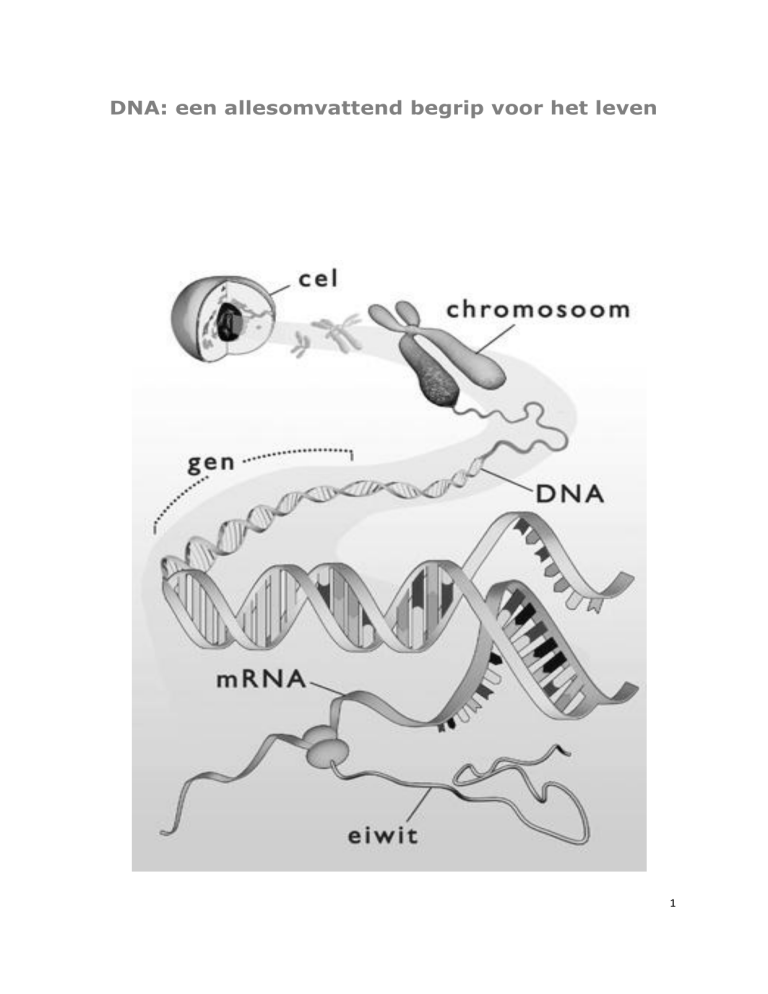
DNA: een allesomvattend begrip voor het leven
1
INHOUD
Inhoud ................................................................................................................................... 2
Les 1 Algemene informatie over DNA , TRANSCRIPTIE en TRANSLATIE ........................... 4
1.1 DNA ............................................................................................................................. 4
1.2 Transcriptie: Van DNA naar RNA ................................................................................. 6
1.3 Translatie: Van RNA naar eiwit .................................................................................... 9
1.4 tRNA ...........................................................................................................................10
1.5 ribosomen ...................................................................................................................10
1.6 Het translatieproces ....................................................................................................11
Opdrachten bij les 1 ......................................................................................................12
Les 2: Eiwitten: AMINOZUREN EN POSTTRANSLATIONELE MODIFICATIES ..................15
2.1 codons ........................................................................................................................15
2.2 aminozuren .................................................................................................................15
2.3 Peptidebindingen ........................................................................................................18
2.4 Posttranslationele modificaties ....................................................................................19
Opdrachten bij les 2 ......................................................................................................22
Les 3: Eiwitten: EIWITVOUWING en MUTATIES .................................................................24
3.1 Eiwitvouwing ...............................................................................................................24
3.2 Transport ....................................................................................................................25
3.3 Retinitis Pigmentosa ...................................................................................................27
3.4 Mutaties ......................................................................................................................27
3.4 Rhodopsine en retinal .................................................................................................28
Opdrachten bij les 3 ......................................................................................................29
BIOINFORMATICA-COMPUTERPRACTICUM ....................................................................30
algemene informatie bioinformatica ..................................................................................31
COMPUTERPRACTICUM MET YASARA ........................................................................31
Les 4 DNA REPLICATIE EN DNA ONDERZOEK.................................................................34
2
4.1 DNA replicatie .............................................................................................................34
4.2 Polymerase chain reaction (PCr) ................................................................................35
4.3 Sequencing .................................................................................................................37
Opdrachten bij les 4 ......................................................................................................39
Definitieblad .........................................................................................................................40
3
LES 1 ALGEMENE INFORMATIE OVER DNA , TRANSCRIPTIE EN
TRANSLATIE
Figuur 1.1: Plek in de cel. Het DNA bevindt zich in de celkern.
Tijdens de ontwikkeling en groei van ons lichaam is de stof DNA betrokken. Dit DNA is
belangrijk in alle processen van ons lichaam. Één van de belangrijkste processen om ons
in leven te houden is de eiwitsynthese. Hierbij wordt DNA overgeschreven naar RNA, dat
weer vertaald wordt naar een eiwit. Wanneer de eiwitten niet goed worden
gesynthetiseerd kan ons lichaam niet goed functioneren en kan het zijn dat we ziek
worden. De besturing van dit proces begint in de celkern met de chromosomen.
Chromosomen bestaan uit een groot aantal genen en ieder gen codeert voor een eiwit
(zie figuur op de voorpagina).
1.1 DNA
Desoxyribonucleïnezuur (DNA) is de belangrijkste drager van erfelijke informatie. In
1953 ontdekten de onderzoekers James Watson en Francis Crick dat het DNA de vorm
heeft van een spiraalvormige draad die met een andere DNA draad is verbonden en die
gedraaid is om zijn lengteas. Dit wordt ook wel de dubbele helix genoemd. Een DNAmolecuul is dubbelstrengs en bestaat uit twee lange ketens van nucleotiden. Deze ketens
zijn polyesters van afwisselend een fosfaat- en desoxyribosegroep (suikergroep). Naast
de fosfaatgroep en de suikergroep bezit het DNA ook nog over base. De twee
nucleotideketens worden met elkaar verbonden door middel van basenparen. De base
waaruit het DNA bestaat zijn adenine (A), thymine (T), cytosine (C) en guanine (G).
4
Figuur 1.2: Structuur van het DNA.
DNA bestaat uit twee ketens nucleotiden
met verschillende basen, die met elkaar
verbonden zijn met waterstofbruggen.
De ketens liggen geordend in een
dubbele helix. Er zijn vier verschillende
basen: adenine, thymine, cytosine en
guanine.
De combinatie van een fosfaatgroep, een desoxyribosegroep en één van de vier basen
heet een nucleotide. De twee nucleotideketens worden door de base gekoppeld op een
specifieke manier. Er is alleen een koppeling mogelijk tussen adenine en thymine en
tussen guanine en cytosine. Deze strengen worden bij elkaar gehouden door
waterstofbruggen (H-bruggen). De binding tussen de basen gebeurt door middel van een
waterstofbrug. Tussen G en C zitten drie waterstofbruggen en tussen A en T twee.
Figuur 1.3: Waterstofbruggen tussen de basenparen. Basenparing is specifiek. Guanine en cytosine
kunnen waterstofbruggen met elkaar vormen en adenine en thymine ook. Tussen guanine en cytosine kunnen
er drie waterstofbruggen gevormd worden en tussen adenine en thymine maar twee. De binding tussen
guanine en cytosine is dus sterker dan die tussen adenine en thymine.
5
De waterstofbruggen geven een zwakke, niet covalente binding tussen twee basenparen.
Deze twee basenparen passen als puzzelstukjes in elkaar. Vanwege het aantal
waterstofbruggen wordt het GC-basenpaar minder gemakkelijk verbroken dan het ATbasenpaar. Verbreking van de basenparen kan relatief makkelijk plaatsvinden door
verhitting of door een mechanische kracht. Qua chemische structuur worden de basen
onderverdeeld in twee soorten: guanine en adenine zijn purinebasen, ze hebben het
skelet van purine, dat bestaat uit een 5-ring en een 6-ring. Cytosine en thymine zijn
pyrimidinebasen, ze hebben het skelet van pyrimidine dat bestaat uit enkel een 6-ring.
Zowel het purine- als het pyrimidineskelet bevatten meerdere stikstof-atomen, waarvan
sommige participeren in de vorming van waterstofbruggen. Elk basenpaar bestaat uit een
purine- en een pyrimidinebase.
De nucleotideketen heeft een 3‟-uiteinde en een 5‟uiteinde. Dit betekent dat aan het 3‟-uiteinde van de
nucleotideketen aan de suikergroep 2 –OH groepen
gebonden zijn aan het 3e C-atoom. Het 5‟-uiteinde van de
nucleotideketen is waar aan het suiker een fosfaatgroep
gebonden zit aan het 5e C-atoom. De DNA strengen zijn zo
aan elkaar gekoppeld dat ze in tegengestelde richting
liggen, waarbij 1 streng van 5‟ naar 3‟ ligt en waarbij 1
streng van 3‟ naar 5‟ ligt. Hieronder zie je een voorbeeld
van een stukje DNA:
5‟ -- A G T C C T G A A G G T T C T A G G 3‟
3‟ T C A G G A C T T C C A A G A T C C -- 5‟
Wanneer de volgorde van één streng bekend is kan de
volgorde van de andere streng worden afgeleid. De
Figuur 1.4: Oriëntatie van nucleotideketens in DNA. De twee nucleotideketens liggen in tegengestelde
richting tegenover elkaar. Dit betekent dat de ene keten van 3‟ naar 5‟ ligt en de andere keten van 5‟naar 3‟.
strengen worden daarom ook wel complementair genoemd. De basen volgen elkaar op in
één lange reeks, met elke keer een andere volgorde. Het aflezen van een DNA streng
gebeurt altijd in de richting van 3‟ 5‟. Het aflezen gebeurt aan de hand van codons.
Drie nucleotiden naast elkaar vormen een codon. De reeks van verschillende codons is de
unieke code voor het ontwikkelen van eiwitten.
1.2 TRANSCRIPTIE: VAN DNA NAAR RNA
Transcriptie is (in de genetica) het proces waarbij het DNA van een gen wordt gekopieerd
naar ribonucleïnezuur (RNA) en is de eerste stap in de eiwitsynthese. Tijdens de
transcriptie wordt de code van het DNA afgelezen en omgezet in een RNA code. Voordat
we het gaan hebben over het proces gaan we eerst wat dieper in op de verschillende
onderdelen van de transcriptie.
6
DNA en RNA verschillen in een aantal opzichten van elkaar:
-
De polyesterketen van het RNA bestaat uit ribose in plaats van desoxyribose. De
moleculen van deoxyribose missen een zuurstofatoom ten opzichte van ribose.
-
RNA bevat het nucleotide uracil (U) in plaats van thymine. Uracil kan net als
thymine alleen maar binden met adenine.
-
RNA moleculen zijn enkelstrengs en DNA dubbelstrengs.
Figuur 1.5: Verschillen tussen DNA en RNA moleculen. RNA en DNA lijken erg op elkaar, maar verschillen
op drie punten. RNA bestaat uit een riboseketen en DNA uit een desoxiriboseketen. Verder heeft RNA de base
uracil in plaats van thymine bij DNA. En RNA bestaat niet uit een dubbele helix, maar uit een enkele helix.
De transcriptie start met een stuk DNA dat geactiveerd wordt om afgelezen te worden.
Hierbij bindt het enzym RNA polymerase op een specifieke plaats, herkenbaar aan de
basenvolgorde TAC. De RNA polymerase bindt aan de DNA keten en vouwt zich
eromheen. Het enzym ontwindt het DNA en verbreekt de waterstofbruggen waardoor het
DNA nu enkelstrengs is geworden en tijdelijk toegankelijk is voor andere actieve
componenten van het enzym. Als alle transcriptiefactoren gebonden zijn kan de
transcriptie beginnen. Deze stap van de transcriptie noemen we de initiatie.
Daarna moet de RNA streng verlengd worden. Dit noemen we elongatie. In de celkern
zwerven losse RNA nucleotiden rond die gekoppeld kunnen worden aan de groeiende RNA
streng. Welk nucleotide dit zal zijn wordt steeds bepaald door de tegenoverliggende DNA
nucleotide.één van de DNA strengen fungeert dus als matrijs of template. Het RNA
7
polymerase beweegt zich in de zogenaamde 3‟ 5‟ richting over het DNA. De RNA
streng groeit in de 5‟ 3‟ richting. Tegelijkertijd wordt het RNA aan de achterkant weer
gescheiden van het DNA.
Wanneer het hele gen gekopieerd is en de RNA polymerase weer een specifieke
sequentie (stopcodon) tegenkomt stopt de RNA synthese. Dit is de terminatie.
Initiatie
Elongatie
Terminatie
Figuur 1.6: Transcriptie in drie stappen (RNAP is RNA polymerase). In de initiatie binden RNA
Polymerase en andere transcriptiefactoren en start de transcriptie. Daarna wordt de mRNA strang verlengd. Dit
is de elongatie. Als de RNA Polymerase een stopcoon tegenkomt wordt de transcriptie beëindigd en laten de
transcriptiefactoren en het mRNA los. Dit is de terminatie.
Het RNA dat nu gesynthetiseerd is wordt ook wel het pre-mRNA genoemd. Het bevat
stukken die wel tot expressie komen (exonen) maar ook stukjes die niet tot expressie
komen (intronen). Door middel van splicing (knippen) worden de intronen verwijderd.
Enzymen plakken de exonen achter elkaar waardoor alleen het gedeelte overblijft dat wel
tot expressie kan komen. Ook wordt de mRNA streng voorzien van een 5‟-methylcap en
een poly-A staart. Een 5‟-methylcap is een methylgroep aan het eerste nucleotide,
waaraan eiwitten kunnen binden. Een poly-A staart is een keten adenines aan het 3‟
uiteinde van het mRNA. Deze bewerkingen zorgen ervoor dat:
de twee uiteinden van het mRNA kunnen worden onderscheiden
het mRNA afgelezen kan worden
transporteiwitten kunnen binden en het mRNA uit de kern kunnen transporteren
het mRNA stabieler wordt
er een controle is dat het mRNA molecuul af is
8
Figuur 1.7: splicing. Het bewerken van het RNA begint met toevoegen aan het 5‟-einde een cap wat
bestaat uit een 7-methylguanosinetrifosfaat groep. Aan het 3‟-einde bevindt zich een knipsignaal. Hier zal het
mRNA geknipt worden en tevens een poly(A)staart aan toegevoegd worden. Als laatste stap worden de
intronen verwijderd uit het pre-mRNA. Alle exonen worden vervolgens aan elkaar geplakt zodat alleen de
gedeelten die afgelezen moeten worden bij elkaar zitten.
Splicing wordt gedaan om van DNA via mRNA naar een functioneel eiwit te komen. De
bouwstenen van een eiwit noemen we aminozuren. Als laatste stap gaan we deze les
kijken hoe een mRNA-streng vertaald kan worden in een reeks aminozuren, dit noemen
we translatie.
1.3 TRANSLATIE: VAN RNA NAAR EIWIT
Splicing wordt gedaan om van DNA via mRNA naar een functioneel eiwit te komen. De
bouwstenen van een eiwit noemen we aminozuren en hier gaan we het volgende les over
hebben. Als laatste stap gaan we deze les kijken hoe een mRNA-streng vertaald kan
worden in een reeks aminozuren, dit noemen we translatie. Van DNA bestaat er maar
een soort, terwijl er van RNA meerdere soorten zijn. Naast mRNA kennen we Transport /
transfer RNA (tRNA). tRNA speelt tijdens de translatie een rol als drager van aminozuren.
Er zijn 20 verschillende tRNA moleculen (omdat er 20 verschillende eiwit-bouwstenen
zijn), elk gespecialiseerd in het vervoeren van een bepaald aminozuur. Ribosomen zijn
vervolgens in staat tijdens de translatie mRNA af te lezen en de aminozuren van het eiwit
aan elkaar te koppelen.
9
1.4 TRNA
Door het translatieproces worden deze aminozuren aan elkaar gebonden. Het tRNA (in
het nederlands „transport RNA‟ en in het engels „transfer RNA‟) speelt daar een
belangrijke rol in. Het tRNA bindt aan een bepaald aminozuur. Wanneer de code van dit
aminozuur overeenkomt met het mRNA zal deze tRNA het aminozuur naar de groeiende
polypeptideketen brengen.
Het tRNA molecuul heeft een aparte
structuur. Anders dan andere RNA
moleculen
is
het
grotendeels
dubbelstrengs, en bezit dus inwendige
waterstofbruggen. Deze zorgen voor een
typische secundaire structuur, die ook
wel
het
klaverbladstructuur
wordt
genoemd. In werkelijkheid wordt deze
structuur nog meer gevouwen waardoor
het geheel een L-vorm krijgt. Een tRNA
molecuul bezit 3 karakteristieke loops.
De linkerarm is de D arm en de
rechterarm is de T arm. De onderste arm
wordt de anti-codon arm genoemd. Een
anticodon is complementair aan een
codon,
bij
een
UAG
codon
zou
bijvoorbeeld een AUC anti-codon horen.
De anti-codon arm bevat een anti-codon
en kan daarmee aan een codon van het
mRNA binden. Dan is er nog de acceptor
stam, waar een aminozuur aan gebonden
kan worden (hij kan een aminozuur
„accepteren‟). Dit is het aminozuur dat bij
het mRNA-codon hoort.
figuur 5.2: Het tRNA molecuul. tRNA heeft een
klaverbladstructuur met drie „armen‟. De anti-codon
arm bindt aan het mRNA, waarna de acceptor stam
het nieuw gevormde aminozuur kan transporteren. Zo
kan het tRNA controleren of de code van het
aminozuur overeenkomt met het mRNA.
Het translatieproces kan niet beginnen voordat een specifieke aminozuur gebonden wordt
aan een tRNA molecuul. Het binden van zo‟n aminozuur aan het tRNA wordt laden
genoemd. Dit proces kost energie, die verkregen wordt van ATP. Er zijn in totaal twintig
enzymen die deze formatie mogelijk maken (één voor elk aminozuur).
1.5 RIBOSOMEN
Naast tRNA moleculen spelen ook ribosomen een belangrijke rol bij het translatieproces.
Nadat het mRNA de celkern verlaat wordt het gebonden aan de ribosomen. Ieder
ribosoom kan maar één mRNA-streng binden, wel kunnen er meerdere ribosomen op één
mRNA-streng actief zijn. Ribosomen bestaan uit twee subeenheden, een groot en een
klein deel. Het ribosoom heeft vier bindingsplaatsen: 1 voor de mRNA-streng en 3 voor
tRNA moleculen. Één tRNA molecuul zit in de wacht op de A-plaats (acceptorplaats) van
het ribosoom. Daarnaast zit één tRNA-molecuul op de P-plaats (werkplaats), die zijn
aminozuur aan de groeiende aminozuurketen koppelt. En achter de P-plaats zit de Eplaats (exitplaats), waar een tRNA molecuul weer wordt losgekoppeld van een ribosoom.
10
1.6 HET TRANSLATIEPROCES
Het translatieproces omvat 3 stappen:
initiatie (start), elongatie (verlenging)
en terminatie (eind).
Initiatie
Translatie begint met het uiteenvallen
van
het
ribosoom
in
twee
subeenheden. Het mRNA bindt aan het
5‟ uiteinde van het kleine subdeel. Het
mRNA verschuift totdat het bij het
startcodon
(AUG)
aankomt
(zie
codontabel in les 4). Het tRNA
molecuul met het anti-codon UAC
bindt aan het mRNA. Vervolgens bindt
het grote ribosoomdeel en kan de
translatie beginnen.
Elongatie
Na binding van het grote subdeel kan
de
verlenging
beginnen.
Een
verlengingsfactor brengt het juiste
tRNA op plaats A naast het al
aanwezige tRNA molecuul (op plaats
P) in het ribosoom.
Enzymen
koppelen het eerste aminozuur los van
het tRNA molecuul waardoor een
polypeptide
ontstaat.
De
peptidebinding tussen de aminozuren
vindt plaats tussen de zuurgroep van
het aminozuur op plaats P en de
aminegroep van het aminozuur op
plaats
A.
Een
tweede
verlengingsfactor verwijdert het “lege”
tRNA molecuul waardoor het ribosoom
drie nucleotiden in de richting van het
mRNA
opschuift
(translocatie:
verschuiving van het ribosoom over
het mRNA door middel van een
verlengingsfactor).
Terminatie
De translatie stopt wanneer het
ribosoom één van de stopcodons
(UAA, UAG of UGA) tegenkomt. Er
bindt een ontkoppelingsfactor aan het
Figuur 1.8: Translatie proces
11
mRNA. Waardoor achtereenvolgens de voltooide polypeptideketen, het laatste tRNA
molecuul en het mRNA van het ribosoom worden ontkoppeld. Het ribosoom valt weer
uiteen in twee subdelen waardoor het klaar is voor een nieuwe cyclus.
OPDRACHTEN BIJ LES 1
1. Waaruit bestaat een DNA-molecuul?
2. DNA is een zuur. Door welk deel van het DNA komt dat?
3. Hieronder is een DNA-molecuul afgebeeld. Een DNA-molecuul is opgebouwd uit
nucleotiden met onder andere vier verschillende basen: A, T, G, en C. Deze basen
komen in het DNA-molecuul paarsgewijs voor.
© Biodoen
In welke afbeelding is een nucleotide weergegeven? En wat zie je in de andere
afbeeldingen?
(A)
(B)
(C)
(D)
4. Wat zijn de belangrijkste verschillen tussen een RNA en een DNA molecuul?
12
5. Voordat de transcriptie van RNA begint, despiraliseren ongeveer de eerste 20
basenparen van het DNA-molecuul. Dit gebeurt met behulp van een `RNApolymerase`enzym dat zich aan één van de twee DNA-strengen hecht. De DNAstreng die met behulp van een `RNA-polymerase`enzym wordt gekopieerd, wordt
de template streng genoemd.
r = RNA-polymerase
i = initiatieplaats
t = terminatieplaats
Kruis de juiste antwoorden aan:
De leesrichting van de template streng van het DNA-molecuul loopt van
5‟
naar 3‟
3‟ naar 5‟ en is de
parallelle
antiparallelle streng van het DNAmolecuul
De leesrichting van de gekopieërde RNA-streng loopt van
3‟ naar 5‟
naar 3‟ en is
parallel
antiparallel aan die van de template DNA-streng.
5‟
6. Hieronder zie je een stuk van een DNA fragment. Geef de nucleotidenvolgorde en
de leesrichting aan van het stukje RNA dat ontstaat na transcriptie.
3‟ G A T C A T T C T A G 5‟
5‟ C T A G T A A G A T C 3‟
13
7.
Label de armen van het tRNA molecuul
8. Hoeveel soorten tRNA moleculen zijn er? Waarom?
9. Door mutatie verandert een codon in het mRNA van 5‟ CAG 3‟ naar 5‟ GAG 3‟.
Welke gevolgen heeft dit voor de eiwitsynthese?
10. Waar in de cel vindt transcriptie plaats? En waar translatie?
Extra informatie over deze les:
Wil je meer weten over één van de volgende onderwerpen bekijk dan de filmpjes!
http://www.bioplek.org/animaties/moleculaire_genetica/transcriptie.html transcriptie
http://www.youtube.com/watch?v=vJSmZ3DsntU transcriptie
http://www.youtube.com/watch?v=YjWuVrzvZYA mRNA processing
http://www.youtube.com/watch?v=FVuAwBGw_pQ mRNA splicing
Wil je het translatieproces nog een rustig bekijken, ga dan naar deze filmpjes:
http://www.youtube.com/watch?v=5bLEDd-PSTQ
http://ihome.cuhk.edu.hk/~z045513/virtuallab/animation/Translation.html
14
LES 2: EIWITTEN: AMINOZUREN EN POSTTRANSLATIONELE
MODIFICATIES
2.1 CODONS
Zoals in les 1 is uitgelegd bestaat mRNA net als DNA uit nucleotiden. De basen van RNA
zijn adenine (A), cytosine (C), guanine (G) en uracil (U). Een set van drie nucleotiden
noemen we een codon, omdat het codeert voor één aminozuur van een eiwit.
mRNA met de sequentie ACU-GGC-AUC
staat
bijvoorbeeld
voor
de
aminozuurketen
threonine-glycineisoleucine. Om zo‟n mRNA sequentie te
vertalen naar een aminozuursequentie
gebruik je een codontabel (zie figuur
4.2). Het eerste nucleotide van een
codon lees je links af, de tweede boven
en de derde rechts. Je kunt dan het
bijbehorende aminozuur aflezen in de
tabel. Er bestaan 64 verschillende
codons. Er zijn namelijk 43 verschillende
manieren om de 4 letters te combineren
in een set van 3. Toch zijn er maar 20
verschillende aminozuren. Dat betekent
dat er in sommige gevallen meerdere
codons
voor
hetzelfde
aminozuur
coderen. Je ziet dan ook dat sommige
aminozuren bij meerdere codons in te
tabel voorkomen. Verder zie je drie keer
STOP staan in de tabel. Dit staat voor
stopcodon, de plaats waar de translatie
translatie start namelijk altijd bij het codon
dan ook altijd een methionine.
Figuur 2.1: Codontabel. De basen worden afgelezen
vanuit de 5‟ naar de 3‟ richting. Met deze tabel kunnen
mRNA-codons vertaald worden naar aminozuren. De
eerste base van het codon staat links in de tabel, de
tweede boven en de derde rechts. Naast de 20
aminozuren zijn ook de start- en stopcodons
weergegeven. AUG is de startcodon (waarbij de
translatie start) en UAA/UGA/UAG zijn de stopcodons
(waarbij de translatie stopt).
stopt. Er bestaat ook een startcodon, de
AUG. Het eerste aminozuur van een eiwit is
2.2 AMINOZUREN
Elk aminozuur bestaat uit een koolstofatoom met een aminogroep, een waterstof, een
zuurgroep en een restgroep. De restgroepen zijn voor ieder aminozuur anders en bepalen
de vorm van het eiwit. Op basis van deze restgroepen, kun je de aminozuren indelen in 5
groepen: de positief geladen aminozuren, de negatief geladen aminozuren, de polaire
aminozuren, de apolaire aminozuren en de aromatische aminozuren. Let op: de lading
van de aminozuren hangt af van de pH van de omgeving! Bij een lage pH (zuur milieu)
worden aminozuren meer positief geladen, omdat ze een H+ kunnen opnemen. Bij een
hoge pH (basisch milieu) worden de aminozuren meer negatief geladen, omdat ze een H+
afstaan aan hun omgeving. De lading wordt gegeven voor pH=7, aangezien de pH op
veel plekken in ons lichaam rond de 7 ligt. De polaire aminozuren zijn niet geladen, maar
15
hebben een ongelijke ladingsverdeling. Dat wil zeggen dat een deel van de restgroep de
elektronen wat meer aantrekt en daardoor negatief geladen wordt. Automatisch zitten
die elektronen dan wat minder in een ander deel van de restgroep en wordt die een
beetje positief geladen. Apolaire aminozuren hebben geen lading en een gelijke
ladingsverdeling. Die zullen dus niet onderhevig zijn aan krachten die met lading te
maken hebben. Dan is er nog een laatste groep aminozuren, waarvan de restgroep een
ringstructuur bevat. We noemen deze aminozuren „aromatisch‟. Hun restgroepen zijn erg
groot en zijn daarom ook erg belangrijk voor de eiwitstructuur.
16
Polaire restgroepen
Glycine
Alanine
Apolaire restgroepen
Serine
Valine
Threonine
Cysteïne
Proline
Leucine
Isoleucine
Asparagine
Methionine
Glutamine
Positief geladen restgroepen
Negatief geladen restgroepen
Aparaginezuur
Glutaminezuur
Histidine
Lysine
Arginine
Aromatische restgroepen
Algemene notatie aminozuren
zuurgroep
Aminogroep
Fenylalanine
Tyrosine
R
Waterstofatoom
Restgroep
Tryptofaan
17
Figuur 2.2: Aminozuren. De 20 aminozuren zijn hier onderverdeeld op basis van hun restgroep. Er zijn
aminozuren met een apolaire restgroep, met een polaire restgroep, met een aromatische restgroep, met een
positief
restgroep zijn
en met
geladen
Naast geladen
deze groepen
er een
ooknegatief
nog een
paarrestgroep.
losse aminozuren die opvallen. Glycine valt
bijvoorbeeld op, omdat hij de kleinste is. De restgroep bestaat uit alleen één
waterstofatoom. Methionine en cysteine bevatten allebei een zwavelatoom. Deze kunnen
zwavelbruggen vormen (hier komen we in les 6 op terug), die ook belangrijk zijn voor de
eiwitstructuur. Zo‟n brug zorgt voor veel stevigheid, en kan alleen gevormd worden als
de zwavelatomen dicht genoeg bij elkaar in de buurt liggen. Proline is ook een bijzonder
aminozuur, want het zorgt voor een knik in de backbone van het eiwit. Normaal
gesproken zitten de aminogroep en de zuurgroep op één lijn en wordt de backbone dus
een lange rechte slinger. Bij proline is dat niet zo, en dat is handig als het eiwit in een
bocht moet liggen.
2.3 PEPTIDEBINDINGEN
Met de aminogroep en de zuurgroep kunnen ze peptidebingen maken met elkaar. Dit
gaat door middel van een condensatiereactie. Hieronder zie je weergegeven hoe zo‟n
reactie verloopt. Aan de linkerkant van de reactievergelijking staan twee alanine
aminozuren. De zuurgeoep van het ene aminozuur wordt aan de aminogroep van het
andere aminozuur gekoppeld. Hierbij wordt een watermolecuul afgesplitst. Dit
watermolecuul bestaat uit OH- van de zuurgroep en een H+ van de aminogroep. Een
reeks peptidebindingen vormt de backbone van het eiwit. De zuurgroepen en
mainogroepen vormen een lange keten, waar de waterstofatomen (erg klein) en de
restgroepen uitsteken.
Figuur 2.3: Peptidebinding. De zuurgroep van het ene aminozuur wordt gekoppeld aan de aminogroep van het
andere aminozuur. Hierbij komt een watermolecuul vrij. We noemen dit een condensatiereactie.
18
2.4 POSTTRANSLATIONELE M ODIFICATIES
Als de complete aminozuursequentie van een eiwit is aangemaakt op een ribosoom, is
het nog niet af. Om het eiwit functioneel te maken moeten er nog twee dingen gebeuren.
Ten eerste moet het eiwit nog gevouwen worden. Daar gaan we het devolgende les nog
uitgebreid over hebben. Maar voor of na het vouwen kunnen er nog wat kleine
veranderingen plaats vinden. Er kan bijvoorbeeld nog een aantal groepen aan de
aminozuren worden gebonden, of er kan een stukje uit de aminozuursequentie worden
geknipt. Al deze dingen samen noem je posttranslationele modificaties. Een moeilijk
woord, maar wel logisch: het zijn veranderingen (modificaties) nadat het eiwit gevormd
is (na de translatie). Deze veranderingen zijn reversibel, en kunnen dus ook weer
ongedaan gemaakt worden. Voorbeelden van groepen die aan de aminozuren worden
gehangen zijn: fosfaatgroepen, suikergroepen, vetgroepen en andere organische stoffen.
Om deze groepen aan een aminozuur te kunnen binden zijn er enzymen nodig. Enzymen
zijn eiwitten die werken als een biologische katalysator. Zij zorgen ervoor dat de reactie
tussen het aminozuur en de extra groep kan verlopen.
Bij fosforylering van een eiwit wordt er een PO43- groep aan een bepaald aminozuur
gebonden. Zo‟n fosfaatgroep is erg groot en hydrofiel, waardoor bindingen in en tussen
eiwitten niet meer mogelijk zijn of er juist nieuwe gemaakt kunnen worden. Het eiwit
verandert dan van vorm en daarmee verandert ook de werking. Meestal zorgen
fosforyleringen voor een activering van het eiwit. Bij fosforyleringen zijn ook enzymen
betrokken, zogenaamde kinases. Een fosfaatgroep kan weer worden verwijderd met
behulp van fosfatases. Dan is het eiwit weer geïnactiveerd.
Figuur 2.4: Fosfaatgroep. „R‟ staat voor „Rest‟ en is in dit geval het eiwit waar de fosfaatgroep aan gebonden
is. De fosfaatgroep zelf bestaat ui één fosforatoom en vier zuurstofatomen. Fosfor is een vijfwaardig atoom en
kan dus vijf bindingen aangaan. Er worden daarom drie enkele bindingen met zuurstofatomen gevormd en één
dubbele binding. Zuurstof is een tweewaardig atoom. De zuurstofatomen die niet dubbel gebonden zijn en geen
binding hebben met het eiwit (R) zijn daarom een beetje negatief geladen (polair). Deze zuurstofatomen binden
daarom snel een waterstofatoom, die een beetje positief geladen is.
Als er een suikergroep aan een eiwit gebonden wordt, noem je dat glycosylering. De
meeste eiwitten worden direct na de translatie geglycosyleerd, aangezien dat vaak
essentieel is voor een goede vouwing. Ze zorgen er dus voor dat het eiwit netjes
gevouwen blijft, en niet weer terug valt in de ongevouwen vorm. Belangrijke
suikergroepen
bij
mensen
zijn
bijvoorbeeld
N-acetylglucosaminegroepen,
mannosegroepen en glucosegroepen. Hieronder zijn deze groepen weergegeven. Valt
jullie iets op aan de notatie? De notatie is in alle drie de gevallen anders. Je kunt zien dat
het allemaal zeshoeken zijn. Op één van de hoeken zit een zuurstofatoom, maar op de
andere vijf hoeken is er niets aangegeven. Voor deze notatie hebben we namelijk
afgesproken dat we alle koolstofatomen in de ring niet weergeven. Elke hoek is dus een
koolstofatoom, tenzij anders aangegeven. Ook de waterstofatomen worden niet
19
weergegeven in deze notatie. De verbindingen naar de zijgroepen hebben een bepaalde
richting gekregen.
Figuur 2.5: Een N-acetylglucosaminegroep (links), mannosegroep (midden) en een glucosegroep
(rechts). De drie groepen zijn in verschillende notaties weergegeven. Het zijn allemaal zeshoeken met op één
hoek een zuurstofatoom. Op de andere hoeken zitten koolstofatomen, maar die worden in deze notaties niet
weergegeven (ook de gebonden waterstofatomen niet!). De oriëntatie van de groepen rondom de ring is in elke
notatie anders weergegeven. (Bekijk ook tabel 67A1 van Binas)
Meestal bestaat een glycosylering uit een keten van reacties, waarbij meerdere
suikergroepen aan een aminozuur worden gebonden. De eerste suikergroep zit vast aan
het aminozuur, en de volgende groepen worden dan aan die eerste vastgekoppeld. Dit
gaat via een condensatiereactie, waarbij een watermolecuul wordt afgestoten (figuur
6.4). Twee gekoppelde suikergroepen heten een disaccharide en meer dan twee
gekoppelde suikergroepen heten een polysaccharide. Om deze disaccharides en
polysacharides weer af te breken tot losse suikergroepen moet water worden
toegevoegd. We noemen deze reactie een hydrolysereactie (lees figuur 3 van rechts naar
links). Een hydrolysereactie is in feite het omgekeerde van een condensatiereactie.
s
Figuur 2.6: Vorming van een disaccharide door middel van een condensatiereactie. Twee
glucosegroepen tot maltose . Bij de condensatiereactie wordt er een watermolecuul afgesplitst. De
glucosegrepen worden met een zuurstofatoom verbonden en vormen nu een disacharide. In deze notatie
worden wel alle atomen weergegeven, vergelijk het maar eens met de glucosegroep uit figuur 2.
20
Sommige eiwitten hebben een functie op een
membraan. Je kunt hierbij denken aan ionkanalen
of receptoren. Het is dan vaak nodig om een eiwit
wat hydrofober te maken, zodat het zich thuis voelt
in het hydrofobe membraan. Dit wordt gedaan door
een aantal vetgroepen aan het eiwit te binden.
Vetgroepen zijn moleculen met een lange
koolstofketen.
Zo‟n
keten
kan
geen
waterstofbruggen vormen met water is daarom erg
hydrofoob. Door deze koolstofketens te verankeren
in het membraan blijft het eiwit op zijn plek zitten.
Deze „lipoproteïnen‟ worden niet alleen gebruikt
voor membranen, maar bijvoorbeeld ook voor
vettransport.
Figuur 2.7: Eiwit met suikergroepen en een vetgroep verankerd in het membraan. Links boven is het
eiwit weergegeven . Er zitten meerdere groepen aan het eiwit gebonden, met aan het uiteinde een vetgroep. De
vetgroep bevat twee koolstofketens die naar binnen in het membraan steken. Het membraan is aan de
buitenkant hydrofiel (de bolletjes) en aan de binnenkant hydrofoob (de staarten). Met behulp van de
koolstofketens zit het eiwit vast aan het membraan.
Naast suikergroepen en vetgroepen zijn er ook andere organische stoffen die gebruikt
kunnen worden voor posttranslationele modificaties. Acetylgroepen zijn bijvoorbeeld van
Figuur 4: eiwit met vetgroepen, verankerd in een membraan (GPI anchor)
groot belang voor histonen. Histonen zijn eiwitten waar het DNA in de kern omheen is
gerold. DNA en histonen samen vormen uiteindelijk
chromatine, het bestanddeel van chromosomen. Belangrijk in
deze les is echten dat histonen normaalgesproken een beetje
positief geladen zijn, waardoor ze binden aan de negatieve
fosfaatgroepen van het DNA (les 1). De acetylgroepen zijn
een beetje negatief geladen, dus als een histon geacetyleerd
wordt dan wordt zijn positieve lading opgeheven. Hierdoor zal Figuur 2.8: Acetylgroep.
In deze notatie zijn de Cde binding met het DNA minder sterk worden en laat het DNA atomen weggelaten. Eigenlijk
dus een beetje los van het eiwit. Dit is erg belangrijk bij staat er CH3-CO-R, waarbij R
transcriptie (les 2), want alleen dit lossere DNA kan in dit geval het eiwit is.
overgeschreven worden naar RNA.
21
Figuur 2.9: Opbouw van chromosomen. Onderaan zie je de dubbele helix van het DNA. Deze dubbele helix
wordt om histonen gerold. Acht histonen met DNA eromheen vormen een nucleosoom. Ook deze nucleosomen
worden weer in een soort spiraalvorm opgerold. Deze spiraal wordt weer opgerold tot een chromosoom, die zich
Figuur
opbouw
chromosomen
in de celkern (nucleus) bevindt.
Op 6:
deze
maniervan
kunnen
wij ongeveer 2 meter DNA per cel opslaan!
Door posttranslationele modificaties kun je van dezelfde DNA-sequentie verschillende
eiwitten maken. Ook kun je door middel van deze modificaties de transcriptie van DNA
reguleren (denk aan de histonen, maar ook aan fosforylering en dus activatie van
transcriptiefactoren). Dit verklaart waarom cellen verschillende functies kunnen hebben,
terwijl ze allemaal hetzelfde DNA bevatten.
OPDRACHTEN BIJ LES 2
1. Een DNA-sequentie kun je vertalen naar aminozuursequentie. Leg uit welke stappen
je hiervoor moet ondernemen.
2. Kun je ook een aminozuursequentie van een eiwit terug vertalen naar de DNAsequentie?
3. Geef aan waarom elk gesynthetiseerd eiwit begint met het aminozuur `methionine`.
4. Hieronder is een DNA keten weergegeven. Vertaal deze sequentie naar een mRNAsequentie en vervolgens naar een aminozuursequentie. Vergeet hierbij niet de 3‟ en
5‟ uiteinden weer te geven. Gebruik voor deze opdracht onder andere de tabel met
de genetische code (Binas 70E)
3‟ --- A G T C A G T C T A G C C T A T G A A C C C T T G A C T A T A T T T A G T --- 5‟
22
5. Teken twee aminozuren die met een peptidebinding aan elkaar zitten. Geef de
restgroepen aan met een „R‟. Deze twee aminozuren zitten in een langere keten,
dus geef aan beide uiteinden aan dat de keten nog verder gaat.
6. Leg uit wat een posttranslationele modificatie is. En waar in de cel vinden
posttranslationele modificaties plaats?
7. Hieronder ze je de structuurformule van sacharose (kristalsuiker). Geef de
reactievergelijking van de hydrolyse van sacharose in structuurformules en
molecuulformules. Krijg je twee dezelfde suikers?
8. Maakt een suikergroep het aminozuur hydrofieler of hydrofober? Hoe komt dat?
9. Zie de mannosegroep in figuur 2.3: 1 OH-groep zit iets verder van de ring af dan de
andere. Welk atoom zit er nog tussen de OH-groep en de ring? En met welke
atomen staat deze in verbinding?
10. Vetgroepen worden gebruikt om een eiwit hydrofober te maken. Waarom zijn de
lange koolstofketens eigenlijk hydrofoob?
11. Als histonen geacetyleerd worden gaat het DNA iets losser om de histonen zitten.
Verloopt de transcriptie makkelijker of moeilijker bij dit lossere DNA? En hoe komt
dat?
23
LES 3: EIWITTEN: EIW ITVOUWING EN MUTATIES
3.1 EIWITVOUWING
Een keten aminozuren is onderhevig aan veel krachten. Positieve ladingen trekken
negatieve ladingen aan, hydrofobe delen zullen zich het liefst van het water afkeren en
de hydrofiele delen willen zich juist in een waterige omgeving bevinden. Onder invloed
van dit soort krachten zal het eiwit zich gaan vouwen.
In les 2 heb je gezien dat er 20 verschillende aminozuren bestaan. Ze hebben allemaal
een aminogroep en een zuurgroep, waarmee ze petidebindingen kunnen vormen. Een
keten van peptidebindingen noemen we de „backbone‟ van het eiwit. Deze ervaart vooral
de aantrekkingskrachten van waterstof- en zuurstofatomen. Dit resulteert in
waterstofbruggen. De aminozuren hebben ook ieder een eigen restgroep. Sommige van
deze restgroepen zijn geladen, hydrofiel of juist hydrofoob. Een aminozuur met een
negatief geladen restgroep zal een aminozuur met een positief geladen restgroep
aantrekken. De restgroepen kunnen dan zoutbruggen vormen. Hydrofiele restgroepen
steken graag naar buiten en zullen ook graag in de omgeving van andere hydrofiele
restgroepen zitten, zodat ze waterstofbruggen kunnen vormen. Hydrofobe restgroepen
zullen zich proberen te clusteren, zodat ze zich een beetje kunnen afschermen tegen de
waterige omgeving. Bij de hydrofiele en hydrofobe interacties zijn de posttranslationele
modificaties natuurlijk ook van belang. Denk maar eens aan de vetgroepen en de
acetylgroepen. Verder zijn er nog restgroepen die zwavel-atomen bevatten. Deze kunnen
zwavelbruggen vormen wat een covalente binding is (-SH HS-, denk aan redoxreacties).
In tabel 1 staat een overzicht van de belangrijkste interacties die een rol spelen bij de
eiwitvouwing.
Backbone
Waterstofbruggen
tussen H (op N) en O
Restgroepen
Waterstofbruggen
Zoutbruggen
Zwavelbruggen
Hydrofobe interacties
bij hydrofiele restgroepen
bij geladen restgroepen
bij restgroepen met zwavel
Bij hydrofobe restgroepen
Tabel 3.1: Aminozuurinteracties bij eiwitvouwing. In de backbone (keten peptidebindingen) kunnen
waterstofbruggen gevormd worden tussen een zuurstofatoom van het ene aminozuur en een waterstofatoom
van het andere aminozuur. Ook hydrofiele restgroepen kunnen waterstofbruggen vormen met elkaar. Verder
kunnen geladen restgroepen zoutbruggen vormen, restgroepen van cysteïnes zwavelbruggen en kunnen
hydrofobe restgroepen hydrofobe interacties met elkaar hebben.
De belangrijkste factor voor de stabiliteit van een eiwit zijn de waterstofbruggen. Het
eiwit zal daarom zo gevouwen worden dat deze bruggen optimaal gevormd kunnen
worden. Deze waterstofbruggen worden voornamelijk gevormd tussen atomen van de
backbone, en die is in ieder eiwit hetzelfde. Je ziet dan ook een aantal structuren die
gunstig zijn voor de waterstofbruggen in alle eiwitten terugkomen. Ten eerste de α-helix,
waarbij de backbone een soort slinger vormt. De restgroepen van de aminozuren steken
allemaal naar buiten. In deze vorm kunnen alle zuurstofatomen van de backbone een
waterstofbrug vormen. Dit doen ze met een waterstofatoom (van een stikstofatoom) één
slinger lager. De tweede structuur is de β-sheet (heet ook wel β-plaat), waarbij de
backbone heen en weer slingert tot een soort plaat. De restgroepen van de aminozuren
zitten ongeveer in het vlak van de plaat en steken om en om naar rechts en naar links.
Bij deze structuur kan de helft van de zuurstofatomen uit de backbone een waterstofbrug
vormen. En om de α-helices en de β-sheets met elkaar te verbinden zijn er turns, een
24
soort kabels. De verschillen tussen eiwitten zitten vooral in de hoeveelheid van deze
structuren en de manier waarop ze tegen elkaar aan liggen.
A
A
B
C
Figuur 3.1: Secundaire structuren (α-helix en β-sheet) en tertiare structuur (rechts). Bij de α-helix
ligt de backbone in een spiraalvorm en steken de restgroepen naar buiten (A). De β-sheet is een soort plaat,
waarin de backbone zigzaggend heen en weer gaat. De restgroepen liggen in het oppervlak van de plaat (B).
De tertiaire structuur aan de rechterkant bestaat uit α-helices, β-sheets en turns (C).
Figuur 3.2: Waterstofbruggen in de α-helix (links) en β-sheet
(rechts). Links is de α-helix weergegeven met alle atomen van de
backbone. Ook zijn de waterstofbruggen getekend, die ervoor
zorgend at de helix zijn vorm behoudt. De restgroepen steken
allemaal naar buiten. In de β-sheet liggen de aminozuurketens
naast elkaar. De Waterstofbruggen zorgen ook hier voor de
stevigheid en de restgroepen steken in het vlak van de sheet.
We noemen de keten aminozuren de primaire structuur van een eiwit, en de α-helices, βsheets en turns de secundaire structuren. Als je kijkt naar de ruimtelijke ordening van
deze secundaire structuren ten opzichte van elkaar (figuur 6.8 rechts), noem je dat de
tertiare structuur van een eiwit. En mocht het eiwit bestaan uit meerdere subunits, dan
noem je de manier waarop deze subunits tegen elkaar aan liggen de quaternaire
structuur.
3.2 TRANSPORT
De translatie, dus de vorming van het eiwit, vindt plaats op een ribosoom op het ruwe
Endoplasmatisch Reticulum (ER). Dit is een membranenstelsel in het cytoplasma, en de
eiwitten zullen dan ook na hun synthese in het cytoplasma terecht komen. Veel eiwitten
moeten daar hun functie uitoefenen, maar er zijn ook eiwitten die hun functie ergens
anders in de cel of buiten de cel uitoefenen. Die eiwitten moeten dan eerst nog naar de
25
goede plaats getransporteerd worden. Maar hoe weet een eiwit nou waar hij heen moet?
Daar kan zo‟n eiwit bijvoorbeeld een signaalsequentie voor gebruiken. Die bestaat
meestal uit de eerste paar aminozuren van het eiwit, en kan herkend worden door
transporteiwitten. Deze transporteiwitten zullen aan het nieuwe eiwit binden en hem
meenemen naar de goede locatie. Je kunt die signaalsequentie dus zien als een soort
treinkaartje, en de transporteiwitten als de trein.
Een andere manier van transport is via posttranslationele modificaties. Glycosylering
heeft bijvoorbeeld veel met transport te maken. De suikergroep aan het eiwit heeft dan
dezelfde functie als een signaalsequentie. Hij kan herkend worden door andere
(transport)eiwitten, die ervoor zorgen dat het eiwit op de goede plek terecht komt. De
suikergroep van het eiwit wordt gebonden aan de transporteiwitten.
De eerste stap in het transport van eiwitten is transport van het ER naar het
golgisysteem. Ook het golgisysteem bestaat uit membranen en je kunt het zien als een
soort postorderbedrijf. Hier worden de transportlabels (signaalsequenties of
suikergroepen) herkend en worden de eiwitten in de goede transportblaasjes gestopt.
Dan komt de tweede stap van het eiwittransport: transport van het golgisysteem naar
een ander organel in de cel. Meestal wordt het eiwit dan naar zijn eindbestemming
gebracht, waar het transportblaasje versmelt met het membraan van het betreffende
organel. Zo komt het eiwit dan in dat organel terecht. Als het eiwit zijn functie buiten de
cel uit moet oefenen zal het blaasje naar het celmembraan gaan, en daarmee
versmelten. Dit proces noemen we exocytose.
Bij het transport van eiwitten zijn veel enzymen betrokken. Er zitten bijvoorbeeld
enzymen op het membraan van het golgisysteem die de transportlabels kunnen
herkennen. Deze zorgen ervoor dat de eiwitten in de goede transportblaasjes terecht
komen.
Figuur 3.2: Transport van eiwitten. Het mRNA wordt naar het ruwe endoplasmatisch reticulum
getransporteerd, waar het met behulp van een ribosoom vertaald wordt naar een eiwit. Na eventuele
posttranslationele modificaties en eiwitvouwing wordt het eiwit naar het golgisysteem getransporteerd. Hier
worden de transportlabels herkend en wordt elk eiwit naar de goede plaats in de cel gebracht in een
transportblaasje.
26
3.3 RETINITIS PIGMENTOSA
We hebben tot nu gezien hoe een DNA sequentie uiteindelijk resulteert in een functioneel
eiwit. Het DNA wordt eerst afgeschreven naar RNA (transcriptie). Dan wordt het RNA
vertaald naar een eiwit (translatie), en wordt dit eiwit gemodificeerd en gevouwen. Pas
dan heeft een eiwit zijn functionele vorm. We hebben net ook gezien dat het eiwit na
vouwing naar de goede plek in de cel wordt getransporteerd, zodat het eiwit daar zijn
functie kan vervullen.
Je kunt je voorstellen dat een foutje in het DNA grote gevolgen kan hebben voor het
functioneren van een eiwit, en dus voor het functioneren van ons lichaam. Na gaan we
een voorbeeld hiervan bekijken: de ziekte Retinitis Pigmentosa.
Retinitis Pigmentosa (RP) is een verzamelnaam voor afwijkingen aan het netvlies (in het
latijn retina). Het komt bij ongeveer 1 op de 4000 mensen voor. Patiënten met RP gaan
steeds minder goed zien. Ze kunnen last krijgen van nachtblindheid, verblinding en een
kokervisus. Bij nachtblindheid kun je in het donker alleen nog maar de lichtpunten zien.
Verblinding houdt in dat je wanneer het licht is alleen contouren ziet. Retinitis betekent
letterlijk ontsteking van het netvlies, maar dat is eigenlijk niet wat er bij de ziekte
gebeurt. Bij RP worden er namelijk pigmentophopingen op het netvlies gevormd.
Hierdoor raken de lichtgevoelige cellen (de kegeltjes en de staafjes) van het oog
beschadigd. De meeste mensen met RP worden niet volledig blind, maar wel
maatschappelijk blind. Dit houdt in dat het gezichtsvermogen kleiner is dan 10% of de
gezichtshoek kleiner dan 10 graden. De leeftijd waarop de ziekte zich gaat uiten verschilt
heel erg. Dat kan al vanaf de geboorte zijn, maar ook pas vanaf het zestigste jaar. Ook
het verloop is niet te voorspellen, het kan erg snel gaan of het kan lang stabiel blijven.
Wel is het zeker dat er geen spontane verbeteringen optreden.
Figuur 3.3: Opbouw van het oog en zicht bij kokervisus. Links is de opbouw van het oog te zien, waarbij
een stukje netvlies is uitvergroot. De lichtgevoelige cellen van het netvlies kunnen onderverdeeld worden in de
staafjes en de kegeltjes
3.4 MUTATIES
RP wordt veroorzaakt door een foutje in het DNA, een mutatie. Mutaties kun je overerven
van je ouders of je kunt ze later krijgen. Ze kunnen dan ontstaan doordat bepaalde
27
processen in je lichaam niet meer zo goed werken of door externe factoren zoals straling.
Als het DNA afgeschreven wordt kan zo‟n mutatie zorgen voor een verandering in de
aminozuurvolgorde van het eiwit. Let op: dat is niet altijd zo! Denk maar eens terug aan
de RNA-codons die coderen voor de aminozuren. Soms zijn er meerdere codons die
coderen voor hetzelfde aminozuur. Het aminozuur Serine wordt bijvoorbeeld gevormd bij
de codons: UCU, UCC, UCA, UCG, AGU en AGC. Als je dan een mutatie zou hebben in de
laatste base van U naar C, dan heeft dat geen gevolgen voor de aminozuursequentie.
Maar een mutatie wardoor UCU verandert in CCU maakt van de Serine een Proline en
zorgt dus wel voor een aminozuurverandering. Als er een aminozuurverandering is in een
eiwit, kan het anders gaan functioneren en kan er een ziekte ontstaan. Dit is ook wat er
gebeurt bij RP.
3.4 RHODOPSINE EN RETINA L
RP kan ontstaan door verschillende soorten mutaties. Deze mutaties kunnen ook nog op
veel verschillende genen liggen. Één van de genen die betrokken zijn bij RP is het
rhodopsine-gen, dat codeert voor rhodopsine. Rhodopsine bevindt zich op het membraan
van lichtgevoelige cellen. Je ziet dat weergegeven in het figuur hieronder. Dat rhodopsine
kan reageren op licht komt doordat het een klein molecuul „retinal‟ bindt. Dat molecuul is
weergegeven in het midden van het linkerfiguur. De fotonen uit het licht kunnen een
verandering veroorzaken van 11-cis-retinal naar trans-retinal. Hierdoor verandert ook
het rhodopsine van vorm, en dit zet een reactie in gang. Uiteindelijk zullen hierdoor
neuronen geactiveerd worden, die er in de hersenen voor zorgen dat je kunt zien.
Figuur 3.4: Rhodopsine met retinal (links) en de structuurformule van retinal (rechts). Rhodopsine
is een membraaneiwit. De helices steken dwars door het membraan heen en vormen een soort cilinder. Hier
binnen in zit het retinal gebonden. Als retinal in de cisvorm aanwezig is, is het rhodopsine inactief. Maar onder
invloed van licht gaat verandert retinal van cis naar trans en wordt rhodopsine actief.
Je kunt je dus wel voorstellen dat de binding tussen rhodopsine en retinal erg belangrijk
is voor het zien. RP kan dan ook veroorzaakt worden door mutaties van rhodopsine op
plekken waar het retinal bindt. Hier gaan we nu naar kijken met een bioinformatica
opdracht.
28
OPDRACHTEN BIJ LES 3
1. Welke verbindingen spelen de grootste rol bij eiwitvouwing? Welke secundaire
structuur zal het meest stabiel zijn?
2. Kijk naar de α-helix en de β-sheet (bron 10 en 11 H-22). Wat valt je op aan de
oriëntatie van de restgroepen?
3. We hebben het kort gehad over de rol van zwavelbruggen bij de eiwitvouwing. Onze
haren zijn opgebouwd uit eiwitketens, waarbij zwavelbruggen een belangrijke rol
spelen. Ze bepalen onder andere of je stijl haar hebt of krullen. Maak hierover
opdracht 31 a-c van paragraaf 18.3 (scheikunde boek).
Wanneer deze zwavelbruggen weer gevormd worden zit het haar om de krulspeld
en zullen de zavelbruggen scheef gevormd worden. Daarom blijft het haar na deze
behandeling nog in de krul zitten.
4. Noem twee soorten „treinkaartjes‟ die eiwitten kunnen hebben, zodat ze op de
goede plek in terecht komen.
5. Beschrijf het transport van een membraaneiwit. Gebruik hierbij de termen: golgisysteem, exocytose, transportblaasje, endoplasmatisch reticulum.
6. Kijk in tabel 67D van je binas. Heeft hemoglobine een quarternaire structuur?
7. Noem drie manieren waarop je een mutatie kunt krijgen.
8. Hieronder staan de eerste 30 basen van het rhosopsinegen. Vertaal deze DNAsequentie naar een eiwitsequentie.
atgaatggca cagaaggccc taacttctac
9. Welke mutaties zijn er allemaal mogelijk in de dertiende base? En welke aminozuren
kunnen er dan ontstaan? Welke mutatie lijkt jou het meest erg?
10. Leg uit hoe een mutatie kan leiden tot een ziekte. Je mag voor je antwoord internet
gebruiken, maar dat hoeft niet.
Extra informatie over deze les:
Soms zegt een filmpje meer dan een stapel papier, dus wie weet steek je nog iets op van
hetvolgende filmpje:
Glycosylatie en transport van eiwitten:
http://www.youtube.com/watch?v=u38LjCOvDZU
29
BIOINFORMATICA-COMPUTERPRACTICUM
celmembraan
Figuur 9.1: Plek in de cel. Het eiwit rhodopsine bevindt zich op het celmembraan.
Voorbereiding:
Yasara downloaden: www.yasara.com
>downloads >Yasara for new users >stage I >click here to download (inschrijven met het gegeven
formulier) >order
Je krijgt na 10 minuten een e-mail van [email protected] (wellicht gelabeld als spam). Bij puntje 2) van deze
mail staat een link die je kunt openen in internet explorer:
http://yasara3.cmbi.ru.nl/go/ER15Sp/DeployYASARA.exe
Eenmaal geopend krijg je een schermpje met uivoeren/opslaan. Kies ‘opslaan’ en selecteer een map waar je
het bestand in wilt zetten. Dit kan even duren. Daarna kun je het bestand deployYASARA.exe openen en klikken
op ‘uitvoeren’. Yasara wordt nu geïnstalleerd en geopend.
Site van het vak bioinformatica 2: http://swift.cmbi.ru.nl/teach/B2/index.html
Site waarop je Opsin.pbd kunt dowloaden: http://www.cmbi.kun.nl/~vgelder/vwo/RP/index_origineel.html
30
ALGEMENE INFORMATIE BIOINFORMATICA
Bioinformatica is een vakgebied waarbij informatica gebruikt wordt om biologische kennis
te verkrijgen. Het is dus een subdomein van de biologie. Je kunt met bioinformatica
bijvoorbeeld verschillende aminozuursequenties vergelijken. Als je de sequentie van een
bepaald eiwit van meerdere diersoorten vergelijkt, kun je bijvoorbeeld iets zeggen over
de evolutie van dat eiwit. De stukken die bij elke diersoort hetzelfde zijn noemen we
geconserveerd. Deze stukken zijn meestal erg belangrijk voor het functioneren van het
eiwit. Andere delen verschillen per diersoort en zijn meestal minder essentieel. Bij dit
soort analyses gebruik je de computer om de sequenties zo goed mogelijk onder elkaar
te zetten. De computer zoekt dus voor je uit welke gebieden van het eiwit sterk overeen
komen.
Een ander deel van de bioinformatica is gericht op het zichtbaar maken van eiwitten. Er
zijn bijvoorbeeld computerprogramma‟s gemaakt die de krachten binnen zo‟n eiwitmolecuul kunnen berekenen. Je voert daar dan een aminozuursequentie in, en de
computer laat je het optimaal gevouwen eiwit zien. Als er in een eiwit bijvoorbeeld
geladen aminozuren voorkomen, dan zullen die krachten ondervinden. Gelijk geladen
aminozuren stoten elkaar af en tegengesteld geladen aminozuren trekken elkaar aan. Als
je al dit soort krachten meeneemt in een computermodel komt je voorspelling erg dicht
bij de werkelijke vorm van het eiwit. Die zal immers ook altijd optimaal gevouwen zitten.
Wij gaan zo met de computer kijken naar rhodopsine. De structuur van dit eiwit kun je
downloaden van de Protein Data Bank (PDB). Dit is een site op internet waar alle
eiwitstructuren, die tot nu toe bekend zijn, opgeslagen zijn. Dit hebben wij al voor jullie
gedaan. Daarna kun je deze PDB-file openen met Yasara. Je krijgt dan een
driedimensionaal beeld van het eiwit. Je kunt het eiwit ronddraaien en voor allerlei
weergaven kiezen. Om te begrijpen hoe zo‟n eiwit in elkaar zit is het wel belangrijk dat je
de verschillende aminozuren kent. We zullen dus eerst even aandacht besteden aan de
verschillende aminozuren en hun eigenschappen.
Figuur 3: structuurformules van de 20 aminozuren
Figuur 9.2: Protein Data Bank (PDB) en Yasara. De Protein Data Bank is een site waarop je vele
driedimensionale eiwitstructuren kunt downloaden. Deze kun je bijvoorbeeld openen in het programma Yasara.
COMPUTERPRACTICUM MET YASARA
Getting started…
31
Open Yasara (dubbelklik op het icoontje en kies „uitvoeren‟) om het opgeslagen
bestand te bekijken. Het kan even duren, maar als het goed is zie je uiteindelijk
een blauw scherm.
Om een beetje wegwijs te worden in dit programma kun je het beste eerst een
instructiefilmpje doorlopen. Die vind je bij >help >play help movie >1.1 working
with Yasara. Dit duurt ongeveer 10 minuten.
Snap je het een beetje? Open dan het eiwit rhodopsine door te klikken naar >file
>load >pdb-file >... >opsin.pdb
Druk op F6, zodat je de secundaire structuren overzichtelijk in beeld krijgt.
The real thing…
1. Beschrijf het eiwit. Hoeveel helices heeft het? En hoeveel sheets? Hoe ziet de
teriaire structuur eruit? Is er een quaternaire structuur?
2. Maak alle glycines en prolines zichtbaar:
>view >show atoms >residue >name: gly, pro
In welke secundaire structuren kom je deze het meest tegen?
Hoe zou dat komen? (hint: vergelijk deze aminozuren met alle andere
aminozuren)
3. Kijk naar alle ladingen in het molecuul:
>view >show atoms >residue >name: arg, asp, glu, lys (houd CTRL ingedrukt om
meerdere residuen te selecteren)
Waar zitten de meeste ladingen in het molecuul?
Kun je hier een verklaring voor geven? (hint: het is een membraaneiwit)
4. Je kunt ook alle waterstofbruggen zichtbaar maken, dit gaat als volgt:
>edit >add >hydrogens to: all
>view >show hydrogen bonds >all
Zijn er ook waterstofbruggen tussen rhodopsine en retinal?
32
Kun je hier een verklaring voor geven?
5. We willen graag weten of er ook zwavelbruggen in dit molecuul voorkomen.
Hiervoor maken we eerst alle cysteïne residuen zichtbaar:
>view >show atoms >residue >name: cys
Er kan alleen een zwavelbrug zitten als de zwavelatomen van twee cysteïnes
tussen de 1.5 Å (Ångström) en de 2.5 Å van elkaar af zitten. Je kunt dit meten
door twee zwavelatomen te selecteren (houd daarvoor CTRL ingedrukt). Dan staat
linksonder bij „marked distance‟ de afstand tussen deze twee atomen.
Zijn er zwavelbruggen in dit molecuul?
6. Kijk naar alle ringstructuren in het molecuul:
>view >show atoms >residue >name: phe, trp, tyr
Valt je iets op?
Door welke soort binding blijft het retinal op zijn plaats?
Waarom staat histidine eigenlijk niet in dit rijtje?
7. Mensen met Retinis Pigmentosa hebben een mutatie in het gen dat codeert voor
rhodopsine. Deze mutaties kunnen een aminozuurverandering in het eiwit
veroorzaken. Bekende aminozuurveranderingen bij RP patiënten zijn: Ser186Pro,
Cys187Tyr, Met207Arg, Ala292Glu en Lys296Glu. Notatie: Bijvoorbeeld bij de
eerste mutatie is aminozuur nummer 186 van een Serine over gegaan naar een
Proline. Maak al deze aminozuren zichtbaar:
>view >show atoms >residue >sequence: Ser186, Cys187, Met207, Ala292,
Lys296
Kun je nu bedenken waarom bij mensen met een of meerdere van deze
mutaties het proces van zien niet goed verloopt?
Welk aminozuur is waarschijnlijk het belangrijkst voor de functie van
rhodopsine? Maak een mooi plaatje van dit aminozuur. Sla het plaatje op met
behulp van „Print Screen‟ en een tekenprogramma.
33
LES 4 DNA REPLICATIE EN DNA ONDERZOEK
Figuur 4.1: Plek in de cel. DNA replicatie vindt plaats in de celkern.
4.1 DNA REPLICATIE
Deze les zal gaan over natuurlijke DNA replicatie en hoe dit in zijn werk gaat in
laboratoria. DNA replicatie is belangrijk voor onze celdelingen. Voorafgaand aan de
celdeling wordt het DNA verdubbeld. Deze replicatie gebeurd in de S-fase van de
celcyclus. Replicatie is het proces waarin DNA verdubbeld wordt. De replicatie begint op
vaste plaatsen op het DNA, de zogenaamde „origin of replication‟ (ORI). Dit gedeelte van
het DNA (sequentie) is AT-basenrijk, er komen dus veel adenine en thymine basen in
voor en is ongeveer 250 basenparen lang. Het enzym helicase ontwindt de DNA dubbele
helix en laat door het verbreken van de waterstofbruggen de twee strengen een stukje
uit elkaar gaan. Het enzym DNA polymerase kan op deze manier een stukje van het
enkelvoudige DNA aflezen. Op de plaats waar de twee strengen een stukje uit elkaar zijn,
hecht zich op de ORI plaats een RNA primer. Deze binding noem je het begin van de DNA
synthese. Het DNA-polymerase zorgt ervoor dat een rondzwevend nucleotide, die
complementair is aan de oude DNA streng, gebonden wordt aan de RNA-primer. Bij
iedere stap wordt een volgend nucleotide hieraan vastgemaakt tot de hele streng is
afgelezen. Het DNA heeft in normale toestand de vorm van een dubbele helix. Bij DNA
replicatie is het DNA in twee strengen verdeeld en is dus tweemaal enkelstrengs. De
replicatie gebeurt dan ook bij beide strengen maar wel op verschillende wijzen. Het DNA
wordt afgelezen vanuit de 3‟ richting naar de 5‟ richting en het opbouwen van het DNA
34
gebeurt altijd in de 5‟ 3‟ richting. Het DNA is maar een klein gedeelte opengeritst en
zijn de DNA replicatie gebeurt in kleine gedeeltes van ongeveer 100 tot 200 nucleotiden
lang omdat de DNA polymerase niet meer nucleotiden aan elkaar kan koppelen. Na deze
streng hecht zich op nieuw een RNA-primer aan de oude DNA streng. Daarna vindt een
koppeling tussen nucleotide en DNA polymerase plaats en zo wordt weer een nieuw
stukje DNA gemaakt. Het stukje RNA (RNA primer) en het DNA samen wordt het
Okazaki-fragment genoemd. Je hebt nu te maken met allemaal losse stukjes DNA
(verschillende fragmenten van 100 – 200 nucleotiden lang). Als laatste fase in de DNA
replicatie worden de verschillende fragmenten aan elkaar gebonden dor het enzym DNAligase.
Figuur 4.2: DNA Replicatie
DNA replicatie gebeurt onder invloed van helicase, RNA primer, DNA polymerase en DNA ligase. Het okasaki
fragment is de RNA primer en het geproduceerde DNA keten.
Het hele proces van DNA replicatie staat onder invloed van de energie die wordt
verkregen uit de hydrolyse van guanosinetrifosfaat (GTP) in ATP (adenosinetrifosfaat).
Niet alleen in het lichaam wordt DNA replicatie gedaan. Ook in het onderzoek aan DNA is
het nodig om grote hoeveelheden van hetzelfde DNA te gebruiken. Hiervoor is een
nieuwe techniek bedacht namelijk de Polymerase Ketting Reactie (PCR).
4.2 POLYMERASE CHAIN REA CTION (PCR)
De volgorde van nucleotiden in het DNA is van groot belang dat wij normaal
functioneren. Daarom worden de specifieke volgordes van nucleotiden in het DNA (de
sequentie)in laboratoria bepaald, met diverse technieken waar de Polymerase ketting
reactie (PCR) er één van is. Omdat één DNA molecuul erg klein is, is het erg moeilijk om
deze te bestuderen. De analyse kan worden vergemakkelijkt met het vermenigvuldigen
van het DNA. De PCR is een techniek om vele kopieën te maken van een kleine
hoeveelheid specifiek DNA. Vaak kun je maar een klein beetje DNA uit weefsel isoleren
wat niet genoeg is voor het analyseren. De PCR reactie is begin jaren 80 uitgevonden en
heeft tot nu toe al veel bijgedragen aan de vooruitgang van de moleculaire biologie. Het
principe van de PCR techniek is gebaseerd op de manier waarop DNA in de natuur wordt
vermenigvuldigd als de cel zich deelt. Bij de PCR techniek wordt het DNA verwarmd,
waardoor de twee strengen uit elkaar gaan en fungeren als mal voor de nieuw te vormen
DNA strengen.
35
De PCR reactie kan kortweg gezegd ingedeeld
worden in drie stappen. De eerste stap is een
temperatuursverhoging naar 94°C, waardoor het
DNA denatureert (DENATURATIE). Dat wil zeggen
dat de waterstofbruggen tussen de DNA-strengen
worden verbroken. Daardoor valt de dubbele helix
van het DNA uit elkaar. De tweede stap houdt in
dat er primers worden toegevoegd. Primers zijn
kleine stukjes chemisch gemaakt DNA, waarvan de
basenvolgorde
complementair
(dus
tegenovergesteld) is met die van de uiteinden van
het te vermeerderen DNA fragment. De primers
zorgen ervoor dat aan de enkelstrengs DNA weer
nieuwe basen gekoppeld kunnen worden (T=72°C)
(ELONGATIE). De laatste stap gebeurt onder
invloed van het enzym polymerase. Polymerase
zorgt ervoor dat de primers worden verlengd tot
een compleet nieuw stuk DNA. Deze verlenging
kan gebeuren omdat je in de mix lossen
nucleotiden toevoegd die door de polymerase op
de juiste plaats gekoppeld wordt. Tegen beide
strengen ontstaat zo een nieuwe streng van DNA
en zo wordt de hoeveelheid DNA verdubbeld. Deze
stappen kun je meerdere keren herhalen binnen
één PCR reactie. Het nieuwe stukje DNA is identiek
aan het origineel. Als je de cycli 30 keer herhaalt
heb je al meer dan één miljard kopieën gemaakt.
De gevoeligheid van de PCR-techniek is zo groot
dat DNA-diagnostiek uitgaande van een enkel
DNA-molecuul mogelijk is. Toepassingen liggen op
verschillende
gebieden,
zoals
de
genetica,
microbiologie, virologie en het kankeronderzoek.
Figuur 4.3: Polymerase Ketting reactie
De PCR begint met het denatureren van het
DNA door verhitting. Primers hechten zich
aan het DNA door de afkoeling. Door het
opwarmen naar 72°C worden de primers
verlengd en ontstaat een identiek DNA
molecuul
als
het
originele.
36
4.3 SEQUENCING
Na de PCR reactie is het gebruikelijk om het DNA te
controleren op lengte en op foutjes. Om de sequentie
te bepalen moeten we nu de lengte vaststellen van de
nieuwgevormde fragmenten. Deze eerste stap wordt
gedaan door elektroforese. De DNA fragmenten
worden op een gel geplaatst en over deze gel loopt
een elektrisch veld. Het DNA zal naar de pluspool gaan
bewegen. De snelheid waarmee de moleculen zich
verplaatsen in de gel is afhankelijk van de lengte van
de fragmenten. Korte moleculen zullen zich sneller
bewegen dan de lange fragmenten en zullen dus
onderin de gel te vinden zijn. De DNA fragmenten
worden op deze manier van elkaar gescheiden in hun
lengte. De volgende stap in het gebruik van DNA in het
onderzoek is het controleren op mutaties en wordt
gedaan door het DNA te sequencen. Sequencen is een
techniek waarbij de basenvolgorde van een DNAstreng wordt bepaald. Deze techniek is in 1975
ontwikkeld en in de loop van de jaren steeds beter en
sneller geworden. In 2001 werd de sequentie van het
menselijk genoom (3 miljard basenparen lang)
gepubliceerd. In de meeste laboratoria wordt gebruik
gemaakt van de Sanger methode. Sequencen heeft
hetzelfde verloop als de PCR reactie alleen worden er
nu enkele dideoxynucleotiden en het meeste van de
gewone nucleotiden aan de reactie toegevoegd.
Dideoxynucleotiden zijn moleculen die lijken op
normale nucleotiden alleen hebben zij geen OH-groep
aan de 3-uiteinde van het DNA. Hierdoor stopt de
replicatie wanneer er een dideoxynucleotide wordt
ingebouwd. Elk van de vier dideoxynycleotides (ddA,
ddC, ddG en ddT) heeft een apart fluorescerend label.
Daardoor is na het sequencen in één opslag te zien
waar de vier verschillende dideoxybasen zich
bevinden. Als de PCR reactie op gang gekomen is,
zullen op een gegeven moment alle kopieën worden
afgesloten met een bepaalde dideoxynucleotide. Het
punt waar het enzym een dideoxynucleotide inbouwt
en dus de plaats waar de keten stopt, is geheel
willekeurig. De ene keer zal een korte keten gemaakt
worden, de volgende keer een lange. Uiteindelijk
komen dus alle mogelijke eindpunten aan de beurt. Je
krijgt dan een verzameling van stukjes DNA van alle
mogelijke verschillende lengtes met ieder een
dideoxynucleotide op het eind. Om er achter te komen
waar alle verschillende dideoxynucleotides in de keten
Figuur 4.4: Sanger Sequencing
37
zich bevinden, moeten we weten wat de verschillende lengtes zijn van al onze
afgebroken ketens. Dit wordt gedaan door middel van gelelektroforese. Als de kortste
keten eindigt met een ddA, weet je dus dat de volgorde van het DNA begint met een A.
Zo kun je voor iedere positie in de DNA volgorde achterhalen welke nucleotide (A, C, G of
T) er zit. Het aflezen van de gel gebeurt automatisch. Met sequentieanalyse kunnen ook
eenvoudige mutaties opgespoord worden door het DNA van een patiënt te vergelijken
met dat van een controlepersoon.
38
OPDRACHTEN BIJ LES 4
In de les
1) In het kernplasma zijn eenentwintig nucleotiden met de volgende stikstofbasen
aanwezig: 6 x adenine, 4 x thymine, 8 x cytosine en 3 x guanine. Hoeveel nieuwe
basenparen kunnen tijdens de duplicatie van DNA maximaal gevormd worden met
deze eenentwintig nucleotiden?
2) Een cel ondergaat 2 keer mitose. Hierbij ontstaan 4 dochtercellen. In hoeveel van
deze dochtercellen zijn nog delen van het oorspronkelijke DNA-molecuul aanwezig?
3) DNA replicatie vindt na/voor/tijdens de celdeling plaats.
4) Geef aan waarom deze `Okazaki`-fragmenten noodzakelijk zijn voor de replicatie van
de antiparallelle DNA-streng.
5) Wat doet een DNA polymerase?
6) Hoe werkt de PCR? Benoem hierbij de verschillende stappen en de functie ervan.
7) Stel dat je met één streng DNA begint. Hoeveel DNA-strengen heb je dan na dertig
verdubbelingscycli?
8) Wat is gelelektroforese?
9) Als je een gelelektroforese uitvoert, naar welke kant zal het DNA bewegen en
waarom?
10) Waarom gebruik je na de PCR de sequencing methode?
Extra informatie over deze les:
Duidelijke weergave van een PCR reactie
http://www.youtube.com/watch?v=eEcy9k_KsDI
DNA sequencing
http://www.youtube.com/watch?v=oYpllbI0qF8
39
DEFINITIEBLAD
Acetylgroep: klein polair molecuul dat als het gebonden zit aan een histon zorgt voor
een open DNAstructuur (euchromatine)
Adenine: base die samen met desoxyribose een DNA nucleotide vormt en samen met
een ribose een RNA nucleotide; basenparing met thymine en uracil
Alfahelix: spiraalvormige keten van aminozuren
Aminogroep: deel van een aminozuur (NH2)
Aminozuur: bouwsteen van eiwitten
Anti-codon: specifieke sequentie van drie nucleotiden in het tRNA, complementair aan
een codon voor een aminozuur in het mRNA
Apolair: niet polair, dus zonder electronegatieve atomen zoals N en O; slecht oplosbaar
in water
Backbone: keten peptidebindingen van een eiwit; het eiwit exclusief de restgroepen van
de aminozuren
Basenpaar: Twee nucleotide die gepaard zijn via waterstofbruggen tussen hun basen,
bijvoorbeeld A met T of U en C met G
Bètasheet: aminozuurketen in een zigzagstructuur
Bioinformatica: vakgebied waarin de informatica wordt gebruikt om biologische
verschijnselen te bekijken en analyseren
Zuurgroep: deel van een aminozuur (COOH)
Celmembraan: membraan (vettige structuur) rondom een cel
Chromatine: bouwstof van chromosome, bestaande uit DNA, hstonen en andere
eiwitten
Chromosoom: een groot DNA molecuul met eiwitten die eraan gebonden zijn; bevat
vele genen
Codon: sequentie van drie opeenvolgende nucleotiden in DNA of mRNA dat codeert voor
een aminozuur
Condensatiereactie: reactie waarbij een (peptide)binding gevormd wordt en er water
vrij komt
Cytoplasma: celinhoud buiten de celkern, bevat organellen zoals mitochondriën
Cytosine: base die samen met desoxyribose een DNA nucleotide vormt en samen met
een ribose een RNA nucleotide; basenparing met guanine
40
Denaturatie: ontvouwing van een eiwit of verbreking DNA dubbele helix
Desoxyribose: suikergroep die samen met een fosfaatgroep en een base (A, C, T,
G)een DNA nucleotide (bouwsteen) vormt
Dideoxynucleotiden: is een molecuul dat lijkt op een normaal nucleotide maar het
heeft geen OH- groep aan het 3‟-uiteinde. Hiermee zorgt dit molecuul ervoor dat de
replicatie gestopt wordt in het sequencing proces.
Disaccharide: Twee gebonden monosachariden (suikergroepen)
DNA: desoxyribonucleic acid, een keten van nucleotiden waarin genetische informatie is
opgeslagen
DNA ligase: is een speciaal enzym dat DNA strengen, die verbroken zijn tijdens de DNA
replicatie weer aan elkaar kan maken.
DNA-methylering: het plaatsen van een methylgroep op een nucleotide van het DNA;
DNA-methylering voorkomt dat histonen geacetyleerd worden en zorgt er zo voor dat de
chromatinestructur compact blijft (heretochromatine). Daarnaast zorgt het ervoor dat
transcriptie factoren belemmerd worden in het aflezen van genen.
Dubbele helix:
zitten
spiraalvormige conformatie waarin twee complementaire DNA ketens
Eiwit: groot molecuul dat bestaat uit één of meerdere keten(s) aminozuren
Eiwitregulatie: zorgen dat het aantal eiwitten op peil blijft door het de synthese,
afbraak, opslag en denaturatie van eiwitten te controleren
Elongatie: verlenging van bijvoorbeeld een mRNA of DNA streng; dit vindt pas plaats
nadat de transcriptie of translatie gestart is (initiatie)
Endoplasmatisch reticulum: stelsel van dubbele membranen in het cytoplasma van
een cel, het ruwe endoplasmatisch reticulum bevat vele ribosomen die nodig zijn voor
eiwitsynthese
Enzym: eiwit of RNA molecuul dat actief is als katalysator (een reactie makkelijker laat
verlopen)
Essentieel aminozuur: aminozuur dat niet zelf aangemaakt kan worden door mensen
en dus verkregen moet worden uit voedsel
Euchromatine: open vorm van chromatine; genen in dit gebied kunnen makkelijk
afgeschreven worden aangezien RNA polymerase en andere belangrijke eiwitten er goed
bij kunnen
Exon: stuk DNA (in een gen) dat codeert voor het uiteindelijke eiwit en in het RNA
aanwezig blijft (zie intron)
Fosfaatgroep: PO4; polaire groep die gebonden aan een aminozuur vaak zorgt voor
activatie van het eiwit
41
Fosfatase: enzym dat de binding van een fosfaatgroep aan een eiwit helpt te verbreken
Fosforylering: het binden van een fosfaatgroep aan een ander molecuul zoals een eiwit
(de fosfaatgroep is afkomstig van ATP)
Gen: een stuk DNA met de genetische informatie voor en bepaalde eigenschap; er liggen
vele genen op een chromosoom
Gelelektroforese: is een scheidingstechniek waarbij DNA moleculen onder invloed van
een elektrisch veld bewegen over een gel. Het negatief geladen DNA beweegt naar de
positieve pool. Hoe groter de moleculen hoe trager ze door de gel bewegen en daarmee
dus de kortste afstand afleggen.
Genregulatie: is het proces waarbij genen onder invloed staan van verschillende
factoren om tot expressie te komen.
Glucosegroep: suikergroep C6H12O6; kan gebonden worden aan een aminozuur en een
rol spelen bij eiwitvouwing, eiwittransport of eiwitstabiliteit
Glycosylering: het proces waarbij er een suikergroep (bijvoorbeeld glucose, mannose,
n-acetylglucosamine) aan een aminozuur wordt gebonden
Golgisysteem: membranenstelsel in het cytoplasma van een cel, waarin
postranslationele modificaties aan eiwitten plaatsvinden en waar de eiwitten gesorteerd
worden voor transport
Guanine: base die samen met desoxyribose een DNA nucleotide vormt en samen met
een ribose een RNA nucleotide; basenparing met cytosine
Helicase: is een groep enzymen die de dubbele DNA helix ontwindt door de
waterstofbruggen te verbreken.
Heterochromatine: open chromatinestructuur, waardoor het DNA toegankelijk is en
overgeschreven kan worden; histonacetylering bevordert deze structuur
Histon: eiwit dat bindt aan DNA en zo mee helpt aan de pakking van DNA
Histon modificatie: histonen zijn eiwitten die het DNA helpen met opvouwen.
Veranderingen aan deze histonen worden histon modificaties genoemd.
Hydrofiel: polair of geladen, kan waterstofbruggen vormen met water en lost daarom
goed op in water
Hydrofobe interactie: meerdere hydrofobe groepen die samenkomen om afgeschermd
te zijn van het water
Hydrofoob: apolair (zonder lading), kan geen waterstofbruggen vormen met water en is
daarom slecht oplosbaar in water
Hydrolyse: verbreken van een (peptide)binding, waarvoor water nodig is
Initiatie: start van transcriptie of translatie
42
Intron: stuk DNA (in een gen) dat niet codeert voor het uiteindelijke eiwit en door
middel van splicing uit het RNA verwijderd wordt (zie exon)
Ionkanaal: eiwit dat transport van een specifiek ion over een membraan faciliteert
Kinase: enzymen die fosforyleringsreacties helpen te verlopen
Kokervisus: symptoom van retinitis pigmentosa; de staafjes op de rand van het netvlies
zijn beschadigd, waardoor er alleen in het midden van het blikveld scherp zicht is
Lipoproteïne: eiwit waaraan een vetgroep gebonden zit
Mannosegroep: suikergroep C6H12O6; kan gebonden worden aan een aminozuur en een
rol spelen bij eiwitvouwing, eiwittransport of eiwitstabiliteit
Monosaccharide: eenvoudigste groep van koolhydraten die niet meer verder te
hydrolyseren is.
mRNA: messenger RNA, RNA molecuul dat complementair is aan een DNA streng, draagt
de genetische code van een chromosoom naar een ribosoom waar het wordt vertaald
naar een eiwit
Mutatie: een verandering in de DNA sequentie
N-aceltylglucosaminegroep: suikergroep C8H15NO6; kan gebonden worden aan een
aminozuur en een rol spelen bij eiwitvouwing, eiwittransport of eiwitstabiliteit
Nachtblindheid: symptoom van retinitis pigmentosa; zicht is erg beperkt bij weinig licht
Nucleosoom: bolstructuur bestaande uit acht histonen en DNA;bouwstof van chromatine
Nucleotide: bouwsteen van DNA of RNA, bestaande uit een fofaatgroep, een suikergroep
(desoxyribose in DNA en ribose in RNA) en een base
Okazaki fragment: is een kort antiparallelle DNA fragment die bestaat uit de RNA
primer en een stukje, door DNA polymerase, nieuw ontwikkelde DNA streng.
Operator: een DNA sequentie waar genen aan of uit geschakeld kunnen worden.
Operon: Een operon bestaat uit op het DNA bij elkaar liggende genen, die bij elkaar
horen voor het tegelijk uitvoeren van één transcriptie van meerdere enzymen voor één
celproces.
Origin of replication: is het begin punt van de DNA replicatie waar de RNA primer zich
hecht.
PCR: is een manier om uit zeer kleine hoeveelheden DNA specifiek één of meer
gedeeltes te vermenigvuldigen tot er genoeg van is om het te analyseren
Peptidebinding: binding tusen de aminogroep van het ene aminozuur en de zuurgroep
van het andere aminozuur, bij de vorming van de binding komt water vrij
43
Polair: met ongelijk verdeelde lading; een molecuul wordt polair doordat een
elektronegatief atoom de elektronen naar zich toe trekt en een beetje negatief geladen
wordt, andere atomen worden dan een beetje positief geladen
Polymerase: het verdubbelt het DNA in de 5‟ 3‟ richting
Polypeptideketen: keten aminozuren die met een peptidebinding aan elkaar gekoppeld
zijn; eiwitten bestaan uit één of meerdere peptideketens
Polysacharide: meer dan twee gebonden suikergroepen
Posttranslationele modificatie: verandering aan een eiwit na de translatie
Primaire structuur: volgorde van aminozuren in een eiwit
Primer: is een stukje DNA of RNA dat gebruikt wordt als startpunt van de PCR. Er zijn
steeds twee primers nodig, één voor de 5‟-streng en één voor de 3‟-streng
Polyadenylatie signaal: is een specifiek RNA sequentie (AAUAAA) die ervoor zorgt dat
bij RNA processing de poly(A)staart eraan wordt gezet
Promoter: is een DNA sequentie voor één gen of meerdere genen dat de expressie van
de genen reguleert.
Protease: enzym dat betrokken is bij eiwitafbraak, katalyseert de hydrolysereactie van
een peptidebinding
Proteasoom: groot complex van afbraakmoleculen dat ervoor zorgt dat onnodige of
beschadigde eiwitten afgebroken worden (bevat onder andere proteases)
Protein Data Bank: site op internet waar je driedimensionale eiwitstructuren kunt
downloaden (www.pdb.org)
Purine: ringstructuur die voorkomt in de basen adenine en guanine
Pyrimidine: ringstructuur die voorkomt in de basen thymine, cytosine en uracil
Quaternaire structuur: driedimensionale structuur van een eiwit dat uit meerdere
aminozuurketens bestaat, vooral de manier waarop deze ketens ten opzichte van elkaar
liggen
Replicatie: synthese van een stuk DNA, identiek aan een ander stuk
Repressoreiwit: eiwit dat voor de startplaats van de transcriptie bindt aan het DNA en
de transcriptie van een gen remt
Restgroep: deel van een aminozuur dat verschilt per aminozuur, kan polair / apolair /
negatief geladen / positief geladen of aromatisch (met ringstructuur) zijn
Retinal: klein molecuul dat verandert van cis naar trans onder invloed van licht; bindt
aan het eiwit rhodopsine, waardoor deze ook lichtgevoelig wordt
Retinitis Pigmentosa: erfelijke aandoening aan het oog, waarbij (delen van) het
netvlies beschadigd zijn door pigmentophopingen
44
Rhodopsine: membraaneiwit dat zich bevindt in lichtgvoelige cellen van het oog; bindt
het molecuul retinal en is kan daardoor op licht reageren
Ribose: suikergroep die samen met een fosfaatgroep en een base (A, C, G, U) een RNA
nucleotide (bouwsteen) vormt
Ribosoom: complex van rRNA‟s en eiwitten op het ruwe endoplasmatisch reticulum, dat
betrokken is bij de translatie (eiwitsynthese)
RNA: ribonucleic acid, een keten van nucleotiden die is afgeschreven van het DNA
RNA polymerase: enzym die de transcriptie (het afschrijven den RNA van DNA) helpt te
verlopen
RNA primer: is een stukje RNA dat gebruikt wordt als startpunt van de PCR. Er zijn
steeds twee primers nodig, één voor de 5‟-streng en één voor de 3‟-streng
RNA processing: is de bewerking van het pre-mRNA streng naar het mRNA waaronder
capping, toevoeging van de poly(A)-staart en intronen splicing
rRNA: ribosomaal RNA, onderdeel van ribosoom
Secundaire structuur: de ruimtelijke structuur van de aminozuurketen (s) van een
eiwit, voorbeelden zijn de alfahelix en de bètasheet
Sequencing: is het bepalen van de nucleïnezuur van een DNA/RNA sequentie
Signaalsequentie: aminozuursequentie (meestal eerste paar aminozuren) die herkend
wordt door transporteiwitten en er zo voor zorgt dat het eiwit op de goede plek terecht
komt
Splicing: verwijdering van intronen uit een mRNA sequentie en koppeling van de exonen
Startcodon: AUG, codeert voor het eerste aminozuur van een aminozuurketen
(methionine)
Stopcodon: UAA, UAG of UGA, signaal voor de terminatie van een aminozuurketen
Synthese: aanmaak (van bv eiwitten)
Template: DNA keten die bij de transcriptie wordt gebruikt om een complementaire
keten te genereren
Terminatie: beëindigen; transcriptie en translatie stoppen bij een stopcodon, het
eiwitcomplex en de nieuw aangemaakte keten laten los
Tertiaire structuur: manier waarop de
bètasheets) ten opzichte van elkaar liggen
secundaire
structuren
(alfahelices
en
Thymine: base die samen met desoxyribose een DNA nucleotide vormt; basenparing
met adenine
Transcriptie: het proces waarbij de genetische informatie van een DNA keten wordt
afgeschreven naar mRNA
45
Transcriptie factoren: zijn eiwitten die binden aan de promoter van een DNA
sequentie.
Translatie: proces waarin de genetische informatie van een RNA keten wordt vertaald
naar een eiwit
Transportblaasje: blaasjes met transporteiwitten die ervoor zorgen dat andere eiwitten
op de goede plek in de cel terecht komen
Transportlabel: een sequentie of posttranslationele modificatie van een eiwit, die
herkend wordt door een transporteiwit en er zo voor zorg dat het eiwit op de goed plek in
de cel terecht komt
tRNA: in het nederlands „transport RNA‟ en in het engels „transfer RNA‟, RNA moleculen
met een gebonden aminozuur en een anticodon, die aan een mRNA codon kan binden;
faciliteert de translatie (eiwitsynthese)
Turn: flexibele secundaire eiwitstructuur die de α-helices en de β-sheets met elkaar
verbindt
Ubiquitinegroep: klein eiwit dat dient als signaal voor eiwitafbraak door het
proteasoom; er kunnen meerdere ubiquitinegroepen aan een eiwit worden gebonden
Uracil: base die samen met een ribose een RNA nucleotide vormt; basenparing met
adenine
Vetgroep: een molecuul met een apolair gedeelte, meestal een paar lange
koolstofketens; als een vetgroep aan een aminozuur bindt kan het eiwit zich bijvoorbeeld
beter in een membraan nestelen
Waterstofbrug: aantrekkingskracht tussen een electronegatief atoom (zoals N of O) en
een waterstofatoom
Yasara: programma waarmee je driedimensionale eiwitstructuren kunt bekijken en
analyseren
Zoutbrug: een binding tussen twee (tegengesteld) geladen aminozuren
Zwavelbrug: binding tussen twee zwavelatomen; komt in eiwitten allen voor tussen
twee cysteïnes
46