www.VETserieus.nl
Beste Student,
De documenten op VETserieus.nl zijn alleen bedoeld als ondersteuning bij het
studeren. De samenvattingen worden nagekeken door studenten tijdens het
volgen van de lessen en waar nodig aangepast. Dit project heeft als doel
foutloze samenvattingen te bieden die met hun tijd meegaan, ondanks dit
streven is er altijd een kans dat er fouten in de documenten staan. Mocht je
tijdens het lezen van de samenvatting fouten vinden kun je dat doorgeven via
de contactpagina op de site of direct een mail sturen naar
[email protected]
De student is verantwoordelijk voor zijn of haar leermethode en voor het
uiteindelijke resultaat. Allemaal veel succes met de voorbereidingen!!
Hartelijke groet,
VETserieus.nl
SAMENVATTING GENOOM TOT POPULATIE: BLOK 4
Zelfstudie 1
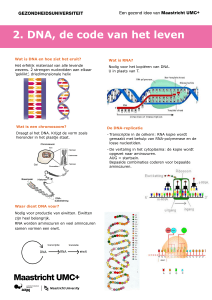
DNA=deoxyribonucleic acid
Genen worden gedragen op chromosomen, welke zowel DNA als eiwitten bevatten. De DNA
componenten bevatten de erfelijke eigenschappen van een cel en de eiwitten zorgen voornamelijk
voor het inpakken en controleren van de enorme DNA moleculen => Watson & Crick Model.
DNA bestaat dan weer uit twee strengen die om elkaar heen gewikkeld zijn. Elk van deze strengen
bestaat uit 4 soorten nucleotiden (ACTG). Deze nucleotiden bestaan uit een 5C-suikergroep
(deoxyribose), een of meer fosfaatgroepen en een stikstof bevattende base. De strengen worden bij
elkaar gehouden door waterstofbruggen tussen deze basen en de nucleotiden zitten covalent
gebonden door middel van de suikergroepen en fosfaat. Hierdoor ontstaat een ruggenmerg in het
molecuul gebonden in afwisselend suiker-fosfaat-suiker-fosfaat. De 4 nucleotiden verschillen alleen
van elkaar in de base, hierdoor kan een DNA molecuul als een ketting (backbone) met 4 soorten
kralen gezien worden.
De manier waarop de nucleotiden gebonden zijn geven een polariteit aan een DNA molecuul,
waardoor de ene zijde met een fosfaat groep eindigt (5’) en de andere kant met een suikergroep (3’).
A bindt altijd aan T en C altijd aan G, waardoor de tussenruimte tussen de twee strengen altijd gelijk
is (A en G zijn bredere nucelotiden dan C en T) . Een purine is een base met twee ringen (G en A)
welke altijd gebonden zit aan een pyrimidine, een enkele ring base (C en T). De dubbele helix draait
per 10 nucleotiden, op energetisch favoriete manier en de losse strengen zijn antiparallel aan elkaar
waardoor de ene streng op 3’ eindigt en de andere op 5’. De base-paring is dus precies
complementair tussen de twee strengen. De manier waarop de basen gebonden worden, door een
waterstofbinding tussen de basen, bevinden de basen zich aan de binnenzijde van de helix. De
waterstofbinding tussen AT zijn minder talrijk dan bij GC en is dus iets zwakker.
Organismen verschillen van elkaar doordat ze verschillende nucleotide sequentie hebben. Deze
sequentie codeert namelijk voor verschillende soorten aminozuren (20 stuks), welke op hun beurt
weer eiwitten kunnen vormen. We kunnen deze codering dus als een soort alfabet zien met 4
verschillende letters, die in ontelbaar veel combinaties kunnen voorkomen. De codering van
aminozuren vanuit het DNA is ook lineair, dat wil zeggen de aminozuren worden in bepaalde
volgorde gecodeerd.
De complete set van informatie in een organisme wordt het genoom genoemd. In eukaryotische
cellen wordt al deze informatie in DNA moleculen en vervolgens in chromosomen opgeslagen. Elk
chromosoom bevat 1 gigantisch DNA molecuul. Deze chromosomen kunnen met de enorme omvang
niet zomaar in een cel passen en worden daarom ingepakt door speciale eiwitten die het DNA
vouwen waardoor deze chromatine (het complex van eiwit en DNA) vormt. Humane cellen (met
uitzondering van de sperma en eicel; en natuurlijk de rode bloedcel welke helemaal geen DNA bevat)
bevatten twee kopieen van chromosomen, afkomstig van papa en mama. Een paar bestaat uit
homologe chromosomen en deze verschillen van de nonhomologe chromosomen bij bijvoorbeeld
een man een X en een Y.
DNA hybridisatie is een techniek die de chromosomen kan kleuren en op die wijze verschillende
chromosomen zichtbaar kan maken. Meer traditioneel wordt echter een kleruingstechniek gebruikt
die een onderscheidt maken tussen DNA rijk in AT of juist GC, waardoor een bandenpatroon op de
chromosomen ontstaat. Het patroon van de banden is voor elk chromosoom uniek, en afwijkingen
kunnen gebruikt worden om chromosomale abnormaliteiten aan te tonen. De gehele set van
chromosomen wordt het karyotype genoemd.
Chromosomen dragen dus over het algemeen de specifieke genen van een individu. Een gen kan
gespecificeerd worden als een bepaalde stuk DNA sequentie welke codeert voor een bepaald eiwit.
Sommige genen kunnen echter coderen voor de productie van een RNA molecuul in plaats van eiwit.
Een groot deel van de chromosomen bevatten echter ook stukken DNA die niet van directe functie
lijken, junk DNA. De waarschijnlijke functie van dit DNA ligt in de evolutie en de juiste activiteit van
normaal DNA.
Over het algemeen geldt dat hoe complexer een organisme, hoe complexer het genoom, hoewel
deze relatie niet altijd juist is. Hoe de DNA verspreid is over de chromosomen, en hoeveel
chromosomen, kan ook verschillen per dier. Er is geen directe relatie tussen genen aantal,
chromosoom aantal en genoomgrootte.
Voor een cel om te kunnen delen is het zo dat de DNA moleculen gerepliceerd kunnen worden en de
gerepliceerde kopieen juist uit elkaar getrokken kunnen worden en verdeeld over dochtercellen. Dit
gebeurd in twee stappen van de celcyclus:
1. interfase: dupliceren van DNA en chromosomen. De chromosomen worden hierin uitegtrokken
tot lange dunne strengen (interfase chromosomen) binnen de nucleus en moeilijk te zien met
de lichtmicroscoop. Op deze chromosomen vinden we delen van DNA sequentie die dienen als
punt van initiatie voor de replicatie (replicatie oorsprong). Op een eukaryotisch chromosoom
vinden we meerdere van deze plekken terug, zodat de replicatie snel kan verlopen. Een andere
DNA sequentie vormt telomeren welke aan elk van de twee uiteinden van het chromosoom
worden teruggevonden. Deze telomeren dienen ervoor om te zorgen dat ook de uiteinden van
een chromosoom juist gerepliceerd wordt en tevens beschermt het de uiteinden van een streng.
(de uiteinden kunnen anders verkeerd geïnterpreteerd worden als kapotte DNA sequentie).
Ondanks de uitgerekte strengen zijn ze goed georganiseerd om verstrengeling te voorkomen.
Elke streng heeft een eigen locatie binnen de nucleus. Deze organisatie wordt bereikt door
binding van de chromosomen aan de nucleaire envelop en de nucleaire laminen.
2. mitose: verdeling onder twee dochtercellen. Tijdens deze fase zijn de strengen volledig compact
opgerold waardoor ze niet in de knoop kunnen raken en makkelijk te scheiden zijn. De
opvouwing wordt bereikt door hulp van eiwitten die de chromosomen in een steeds hogere
organisatie plaatsen. Tijdens deze fase binden de filamenten aan een derde specifieke DNA
sequentie, centromeer, waaraan ze uit elkaar getrokken worden.
Ondanks dat de interfase chromosomen minder gecondenseerd zijn is het geen bende van
ongeorganiseerd DNA. Elk chromosoom heeft zijn eigen plekje in de nucleus waardoor ze niet door
elkaar raken. Het meest duidelijke voorbeeld van chromosoom organisatie is die van de nucelolus,
waar delen van de chromosomen die genen dragen voor ribosomaal RNA samen clusteren.
Omdat tijdens het DNA dat ingepakt is in chromosomen toch bereikbaar moet blijven voor eventuele
reparaties en natuurlijk replicatie zijn er specifieke eiwitten die deze functies kunnen waarborgen.
Het DNA is dus flexibel ingepakt en kan niet alleen in en uitpakken, maar ook op bepaalde locaties dit
juist wel of niet doen. Er zijn twee typen eiwitten die een belangrijke rol spelen bij het vormen van de
chromatine (verzamelnaam voor de DNA + eiwitten (bijvoorbeeld Histonen) en het inpakken van
DNA.
1. histonen: deze zijn aanwezig in enorme aantallen en zorgen voor ongeveer de helft van de massa
van chromosomen. Ze zijn verantwoordelijk voor de eerste stap van chromatine inpakken, de
nucleosoom. Het eiwit vormt een soort van kraaltje of jojo achtige structuur waar het lange DNA
molecuul omheen gewonden wordt, er wordt een nucleosoom core partikel gevormd. De
kraaltjes worden aan elkaar gebonden door zogeheten linker DNA. de onderdelen van een zo’n
partikel bestaan uit positieve aminozuren welke stevig binden aan de negatief geladen DNA
ruggengraat van suiker en fosfaat. De kraal op zich, nucelosoom, bestaat uit een octamerisch
histon deel (8 histonen) en een stuk DNA van ongeveer 146 nucelotiden paren lang.
Nadat deze kralenketting is gevormd worden de nucleosomen vrijwel altijd verder ingepakt in de
30nm-vezel. Ook hierbij is een histoneiwit nodig. Er zijn meerdere modellen over hoe zo’n
nucleosoom in een 30nm-vezel wordt ingepakt maar het zigzag model is wel de meest gangbare.
Een mozaik van verschillende zigzag conformaties vormen samen de vezel. Deze 30nm-vezels
worden weer verder georganiseerd in een chromosoom, waarschijnlijk door het vormen van
lussen, welke uiteindelijk na verdere verpakking het daadwerkelijke chromosoom vormen.
2. niet histonen
over het algemeen zijn de chromosomen die tot uiting worden gebracht in een cel minder verpakt
(euchromatine) dan de chromosomen die niet tot uiting komen (heterochromatine). Dus ook de
exacte structuur van een interfase chromosoom kan van celtype tot celtype verschillen afhankelijk
van de genen die tot expressie moeten komen. Heterochromatine wordt gewoonlijk meer
gelokaliseerd rond het centromeergedeelte en de telomeergebieden van een chromosoom.
Ondanks dat de meeste chromosomen een combinatie van heterochromatine en euchromatine
bevatten bestaan er een aantal belangrijke uitzonderingen. Vrouwen bevatten twee X chromosomen,
maar omdat een dubbele hoeveelheid hieraan dodelijk is moet een van de twee chromosomen niet
tot expressie komen. Er is dus een mechanisme ontwikkeld om permanent 1 chromosoom uit te
schakelen door ontzettend dichte inpakking van dit chromosoom tijdens de embryonale
ontwikkeling. Vanaf dat punt hebben de dochtercellen van deze cellen ook hetzelfde dichtbepakte X
chromosoom.
Soms is het echter nodig om wel bij dichtgepakt DNA te kunnen en een manier om de nucleosomen
dan wat minder te bepakken is met behulp van zogenaamde chromatine remoddeling complexen.
Dit zijn eiwitmachines die de energie van ATP gebruiken om de structuur van nucleosomen aan te
passen. Tijdens de mitose is ten minste een deel van deze complexen uitgeschakeld omdat het juist
nodig is om de strakke structuur te behouden.
Een andere methode om de structuur van de nucleosomen aan te passen is met behulp van een
reversibele verandering in de structuur van histonstaarten. Deze N-termini kunnen met behulp van
enzymen aangepast worden door middel van covalente bindingen. Hoewel deze vrom van
modificattie geen directe invloed lijtk te hebben op de centrosoom structuur heeft deze wel degelijk
invloed op de hogere orde organisatie zoals de 30nm-vezel en hoger. Er zijn veel verschillende
soorten van modificaties mogelijk aan deze staarten en de combinatie van de bindingen en brekingen
van bindingen signaleren aan de cel wat er moet gebeuren. Net zoals bij de chromatine remoddeling
complexen worden ook de enzymen die van invloed zijn op de histonstaarten sterk gereguleerd
Zelfstudie 2 (H6 Alberts):
Elke DNA streng bevat dus een nucleotide sequentie die complementair is aan de andere streng en
elke streng kan daarom ook als een template dienen voor de synthese van een nieuwe streng.
Genetisch materiaal kan dus gekopieerd worden indien de twee strengen van elkaar worden
getrokken en beiden dan als template voor een nieuwe streng dienen. Dit wordt uitgevoerd door een
cluster van eiwitten die samen de replicatie machine worden genoemd. Elk van de dochter DNA
helixen eindigen met een oud kopie (de template) en een nieuw kopie. Deze vorm van replicatie heet
semiconservatief.
De DNA replicatie wordt begonnen door initiator eiwitten die aan het DNA binden en de
waterstofbindingen tussen de basen afbreken. Het punt waar het DNA als eerste wordt geopend
heet de origine van replicatie en wordt meestal gekenmerkt door een bepaalde volgorde van
nucleotiden. A-T basen worden door minder waterstofbruggen bijeen gehouden dat C-G basenparen
en daarom worden A-T paren vaker op deze replicatie beginpunten gevonden, ze zijn immers
makkelijk uit elkaar te trekken. Nadat de initiator aan het DNA gebonden is trekt het een andere
groep eiwitten aan, een eiwitmachine. Deze groep draagt de werkelijke replicatie uit.
DNA moleculen kunnen tijdens het proces van replicatie geobserveerd worden in een
elektronenmicroscoop. Hier kun je Y-vorm junctie zien in het DNA, de replicatievork genoemd. Twee
replicatievorken worden gevormd vanaf elke replicatieorigine, één richting de 3’ kant en één richting
de 5’ (bidirectioneel).
Essentieel binnen de replicatiemachine is DNA polymerase, welke vanaf een oude streng een nieuwe
streng synthetiseert. Dit enzym katalyseert de toevoeging van nieuwe nucleotiden aan de 3’ van de
groeiende streng, de synthese gaat dus ook van 5’ 3’. De nucleotiden beginnen de reactie als
energierijke trifosfaten en geven dus zo energie voor de condensatiereactie welke de nucleotiden
aan de keten zet. DNA polymerase is belangrijk om deze reactie te koppelen. Doordat de ontstane
pyrofosfaat (PPi) verder gehydrolyseerd wordt tot anorganisch fosfaat is deze reactie onomkeerbaar.
DNA polymerase blijft geassocieerd met het DNA en beweegt langs de streng stapsgewijs voor vele
cycli van de reactie.
Het feit dat polymerase slechts van 5’ naar 3’ synthetiseert geeft een probleem bij de vorken van
replicatie. Deze zijn namelijk asymmetrisch en gaan in beide richtingen. Dit probleem wordt opgelost
door de ‘achterwaartse steken manoeuvre’. In deze methode wordt het stuk DNA dat van 3 naar 5
gesynthetiseerd moet worden opgedeeld in kleinere stukken waarbij het polymerase in elk nieuw
stuk achterwaarts van 5 naar 3 synthetiseert. Deze stukjes DNA, Okazaki fragmente, worden later
aan elkaar ‘genaaid’ om een continue streng te maken. Het DNA dat op deze wijze gesynthetiseerd
wordt heet de lagging strand, de andere normale heet de leading strand.
DNA polymerase kan fouten corrigeren (bijvoorbeeld G-T paren), genaamd proofreading. Voordat
het enzym een nieuwe nucleotide aan de streng plaatst, kijkt het eerst even of de vorige goed is
aangeplakt. Zo niet, dan haalt polymerase deze er weer af en probeert het opnieuw. Deze zogeheten
exonuclease mogelijkheid gaat uiteraard de andere kant op van 3’- 5’. Dit proofreading mechanisme
verklaart direct waarom polymerase alleen van 5’- 3’ synthetiseert, andersom geeft namelijk niet
meer de mogelijkheid tot proofreading. Bij het afhalen van een foute nucleotide ontstaat een dood
einde waarbij polymerase niet verder kan.
Omdat DNA polymerase alleen maar nucleotiden kan toevoegen aan een groeiende dubbele helix is
een ander enzym nodig om een geheel nieuwe streng te beginnen. Dit enzym maakt echter niet DNA
maar korte stukken van een ander gerelateerd type nucleide zuur, RNA, waarbij DNA als een
template wordt gebruikt. Dit stukje zorgt voor en 3’ voor DNA polymerase om op verder te bouwen,
het is dus een primer. Het enzym dat deze stukken maakt staat dan ook bekend als primase. RNA
lijkt in principe heel erg op DNA alleen is het suiker ribose en geen deoxyribose, en het bevat Uracil
i.p.v. Thymine. U paart wel met A. In de leading streng is RNA alleen nodig op de beginpunten van
replicatie, in de lagging streng zijn nieuwe primers continue nodig op alle gebroken stukken. Om van
deze stukken DNA en RNA uiteindelijk een complete streng te maken zijn een drietal extra enzymen
nodig. Deze verwijderen de RNA primer (nuclease), vervangen het met DNA (repair polymerase) en
plaatsen de losse DNA fragmenten aan elkaar (DNA ligase). ATP of NADH is nodig voor de ligase
activiteit. Primases kunnen nieuwe DNA strengen beginnen omdat ze niet aan proofreading doen, ze
maken dus ook veel fouten. Alleen ook omdat dit RNA in plaats van DNA bevatten zijn ze sowieso
verdachte kopieën en worden altijd vervangen door DNA.
Ondanks dat het logisch is te denken dat bovengenoemde 3 systemen onderdeel zijn van de
replicatie machine is het onduidelijk of dit werkelijk het geval is. Aan de kop van deze machine
vinden we sowieso wel de helicase, welke door het gebruik van ATP de helix los ritst tijdens het
bewegen. de single strand binding eiwitten zorgen door binding ervoor dat de helixen tijdelijk uit
elkaar blijven. De sliding clamp is vervolgens verantwoordelijk voor het gebonden houden van de
polymerase aan de DNAstreng. Aan de lagging kant houdt deze de polymerase gebonden tot een
Okazaki frament gecompleteerd is en laat dan los. Deze clamping eiwitten vormen een ring rond het
DNA en glijden zo langs de streng. Alle eiwitten die betrokken zijn bij de replicatie worden gezien als
één machine die als het geheel langs het DNA glijdt.
Op de uiteinden van de chromosomen komen we een probleem tegen, er is namelijk geen plek meer
om de RNA primer neer te leggen en een Ozakaki fragment te starten. Eukaryoten lossen dit
probleem op door aan deze uiteinden punten te hebben die telomeren heten. Deze teeomeren
hebben een bepaalde DNA sequentie welke telomerase aantrekt. Deze prodceren een aantal
kopieën van deze segementen aan de uiteinden waar de primer op kan landen. Deze telomeren
hebben tevens de functie ervoor te zorgen dat deze delen van de chromosomen de werkelijke
uiteinden zijn en niet zomaar gaten in het midden.
Zelfstudie 3 (H6 Alberts, H7 Russel):
Veel van de fouten en veranderingen die in DNA plaatsvinden worden direct door DNA herstel
ongedaan gemaakt. Echter permanente veranderingen kunnen ook plaatsvinden en heten dan
mutaties. Deze mutaties leiden uiteindelijk in sommige gevallen tot andere aminozuurvolgorden in
eiwitten en daarmee vaak tot verlies van functie. Bij Sikkelcel anemie is dit bijvoorbeeld het geval
waardoor de bèta globine keten niet meer goed functioneert. Er zijn verschillende soorten mutaties
mogelijk:
1. depurinatie. Dit is het verlies van purinen (A en G). het lijkt op missende tanden in de streng
omdat de backbone van het DNA wel normaal blijft. Aangezien er geen aanbindt punt meer
is voor polymerase stopt deze hier ook.
2. deaminatie: het verlies van een aminogroep van Cytosine waardoor Uracil wordt
geroduceerd. Als dit niet gerepareerd wordt ontstaat U-A en uiteindelijk A-T binding in plaats
van C-G Een transitiemutatie is het gevolg.
3. thymine dimeren: o.a. veroorzaakt door UVlicht, zorgt voor een covalente binding tussn
twee pyrimidinen en voornamelijk thymine.
Wanneer deze fouten onveranderd blijven leiden ze vaak tot substitutie van één base in een andere,
of deletie van paren. Sommige van deze veranderingen stoppen de polymerase reactie, bijvoorbeeld
de thymine dimeren, waardoor replicatie niet vervolgt.
In russel wordt vervolgens dieper ingegaan op deze stof.
Mutagenese is de creatie van mutaties deze kunnen spontaan optreden maar ook geïnduceerd
worden door bijvoorbeeld mutagenen. Geïnduceerde mutaties komen over het algemeen veel vaker
voor dan spontane mutaties.
1. spontane mutaties: puntmutaties kunnen spontaan voorkomen tijdens de replicatie en
tijdens de G1 en G fase. De meeste spontane mutaties worden door herstelwerkzaamheden
wel weer gecorrigeerd.
DNA replicatiefouten: base paar substitutie fouten ontstaan wanneer één base paar wordt
veranderd in een ander base paar. Chemisch gezien bestaat een base uit verschillende
chemische varianten, tautomeren. Wanneer een base tussen deze vormen wisselt wordt het
gezegd een tautomerische shift te ondergaan. Een niet Watson-Crick paring (dus geen AT;CG)
kan ontstaan wanneer een base zich in een zeldzame tautomerische staat is, de enol vorm.
Kleine toevoegingen of deleties kunnen ook spontaan ontstaan wanneer er een base ‘looping
out’ doet van de template of de groeiende streng. Aan de template kant skipt de polymerase
de base (deletie) aan de groeiende streng maakt de polymerase de base twee maal additie).
Wanneer dit in een coderend gebied voor een gen plaats vindt ontstaan frameshift mutaties.
Zowel het DNA van eukaryoten als prokaryoten bevatten kleine hoeveelheden 5methylcytosie (5m-C) in plaats van normale cytosine. Deaminatie van 5mC zorgt voor
thymine. Als gevolg ontstaat er een GC naar AT transitie. Locaties waar 5mC in het genoom
zitten worden ook wel mutational hotspots genoemd.
2. geïnduceerde mutaties: röntgen straling produceren mutaties doordat deze de covalente
binding in de backbone van DNA breken. Dit kan dus leiden tot grote chomosomale mutaties.
UV licht produceert mutaties doordat ze fotochemische veranderingen teweeg brengen
waardoor abnormale covalente bindingen ontstaan. Met name aangrenzende pyrimidinen of
overstaande pyrimiden zijn hier de dupe van, thymine dimeren. Dit ongewenste paren zorgt
voor een bult in het DNA en voorkomt normaal paren van deze T’s met A’s. polymerase komt
niet voorbij deze leasie en bij genoeg leasies gaat de cel dood. Chemische mutagenen
kunnen ook mutaties veroorzaken en worden meestal ingedeeld op basis van hun
mechanismen van actie:
a. base analogen: ook deze bestaan net als normale basen in tautomerische staten en
zijn zo gelijkend op normale basen dat ze in het DNA gecorporeerd kunnen worden.
5BU is een bekende base analoog. In de normale staat lijkt deze op thymine, in de
zelzame staat lijkt het op C. wanneer 5BU wisselt van tautomeer nadat het in het
DNA is geplaatst kan het mutaties veroorzaken, transitie van TA naar CG, andersom
kan ook. Niet alle base analogen zijn mutageen, zolang ze geen base paar
veranderingen teweeg brengen.
b. Base modifying agents: deze kunnen naast tijdens de replicatie ook op andere
momenten actie ondernemen. Ze veranderen namelijk de chemische samenstelling
en eigenschappen van basen. Ze kunnen bijvoorbeeld deamineren, wat uiteindeljik
een transitie als gevolg kan hebben.
c. Intercalating agenten: deze plaatsen zichzelf tussen basen in één of beide strengen,
waardoor de helix ontspant. Wanneer dit in de template is dan vindt een insertie
plaats aan de nieuwe streng omdat deze opaart met de agent. Na een extra ronde is
heeft een additie plaatsgevonden, ook al is de agent inmiddels weer verdwenen.
Wanneer de agent in de nieuwe DNA streng gaat zitten ipv de template is een deletie
het gevolg omdat de agent op de plaats van een goede base zit. Frameshift mutaties
kunnen van beiden het gevolg zijn.
Site specifieke mutagenese is nu mogelijk, waarbij op speciaal gekozen plaatsen een mutatie
geïnduceerd wordt. Deze gemuteerde genen kunnen terug gebracht in een cel en op de gevolgen
onderzocht.
Carcinogenen zijn specifieke chemicalieën die door middel van mutaties kanker kunnen veroorzaken.
De mutaties zijn over het algemeen base-paar substituties welke missense of nonsense mutaties
produceren, of addities en deleties welke frameshifts als gevolg hebben. De Ames test is speciaal
ontwikkeld om te testen of stoffen al dan niet mutageen zijn. Er worden hierbij bacteriën in een
kweek geplaatst die al dan niet auxotroof zijn voor histidine, de mutanten hebben histidine nodig om
te kunnen groeien, de normalen niet. De kweek bevat geen histidine, maar wel lever enzymen omdat
deze stoffen die getest worden mutageen kunnen makendoor activatie. Controle platen worden ook
ingezet die de teststof niet bevatten. Na een incubatieperiode bevatten de controle platen weinig
bacteriekolonies, omdat deze zelf geen histidine aanmaken. Wanneer de kolonie histidine kan
maken, zal een grotere kolonie te zien zijn en moet er dus een mutatie zijn opgetreden, wat dan door
de hulp van de chemische stof moet zijn gebeurt.
Echter, sommige carcinogene stoffen zijn toch negatief.
De duizenden random ontstane mutaties in het DNA worden gerepareerd door verschillende
mechanismen. Bijna al deze mechanismen zijn gebaseerd op het bestaan van 2 DNA kopieën, twee
strengen in een helix, en zorgt dus voor een back-up van de informatie. De meeste schade aan DNA
brengt constructies teweeg die normaal in het DNA helemaal niet voorkomen en worden dus
makkelijk herkend en hersteld. (herkend door nucleasen en uitknippen, terugzetten van DNA
polymerase, inkeping herstellen door ligase) De nucleasen kunnen gespecialiseerd zijn voor een
bepaald type DNA schade. Het reparatie systeem moet natuurlijk wel alleen de verkeerde kant
vervangen, maar hoe weet hij welke de verkeerde is? Het is niet helemaal zeker hoe dit gebeurt maar
wel bewijs dat de nieuwe gesynthetiseerde strengen kleine kiertjes in de streng bevatten die herkend
kunnen worden. Het reparatiesysteem komt voort uit een bepaald gen waar mensen normaliter twee
kopieën van hebben.
In russel wordt nog een aantal reparaties specifiek besproken:
1. alkylerende schade: deze ontstaat wanneer een agent een aklyl groep aan een base
plaatst op verschillende lokaties. Deze schade kan hersteld worden door een enzym O6methylguanine-methyltransferase welke de methylgroep weer kan verwijderen. Ook
voor gealkyleerde thymine bestaat een soortgelijk systeem. B
2. beschadigde basen worden bijna altijd gerepareerd door deze base te verwijderen en de
juiste te plaatsen, excision repair.
3. excisie repair (NER): is een systeem wat GEEN zonlicht nodig heeft om te kunnen
functioneren, en hersteld voornamelijk thymine dimeren en distorties van de helix. Dit
gebeurt door een heel stuk van DNA weg te knippen. Het geknipte stuk wordt
losgekoppeld van de complementaire streng en een nieuw stuk wordt aangezet.
Zelfstudie 4 (H7 Alberts):
de functies en eigenschappen van een eiwit wordt bepaald door de lineaire volgorde van aminozuren
en deze worden weer gecodeerd door DNA (of RNA beter). Het gaat van DNA RNA aminozuurvolgorde eiwit. Dit wordt ook wel het centrale dogma van genetica genoemd. DNA naar
RNA heet transcriptie, RNA naar eiwit heet translatie.
Veel identieke RNA kopieën kunnen gemaakt worden van de transcriptie van één gen en elk van deze
RNA moleculen kunnen weer een eiwit maken. Dit zorgt voor snelle en efficiënte synthese. Elk gen
kan tevens ook getranscribeerd worden op verschillende frequenties, waardoor verschillende
hoeveelheden van verschillende eiwitten mogelijk zijn.
Ondanks dat de chemische verschillen tussen DNA en RNA klein zijn is de algehele structuur heel erg
verschillend. RNA heeft slechts één streng en kan om deze reden in verschillende vormen opvouwen
door vorming van base-paren tussen verschillende gebieden van het streng . RNA kan naast het
opslaan van informatie ook een katalytische en structurele functie benutten. Alle RNA in de cel wordt
gemaakt door transcriptie wat begint met de opening van een klein stuk DNA. Eén van de twee DNA
strengen dient dan als template voor de transcriptie waardoor met één nucleotide per keer een
streng wordt gevormd, die complementair is aan het DNA. Deze transcriptie verschilt echter van DNA
replicatie op een aantal cruciale punten:
1. de nieuw gevormde streng RNA blijft niet aan het DNA gebonden. De helix hervormt en
vervangt het RNA, de RNA wordt losgelaten.
2. doordat RNA van een beperkt gedeelte wordt gekopieerd zijn deze moleculen korter dan
DNA.
3. RNA polymerase voert de transcriptie uit in de 5’ 3’ richting. Deze kan de transcriptie
zonder primer beginnen en maakt hierdoor wel meer fouten. Dit is ook niet erg aangezien
één RNA molecuul slechts een aantal eiwitten codeert.
RNA moleculen die van genen worden gekopieerd worden en gebruikt voor de sysnthese van
eiwitten worden mRNA’s genoemd. Andere genen die niet voor een eiwit dienen hebben als
eindproduct slechts RNA, oftewel nonmessenger, RNA;s. Ribosomaal RNA (rRNA) is hier een
voorbeeld van welke de onderdelen van ribosomen vormt waaraan de mRNA kan binden; tRNA,
transfer, vormt adaptoren die de aminozuren selecteren en op z’n plek houden tijdens de synthese in
een eiwit aan de ribosomen.
De manier waarop RNA het punt herkent waar de transcriptie moet beginnen verschilt een beetje. Bij
prokaryoten is het systeem echter simpeler en beginnen dus daarmee.
RNA polymerase blijft zwak plakken aan het DNA in de cel op willekeurige plaatsen door de
bewegingen. He RNA polymerase glijft dan langs het DNA tot het een regio promotor genoemd tegen
komt, waarna het stevig bindt. Deze promotor bevat een bepaalde sequentie welke de RNA synthese
indiceert. De DNA kan door RNA polymerase herkend worden in de dubbelhelische vorm door basen
aan de buitenzijde, na cotact opent de helix zich. Het verlengen van de DNA keten gaat vervolgens
door totdat er een andere sequentie wordt gevonden, de terminator. Bij bacterieën is een deel van
de polymerase, de sigma factor, verantwoordelijk voor het herkennen van de promotor, welke factor
van het DNA wordt losgelaten nadat ongeveer 10 nucleotiden zijn gesynthetiseerd. Hierdoor kan de
polymerase voorwaarts bewegen. Naat de polymerase bij een terminator wordt logelaten associeert
het weer met een vrije sigma factor, en zoekt opnieuw naar een promotor. Hele riedeltje opnieuw.
Omdat het DNA twee strengen bevat zou van elk gen twee soorten RNA moleculen gemaakt kunnen
worden, complementair aan elkaar. Echter, de promotor is asymmetrisch en bindt de polymerase in
één enkele oriëntatie, van 5’ 3’. Wanneer de polymerase correct aan de promotor gebonden is kan
het slechts op één streng plaatsvinden. Kijkend naar een chromosoom in het geheel kan de
transcriptie wel in beide kanten, afhankelijk van hoe het gen georiënteerd is. Een deel van DNA kan
echter wel alleen getranscribeerd worden wanneer het voorafgegaan wordt door een promotor.
De manier waarop een RNA transcript behandeld kan worden verschilt erg veel tussen eukaryoten en
prokaryoten. Bacterieël DNA ligt direct aan het cytoplasma terwijl in eukaryoten deze zich in de
nucleus bevinden. Zodra mRNA in beacterieën is gemaakt kunnen deze direct aan de ribosomen
binden. Bij eukaryoten moet het mRNA dus eerst uit de nucleus getransporteert worden. Daarnaast
moet het RNA eerst door een aanal verwerkingsstappen voordat het uit de nuceleus getransporteert
kan worden. De enzymen die hier verantwoordelijk voor zijn zitten eigenlijk op de staart van de RNA
polymerase en springen vervolgens op de nieuw gevormde RNA om de stappen uit te voeren. Twee
stappen die op mRNA moeten gebeueren zijn:
1. RNA capping: dit bevat een modificatie van het 5’einde van het transcript (het eerste
gesynthetiseerde deel). De RNA wordt gekapt door additie van en atypisch nucleotide (G)
welke een methylgroep bevat. Dit gebeurt al wanneer de streng zo’n 25 nucleotiden lang is.
2. polyadenylatie: voorziet het nieuwste gesynthetiseerde gedeelte (3’) met een speciale
structuur. Eerst wordt dit uiteinde getrimd en vervolgens afgemaakt met een herhaalde serie
van adeninen (A’s). om deze reden wordt dit uiteinde ook wel de Poly-A-staart genoemd.
Deze twee modificatie hebben waarschijnlijk als functie het RNA te stabiliseren voordat het het
cytoplasma induikt. Eveneens kan op deze manier het RNA als mRNA geïdentificeerd worden.
De meeste eukaryotsiche RNA’s moeten nog een andere modificatie ondergaan voordat ze werkelijk
functioneel zijn. De intronen moeten namelijk van de exonen onderscheiden worden en uitgeknipt,
terwijl deze wel tijdens de transcriptie in het DNA terecht zijn gekomen. Dit gebeurt door middel van
RNA splicing. Elk intron bevat en aantl nucelotide sequenties die als sleutel dienen voor verwijdering.
Deze worden bij of op het einde van het intron gevonden en de intro wordt vervolgens in een lasso
gevormd. De gehele lasso is vervolgens verwijderd van het RNA. Dit splicen van RNA wordt
voornamelijk door RNA moleculen zelf gedaan dan door eiwitten. Deze snRNA’s, smalle nucleaire
RNA, binden aan kleinere eiwitpartikelen (snRNP’s) welke het hart van het spliceosoom vormen,
welke de uiteindelijke splicing uitvoert. Ondanks dat de snRNPs cruciaal zijn in de reactie zijn
toevoegingen van eiwitten ook noodzakelijk.
De intron-exon structuur lijkt handig in de evlutie geweest te zijn waar het gedacht werd de vorming
van nieuwe enen sneller mogelijk te maken. De manier van splicing biedt eveneens een methode om
verschillende RNA producten te creëren uit eenzelfde gen, door verschillende manieren van splicing
afhankelijk van celtype.
Een klein deel van de RNA is uiteindelijk maar bruikbaar voor de cel en de overige delen moeten dan
ook afgebroken worden om niet schadelijk te worden. Om dit te kunnen doen moet er natuurlijk wel
een onderscheid gemaakt kunnen worden tussen de goede en afvalproducten. Dit gebeurt doordat
het transport van de nucleus naar het cytoplasma hoog selectief is en alleen complete mRNA
transporteert. Om export klaar te zijn moeten mRNA moleculen aan een geschikte set eiwitten
gebonden zijn. Naast het cappen en toevoegen van een poy-A-staart zetten deze ook een signaal op
complete mRNA. Alleen bij de aanweizgheid van de hele set wordt het mRNA getransporteerd.
Omdat een mRNA molecuul meerdere malen voor een eiwit kan coderen is de hoeveelheid tijd die in
het cytoplasma wordt doorgebracht cruciaal voor de hoeveelheid geproduceerd eiwit. Elk mRNA
molecuul wordt uiteindelijk wel door RNases afgebroken. De verschillende levenslengtes worden
onder andere bepaald door nucleotide sequenties die in het mRNA zelf zitten en heten de
ongetransleerde ruimte. Deze coderen dus niet voor een eiwit maar zijn wel aanwezig in volwassen
mRNA.
Zelfstudie 5 (7en 20 Alberts; H7 Russel):
De regels waarbij een DNA/RNA sequentie wordt omgezet in een eiwit wordt de genetische code
genoemd. De nucleotiden worden gelezen in groepen van 3 wat 64 verschillende combinaties maakt,
terwijl er slechts 20 aminozuren zijn. Meer dan één triplet kan dus voor een aminozuur coderen. Elke
groep van 3 nucleotiden wordt een codo genoemd, en zijn universeel, zelfs in mitochondria komen
deze voor hoewel daar kleine veranderingen voorkomen.
In principe kan elk stuk RNA in 3 afzonderlijke niet overlappende leesframes geplaatst worden,
afhankelijk waar de codering begint. Deze codons reageren niet direct op een aminozuur waar ze
voor coderen maar binden in plaats daarvan aan een adaptor molecuul welke aan de ene zijde aan
het codon bindt en aan de andere zijde aan een aminozuur. Deze adaptoren bestaan uit een set
kleine RNA moleculen, tRNA’s.
We zagen eerder dat RNA een 3d vorm kan maken en zelfs in een helische structuur kan vouwen en
dit is ook precies wat er in tRNA gebeurt. 4 segementen van tRNA zijn dubbel helisch en produceren
zo een soort van klavertje. Dit gebeurt eveneens door een basepaar vorming tussen stukken van het
tRNA.het klavertje ondervindt verdere vouwing in een L-vorm structuur welke door
waterstofbruggen bij elkaar gehouden wordt. De twee uiteinden van deze L vorm welke ongepaarde
basen bevat vormt de essentiële fnctie van tRNA in eiwitsythese. Eén van deze gebieden vormt het
anticodon, een set van 3 of meer nucleotiden die complementair aan het mRNA binden. De andere
kant is op het 3’einde van het tRNA en de plek waar een aminozuur aan het tRNA bindt.
Aangezien er meerdere codes zijn voor één enkel aminozuur zijn kunnen meerdere tRNAs voor één
aminozuur zijn en tRNA’s kunnen binden aan meerdere soorten codons. Vaak gebeurt dit laatste zo
dat een tRNA bindt als de eerste twee letters van het codon kloppen, de derde letter maakt dan vaak
niet meer uit (wobble base pairing).
Het correct herkennen en binden van het juiste aminozuur aan de tRNA gebeurt met behulp van
aminoacyl-tRNA-synthetase, welke het juiste aminozuur covalent aan het tRNA binden. Voor elk
aminozuur bestaat een ander sythetase. Specifieke nucleotiden in het anticodon en de aminozuurontvangende arm maken het mogelijk dat de correcte tRNA aan het synthetase gebonden wordt.
Door de afbraak van een hoge energieverbinding door de synthetase kan het amiozuur stevig en
hoog energetisch gebonden worden aan het tRNA. De energie van deze verbindin wordt later
gebruikt om het amnozuur aan een groeiend eiwit te binden.
Een ribosoom is eigenlijk het belangrijkste component die voor de eiwitsynthese zorgt. Het
beweegtover het mRNA, laat de tRNA op de juiste plaatsen binden, houdt de gebonden aminozuren
op de juiste plaats en zet uiteindelijk een eiwit in elkaar. Ook verschillende RNA moleculen, rRNA’s,
zijn hierbij betrokken. Ribosomen bestaan uit een groot en een klein deel. Het kleine deel matcht de
tRNA met het juiste codon op het mRNA en het grote deel katalyseert de vorming van peptide
bindingen tussen de aminozuren. De twee delen komen op en mRNA molecuul samen, vaakvlak bij
het 5’eind, het begin van translatie. Het beweegt vervolgens langs het mRNA, één codonper keer,
gebruikt de tRNA als adaptoren. Wanneer de synthese is afgelopen laten de twee ribosoomdelen
weer los.
Elk ribosoom beat drie bindingsplaatsen voor een tRNA molecuul, de A, P en E plaats. Een tRNA
molecuul wordt op de A en P plaats aan het mRNA gebonden wanneer deze een anticodon heeft die
past (met inbegrip van wobbling). Bij het plaatsen van een nieuw aminozuur wordt in eerste instantie
het tRNA op de A plaats vastgehouden. Het carboxyleinde van het groeiende eiwit wordt op de P
plaats losgemaakt om een peptidebinding aan te kunnen gaan met het aminozuur welke aan A
gebonden zit. Deze reactie wordt gekatalyseert door peptidyltransferase, welke een onderdeel van
het ribosoom is. Door een verschuiving tussen het kleine en grote deel van het ribosoom wordt het
groeiende eiwit naar P en E geduwd. Het kleine deel beweegt precies 3 nucleotiden lang het mRNA
waardoor het tRNA op de E plaats weer loslaat.
Het wordt altijd al gedacht dat de rRNA’s in een ribosoom een grote rol spelen in de algehele
structuur van het ribosoom en daarmee de mogelijkheid tot positionering van tRNA en mRNAs en
zelfs de katalytische functie voor het vormen van peptidebindingen. 2/3 van het ribosoom bestaat
dan ook uit rRNA, 1/3 uit eiwitten. Deze eiwitten liggen vaak aan het oppervlakte en vullen zo de
gaten van het gevouwen RNA op de voornaamste rol van deze eiwitten lijkt dus ook het stabiliseren
van de RNA kern en het toestaan van veranderingen in de rRNA conformatie welke nodig is voor
eiwitsynthese. Niet alleen de A,P en E bindingsplaatsen bestaan uit rRNA ook de voornaamste
katalytische functies worden uitgevoerd door RNA op de grote subunit. De RNA moleculen die dit
katalytisch vermogen bevatten heten ribozymen.
De locatie waar de eiwitsynthese begint is essentiëel omdat het het leesframe bepaalt voor de
gehele lengte van het mRNA. De stap van inititaie is eveneens belangrijk om een andere reden, het is
dé laatste locate waar bepaald kan worden of het mRNA überhaupt getransleert moet worden. De
frequentie van inititaie bepaald dus de frequentie van synthese.
De translatie van een mRNA begint bij de codon AUG, initiator tRNA bindt aan dit codon en draagt
altijd een methionine bij zich. Nieuw gesynthetiseerde eiwitten beginnen dus ook altijd met
methionine aan de N-zijde. Later wordt dit methionine door een protease vaak verwijderd. Het
initiator tRNA is wel verschillend van he normale tRNA dat methionine draagt.
De initiator tRNA wordt eerst in het kleine deel van het ribosoom gesleept samen met translatie
initiatie factoren. Van alle geladen (dus een aminozuurdragende) tRNA’s kan alleen de initiator strak
aan de P-plaats van het kleine deel binden.. de volgende stap is dat dit geladen kleine deel van het
ribosoom aan het 5’ van een mRNA bindt welke onder andere door de cap wordt herkend. Het kleine
deel zoekt nu verder naar de eerste AUG en vervolgens laten een aantal van de initiatorfactoren los
zodat het grote ribosoomdeel kan binden.
Dit mechanisme om een startcodon te vinden is anders in bacteriën omdat deze geen caps hebben
op het mRNA en zo dus niet weten aan welke kant te beginnen. Zij hebben dus andere soorten
codons die specifiek aan ribosomen binden. Een prokaryotisch ribosoom kan dus ook direct binden
aan een startcodon zolang er een aantal nucleotiden voorafgaan voor de plaatsing. De ribosoom
bindingsplaatsen zijn in prokaryoten ook nodig omdat ze vaak polycistronisch zijn, wat betekent dat
één mRNA vaak voor meerdere eiwitten codeert.
Het einde van een eiwitcodering wordt aangeven met een stopcodon, UAA, UAG, UGA. Deze worden
niet door een tRNA herkend maar geven wel een signaal aan het ribosoom dat de translatie moet
stoppen. Relese factoren binden aan het stopcodon welke dan de A-plaats van het ribosoom bereikt
en dit verandert de activiteit van peptidyltransferase. Hierdoor wordt een watermolecuul
gekatalyseerd in plaats van een aminozuur waardoor de tRNA het carboxyleinde van het eiwit loslaat,
het eiwit wordt in het cytoplasma losgelaten.
De synthese van een eiwit kost overigens veel energie 2 ATP per tRNA binding, dit is nog afgezien van
de samenstelling van het ribosoom en alle gebruikte factoren en enzymen.
Een nieuw ribosoom springt bijna direct op een mRNA deel zoda de vorige ribosoom net ver genoeg
is om die ruimte te bieden. De mRNA wordt daarom vaak als polyribosomen gezien, ze bevatten
meerdere ribosomen tegelijk (polysomen). Zowel prokaryoten als eukaryoten bevatten deze
polysomen maar bacteriën kunnen dit proces zelfs nog net iets meer versnellen. Deze kunnen
namelijk zelfs al aan het mRNA binden terwijl deze nog in transcriptie is. Dit door vrije binding van
ribosomen aan mRNA en het niet nodig zijn van modiicaties, die we wel bij eukaryoten terug zien
(capping).
Deze verschillen tussen eukaryoten en prokaryoten zijn overigens wel handig, aangezien bijvoorbeeld
veel antibiotica hierop werken. Antibiotica inhiberen vaak verschillende stappen van de
eiwitsynthese in bacteriën die eukaryoten niet treffen.
Nadat een eiwit wordt logelaten uit het ribosoom wordt het onderwerp van aandacht voor een groot
aantal controlestappen van de cel. Onder andere kan de cel het eiwit weer afbreken om zo
deconcentratie op peil te houden. Hier zijn gespecialiseerde paden voor die afbraak (proteolyse)
bepalen. De eiwitten die snel door proteasen worden afebroken zijn bestemd tot een korte
levensverwachting, zijn verkeerd gevouwen of beschadigd. De meeste eiwitten worden zelfs specifiek
door proteasomen gedaan, welke een centrale cilinder vormt waar de eiwitten als het ware
doorheen gaan. In deze tunnel zitten ook de proteasen, welke logisch handig in het midden van de
cilinder zitten en zo niet lukraak de hele el afbreken. Een bekende manier om een eiwit te merken
voor destructie en welke dus ook door proteasomen herkend wordt is het merken met ubiquitine.
Eiwitten die ook kort leven dienen te hebben bevatten vaak ook specifieke ubiuitine bindende
sequenties, ook beschadigde eiwitten maken gebruik van dit systeem.
Nu gaan we verder op de verschillende definities van mutaties. Ik ga ervan uit dat iedereen
somatische mutatie en geslachtscel mutatie wel kent.
Mutatie-rate: de waarschijnlijkheid dat een mutatie voorkomt als functie van tijd.
Mutatiefrequentie: de werkelijke voorkoming van een specifieke soort mutatie.
Punt mutaties veranderen sechts één of enkele base paren en kunnen grofweg ingedeeld worden in
twee typen:
1. base paar substitutie: één base paar wordt vervangen door een andere. Deze zijn weer
onder te scheiden in twee soorten.
a. Transitie: één basepaar wordt vervangen door het andere paar
b. Transversie: een purine-pyrimidine wordt omgedraaid naar een pyrimidinepurine, bijv. AT naar TA.
2. base-paar inserties of deleties. Hierbij worden stukken uit het DNA weggehaald of
toegevoegd. .
Afhankelijk van het effect van de mutatie kan deze ook nog ingedeeld worden.
1. missense: een ander aminozuur wordt gecodeerd en er ontstaat en ander eiwit.
Fenotypische veranderingen kunnen optreden.
2. nonsense: een mRNA stuk wordt veranderd naar een sopcodon, hierdoor stopt de eiwitketen
voortijdig en kortere peptideketens ontstaan. Vaak zijn deze niet-functioneel.
3. neutraal: ondanks dat er een nieuw aminozuur in de keten wordt gezet maakt dit niet uit
voor de functie van het eiwit, het is dus eigenlijk een subset van missense mutatie met
neutrale uitwerking.
4. silente mutatie: het veranderde codon codeert voor hetzelfde aminozuur en dus heeft de
mutatie geen enkel effec.
5. framesshift: bij toevoegingen of deleties van het DNA kunnen frameshift mutaties ontstaan.
Vaak resulteert dit in een niet functioneel eiwit.
Naast de indeling voor de gevolgen van aminozuren kun je mutaties ook indelen naar de
functiegevolgen voor een eiwit. Hierbij kijk je naar verlies van functie, welke vaak recessief
overgeërfd wordt omdat één goed allel vaak voldoende is om te compenseren voor het verlies. Het
erbij krijgen van een extra functie wordt vaak dominant overgeërfd omdat de extra overactiviteit niet
gecompenseerd kan worden, het andere allel gaat niet zijn activiteit aanpassen.
Zelfstudie 6 (H 6 en9 Alberts):
Van alle recombinatie mechanismen is waarschijnlijk homologe recombinatie het meest belangrijk.
Er zijn hier een aantal karakteristieken aan verbonden:
1. twee dubbel strengen DNA moleculen met sterk gelijkende delen (homologen)gaan zodanig
naast elkaar liggen dat deze homologe sequenties gelijk liggen. De DNA moleculen kunnen
dan over kruisen
2. het punt van overkruising kan overal plaatsvinden.
3. er worden geen sequenties veranderd, alleen op een ander DNA deel gezet
een groot aantal DNA sequenties kunnen zich ook binnen een genoom zelf verplaatsen en
vermenigvuldigen, de transposonen. Deze zijn dus niet afhankelijk van homologe DNA sequenties en
zijn mobiele sequenties die zich vrijelijk door het DNA kunnen bewegen. Virussen maken ook gebruik
van dit mehanisme om zich in en uit een host cel te nestelen. De meeste mobiele elementen kunnen
zich echter slechts binnen één cel verplaatsen maar hebben wel de neiging tot replicatie. Deze sitespecifieke recombinati wordt geholpen door speciale enzymen die de sequenties herkennen,
meestal gecreeërd door deze mobiele elementen zelf. Veel elementen dragen daarnaast ook andere
genen. De meeste transposonen bewegen door een ‘cut-and-paste’ mechanisme, simpel uitsnijden
op één plek en hervestigen op een andere plek. Sommigen doen aan replicatie. Er is echter nog een
bijzondere groep transposonen die via een RNA intermediair bewegen, de retrotransposonen. Deze
zijn uniek voor eukaryoten. Deze gaan van het donor DNA via transcriptie naar RNA. Via reverse
transcriptase kunnen ze op een andere plek in het DNA worden ingebouwd. Sommige van deze
coderen voor hun eigen reverse transcriptase (L1element) anderen zijn hiervoor van andere enzymen
afhankelijk (Alu).
Om de genetische veranderingen in evolutie te volgen, kijken we naar de geslachtscellen, deze
worden immers aan volgende generaties doorgegeven. Somatische cellen ondersteunen deze functie
alleen maar. Er zijn ten minste 5 belangrijke genetisce veranderingen die en rol spelen in de evolutie:
1. mutatie binnen een gen. Er is een kans van ongeveer 1 op de 10^10 dat er een puntmutatie
op een locatie plaatsvindt. Deze mutaties kunnen de functie van een gen door kleine
aanpassingen verbeteren, maar ook uitschakelen. Vaak doen ze echter geen van beiden en
zijn ze silent. Deze variant van mutaties kunnen in een genoom opstapelen over generaties
en op die manier soorten creëren.
2. gen duplicatie: een gen, een stuk DNA of zelfs een heel genoom kunnen gerepliceerd worden
waardoor een set van gerelateerde genen binnen één cel ontstaan. Dit is de belangrijkste
methode voor het genereren van nieuwe genen. De kopieën kunnen namelijk afzonderlijk
weer divergeren door mutatie. Het mechanisme dat de duplicatie op gang zet gebeurd door
een type recombinatie tussen twee homologe segmenten van chromosomen of delen van
chromosomen, welke identiek of vergelijkbaar in sequentie zijn. Normaal vindt deze
recombinatie ook plaats op gepaarde chromosomen, vaak de identieke twee chromosomen.
Soms echter vindt deze recombinatie plaats op korte herhalende DNAsequenties in plaats
van chromosomen, aan de tegenovergestelde zijden van een gen. Eén van de twee
chromosomen eindigt dus met een extra kopie van dat gen, terwijl de andere een stuk
minder heeft. Zodra deze gedupliceerd is kunnen onevenredige cross-overs ontstaan welke
extra kopieën aan de gedupliceerde set kunnen toevoegen door een gen te paren met het
gelijke gen op een ander chromosoom. (kijk naar fig. 9-5). Een goed voorbeeld van een famile
van genen waar dit ooit gebeurt is, is de globine familie. Er zijn tal van globine genen die van
een gemeenschappelijke voorouder afstammen door duplicatie. Er zijn meerdere van deze
globine genen die zogenaamde pseudogenen vormen, deze komen niet meer tot expressie.
Niet elke duplicatie leid dus tot een functioneel gen.
Bijna elk gen in het genoom bevat meerdere versies, wat indiceert dat een groot aantal
ronden van duplicaties ons op de huidige genen heeft gebracht, of dit het hele genoom in
één keer betreft of slechts delen is onduidelijk, hoewel het laatste waarschijnlijker is. Het
eerste is echter zeker weten wel mogelijk. Ook een exon kan door onevenredige overkruising
gedupliceerd worden.
3. gen deletie: omdat het bovenstaande mechanisme er ook voor zorgt dat er deleties plaats
vinden op het andere chromosoom dat juist tekort kwam bij onevenredige cross-over is het
logisch dat er vaak gelijktijdig ook een gendeletie voorkomt op een ander chromosoom.
4. exon shuffling: het proces van duplicatie die we gezien hebben bij exonen kan ook
voorkomen tusse verschillende genen. Dit heet dan exon shuffling. Recombinatie
gebeurtenissen die een zekere onnauwkeurigheid kunnen hebben door de aanwezigheid van
intronen, vergroten de kans op deze gebeurtenis aanzienlijk, omdat hierbij soms ook exonen
worden gekoppeld. De fout wordt dan niet hersteld.
5. horizontale genen transfer: de mobiele DNA fragmenten zoals hierboven beschreven
hebben een belangrijke functie in genoom veranderingen. Ze kunnen functies van bestaande
genen verstoren of veranderen, de regulatie ervan aanpassen of zelfs nieuwe genen creëren.
Een insertie van een trasposon kan leiden tot een spontane mutatie (insertie mutatie) .
eveneens kunnen ze dienen bij homologe recombinatie door als target te fungeren. Ondanks
dit hebben transposonen de meeste functie bij de evolutie van het genoom. Naast het
bewegen van zichzelf kunnen ze namelijk ook omliggende delen bewegen door samen te
werken met een ander transposon. Het stuk DNA dat tussen de twee transposonen inl ligt
wordt namelijk door transposase meegenomen. Een voorwaarde is wel dat de beide
transposonen van dezelfde transposase gebruik maken. Exonen kunnen op deze wijze
bijvoorbeeld heel makkelijk getransporteert worden, waardoor nieuwe genen uit bestaande
exonen gevormd kunnen woden. Onder bacteriën kunnen genen ook tussen organismen
worden uitgewisseld, maar dit is zeldzaam bij bacteriën. Dit kan echter wel in het begin van
leven op aarde een grote rol gespeeld hebben.
Sommige DNA delen kunnen vrijelijker muteren dan andere delen, namelijk de delen die niet
belangrijk zjn bij coderingen. Genen die een belangrijke functionele rol spelen zijn daarom zeer goed
bewaard gebleven (highly conserved). Deze goed bewaarde genen kunnen we het beste gebruiken
om relaties vast te stellen tussen verder gedisantieerde diersoorten. Dichter gerelateerde
diersoorten kun je beter focussen op neutrale veranderingen, deze hopen vrij gestaag op en kunnen
zo gebruikt worden om een schatting van de evoutionaire klok te maken. De rest van dit deel is
doorlezen, dus die vat ik niet samen.
2% van het genoom codeert slechts voor eiwit en structurele RNA . De rest is voornamelijk gebouwd
op transposen (45%) waarvan een groot deel niet meer kan bewegen door opvolgende mutaties en
hebben dus geen functie. Het zijn wel overblijfselen van de evolutie.
Een tweede eigenschap van het menselijk genoom is dat het heel erg groot is. Naast het bestaan van
intronen en exonen zijn er ook nog delen die de genexpressie beïnvloeden in tijd en celtype.
Daarnaast bevat het genoom toch een ontzettend groot aantal genen. Tot slot is het eigenlijk één
grote bende.
Zelfstudie 7 (H10 Alberts):
Grote stukken DNA kunnen worden afgebroken in kleine stukjes door mechanische apparaten; maar
de fragmenten die een specifiek gen bevatten zijn één van de vele fragmenten hierin.
Het isoleren en zuiveren gebeurt door restrictie nucleasen. Een nuclease katalyseert de hydrolyse van
een fosfodiester binding in een nucleïne zuur. Ze kunnen de dubbelstrengen DNA op specifieke
plaatsen doorknippen, welke worde vastgesteld door korte sequenties van nucleotide paren.
Bacteriën bevatten deze restrictie nucleasen. Hun eigen DNA wordt beschermd door chemische
modificatie van deze sequenties. Omdat deze enzymen de transfer tussen bepaalde bacteriesoorten
voorkomen, worden ze restrictie nuclease genoemd. Elke bacterie bevat een eigen restrictie nucleas.
Omdat de doelsequenties vrij kort zijn kan het DNA op basis van kans door het hele molecuul op
plaatsen worden doorgekipt. Wel zullen de verschillende nucleasen op steeds dezelfde plek knippen,
wat ze ontzettend bruikbaar maakt. Er komt altijd dezelfde set fragmenten uit.
Als het DNA in kleinere fragmenten is geknipt, moeten de DNA fragmenten nog gescheiden worden
van elkaar. Dit kan gebeuren door gel electroforese, welke de fragmenten scheidt op basis van de
lengte. Aan de een kant wordt DNA toegevoegd in agarose of polyacrylamide gel, welke poriën
bevatten. Vervolgens wordt er spanning op gezet. Omdat DNA negatief geladen is, zullen de
fragmenten bewegen naar de positieve electrode; de grotere fragmenten migreren langzamer door
de matrix. Zo worden de DNA fragmenten verspreid over de gel volgens de grootte, waardoor
banden gevormd worden. Deze banden kunnen met een klein mesje uitgesneden worden om het
specifieke stuk DNA te verkrijgen. DNA banden zijn onzichtbaar en moeten daarom gelabeld worden.
Dit kan door verf dat fluoresceert onder ultraviolet licht. Een andere manier is een radioistoop toe te
voegen. Bv. 32P; kan in fosfaten van DNA en zendt β straling uit dat gedetecteerd kan worden door
autoradiografie. Door knippen en scheiding van DNA fragmenten in banden ontstaan fysische
mappen van kleine delen DNA. Een fysieke map karakteriseert een deel DNA door de positie te
bekijken van verschillende landmarken; restrictie nuclease sites zijn een type landmark. Door de
grootte van DNA fragmenten te vergelijken na behandeling met verschillende combinaties van
nucleasen, kan een fysieke map gemaakt worden van ieder gebied waar de plaats van iedere knipsite
te zien is. Dit is een restrictie map.
Nadat we stukken DNA hebben kunnen we deze gerbuiken om de volgorde van de nucleotiden vast
te stellen. De dideoxy methode wordt het meest gebruikt om DNA te sequencen. DNA synthese
wordt in vitro gedaan met dideoxyribonucleoside trifosfaten. DNA polymerase wordt gebruikt om
gedeeltelijke kopieën te maken van de DNA fragmenten. Deze DNA replicaties worden onder
condities gedaan die zorgen dat de nieuwe DNA strengen eindigen als een bepaalde nucleotide
bereikt is (wanneer een dideoxyribonucleoside zich voegt). Deze methode zorgt voor een collectie
van verschillend DNA dat eindigt op iedere specifieke positie in het DNA. Deze DNA strengen kunnen
gescheiden worden door gel elcetroforese en de nucleotidesequentie kan bekeken worden door de
volgorde van de fragmenten in de gel.
Tegenwoordig gaat dit alles automatisch. Dit proces wordt vergemakkelijkt door keten-eindigende
nucleotides die worden gekleurd met verschillend gekleurde fluorescente verf.
Door overlappingen te creëren van herhalingen van de proef, kan de sequentie uiteindelijk gevonden
worden
Als we een gen kennen, willen we weten wanneer in de ontwikkeling en waar het gen actief is. We
willen soms ook weten of een organisme een gerelateerd gen bezit. Soms willen we onderzoeken of
er een mutatie in het gen zit. Aangezien DNA stukken specifiek binden aan complementaire dele
kunnen we hier gebruik van maken om specifieke sequenties op te spoeren. Hybridisatie is de
techniek die hier gebruik van maakt. De DNA dubbele helix kan worden verbroken bij 90 graden of
een extreme pH. Wanneer de temperatuur weer verlaagd wordt, hervormt zich de dubbele helix. Dit
heet hybridisatie of renaturatie. Deze reactie kan ook plaatsvinden tusen DNA/RNA, als ze
complementair zijn. Na het verbreken kan men dus ook een complementaire sequentie toevoegen
om te kijken of deze in het stuk DNA aanwezig is.
Om een nucleotide sequentie door hybridisatie op te zoeken, moet men op zoek zijn naar een
bepaalde sequentie. Deze DNA probe is een enkelstrenging DNA molecuul, dat gebruikt wordt bij
hybridisatie om een complementaire sequentie te herkennen. Tegenwoordig kan men elke sequentie
maken die men wil. Men kan door deze methode in de vroegontwikkeling erfelijke ziektes
ontdekken, wat helemaal makkelijk is wanneer deze recessief is. Eén enkel recessief allel wordt
namelijk niet in fenotype herkend.
Bij de herkenning van een mutatie worden twee probes gebruikt: eentje die het normale gen bevat
en eentje die de mutatie bevat. Hierdoor kan men DNA onderscheiden in de genen. Een manier om
hybridisatie weer te geven is Southern blotting. Hierbij maakt men gebruik van gel-transfer
hybridisatie. De aan een probe gebonden stuk DNA zal na autoradiografie als banden zichtbaar
worden. De techniek die dit doet met RNA heet northern blotting.
Hybridisatie kan ook gebruikt worden om de expressie waarde van bepaalde genen te bepalen, silent
of getranscribeerd. DNA microarrays zorgt voor het analyseren van de expressie van vele genen
tegelijk, dus deze techniek is inmiddels veel populairder.
DNA microarrays zijn microscoop glaasjes met grote hoeveelheden DNA fragmenten, waarvan ieder
fragment een nucleotide sequentie bevat die als probe van het specifieke gen dient. De arrays
bevatten vele malen dezelfde sequentie en hierdoor kunnen hybridisatie reacties parallel lopen. Om
dit te kunnen doen, moet men mRNA van de cellen extraheren en kopiëren in een complementaire
DNA vorm. Dit complementaire DNA, cDNA, wordt gelabeld met een fluorescente probe. De
microarray bindt aan het cDNA en hybridisatie vindt plaats. Het glaasje wordt gewassen om
ongebonden moleculen te verwijderen en de positie waar het cDNA heeft gebonden wordt herkend
door fluorescente stippen. Deze positie wordt dan bekeken in vergelijking met welk gen het was.
Wanneer genen niet tot expressie komen zal er ook geen RNA van verkregen worden en laten ze zich
niet zien op het array.
Nucleïnezuur probes kunnen ook gebruikt worden om specifieke nucleïnezuur sequenties op te
zoeken in de cel of chromosoom zonder deze eruit te halen. Dit heet in situ hybridisatie (in de
plaats). Dit kan gebruikt worden om DNA op chromosomen of RNA sequenties in de cel te
herkennen. Door de DNA strengen aan een hoge pH bloot te stellen kan vervolgens hybridisatie
plaatsvinden. De strengen gaan los van elkaar en binden vervolgens aan een fluorescerende probe,
welke dan zichtbaar wordt. Dit zorgt voor de weergave van de positie van een gen op het
chromosoom. Ook kan het gebruikt worden om de distributie van genexpressie te bestuderen in
verschillende weefsels en cellen.
PCR (polymerase chain reaction) zorgt voor een snelle en goedkope manier om specifieke
sequenties in vitro te dupliceren, met name als de gehelegenoom sequentie bekend is. Hierdoor kan
van een bepaalde sequentie een enorm aantal klonen goedkoop en snel gemaakt.
PCR is gebaseerd op het gebruik van DNA polymerase om een DNA template te kopiëren in
herhaalde replicaties. Door een korte primer van oligonucleotides dat aan het template DNA
hybridiseerd aan het begin en einde wordt de polymerase naar een sequentie geleid. Deze primers
kunnen op beide strengen hechten. Het kan dus alleen gebruikt worden wanneer zowel het einde als
het begin van de sequentie van een gen bekend is. Het kan zowel op DNA als RNA (verkregen wordt
cDNA) gebruikt worden. Ook de infectie van pathogenen kunnen in een vroegstadium gedetecteerd
worden, waarbij primers complementair aan het pathogene genoom gebruikt worden. Verder wordt
PCR gebruikt bij forensisch onderzoek. Doordat kleine beetjes bloed of weefsel toch een DNA
fingerprint achterlaten kunnen deze makkelijk vermenigvuldigd worden voor uitgebreider onderzoek.
Zelfstudie 8 (H8 Alberts):
Cel differentiatie wordt bereikt door verschillen in gen expressie, niet verlies van de genen zelf.
Vandaar dat regulatie in gen expressie van groot belang is. Als DNA irreversibel veranderde tijdens de
ontwikkeling, zouden de chromosomen van gedifferentieerde cellen niet kunnen helpen bij de
ontwikkeling van een organisme. Om dit te testen, wordt de nucleus van een huidcel van een
volwassen kikker geïnjecteerd in een kikkerei waar de nucleus van verwijderd is. Soms ontwikkelde
het ei zich normaal, wat aangaf dat de nucleus van de huidcel niet zijn DNA verloren had. Deze soort
transplantaties zijn succesvol bij verschillende soorten cellen van verschillende dieren.
Door twee-dimensionale gel electroforese kan men het verschil in genexpressie tussen verschillende
celtypes globaal vergelijken door eiwitcomposities te vergelijken.
Veel eiwitten zijn algemeen in alle cellen van het organisme. Deze housekeeping eiwitten zijn
inclusief structurele eiwitten van chromosomen, RNA polymerases, DNA reparatie enzymen,
ribosomale eiwitten, enzymen betrokken bij glycolyse en andere basis metabolische processen en de
meeste eiwitten die het cytoskelet vormen. Daarnaast produceert ieder celtype zijn eigen
gespecialiseerde eiwitten die verantwoordelijk zijn voor de uiteenlopende eigenschappen van een
cel.
Veel eiwittten in een cel worden in zulke kleine aantallen geproduceerd dat ze niet door gel
electroforese onderzocht kunnen worden. De mRNAs die voor deze eiwitten coderen kunnen echter
wel worden gedetecteerd door gevoeligere technieken. Schattingen naar het aantal verschillende
mRNA sequenties suggereert dat een typisch gedifferentieerde humane cel rond de 10 á 30 000
genen tot expressie brengt. Het is de expressie van een verschillende collectie van genen in ieder
celtype dat de grote variatie maakt in grootte, vorm, gedrag en functie.
De meeste gespecialiseerde cellen in een multicellulair organisme kunnen hun patroon van
genexpressie veranderen door extracellulaire signalen. Verschillende celtypen reageren verschillend
op deze signalen.
De regulatie van ge expressie is op verschillende niveaus:
1. Regulatie wanneer en hoe vaak een gen getranscribeerd wordt.
2. Regulatie hoe het primaire RNA transcript gesplitst wordt of afgerond.
3. Selecteren welk mRNA getransleerd moet worden door ribosomen.
4. Selectief activeren of inactiveren van eiwitten na transcriptie.
De genregulatie wordt het meest beïnvloedt door stap 1. Dit komt omdat alleen transcriptie controle
kan zorgen dan geen onnodige intermediaire stoffen worden gemaakt.
Regulatie van transcriptie wordt meestal gedaan op het moment dat het proces geïnitieerd wordt.
Het promotor gebied van een gen trekt het RNA polymerase enzym en geeft de plaats voor het
enzym aan om te beginnen. Deze promoters bevatten een initiatie site, waar transcriptie begint en
een sequentie van rond de 50 nucleotiden waar RNA polymerase aan de promotor bindt. Verder
bevatten haast alle genen een regulatie DNA sequentie, die nodig is het gen uit en aan te schakelen.
Sommige regulatie DNA sequenties zijn kort (10N) en zijn simpele gen schakelaren die op één signaal
reageren. In eukaryoten zitten echter veel complexere schakelaren (10.000N), die reageren op
meerdere signalen, en zo tot een geïntegreerde microprocessor werken die instructies geeft.
Deze regulatie DNA sequentie kunnen niet alleen werken en moeten herkend worden door eiwitten
genaamd gen regulatie eiwitten die aan het DNA binden. De combinatie van deze twee bepaald de
schakelaar van transcriptie.
De eiwitten herkennen de specifieke DNA sequenties, omdat het oppervlak van de eiwitten sterk
past op het speciale oppervlak van de dubbele helix in dat gebied. Dit hangt af van de nucleotide
sequenties, zodat dus verschillende eiwitten op verschillende sequenties passen. Over het algemeen
gaan de eiwitten binnen in de helix zitten en maken moleculaire contacten met de base paren. Het
eiwit vormt waterstofbindingen, ionische bindingen en hydrofobe interacties met de randen van de
basen, zonder de waterstofbindingen tussen de basen te verstoren. Elke afzonderlijke binding is zwak
maar samen een van de sterkst bekende bindingen in de biologie.
Hoewel ieder eiwit-DNA herkenning uniek is in detail, bevatten de meeste eiwitten een van de
volgende stabiele e vouwpatronen, DNA-binding motifs:
A. Homeodomain (helix-turn-helix) cylinders
B. Zinc finger zink + alfa helix & beta sheet
C. Leucine zipper twee alfa helixen dimeren
Vaak binden DNA-binding eiwitten in paren (dimeren) aan het DNA. Dimerisatie verdubbeld het
gebied van contact met DNA, zodat de sterkte en specificatie vergroot van de interactie. Omdat
verschillende eiwitten gecombineerd kunnen worden, kunnen verschillende sequenties met slechts
een klein aantal eiwitten worden herkend.
Bacteriën reguleren hun genexpressie door de voedselbronnen die beschikbaar zijn in de omgeving.
De genen voor een bepaald enzym zijn geordend op het chromosoom en worden getranscribeerd
van één promoter als één lang mRNA molecuul (de genen zijn een operon) waaruit de eiwitten
worden gemaakt. Hierdoor worden meerdere genen dus gelijktijdig gereguleerd.
In de promoter zit een korte DNA sequentie (15 nucleotide lang) welke herkend wordt door een
genregulatie eiwit. Als het regulatie eiwit bindt aan de sequentie, de operator, blokkeert het de
toegang voor RNA polymerase; dit voorkomt transcriptie van de operon en productie van het enzym.
Het eiwit heet een repressor; dit eiwit kan gereguleerd worden door producten. De repressor is dan
een allosterisch eiwit: er kan een conformatieverandering optreden, zodat deze kan binden aan de
operator. Als de concentratie daalt van de producten, bindt de repressor niet langer aan het DNA en
wordt de operon getranscribeerd. De repressor kan dus de genexpressie in en uitschakelen.
De repressor is altijd aanwezig in de cel; het gen dat hiervoor codeert is altijd aanwezig op een laag
niveau, zodat kleine hoeveelheden aanwezig zijn. Deze ongereguleerde genexpressie heet
constitutieve genexpressie.
Repressoren zetten dus genen uit. Andere regulatie eiwitten schakelen juist genen aan, activeren ze.
Deze activator eiwitten beïnvloeden promotoren, door een binding met de RNA polymerase. De
promotoren worden soms slechts zwak gebonden met de polymerase. De activator eiwitten maken
de promotoren volledig funtioneel en binden met het RNA polymerase op een manier die helpt bij de
transcriptie. De activator’s mogelijkheid tot binden wordt vaak beïnvloed door interactie met andere
moleculen, bijvoorbeeld cAMP.
In veel gevallen wordt de activiteit van een enkele promotor gereguleerd door twee verschillende
signalen. Vb. lac door lac repressor en CAP activator. Bij afwezigheid van glucose, wil CAP het gen
aanzetten. Als echter lactose afwezig is, blokkeert de lac repressor de operator. Dus werkt alleen als
lactose in grote hoeveelheid aanwezig en glucose afwezig is.
De genetische schakelaren in eukaryoten zijn meer complex omdat een gen op meerdere signalen
reageert. Er zijn vier verschillen tussen eukaryoten en bacterieën:
- Verschil in RNA polymerase zelf. Bacteriën bestaan uit een type RNA polymerase,
eukaryoten in drie. Deze polymerasen zijn verantwoordelijk voor de transcriptie van
verschillende genen. I en III zorgen transcriberen de genen die coderen van tRNA, rRNA en
sRNA die een structurele en katalytische rol spelen in de cel. II transcribeert het merendeel
van de genen.
- Bacterieel RNA polymerase kan transcriptie initiëren zonder hulp van andere eiwitten.
Eukaryotische RNA polymerase hebben algemene transcriptie factoren nodig, welke moeten
binden aan iedere promotor met de polymerase voordat de polymerase met transcriptie kan
beginnen.
- De genregulatie eiwitten kunnen de initiatie van transcriptie beïnvloeden, zelfs als ze
duizenden nucleotiden van de promotor verwijderd zijn. Een enkele pronotor wordt dus
beïnvloed door duizende regulerende stukken in het DNA.
- Eukaryotische transcriptie initiatie moet rekening houden met de pakking van DNA in
nucleosomen en de meer compacte vormen van chromatine structuren.
Eukaryotische RNA polymerase kan niet zonder algemene transcriptiefactoren initiëren. Deze
factoren geven de positie voor de polymerase aan op de promotor, en helpen met het uit elkaar
trekken van de twee strengen DNA voor de transcriptie en te zorgen dat de polymerase de promotor
kan verlaten om transcriptie te starten. Deze factoren binden op alle promotoren (met RNA
polymerase II) en vandaar ook de term algemeen. Ze verschillen dus van repressoren en activatoren
in bacteriën en de gen regulerende eiwitten in eukaryoten die genspecifiek handelen.
Een voorbeeld van hoe dit werkt is met TFIID, welke bindt aan een specifieke korte sequentie die
voornamelijk uit T en A’s bestaat, vandaar ook wel TATA box. Hierdoor ontstaat een dramatische
lokale verandering in het DNA, wat helpt als een signaal zodat andere eiwitten kunnen binden aan de
promotor. Als eenmaal een factor gebonden is, binden andere factoren waaronder RNA polymerase,
om een transcriptie initiatie complex te vormen.
Hierna moet de polymerase losgelaten worden van het complex om te kunnen beginnen. Door
toevoeging van fosfaatgroepen aan de polymerase (door TFIIH), kan de polymerase ontbinden van de
rest. De fosforylering helpt ook andere eiwitten die het uiteindelijke RNA modificeren te binden aan
de polymerase. Als de transcriptie begint, laten de meeste transcriptiefactoren los, zodat ze weer
beschikbaar zijn voor een nieuwe transcriptieronde.
Ondanks dat de transcriptie factoren voldoende kunnen zijn in vitro, gebruiken eukaryoten in de cel
zelf ook activatoren die noodzakelijk zijn om de transcriptie te initieren.
De DNA sites waar activators binden heten enhancers, omdat hun aanwezigheid de snelheid van
transcriptie vergroot. Deze activator eiwitten kunnen op duizenden nucleotiden ver van de promotor
zitten. Bovendien maakt het niet aan welke kan van de promotor de enhancer zit.
Het DNA tussen enhancer en promotor wenkt uit om te zorgen dat het activator eiwit de promotor
direct kan beïnvloeden. Vaak zijn er ook eiwitten die dienen om de afstand tussen de twee te linken;
de bekendste hiervan is de mediator.
Een van de manieren dat activators helpen is bij de binding van algemene transcriptiefactoren en
polymerase aan de promotor.
Repressors kunnen het tegenovergestelde: zijn verminderen transcriptie door de binding van de
eiwitcomplexen tegen te houden.
Activators en repressor hebben nog een mechanisme: ze trekken eiwitten aan die de chromatine
structuur kunnen moduleren en zo dus de toegankelijkheid van een promotor voor de factoren en
polymerase beïnvloeden.
Hoewel alle cellen genen moeten kunnen in- en uitschakelen door de omgeving, kunnen cellen van
multicellulaire organismen zich definitief specialiseren. De bepaling van een bepaalde
celdifferentiatie blijft hetzelfde gedurende veel celgeneraties, ofwel de veranderingen in
genexpressie worden onthouden. Dit celgeheugen zorgt voor de creatie van georganiseerd en stabiel
weefsels. Kleine veranderingen in gen expressie zijn echter wel tijdelijk.
Omdat genregulatie eiwitten de transcriptie kunnen reguleren wanneer ze ver weg gebonden zijn
aan het DNA, kunnen de nucleotide sequenties die betrokken zijn bij de genexpressie verspreid
liggen op lange einden van het DNA. De stukken tussen deze sequenties zijn wel vaak niet funtionele
spacer DNA
De meeste eukaryotische genregulatie eiwitten werken samen, welke allemaal nodig zijn om een gen
tot expressie te brengen, bij juiste condities, tijd en juiste hoeveelheid.
Combinatorial control is de manier waarop groepen eiwitten samenwerken om een gen tot
expressie te brengen. Vele verschillende eiwitten binden aan de regulator sequentie om de
transcriptie te beïnvloeden. Vaak zijn sommige van deze eiwitten repressoren en andere activators.
De optelsom van deze effecten bepaald wat er uiteindelijk gebeurt.
Zelfstudie 9 (H5 en 8 Alberts; H12 en 20 Russel):
De cellen moeten de expressie van verschillende genen bij elkaar regelen. Als een cel een signaal
ontvangt van buitenaf om te delen, bijv., worden genen die normaal niet tot expressie komen samen
aangezet om de celdeling op gang te brengen. Een manier voor bacteriën is de genen samen te
clusteren onder een enkele operon, binnen de controle van een enkele promotor. Eukaryoten doen
dit anders, waar elk gen apart gereguleerd wordt.
Een eukaryotische cel gebruikt een groep van regulatie eiwitten om ieder gen te reguleren. Ook al is
de regulatie van genexpressie combinatoriaal, het effect van één genregulatie eiwit kan beslissend
zijn in het schakelen van een gen, door de combinatie compleet te maken die nodig is voor activatie
of repressie (analoog: cijferslot laatste invoer cijfer). Als een aantal verschillende genen een regulator
site bevatten met hetzelfde genregulatie eiwit, kan dit ookde regulatie van al deze genen
beïnvloeden.
Bv. Glucocorticoid receptor eiwit. Dit eiwit moet eerst binden aan een hormoon. Verschillende genen
worden vervolgens gereguleerd door de binding van het hormoon-receptor complex aan de regulatie
site van de genen.
De mogelijkheid om verschillende genen met een enkel eiwit in en uit te schakelen is ook van belang
bij de celdifferentiatie tijdens de embryonale ontwikkeling.
Bv. Ontwikkeling van spiercellen. Een spiercel wordt gevormd door de fusie van myoblasten. Deze
maakt grote aantallen van bepaalde eiwitten, zoals actine en myosine. De genen die coderen voor
deze spierspecifieke eiwitten worden gezamenlijk aangezet tijdens de fusie. Er zijn genregulatie
eiwitten die alleen in spiercellen tot expressie komen. Ze activeren de transcriptie van genen die
coderen voor de spierspecifieke eiwitten door te binden op de regulator gebieden. Deze eiwitten
kunnen nonmusculaire cellen omzetten in myoblasten door de veranderingen in genexpressie te
activeren.
Multicellulaire organismen hebben speciale genschakel mechanismen nodig om de verschillende
celtypes te genereren en te handhaven. De veranderingen in genexpressie die de celdifferentiatie tot
stand brengen worden doorgegeven aan de dochtercellen bij de celdeling, terwijl tijdelijke
veranderingen niet worden doorgegeven. De manier waarop cellen hiervoor zorgen is o.a. de
positieve feedback loop. Deze activeert een sleutel gen regulatie eiwit voor de transcriptie van zijn
eigen gen en die van de andere celspecifieke genen. Een andere manier is door de celspecifieke
chromatine structuur aan dochtercellen door te geven. Hoe dit precies gedaan wordt is onbekend.
Hoewel combinatorial control een norm is in eukaryotische genen, kan een enkel genregulatie eiwit,
als het de juiste combinatie compleet maakt, beslissend zijn bij het in- of uitschakelen van genen. Bv.
Ey kan heel het oog vormen. Ey reguleert de expressie van vele genen door te binden op de regulator
site. Sommige van deze genen coderen voor extra genregulatie eiwitten, die weer andere genen
kunnen beïnvloeden. Sommige eiwitten geven positieve feedback op de ey.
Initiatie van transcriptie moet altijd rekening houden met de pakking van DNA in chromatine. Het
genetische materiaal is gepakt in nucleosomen, welke in een hogere-orde structuur gevouwen zijn.
Nucleosomen kunnen de initiatie van transcriptie inhiberen als ze op de plaats van een promotor
zitten, omdat ze de binding van de algemene transcriptiefactoren of RNA polymerase blokkeren.
Chromatine voorkomt verkeerde genexpressie – initiatie van transcriptie in afwezigheid van de juiste
activator eiwitten.
Activator en repressor eiwitten benutten de chromatine structuur om genen aan of uit te zetten.
Cellen kunnen de chromatine structuur veranderen door chromatine remodeling complexes of de
histon eiwitten te veranderen die de kern van de nucleosomen vormen. Veel genactivators gebruiken
deze mechanismen om anders verborgen genen te activeren. De veranderingen in chromatine
structuur zorgen voor betere bereikbaarheid van het onderliggend DNA.
Gen repressor eiwitten kunnen de chromatine veranderen waardoor de efficiëntie van de
transcriptie initiatie verminderd wordt. Ze trekken enzymen aan (histon deacetylase) die de
acetylgroep van histonstaarten haalt, waardoor het onderliggend DNA minder toegankelijk wordt.
Na een complete deling ontvouwen de mitotische chromosomen in een meer uitgebreide vorm – de
interfase chromosomen. Gebieden waar de chromosomen genen bevatten die expressie tonen, zijn
meer uitgezet, terwijl andere genen meer compact zijn. De anier van condensatie verschilt dus ook
per celsoort, elke cel heeft een andere genexpressie.
De meest gecondenseerde vorm van de interfase chromatine is heterochromatine. Het zit meestal
rond het centromeer en bij de telomeren en beslaat ongeveer 10% van het DNA. Het meeste DNA
dat gevouwen is in heterochromatine bevat meestal geen genen. Genen die heterochroom worden
komen niet meer tot expressie omdat ze ongewoon compact zijn geworden en ontoegankelijk zijn.
De rest bevindt zich in een meer uitgezette staat, euchromatine. Ondanks dat de meeste
chromosomen een combinatie van beide soorten bevat zijn er uitzonderingen Vrouwelijke
zoogdieren schakelen één X chromosoom uit (anders dodelijk); één chromosoom wordt hoog
gecondenseerd in de heterochromatine, tijdens embryologische ontwikkeling. Deze inactieve staat
van het X-chromosoom is erfelijk voor alle dochtercellen.
De structuur van chromatines moeten veranderd worden om bij het DNA te kunnen komen en is dus
ook dynamisch. De toegankelijkheid is meestal beperkt tot die regio’s die op dat moment nodig zijn.
Er bestaan chromatin remodeling complexes, welke energie van ATP hydrolysering gebruiken om de
structuur van nucleosomen te veranderen. Tijdens mitose worden in ieder geval delen van deze
complexen gedeactiveerd, om zo de compacte structuur te kunnen behouden.
Er is ook een reversibele modificatie mogelijk van de histonstaarten. De N-terminus staarten van
ieder van de 4 kern histon eiwitten fungeren als regulator voor de chromatine structuur. Iedere
staart kan bepaalde covalente modificaties aangaan, welke toegevoegd en verwijderd worden nadat
de nucleosoom in elkaar is gezet door enzymen. Ondanks dat de mocificaties vaak niet direct effect
hebben op het nucleosoom heeft deze het wel op hogere orde structuren zoals de 30nm-vezel. Het
meeste effect zien we echter ergens anders. Door modificatie krijgen ze de mogelijkheid te binden en
hierdoor bepaalde eiwitten aan te trekken, om de chromatine uit te rekken. Verschillende
combinaties van de staartmodificaties en verschillende histon-bindende eiwitten kunnen
verschillende signalen geven: bijv. bepaald deel van chromatine is gekopieerd of genexpressie moet
plaatsvinden. De enzymen die de histonstaarten modificeren zijn sterk gereguleerd.
Naast modificatie van histonen kunnen genen ook gereguleerd worden door methylering van
cytosine. Gen silencing is het fenomeen waarbij op een gen geen transcriptie kan plaatsvinden door
zijn locatie, niet door de actie van specifieke repressoren. Gen silencing is een kenmerk van
heterochromatine en omsluit grote delen DNA en vele genen. Heterochromatine zit bijvoorbeeld bij
de telomeren en centromeren.
Bij de telomeer van gist is gen silencing die makkelijk uit te leggen is. Normaal bevinden zich daar
geen eiwitgenen. Maar als die er naar toe bewegen, worden ze gesilenced, door het telomeer positie
effect. Dit komt doordat de telomeer vier tot vijf keer verbonden is met de nucleaire envelop.
Door genverplaatsing naar een silenced gebied, kunnen mutanten gevonden worden. Zulke
mutanten zijn de silent informatie regulatie genen, SIR. Een ander eiwit, Rap1p, bindt aan de
telomeer herhaalsequentie. Eenmaal gebonden trekt deze Sir silencing eiwitcomplex aan. Dit Sir
complex staat in contact met de histonen en een histon deacetylase katalyseert de verwijdering van
de acetyl groepen van de histonstaarten. Er gaat vervolgens een golf van deacetylering langs het
chromosoom, wat een heterochromatine structuur creëert.
Hij wordt echter gestopt door de methylering van de histon H3 staarten. Deze wordt gekatalyseerd
door histon methyl transferase.
Transcriptie kan ook gesilenced worden door methylering van DNA sequenties. Dit kan ook grote
delen DNA bevatten. Hierbij gaat het om DNA methylering door DNA methylase modificerende
cytosines die 5-methylcytosine 5mC maken.
Genomic imprinting is een ander voorbeeld van methylering; hierbij wordt de genexpressie bepaald
door af het gen van de vader en moeder afkomstig is (epigenetisch!). Vb. van Igf2 en H19: Een enkele
enhancer reguleert de genexpressie van beide genen. Als een activator bindt aan de enhancer, kan
de transcriptie bij beide beginnen. Er bevindt zich echter een element tussen beide genen in,
insulator, die de activatie van de promotor aan de ene kant blokkeert en de enhancer zich aan de
andere kant bevindt. Door aan of uitzetten van insulator wordt ook de schakeling tussen Igf2-H19
gereguleerd.
De sleutel van imprinting is de methylering van specifieke DNA sequenties en de erfelijkheid van deze
gemethyleerde sequenties. Bij mitotische delingen is deze erfelijkheid duidelijk. Na DNA replicatie, is
iedere dochter DNA dubbele helix halfgemethyleerd; een streng wel en de andere niet. De
onderhoud methylasen herkennen deze halfgemethyleerde stukken en methyleren nu ook de andere
kant.
Bij meiose is dit echter anders. De methylering imprint is gevormd bij de parentale germ line en is
gereset iedere generatie. De moeder reactiveert de allelen, maar de vader geeft imprinting door.
Epigenetica is het doorgeven van een eigenschap, bijvoorbeeld het expressieniveau van een gen,
onafhankelijk van de genetische code. Dit gebeurt bijvoorbeeld ook bij het bovenbeschreven
inhiberen van één X-chromosoom in vrouwen. Dit laatste wordt ook wel dosis compensatie
genoemd. Organismen met sex chromosomen hebben ongelijke genhoeveelheid tussen de seksen:
twee X-en in vrouwen, 1 in mannen. Als de genexpressie van deze X chromosomen niet gehalveerd
wordt, heeft dit letale gevolgen. Er zijn verschillende dosis compensatie systemen. In vrouwelijke
zoogdieren bevatten de nuclei een hoog gecodenseerde massa chromatine – het Barr lichaampje,
wat niet bij mannen wordt teruggevonden. Dat houdt in, vrouwen hebben één Barr lichaam, mannen
geen. Dit wordt uitgebreid met de Lyon hypothese:
1. Het Barr lichaampje is hoog gecondenseerd en is een (grotendeels) inactief X chromosoom.
(Lyonisatie) Hierdoor blijft één X chromosoom over.
2. Het X chromosoom dat geinactiveerd is, is bij toeval gekozen, in een proces dat onafhankelijk
is van cel tot cel.
X inactivatie is een voorbeeld van een epigenetisch fenomeen; een overerfbare verandering in
genexpressie die gebeurt zonder verandering in de DNA sequentie.
Vrouwelijke zoogdieren die heterozygoot zijn voor bepaalde eigenschappen op het X chromosoom
hebben genetische mozaïeken; hierbij tonen sommigen cellen dus het ene X chromosoom en andere
dus het andere X chromosoom. Een goed voorbeeld hiervan is de lapjeskat, welke dus ook altijd
vrouwelijk zijn.
Bovengenoemd systeem bestaat niet voor afwijkende hoeveelheden autosomen.
Ook nadat RNA getranscribeerd is, is nog regulatie mogelijk. Individuele genen kunnen gesilenced
worden door een mechanisme genaamd RNAi. In RNAi, een dubbelstrengs RNA is geknipt in kleinere
fragmenten RNA (siRNA), welke door een complex van eiwitten binden aan complementaire
sequenties in mRNA en de deling van deze mRNAs triggeren. Een knockout of knockdown van de
genexpressie is het resultaat. RNAi geeft bescherming tegen virale infecties en regelt de
ontwikkelingsprocessen.
Zelfstudie 10 (H10 Alberts):
de eerder geleerde technieken van hybridisatie kunnen ook gebruikt worden om DNA te kloneren.
DNA moet in kleine stukjes kunnen worden geknipt en vervolgens weer bij elkaar gevoegd kunnen
worden. DNA ligase verzegelt de nicks in de DNA backbone die ontstaat bij DNA replicatie en
reparatie. Wanneer twee stukken DNA aan elkaar gezet zijn kan de cel niet meer zeggen dat ze ook
niet bij elkaar hoorden en beschouwt het dus als een enkel DNA molecuul.
Een manier om DNA te kopiëren, is het DNA in een bacterie te doen, door middel van transformatie.
Sommige bacteriën duwen het DNA door hun membraan. Door recombinatie wordt vervolgens het
DNA in het genoom toegevoegd.
Normaal wordt het DNA opgenomen in het genoom van de bacteriën. Soms wil men dit echter niet
en het DNA zuiver houden. Een oplossing hiervoor is het gebruik van plasmiden oftewel vectoren.
De plasmide gebruikt voor klonen zijn kleine circulaire DNA moleculen die in een bacterie kunnen
repliceren. Deze vector bevat een replicatie origin, welke replicatie mogelijk maakt onafhankelijk van
het bacteriële chromosoom. Het heeft ook een knipsite voor restrictie nucleasen, zodat de plasmide
geopend kan worden en vreemde DNA fragmenten toegevoegd kunnen worden. Verder bevatten ze
ook vaak een gen voor selectie, zoals antibiotica resistentie, waardoor de bacteriën die het DNA
opnemen makkelijk identificeerbaar zijn.
Om een stuk DNA dat gekloond moet worden in het plasmide te zetten, wordt het plasmide DNA
geknipt door een restrictie nuclease dat het slechts op één plek knipt en wordt het DNA fragment dat
gekloond moet worden covalent toegevoegd door DNA ligase. Door transformatie wordt dit
recombinant DNA molecuul toegevoegd in de bacterie (E. coli) en de kolonie moet vervolgens
groeien. Na een grote hoeveelheid replicatie wordt de bacterie gelysed en het plasmide DNA
gezuiverd van de rest van de cel. Het DNA fragment kan weer gepakt worden door knippen door
restrictie enzymen en scheiden door gel electroforese.
Het DNA dat ingebracht moet worden voor cloning moet uiteraard eerst gekozen en bewerkt
worden.
1. Het DNA wordt geknipt in kleinere fragmenten.
2. Het mengsel wordt dan in plasmide vectoren geplakt.
3. Deze verzameling van gekloonde DNA fragmenten in een bacteriële cultuur heet een DNA
library.
4. Via zuivering van eiwit wordt DNA probe gemaakt.
5. De DNA stukken met hybridisatie met probe worden herkend.
Het is vrij makkelijk om een gen vrij te maken van alle intronen. Wanneer dit is gebeurd noemen we
het cDNA (library). Het DNA in cDNA is niet genomisch DNA, maar een DNA kopie van het mRNA. Om
een cDNA library te maken, wordt het totale mRNA geëxtraheerd van de cel en DNA kopieën worden
gemaakt door reverse transcriptase met mRNA. Deze cDNA moleculen worden vervolgens gekloond.
Genomische klonen zijn een toevals product van al het DNA in het genoom. Ze bevatten grote delen
herhalings DNA sequenties, intronen, genregulatoren en vul DNA en exonen. cDNA bevat alleen
coderende sequenties en alles voor genen die getranscribeerd worden in mRNA in het weefsel waar
het RNA vandaan kwam.
Dus er ontstaan zo verschillende cDNA library’s voor verschillende weefselcellen. Ze kunnen dus ook
gebruikt worden om de genexpressie op verschillende ontwikkelingsstadia te onderzoeken. Het
allerbelangrijkste is echter dat cDNA een onverstoorde sequentie van het gen bevatten. Het grootste
voordeel van normale genomische klonen is echter dat de complete genoom sequentie kan worden
vastgesteld met alle intronen en regulerende gebieden.
Recombinante DNA moleculen kunnen gemaakt worden door DNA ligase. Bij productie van een DNA
library, is de ene DNA molecuul een vector en de andere een cDNA molecuul of een fragment van het
chromosoom. Zo’n DNA molecuul kan zorgen als vector voor de insertie en klonen van extra DNA en
bij herhaling van deze procedure, kan gekloond DNA gemaakt worden dat verschillend is van
‘normaal’ DNA. Deze kunnen gemaakt worden van natuurlijke DNA moleculen, maar ook van
sequenties door chemie gemaakt. Zo kunnen zeer zeldzame eiwitten makkelijker gemaakt worden.
Door het gebruik van speciale expressie vectoren kunnen eiwitten gemaakt worden. Deze vectoren
hebben een gen regulator en promotor DNA sequentie om transcriptie mogelijk te maken.Deze
vectoren kunnen de productie van grote aantallen mRNA, welke vervolgens omgezet kunnen worden
naar eiwitten, tot stand brengen. Anders dan bij klonen kunnen door deze expressie eigenschappen
veranderingen in de cel teweeg gebracht worden.
Er zijn verschillende expressie vectoren voor de verschillende soorten cellen en organismen. De
expressie vector wordt bij iedere rond van celdeling gerepliceerd, waardoor hele kolonies deze
exppressieeigenschappen kunnen ontwikkelen en het zeldzame eiwit produceren.
Omdat het eiwit in de cel zit, moet het gezuiverd worden uit de host cel na cellyse door
chromatografie.
DNA database
zoeken voor
gensequentie
DNA probe
synthetiseren
Aminozuur
sequentie van
peptide fragment
onderzoeken
Klonen door PCR
of onderzoek
cDNA of genomic
DNA library
Eiwit
In een host cel
voegen
Gen of
cDNA
In expressie
vector voegen
Over sommige eiwitten is slechts zeer weinig bekend. Door onderzoek naar waar en wanneer een
gen tot expressie komt kan meer informatie naar de functie van een eiwit worden gevonden. Het
patroon en de tijd van genexpressie te onderzoeken kan door het samenvoegen van een regulatie
gebied van een gen en een reporter gen – waarvan de activiteit makkelijk gezien kan worden.
Genexpressie wordt gereguleerd door regulator DNA sequenties. Deze sequenties, die reguleren
welke cel het gen tot expressie brengt en onder welke condities, kunnen ook de expressie van het
reporter gen reguleren. De hoeveelheid, tijd en cel-specifiekheid van het eiwit zal de functie van het
originele gen duidelijk maken. Het reporter gen wordt vaak onderzocht door middel van
fluorescentie of enzymactiviteit van het eiwitproduct. De meest gebruikte is het groene fluorescente
eiwit (GFP).
Mutante organisme geven de functie van genen het beste weer en beiden eveneens de mogelijkheid
het gen in het organisme zelf te onderzoeken.
Men begint door het isoleren van een mutant die een speciaal fenotype heeft. Hierna wordt het
genotype onderzocht. Recombinant DNA techniek maakte meer mogelijk. Men begint dan met een
gekloond gen en maakt mutaties in vitro. Door dit gen terug te plaatsen in het organisme, kan een
mutant organisme ontstaan, zodat de genfunctie kan worden weergegeven. Naast het gen zelf te
veranderen, kan ook het regulator gebied veranderd worden zodat het aantal geproduceerde
eiwitten veranderd, zodat expressie verschilt tijdens volwassen of ontwikkeling. Door manipulatie in
vitro is precieze mutatie mogelijk. Dit kan door site-directed mutagenese; door toegevoegd
gemuteerde primer en vervolgens replicatie ontstaat een gemuteerde versie. Door specifieke
aminozuren binnen een eiwit te veranderen kan men bijvoorbeeld vaststellen welke hiervan
noodzakelijk zijn bij eiwitvouwing.
Transgene organisme zijn organismen waarin een nieuw gen zit, of het genoom veranderd is door
recombinant DNA techniek. Homologe recombinatie met DNA dat een deletie in het gen bevat kan
leiden tot een grote deletie in het normale gen en complete inactivatie van het gen; knockout. Een
andere mogelijkheid is dat het gemuteerde gen toegevoegd wordt in het genoom zonder
verandering in het normale werkende gen.
Het is moeilijker genen te veranderen in diploïde organisme met grote en complexe genomen, zoals
muizen. Als het veranderde gen geïnduceerd kan worden in de cellen die in de germ line liggen,
kunnen zulke transgene dieren het doorgeven aan hun nageslacht. Bij mensen wordt dit om ethische
redenen niet gedaan. Somatische cellen, zoals in de lever, pancreas, been of huid reproduceren in
een individu maar niet in het nageslacht. Zo kunnen wel ziekten binnen één persoon aangepast
worden, maar niet doorgegeven aan afstammelingen. Het probleem blijft in de volgende generatie
dus wel bestaan.
Er is recent een gemakkelijkere manier ontdekt. RNA interference (RNAi). Bij deze techniek wordt een
dubbelstrengs RNA molecuul geïntroduceerd, waarvan de nucleotide sequentie overeenkomt met
het te inactiveren gen. Het RNA hybridiseert met het mRNA en laat het afbreken. Kleine fragmenten
van dit afgebroken RNA worden gebruikt om meer dubbelstrengs RNA te produceren welke continu
mRNA afbreekt. Omdat dit RNA door geslachtscellen kan, kan RNAi erfelijke veranderingen in
genexpressie veroorzaken.
RNA fragmenten, na afbraak in het cytosol, kunnen de nucleus binnentreden en interacties aangaan
met het gen zelf, zodat de pakking van transcriptie in een repressieve vorm van chromatine komt. Zo
kunnen dus genen ook individueel uitgezet worden
Zelfstudie 11 (H11,12,13 Russel):
Een testcross is een kruising tussen een individu en een homozygoot recessief individu.
Mendel’s wetten:
1. De twee homologe genen worden gescheiden bij de vorming van gameten.
2. Genen van verschillende genenparen worden onafhankelijk van elkaar gestuurd bij de
vorming van gemeten.
Eigenschappen die doorgegeven worden heten erfelijke eigenschappen. Deze eigenschappen liggen
in de genen. De genetische constitutie van een organisme het genotype, het fenotype zijn de
waarneembare eigenschappen, door interactie tussen genotype en omgeving (milieu).
Genen zijn een startpunt voor het onderzoeken van de structuur en functie van en organismen.
Organismen kunnen door zelfbevruchting voortplanten, vaak echter gebeurt dit door kruisbevruchting waar de mannelijke gameet fuseert met de vrouwelijke gameet.
True breeding houdt in dat een bepaalde eigenschap door allelel generaties lang ongewijzigd blijft.
De oudergeneratie heet de P generatie. De volgende generatie heet F1, daarna F2 enzovoorts.
Bij een monohybride kruising is slechts één gen betrokken.
Wanneer de eigenschappen bij zowel mannen als vrouwen op beide manieren gekruist worden
spreken we van reciprocale kruisingen (A man x a vrouw en a man x A vrouw). Als de resultaten
hetzelfde zijn, hangt de eigenschap niet van het geslacht af.
Als er sprake is van uniformiteit bij F1 (vaak allemaal heterozygoot), wordt er met deze doorgekruist.
Als hierbij beide fenotypen tot uiting komen, is er sprake van een dominant en recessief allel.
De specifieke locatie van een gen op het chromosoom heet het locus.
Gen scheiding is de loopt parellel aan de scheiding van de homologe paren chromosomen bij anafase
I tijdens meiose.
Een wild-type allel is meestal dominant over de gemuteerde (loss-of-function) allelen.
Kruisingen waarbij twee genen betrokken zijn, heet een dihybride kruising. Volgens Mendel zouden
deze dus onafhankelijk moeten overerven. Zelfs trihybride kruisingen zijn mogelijk, met 3
verschillende allelen.
Kijk voor jezelf nog even naar de chi-kwadraattest, deze zal ik hier niet behandelen. Onthouden is dat
wanneer de chi-uitkomst niet significant is (dus P is groter dan .05) dan zal de 0 hypothese behouden
worden. Dit stelt dan weer dat het verschil tussen de verwachte en de geobserveerde waarde geen
statistisch verschil is en Mendel’s wetten in de overerving gehanteerd worden.
Recessieve aandoeningen ontstaan door loss-of-function.
Dominante aandoeningen ontstaan door gain-of-function.
De chromosoom theorie van erfelijkheid zegt dat genen op chromosomen gelokaliseerd zijn en ook
middels die chromosomen overgeërfd worden. Seksechromosomen verschillen echter per sekse. In
mensen zijn mannen heterogametisch en de vrouw homogametisch, omdat mannen de helft van het
aantal X-en hebben t.o.v. de vrouwen worden ze voor de genen die zich op dit X chromosoom
bevinden ook wel hemizygoot genoemd. Deze genen die zich op de X bevinden worden ook wel
sekse gelinkt genoemd.
Bij meiose I: twee homologe chromosomen worden in twee verschillende geslachtscellen gesplitst
waardoor haploïde cellen ontstaan.
Bij meiose II: één chromosoom wordt in twee chromatiden gesplitst en verdeeld onder twee
gameten.
Wanneer de scheiding van één van bovenstaande stappen niet lukt spreken we over non-disjunctie.
Dit kan in zowel sekse als autosomen plaatsvinden. Primaire non disjunctie is wanneer het plaats
vindt in een individu met een normale set chromosomen. Secundaire is in nakomelingen van ouders
met reeds een non-disjunctie. Deze non-disjuncties kunnen leiden tot abnormale hoeveelheden
chromosomen, aneuploïditeit.
Het Y chromosoom bevat een gen of enen die de mannelijke seksuele differentiatie uitmaken. Dit
genproduct heet de testis-determinerende factor.
Vroege evidentie voor dit fiet komt voort uit de zeldzame individuen die één enkele X bevatten en
geen Y, het Turner Syndroom. Deze zijn steriel mensen die geen secundaire geslachtskenmerken
hebben.
Bij het klinefelter syndroom hebben mensen een Y en 2 X-en, XXY. Deze hebben van beide
geslachtskenmerken een beetje. Voor mannelijke ontwikkeling is dus een X en een Y nodig, voor
vrouwen 2 X-en.
Het vaststellen van sekse gebeurt in andere organismen ook wel eens anders, vogels hebben ZW,
waarbij het vrouwtje de heterozygoot is en man ZZ, dus andersom. Bij Drosophila wordt bijvoorbeeld
de ratio van het aantal X-en ten opzichte van de normale autosomen bepalend voor de sekse. Het Y
chromosoom heeft geen effect en wordt dus ook wel het X-chromosoom-autosoom balans systeem
genoemd. Een ratio van minder of gelijk aan .5 is man, daarboven vrouw. Soms gaat het zelfs niet om
een heel chromosoom bij organismen, maar gewoon het bevatten van een klein aantal genen die de
sekse bepalen.
Ook de dosis compensatie kan verschillen, bijvoorbeeld door verminderde transcriptie van het X
chromosoom.
Er zijn eigenschappen die via Xchromosomen worden overgedragen, X gebonden. Vaak zijn deze
recessief, vrouwen moeten er dus twee bevatten. Aangezien mannen slechts één X hebben komt
deze vaker tot expressie. Veel van deze aandoening komen dus ook vaker bij mannen voor. Sommige
aandoeningen worden dominant overgedragen. Bij Y gelinkte aandoeningen kunnen alleen mannen
het krijgen, maar dat lijtk me duidelijk. Deze worden ook wel holandrisch genoemd. Deze komen
echter wel heel erg weinig voor. Wat wel kan is dat het lijkt alsof dit het geval is maar dat het
eigenlijk voortkomt uit de interactie tussen het gen en mannelijke hormonen zoals testosteron.
Incomplete dominantie: het fenotype van heterozygoten ligt tussen de volledige eigenschappen van
homozygoten in.
Codominantie: het fenotype van heterozygoten is een optelsom van de twee allelen. Dit is anders
dan een tussenliggend fenotype maar is meer een optelsom van de fenotypen. Bijvoorbeeld Abloedgroep, B- bloedgroep en dan is AB een optelsom. O is als beiden ontbreken.
Zelfstudie 12:
Bij een complementatie test worden twee mutaties met hetzelfde fenotype gekruist. Het nageslacht
wordt geobserveerd, wanneer de mutaties op verschillende genen zijn zal het nageslacht wildtype/mutatie heterozygoot zijn en het fenotype dus wild type. wanneer het echter om hetzelfde gen
gaat zal het nageslacht twee mutante allelen bevatten en fenotypisch mutant zijn. Op deze manier
kun je onderzoeken of mutaties die in hetzelfde fenotype resulteren op hetzelfde gen liggen.
Een gen kan ook meer dan twee allelen bevatten die een genotype vormen. Echter moet je je hierbij
realiseren dat een diploide organisme slechts twee van deze meerdere allelen kan hebben. Een
voorbeeld hiervan is wederom bloedgroep type welke 3 allelen heeft A, B en O.
Wanneer twee allelenparen een fenotype beïnvloeden en op die manier een nieuw fenotype samen
creëren spreken we van een interactie tussen de genen. Bijvoorbeeld de interactie van twee
dominante allelen die normaal een bepaald fenotype produceren, produceren samen een heel ander
fenotype. De mendeliaanse ratio’s blijven wel gelijk.
Bij Epistase is er een interactie tussen twee of meer genen om een enkel fenotype te beïnvloeden.
De interactie betreft een (epistatisch) gen die de fenotypische expressie van een ander (hypostatisch)
gen maskeert. Bijvoorbeeld albinogen (geen pigment) maskeert de expressie van vachtkleur omdat
zonder pigment deze toch al niet tot uiting kan komen. Er worden geen nieuwe fenotype
geproduceerd maar de mendeliaanse ratio’s kunnen wel aangepast worden.
Het gen dat maskeert is epistatisch, het gen dat gemaskeert wordt is hypostatisch. Eveneens heb je
een onderscheid tussen dominante en recessieve epistase, afhankelijk van of één of twee allelel van
het gen voldoende zijn om epistatisch te worden.
Een allel dat resulteert in de dood van een organisme het een letaal allel en het gen hierbij heet een
essentieel gen, wild type nodig om te overleven. Ook is hier verschil in recessief en dominant.
Karakteristeik om de recessieve versie te herkennen is het 2:1 ratio van de afstammelingen. Immers
¼ is dood, de homozygoot recessieven. 2:1 = heterozygoot: homozygoot dominant. Letale allelen
kunnen ook sekse gelinkt zijn.
De penetrantie is de frequentie waarbij een dominant of homozygoot recessief gen tot uiting komt in
het individu in een populatie. Penetrantie hangt af van genotype en milieu. De penetrantie is
compleet wanneer beide homozygote varianten een ander fenotypen hebben en de heterozygoten
allemaal hetzelfde fenotype. Het wordt in procenten weergegeven als percentage individuen die het
fenotype van verwachting hebben.
Expressiviteit is de mate waarbij een penetrant gen of genotype fenotypisch tot expressie komt in
een individu. Dus de invloed van het gen op het fenotype. Ook dit hangt van de omgeving en het
genotype af en kan constant of variabel zijn. De combinatie van incomplete penetrantie en variabele
expressiviteit is ook mogelijk.
De Milieufactoren zijn:
• Leeftijd (verschillende interne veranderingen met leeftijd)
• Geslacht (sekse gebonden en door sekse beïnvloed)
• temperatuur
• chemicaliën
In hoeverre het milieu of het genotype het fenotype beïnvloeden blijft natuurlijk de bekende
nature/nurture discussie. Het potentiaal dat een genotype heeft om een bepaald fenotype neer te
zetten wordt de norm van de reactie genoemd. Binnen deze range hebben de milieufactoren invloed
op welk punt van de schaal het fenotype terecht komt.
Vaak worden genen samen geërfd omdat ze op hetzelfde chromosoom liggen. Deze zijn dan gelinkt
en erven dus niet onafhankelijk van elkaar over. Ondanks dit kunnen er toch combinaties van
gelinkte genen voorkomen die niet bij de ouders voorkomen, de zogenaamde recombinanten.
Een marker, of genetische marker is een andere naam voor een mutatie die een onderscheidbaar
fenotype geeft en een DNA marker laat de locatie van een gen op een chromosoom zien.
De locatie van genen is van belang omdat hoe groter de afstand tussen twee genen op een
chromosoom, hoe groter de kans op crossing-over en daarmee recombinatie van gelinkte genen.
Met markers kunnen deze locaties en afstanden tussen genen bepaald worden, een genetische map
gemaakt. Genetische recombinantie resulteert door fysieke uitwisseling tussen homologe
chromosomen tijdens de meiose. Een chiasma is de site van crossing-over. Crossing-over is een
wederzijdse gebeurtenis, tijdens profase I. Crossing-over wordt echter ook gebruikt om recominatie
te definiëren.
Wanneer genen niet gelinkt zijn hebben ze afzonderlijk in verschillende combinaties evenveel kans
om overgeërfd te worden, wanneer ze wel gelinkt zijn vinden we veel meer (te veel) van de ouder
combinaties terug, de recombinanten (andere combinaties dan bij de ouders terug gevonden) zijn
minder talrijk.
Koppeling is dat de wild type allelen op éénchromosoom voorkomenen de recessieve mutante
allelen op het andere chromosoom (w+ m+ // w m). Repulsie daarentegen is de organisatie waarbij
beide chromosomen een wild type en een mutant type dragen voor de verschillende allelen. (w+ m
// w m+ )
De genetische afstand tussen genen wordt gemeten in map units, waarbij één map uni wordt
gedefiniëerd als een afstand waarbinnen 1% crossing-over plaatsvindt. Dit heet ook wel centimorgan
(cM). Crossver en recombinatie frequenties geven een lineaire relatie tussen genen op een
chromosoom en kunnen dus ook gebruikt worden om de volgorde van de genen te bepalen. Voor
elke testkruising kan de recombinatiefrequentie niet meer zijn dan 50% omdat meer zou betekenen
dat ze onafhankelijk overerven omdat ze niet op hetzelfde chromosoom zitten of zo ver weg dat ze
haast niet meer gelinkt zijn.
Zelfstudie 13 (H15 Russel):
Om aan gene mapping te doen kan een testkruising gedaan worden bij diploïde organismen met 3
genen op een chromosoom, driepunts testkruising. Vaak wordt dit gedaan met een heterozygoot
voor alle allelen en een recessief homozygoot voor alle allelen. Bij een sekse gelinkt gen is de vrouw
altijd heterozygoot (bij mensen), de haan bij kippen.
Wanneer elk allel 2 mogelijkheden heeft zijn er voor alle drie samen 2^3 = 8 genotypen te bedenken.
1. De eerste stap is het uitzoeken van de volgorde van de genen op het chromosoom. De
parentale combinaties komen het meeste voor, dat levert bij bovengenoemde kruising twee
mogelijke genotypen. Aangezien één ouder homozygoot is ligt de informatie bij de
heterozygote ouder, daar namelijk kun je aan af zien hoe het doorgegeven wordt. Naast de
ouderlijke combinaties kunnen recombinanten door crossing over ontstaan waarbij een
enkele cross-over mogelijk is, of een dubbele (tussen gen 1-2 en tussen gen 2-3) Dubbele
cross-overs komen het minste voor, en deze veranderd alleen de oriëntatie van het
middelste gen.Je zoekt dus naar het gentype met de minste frequentie en kijkt dan welke van
de 3 genen afwijkt van wat je zou verwachten. Dit is het middelste gen. Aangezien een
chromosoom ruimtelijk is, maakt de positie van de andere twee niet uit, als je het
chromosoom omdraait wordt het toch weer anders.
2. nu kun je de recombinatie frequenties voor twee genen tegelijk uitrekenen. Alle cross-overs
(enkel en dubbel) in bijvoorbeeld regio 1-2 kun je optellen en delen door het totale aantal
nageslachten. Aan de hand van de gevonden percentages kun je nu de mapafstand tussen de
twee genen vinden.
3. De afstand tussen de twee buitenste enen (dus 1-3) is iets lastiger te doen. Dit is niet een
simpele optelsom omdat je de dubbele crossovers niet meegeteld hebt en deze als
oudertype ziet. Wat je echter wel moet doen is het percentage dubbele cross-overs van het
cM aftrekken. Dus stel chromosoom: A -------18 -------B --------9 -------C. Hier zou je denken
dat de totale afstand is 27. maar er heeft 5% dubbele crossover plaatsgevonden, dus is de
eigenlijke afstand 27 – 5 = 22. ( niet helemaal zeker, dus let op!!!)
je zou verwachten dat de kans op een dubbele cross-over de kans op crossover in 1-2 maal de kans
op cross-over is 2-3. dit is echter meestal niet het geval. Dit komt omdat de eerste cross-over
interfereert met een tweede cross-over. Het beïnvloedt de kans erop. De mate van inferentie wordt
ook wel uitgedrukt in het coëfficiënt van toeval en wordt berekend door het geobserveerde aantal
d.o. te vergelijken met de te verwachten c.o.’s. de interferentie is vervolgens 1 – het coëfficiënt.
De C.O. frequentie en de recombinatie frequentie is niet hetzelfde. Dit is makkelijk uit te leggen door
en voorbeeld te nemen van een chromosoom met twee genen (twee allelenparen) die enige afstand
van elkaar liggen. Bij een enkele c.o. wordt het genotype veranderd en bij elk ander oneven aantal
c.o.’s ook. Echter wanneer we een dubbele C.O. hebben op deze afstand blijft het genotype wel
gelijk. Hierdoor onderschat je de werkelijke afstand omdat de recombinatiefrequentie lager is dan de
frequentie aan werkelijke cross-overs. Hoe groter de afstand tussen twee allelenparen, hoe groter de
kans op zo’n onderschatting. Gelukkig zijn er wel wiskundige functies ontwikkeld die hiervoor
corrigeren. Deze doen wel veel aannames om dit mogelijk te maken en dus niet altijd even bruikbaar.
Zelfstudie 15 (H17 Russel (12 is in de rest van deze samenvatting terug te vinden):
De mitose is de normale celdeling, de meiose waarbij haploïde gameten worden gevormd.
Chromosomale mutaties zijn variaties in chromosoom aantal of structuur.
Er zijn vier soorten chromosomale mutaties in de structuur: deletie, duplicatie, inversie, translocatie.
Al deze mutaties beginnen met een breuk in het chromosoom. Polytene chromosomen zijn speciale
chromosomen in bepaalde weefsel. Ze bestaan uit chromatide bundels door duplicatie zonder
nucleaire deling en celding, endoreduplication.
1. deletie: Bij een chromosomale deletie mist een stuk van het chromosoom. Omdat er een
stuk mist kan deze mutatie niet weer omgedraaid worden naar het wilde type gen. De breuk
kan door allerlei externe factoren en interne factoren veroorzaakt worden. Wanneer de
deletie een centromeer bevat is het gevolg een acentrisch chromosoom, welke vaak tijdens
de meiose verloren gaat. De deletie van en heel chromosoom heeft uiteraard ernstige
gevolgen voor het organisme en kunnen zelfs dodelijk zijn. Er zijn geen levende mensen met
een heel missend chromosoom (teveel daarentegen wel). Alleen door het sekse
chromosoom dosis compensatie kunnen XO individuen bestaan, maar voor alle andere
autosomen bestaat dit niet. Wanneer door de deletie een dominant allel verdwijnt kan het
zo zijn dat één enkel recessief allel tot expressie komt, pseudodominantie.
2. duplicatie: hierbij is er een segment van het chromosoom verdubbelt en veroorzaakt dus
extra genen. Er zijn meerdere soorten duplicaties, bijvoorbeeld bij chromosoom ABCDEF:
Tandem: op de juiste volgorde t.o.v. elkaar: BCBC
Reverse tandem: omgekeerd t.o.v. elkaar: BCCB; Terminal tandem: aan het einde van het
chromosoo noemen we een tandem een terminale tandem: ABAB. Duplicaties hebben een
grote rol gespeeld in de evolutie zoals we eerder zagen.
3. inversie: bij deze mutatie wordt een fragment van het chromosoom eruit gehaald en
verkeerd om terug geplaatst. Dit is bijvoorbeeld bij ABCDEF ABEDCF. Er zijn twee typen
inversies: paracentrisch bevat het centromeer NIET, pericentrisch WEL. Over het algemeen
gaat er geen informatie verloren bij inversies, tenzij het breekpunt net essentiëel is voor gen
expressie. Wanneer de inversie voor beide allelen, homozygoot, is EN er vindt geen crossingover plaats dan zijn er geen consequenties voor de meiose. Wanneer er wel crossing-over
plaats vindt zijn er echter ernstige gevolgen! Bekijk hiervoor heel erg goed blz. 460 en 461 in
het boek, is niet in woorden te zetten!
4. translocatie: hierbij is er is er een verandering in de positie van een chromosoomfragment.
Er gaat geen genetsiche informatie verloren. Er zijn meerdere soorten trasnlocaties.
Nonreciprocal intrachromosomal translocatie: Verandering positie in hetzelfde
chromosoom.
Nonreciprocal interchromosomal translocatie: Tussen chromosomen.
Reciprocal interchromosomal translocatie: Uitwisseling tussen twee chromosomen.
Het gevolg van translocaties tussen chromosomen is een verandering in de gelinkte genen.
Tijdens de meiose zijn er wel een aantal problemen omdat de chromosomen niet gepaard
kunnen worden. Er ontstaan meer niet levensvatbare gameten door deleties of duplicaties.
Kijk hiervoor ook weer goed naar blz. 463, moeilijk in woorden te zetten.
De meeste kwaadaardige tumoren hebben chromosomale mutaties en met name translocatie komt
heel veel voor. Naast de problemen die we tegenkomen bij heterozygoten voor de mutatie tijdens de
meiose, is er nog een ander probleem. Het positie effect is een fenomeen waarbij een verandering in
de fenotypische expressie door een of meer genen veroorzaakt wordt door een verandering in
positie in genoom. Dit is een epigenetisch fenomeen – een overerfbare verandering in genexpressie
waarbij geen verandering in DNA sequentie plaatsvindt. Bv. gen in euchromatine heterochromatine.
Er kunnen eveneens fragile sites ontstaan, waardoor het chromosoom spontaan kan afbreken, wat
een deletie van het chromosoom resulteert. Dit is bijvoorbeeld te zien bij het fragiel X syndroom.
Een non-disjunctie tussen chromosomen is anders in meiose I dan in meiose II tijdens meiose I
worden namelijk 4 abnormale gameten gevormd, terwijl dit in meiose II slechts 2 abnormalen betreft
en 2 normalen. (terug te zien op p. 319)
Er zijn verschillende variaties in het chromosoomgetal die met vershillende termen worden
aangeduidt.
Euploidy: het organisme heeft een complete set (gezond)
Aneuploidy: een veranderd aantal chromosomen t.o.v. normaal, onder te verdelen in:
Nullisomy; verlies van een homoloog chromosomen paar. 2N-2
Monosomy; verlies van een chromosoom 2N - 1
Trisomy; extra chromosoom 2N + 1
Tetrasomy; twee extra chromosomen 2N + 2
Het kan ook voor meerdere chromosomen tegelijk plaatsvinden, dan spreken we van dubbel
monosomy bijvoorbeeld. Eigenlijk zijn alle monosomiën letaal,trisomieën komen nog wel eens voor.
Wanneer er een verlies is voor het gehele genoom kunnen we ook meerdere termen onderscheiden:
Monoploidy: verlies van 1 chromosoom van alle chromosoomparen. Vanwege recessieve
aandoeningen wordt dit vaak niet overleefd.
Polyploidy: van elk chromosoom meer dan de gebruikelijke 2. vaak is dit het gevolg van een kapotte
spindle tijdens de deling. Hierbij kan sprake zijn van een even aantal chromosomen of een oneven
aantal. Een even aantal heeft een betere vruchtbaarheid omdat er nog tijdens de meiose homologen
gescheiden kunnen worden. Ook hier is het echter niet optimaal. Triploidy bij mensen is het meest
voorkomend en altijd letaal.
Zelfstudie 18 (H9 en 10 Russel):
Men gebruikt genen als markers voor genetische analyse. Genen hebben verschillende allelen welke
verschillende fenotypen tot uiting brengen.
Om een genetische map te maken van de volgorde van de genen in het genoom, worden kruisingen
gemaakt van ouders verschillend in allel en de fractie nakomelingen met recombinanten fenotypes
worden onderzocht. Door deze mappen kan men de plaats, locus van ieder gen determineren.
Genetische mappen zijn handig bij het sequencen van het genoom, omdat ze aangeven waar de
genen liggen op het chromosoom.
Omdat sommige allelen op genen niet goed vindbaar zijn en een groot deel niet coderend is, is het
niet mogelijk een hoog gedetailleerde map te maken.
Men ging over op DNA polymorfisme; dit is een van de twee of meer alternatieve vormen (allelen)
van een chromosomale locus die of verschillen in nucleotide sequentie of verschillende tandem
herhalingen hebben. Een allel is in deze context dus wat anders dan een onderdeel van een gen,
want een DNA polymorfisme kan ook buiten een gen voorkomen.
Om de locaties van genen en DNA polymorfisme te vinden, moeten we een locus definiëren als een
plaats op het chromosoom met variatie.
DNA polymorfismes zijn handig bij genetisch studies en worden DNA markers genoemd. Omdat deze
markers niet tot uiting komen in het fenotype, zijn de allelen van DNA markers codominant. Ze
worden onderzocht door te richten op het DNA zelf en niet op het genproduct of fenotype. Door
genen en DNA markers kunnen map afstanden berekend worden tussen genen, tussen DNA markers
of tussen een gen en DNA marker.
Genoom sequenties van mensen bevatten een grote verscheidenheid aan variatie.
Er zijn verschillende soorten DNA polyorfismen:
Single nucleotide polymorfisme (SNP):
Dit is een enkele basepaar verandering – puntmutatie – op een site, SNP locus. SNPs komen het
meeste voor. Nieuwe SNPs ontstaan door spontane mutaties, zoals replicatie fouten. Omdat deze
zeldzaam zijn, komen nieuwe SNPs ook zeldzaam voor.
Het grootste gedeelte bevindt zich in het noncoding gebied van het genoom; noncoding SNPs. SNPs
in coding gebied zijn coding SNPs (cSNP). Ieder gen heeft ongeveer vier cSNPs, waarvan de helft
missense mutaties veroorzaakt in het eiwit en de andere helft silent mutaties veroorzaakt. Of een
cSNP het fenotype beïnvloedt, hangt af welk aminozuur het polymorfisme verandert in het eiwit. De
genfunctie kan ook worden beïnvloedt door noncoding SNPs als ze in een promotor of ander
genregulatie gebied zitten.
SNPs die dicht bij elkaar liggen hebben een band met elkaar: de ene variant van een SNP wordt
samen gevonden met één variant van de andere SNP; ze zijn samen verspreid in de populatie. Dit
wordt linkage disequilibrium genoemd. Een klein deel van de SNP’s beïnvloedt restrictie sites, door
ze te creëren of te verwijderen. Zulke SNPs kunnen gedetecteerd worden door restrictie enzymen op
de site en Southern blot analyse of PCR. De verschillende patronen van restrictie sites in
verschillende genomen leidt tot restrictie fragment lengte polymorfisme (RFLPs); restrictie
enzymcoderende fragmenten van verschillende lengtes.
Southern blot: Hierbij kan men dus onderzoeken of een bepaalde site in een fragment doorgeknipt
wordt of niet (door een mutatie). Dit wordt dus gedaan met een daarvoor specifiek restrictie enzym,
waardoor de fragmenten met intacte allel in kleinere fragmenten worden opgedeeld. Na hybridisatie
met een gelabelde probe worden de fragmenten door autoradiografie onderzocht.
PCR-RFLP analyse methode: Eerst wordt het fragment door primers geïsoleerd en door PCR
vermeerderd. Dit wordt geïsoleerd en geknipt door een restrictie enzym. Vervolgens wordt door
gebruik van agarose gel electroforese de grootte van de fragmenten onderzocht.
Omdat de meeste SNPs geen restrictie sites omvatten, zijn andere methode van analyse nodig.
Door allelspecifieke oligonucleotide (ASO) hybridisatie analyse kunnen individuele SNPs
geanalyseerd worden. Een korte oligonucleotide die complementair is aan een SNP allel wordt
gemaakt. Deze oligonucleotide wordt vermengd met het DNA en hybridisatie wordt onder de beste
condities met elkaar verbonden. Als hybridisatie plaatsvindt, is dit DNA het normale allel. Vindt er
geen hybridisatie plaats, dan is er geen locus met dit stukje goede code en dus gemuteerd.
Door DNA microarrays, DNA chips, GeneChip arrays en oligonucleotide arrays kunnen vele SNPs
tegelijk onderzocht worden. Een DNA microarray is een geordend rooster met DNA moleculen met
bekende sequentie, gefixeerd op bekende posities op het substraat; een silicon chip, glas of nylon
membraan. Het DNA wordt geplaatst, arrayed, op het substraat op verschillende manieren. Bij DNA
microarrays zijn probes en target nucleïne zuren betrokken. De probes zijn echter niet gelabeld en de
targets zijn de gelabelde vrije DNA moleculen waarvan de identiteit of quantiteit onderzocht wordt.
Dit heet dus ook wel probe array. De labels zijn meestal fluorescente tags, groen of rood.
Omdat de oligonucleotide locaties van probes voor ieder allel bekend zijn, geeft het patroon van
fluorescentie aan welk SNP allel het individu heeft, inclusief homozygoot/heterozygoot.
Omdat een probe array een groot aantal oligonucleotide probes kan hebben, kunnen SNP allelen op
duizenden loci onderzocht worden door één experiment.
Short Tandem Repeats (STRs)
STR of microsatellieten of simple sequence repeats (SSR), zijn DNA sequenties die tandem herhaald
worden. Voorbeeld is zes-nucleotide herhaler telomeer. Veel STRs zijn polymorfisch in een populatie,
zodat ze voornamelijk bij genetische mapping en forensisch onderzoek een belangrijke waarde
hebben. Omdat ze relatief kort zijn, wordt meestal PCR toegepast. De analyse maakt gebruik van
primers. Door lengte verschil kan het aantal herhalingen worden bekeken.
Variabel-nummer tandem repeats (VNTRs)
Ook wel minisatellieten hebben langere herhalingen, vanaf 7 tot tientallen basenparen. Er zijn
minder VNTR loci dan STR loci. Deze bevatten ook polymofismen. Omdat ze langer zijn, wordt geen
PCR, maar restrictie afbraak en Southern blot toegepast. De fragmenten worden gescheiden door gel
electroforese en in een membraan filter gebracht. De lengte wordt geanalyseerd door een probe te
gebruiken voor specifieke herhalingen op het locus. Het resultaat geeft de aanwezige allelen weer in
het genoom.Er zijn twee type VNTR loci: unieke loci en multicopy loci. Een VNTR locus kan dus uniek
zijn, maar ook meerdere keren voorkomen in het genoom. Een probe detecteert slecht een VNTR
locus, een monolocus of single-locus probe. Een probe dat meerdere VNTR loci detecteert heet een
multilocus probe.
Bij whole-genoom shotgun, WGS, is het hele genoom gebroken in gedeeltijk overlappende
fragmenten, ieder fragment wordt gekloond en gesequenced en de genoom sequentie wordt
gemaakt door een computer. Eerst worden gedeeltelijk overlappende fragmenten van genomisch
DNA gemaakt door mechanische machines en de fragmenten worden gekloond. Deze fragmenten
zijn vrij kort, waardoor klonen met vectoren mogelijk is. Door overlappingen kan de sequentie
onderzocht worden. Vervolgens gaat men willekeurige, gedeeltelijk overlappende fragmenten
gebruiken in een plasmide vector. Dit is om gebieden te sequencen die herhaalde sequenties
bevatten. Deze fragmenten moeten dus langer zijn dan de maximale lengte van een herhalende
sequentie. Door herhalingen is het moeilijk te kijken wat bij wat hoort, omdat die herhalingen vaker
voorkomen in het genoom. Door een 10 kb kloon library kan dit probleem worden opgelost.
Er zijn verschillende eukaryotische genomen bekend waarvan een aantal relevante:
Gist De S. cerevisiae is vele jaren gebruikt als model voor eukaryoten, omdat het makkelijk op een
media een cultuur vormt, zich makkelijk laat genetisch analyseren en moleculaire manipulaties
makkelijk traceerbaar zijn. Bovendien lijkt het veel op zoogdieren. Het heeft 16 chromosomen.
Rondworm De C. elegans, was de eerste multicellulaire eukaryoot die gesequenced werd.
Rondwormen zijn gladde, niet gesegmenteerde wormen met langen, cylindrische lichamen. Het leeft
in de grond waar het zich voedt met microben. Er zijn twee geslachten: XX en XO. De worm is een
belangrijk model voor genetische en moleculaire aspecten van embryogenese, morfogenese,
ontwikkeling, zenuwontwikkeling en functie, veroudering en gedrag.
De fruitvlieg De D. melanogaster is bij veel genetisch onderzoek gebruikt en heeft meegewerkt aan
ons begrip over moleculaire genetica ontwikkeling. De sequentie werd ontdekt door de wholegenoom shotgun.
Het aantal genen zegt niet zoveel over de complexiteit van organismen. De fruitvlieg heeft ook
bijgedragen bij de kennis over kanker.
De bloemenplant Het genoom van de A. thaliana was het eerste bloemenplant genoom dat
gesequenced werd. Ze was een belangrijk model voor het studeren van genetische en moleculaire
aspecten van de plantontwikkeling. Bovendien bevat ze genen voor de genetische humane ziektes,
zoals borstkanker en cystic fibrosis.
Homo sapiens Voor het sequencen van het genoom werd de whole-genome shotgun gebruikt, met
behulp van humane genoom fysische map data. Het hele genoom is nu gesequenced. De volgende
stap is echter de genen te herkennen in het genoom. Data mining = zoeken door genoom sequenties
voor informatie.
Verder zijn twee andere zoogdieren genomen gesequenced, de muis en de rat. De muis is een model
van genetica van zoogdieren door de traceerbaarheid van genetica, zoals gen knockout mutaties.
Door een aantal genomen gesequenced te hebben kunnen genomen organisaties vergeleken
worden, voornamelijk de ordening van genen en intergenetische regio’s.
De C waarde paradox zegt dat er geen direct verband is tussen de hoeveelheid DNA in het genoom
en de structurele of georganiseerde complexiteit van een organisme. Met de genoomsequenties
kunnen we zien dat er verschil is in genoomorganisatie die zorgen voor deze paradox, inclusief de
gendichtheid; het aantal genen in een gegeven lengte DNA.
Je kunt overzichtelijk per organisme de genoom groottes, aantal genen en genendichtheid
weergeven.
Genomen van bacteriën De genomen verschillen erg veel. (Van 0,5 tot 9 Mb is gesequenced) Ze
hebben gelijke gendichtheid van 1 gen per 1-2 kb. De ruimtes tussen genen zijn relatief klein, wat
betekent dat de genen dicht op elkaar liggen.
Genomen van Archaea De meeste Archaea zijn extremofielen; ze leven in extreme omgevingen. Ze
kunnen leven in hoge temperaturen, hoge druk, extreme pH, hoge metaalion concentratie, hoog
zoutniveau. Ze komen overeen met bacteriën in morfologie. Hoewel fysiologisch en moleculair lijken
ze erg op eukaryoten. Er zijn genen voor DNA replicatie, RNA transcriptie en eiwit synthese systemen
in Archaea meer gerelateerd aan eukaryoten dan aan bacteriën. Er zijn echter geen intronen in eiwitcoderende genen, maar er zijn wel intronen in tRNA genen. De genomen lopen uiteen in grootte (van
1.56 tot 5.75 Mb). De genen zijn dicht op elkaar gepakt in het genoom. (rond 1-2 kb)
Genomen van eukaryoten Eukaryoten verschillen enorm in vorm en complexiteit; van enkelcellige
organismen zoals schimmel tot multicellulaire organisme zoals de mens. Er is geen verband tussen de
toenemende hoeveelheid DNA en de complexiteit. Dit wordt veroorzaakt door verschillen in
genendichtheid. De genendichtheid in eukaryoten is lager en geeft meer verscheidenheid. Er is een
trend met toenemende genendichtheid en toenemende complexiteit (evolutie?). Gist heeft een
dichtheid die het meest overeenkomt met die van prokaryoten. Organismen met grotere genomen
hebben een lagere genendichtheid.
Natuurlijk is dit het gemiddelde en zijn er gen-rijke regio’s en gen woestijnen.
Complexe organismen hebben dus de minderheid van het genoom gewijd aan genen, de rest zijn
intergenetische regio’s, bestaande uit bijvoorbeeld herhaald DNA. Hierdoor is het soms moeilijk
belangrijke genen te vinden. Door een andere gewervelde met een hoge genendichtheid te
gebruiken kan helpen. Zo eentje is Fugu rubripes, de putterfish (kogelvis). Deze vis heeft ongeveer
net zoveel genen, maar heeft een kleiner genoom dan dat van mensen. De genendichtheid is dus
kleiner dan dat van de mens. Dit komt door de kleinere en minder intronen in de genen; homologe
genen in mensen zijn langer. Ook bezit het weinig herhalend DNA en minder intergenetisch DNA.
Door de kleinere dichtheid is de vis makkelijker te onderzoeken. Omdat veel Fugu genen homoloog
zijn aan mensengenen, kunnen ze, eenmaal geïdentificeerd in de vis, herkend worden in de
homologe genen van de mens. Men hoopt dat hierdoor het onderzoek naar de functies van genen
van de vis helpen bij de identificatie van humane genen die ziektes veroorzaken.
Zelfstudie 19 (H24 Russel):
Populatiegenetica houdt zich bezig met de overdracht van genen op grotere groepen, populatie. Een
medeliaanse populatie is een groep van voortplantende organismen, interbreeding, die dus
eenzelfde set genen bevat. De gedeelde genen heten ook wel de genenpool. Er wordt gekeken naar
deze genenpool meer dan de genotypen van elk individu. Hoe deze veranderd over generaties en tijd
en hoe de variatie is kunnen we een algemene wet aannemen, de hardy-weinbergwet.
We kunnen hierbij onderscheid maken tussen een genotype frequentie (voorkomen van combinaties
van allelen) en een allelfrequentie (voorkomen van een enkel allel). Vaak biedt het laatste wat
voordelen over het gebruik van de eerste. Genotypen worden in gameten afgebroken en allelen
worden dus overgedragen. En ten tweede zijn er altijd minder allelen dan genotypen, wat het
rekenen makkelijker maakt. Omdat elk individu twee allelen heeft is het aantal allelen twee keer zo
groot als het aantal individuen.
Wanneer een genotype bekend hier en de frequentie hiervan kunnen we vanuit daar de
allelfrequentie uitrekenen. Dit is altijd de helft maar de heterozygote frequentie + de frequentie van
de homozygoten. De allelen duiden we aan met p (wild type, dominant) en q (recessief, mutant). Ook
bij 3 allelen kun je dit doen, dan heet het derde allel R.
Voor X-gelinkte allelen is deze procedure iets ingewikkelder maar kunnen in principe dezelfde
bovenstaande regels vormen.
De Hardy-Weinberg wet wordt in 3 delen opgedeeld, een verzameling van aannames en twee grote
resultaten.
1. aannames: een oneindig of heel grote populatie die random seksen, geen mutatie, geen
selectie, en geen migratie.
2. resultaat 1: de frequenties van de allelen veranderd onder deze aannames niet.
3. resultaat 2: zolang het seksen random gebeurd blijven de genotypen in dezelfde
verhoudingen.
De grote populatie is nodig om genetic drift tegen te gaan. Het is een foute veronderstelling dat het
seksen voor alle eigenschappen random moet zijn, iets wat niet kan omdat mensen bijvoorbeeld nu
eenmaal andere mensen kiezen die bij hun passen. Voor de wetten om te handhaven moet er alleen
op het allel van interesse random gepaard worden. De overige aannames zijn er om ervoor de zorgen
dat de genenpool vrij is van toevoeging of verdwijning van allelen. Wanneer deze condities goed zijn
is de populatie in een zogenaamd genetisch equilibrium en mogen de twee genoemde resultaten
verwacht worden. Wanneer er tijdelijk afgeweken wordt van het evenwicht is deze na één ronde van
reproductie al weer teruggekeerd. Dus ook als er letale recessieve allelen zijn, zal de volgende
generatie volgens de wetten van hardy weinberg geboren worden, of dood gaan en de
allelfrequenties van mixing blijven gelijk. Wat er blijft leven is een ander verhaal, dus wat je ziet kan
afwijken omdat een aantal dood gaat.
Ook met meer dan twee allelen kan de formule gewoon uitgebreid worden en de wetten toegepast.
Bij X-gelinkte genen zijn die voor de vrouw gewoon normaal, met p en q voor beide allelen op het X
chromosoom. Voor mannen werkt het echter anders omdat ze maar één X hebben en dus één allel.
Hierbij is de frequentie van het genotype gelijk aan de frequentie van het allel.
Als gevolg hiervan is de frequentie van het allel in mannen gelijk aan de frequentie in de moeder, bij
vrouwen is dit het gemiddelde tussen de moeder en de vader.
Wanneer de hardyweinberg wet mogelijk niet aan kan houden omdat toch een van de aannames
verworpen is moet men voorzichtig zijn met het berekenen van de allelfrequenties uit de genotype.
Men moet dan altijd gebruik maken van de bovenstaande formule (1/2hetero + 1 homo) en niet
zomaar voor p de wortel van de homozygote voor p trekken. Hierbij ga je er namelijk al vanuit dat
het evenwicht opgaat terwijl je juist wilt kijken of dit zo is. De chi-kwadraat test kan uiteindelijk
uitsluitsel geven of hardy-weinberg wordt gehandhaafd of niet. Lees dit nog even door. Het aantal
vrijheidsgraden is het aantl klassen – 2.
Verder moet je er aan denken dat proporties niet met de chikwadraat getest kunnen worden en in
hele getallen omgezet moeten worden. Dit doe je door de proporties simpel met het totale aantal
allelen (2x antal individuen) te vermenigvuldigen.
Zelfstudie 20 (H24 Russel + extra tekst syllabus):
Een belangrijke afwijking van random paren is inteelt. Dit gaat op paring tussen verwanten van
elkaar. Kleine populaties hebben sowieso al snel te maken met inteelt, omdat deze maar een beperkt
aantal paringsmogelijkheden hebben. De mate van inteelt is te berekenenmet de inteeltcoëfficiënt F.
Hoe groterF, hoe meer inteel. Inteelt betekent ook reductie in heterozygositeit, of ook toename in
homozygositeit. Dit betekent ook afname in genetische variatie. Daarom is de formule voor F ook
verwachte heterozygositeit min de geobserveerde heterozygositeit gedeeld door de verwacht
heterozygositeit. Let hierbij op dat de allelfrequentie niet perse hoeft te veranderen!!!
Het bekendste geval van inteelt is natuurlijk zelfbevruchting.
Inteelt lijkt op genetische drift, hoewel drift slechts kleine afwijkingen van hardy-weinberg als gevolg
heeft en inteelt grote.
Een andere manier om inteelt te bepalen is aan de hand van een stamboom. Hiervoor kijk je naar de
additief genetische relatie. Dit is de relatie tussen individuen en meet het propotie allelen dat dieren
gemiddeld gelijk hebben in die relatie. Tussen ouders en kinderen is dit .5, broer en zus ook .5, en
grootouders-kleinkind .5 x .5 = .25. binnen een stamboom kun je dit tellen door het aantal ouderkind relaties tussen de persoon en de persoon van interesse te tellen. De additief genetische relatie
zegt iets over de relatie tussen dieren, de inteeltcoëfficiënt binnen een dier. Uit paring tussen twee
dieren is de kans af te leiden dat een nakomling homozygoot wordt voor een allel. Als regel geldt dat
de inteeltcoefficient de helft is van de additief genetische relatie VAN DE OUDERS van dat dier.
Bij eenvoudige stambomen is het makkelijk om deze relatie te bepalen, zowel de additief genetische
relatie en de inteelt. Deze methode heet de paden-tel-methode. Hierbij is één pad een oudernakomeling relatie.
Het is gebaseerd op de kans dat twee nakomelingen hetzelfde allel krijgen, dus .5 van de ene ouder +
.5 van de andere ouder is 25 (of een half maal de additief genetische relatie). Om nu aan de hand
hiervan d inteelt coefficient te bepalen tel je van een nakomeling het pad vanaf d ene OUDER tot de
gemeenschappelijke voorouder, plus het pad van de andere ouder tot de gemeeschappelijke
voorouder. Elk pad telt voor een half additief genetische relatie, dus .5 in de macht van het aantal
paden. Bij het aantal paden moet echter nog 1 opgeteld worden, omdat het om de kans gaat dat het
allel nog aan de nakomeling doorgegeven wordt (ook weer een relatie van een half). Omdat het gaat
om het doorgeven van allelen en de voorouder er twee heeft moet dit geheel nog maar 2. omdat je
ervan uitgaat dat het per gemeenschappelijke voorouder onafhankelijk van elkaar gebeurt mag je de
gevonden waarden per voorouder bij elkaar optellen!
Wanneer de gemeenschappelijke voorouder zelf al ingeteeld was komt er een andere som bij kijken.
Hierbij doe je nog de inteeltcoëfficient van die ouder + 1 maal de som van net. Dit alles staat ook op
het formuleblad.
De toename in inteelt per generatie kun je ook berekenen. Dit is het verschil in gewogen gemiddelde
inteelt van één generatie tot de volgende. De toename kan niet lineair zijn omdat deze 1 niet kan
overstijgen en is dus ook asymptoot tot 1. In het algemeen wordt aangenomen dat een toename van
inteelt per generatie tot 1%, .01 acceptabel is. Alles daarboven is teveel.
Zelfstudie 21 (H24 Russel + tekst syllabus):
Selectie kan worden toegepast om een toename, een optimum of een afname van een eigenschap te
bereiken in een populatie.
Gen frequenties kunnen door een aantal factoren veranderd worden:
1. mutatie: je kunt hier onderscheid maken tussen een voorwaartse mutatie (van A naar a) of
een achterwaartse mutatie (van a naar A). het laatste gebeurd minder vaak. Uiteindeljk
bereikt een poulatie een evenwicht tussen deze beide mutaties, waardoor de allelfrequentie
niet meer veranderd. Het muteert even vaak naar voren als naar achteren. Vaak echter is de
mate van mutatie zo langzaam dat er vaak geen evenwicht komt, maar dat invloed op de
populatie ook minimaal is.
2. genetische drift: dit is de verandering in allelfrequentie als gevolg van toeval. dit speelt
vooral een rol bij kleinere populaties. Dit kan door de toevallige dood van een deel van de
populatie komen, kansafwijkingen van de indeling in gameten (door normale sampling error).
De verwachting van de allel frequentie is gelijk, maar de geobserveerde kan door klein aantal
samples afwijken(3 keer met dobbelsteen gooien, of 100x). om de grootte van genetische
drift vast te stellen kijken we naar de effectieve popuatiegrootte (Ne), wat gelijk is aan het
aantal individuen die gameten voor de volgende populatie creeërt. Dit hangt dus af van het
aantal reproducerende mannen en vrouwen. Wanneer deze heel erg ongelijk zijn is ook de
effectieve populatie kleiner. De formule staat op het formuleblad. alle genetische drift komt
van steekproeffouten (samplingerror) maar de manier waarop deze ontstaat kan door een
aantal effecten worden beïnvloed. Allereerst blijft de genetische drift alleen groot als de
populatie constant klein is. Een andere manier is door het founder effect, wat optreedt als
het begin van de populatie door een kleine groep individuen is gestart. De genenpool in de
populatie stamt namelijk van deze individuen af. De bottleneck is het effect op genetische
drift wanneer de populatie drastisch in grootte afneemt, waarbij genen van de genenpool
verloren gaan. het kan gezien worden als een founder effect omdat de resterende populatie
door de overlevende individuen in stand gehouden wordt. Genetische drift produceert
veranderingen in allelfrequentie. Wanneer één allel compleet verloren gaat is de populatie
gefixeerd voor het overblijvende allel. Dit kan alleen door migratie of mutatie weer
veranderen. Vaak is het zeldzaamste allel verloren. Het aantal heterozygoten wordt hierdoor
ook minder en is na fixatie 0.
3. migratie: wanneer nieuwe individuen migreren naar een nieuwe populatie kunnen ze nieuwe
allelen in de genenpool toevoegen de allelfrequenties aanpassen. Migratie heeft dus de
potentie om hardy-weinberg te verstoren. We zijn echter wel geïnteresseerd in de beweging
van genen, niet zozeer organismen omdat deze zich niet altijd voortplanten en daarom
genetisch niet bijdragen aan de populatie.
4. natuurlijke selectie: dit kan gedefinieerd worden als de verschillen in reproductie van
genotypen, bepaalde genotypen produceren meer nakomelingen dan anderen. Natuurlijke
selectie kan gemeten worden in termen van fitness, wat de reproductieve mogelijkheden
stelt van een genotype. 1 is de beste fitness van alle genotypen, de andere genotypen
worden relatief gemaakt ten opzichte van di genotype. Pleiotropische effecten (enkele
genen hebben een grote invloed op de levenscyclus) hebben effect op fitness van genotypen.
De mate van verandering in allelfrequentie als gevolg van fitness van een allel en natuurlijke
selectie kan berekend worden. Dee formule staat op het formuleblad. De P is altijd het allel
met de beste fitness, en de W is de relatieve fitness van Q (immers P heeft een fitness van 1).
Heterose leidt tot een balans in allelfrequentie en dit is het gevl wanneer de heterozygoot
een betere fitness heeft dan elk van de homozygoten.
In de syllabus laten ze nog even zien dat genetische drift gelijk is aan de afname in heterozygotie
door een eindige populatie omvang hetzelfde zijn. Een geïdealiseerde populatie ontstaat wanneer er
geen selectie, migratie of mutatie is met maar één geslacht. In deze populatie neemt de
heterozygotie af met 1/2N. de toename in inteelt is dus ook direct gelijk aan het aantal dieren in de
GEIDEALISEERDE POPULATIE. Er is echter niet zo’n populatie en het verhaal zit dus heel anders in
elkaar en moet de effectieve populatieomvang berekend, Ne.
Wanneer er geen selectie is kan de toename in inteelt berekend worden de formule staat op het
formuleblad met mannen en vrouwen apart.
Zelfstudie 22 (H25 Russel en syllabus):
Homologe eiwitten hebben een zelfde voorvader. Mutaties zijn veranderingen in
nucleotidevolgorde, substituties zijn mutaties die op één of andere manier door de natuurlijke
selectie zijn heen gekomen. Ze zijn niet echt een goede maat voor het werkelijke aantal mutaties die
geweest zijn. Substituties zijn het meest waarschijnlijk door te komen wanneer ze weinig effect
hebben op de genen of de genen in positieve zin beïnvloeden. Neurale mutaties zien we dus vaker
terug dan missense mutaties in substituties. Pseudogenen hebben natuurlijk de hoogste mate van
evolutie. Mutaties in het 3’einde van een gen hebben geen invloed op de aminozuurvolgorde, die in
de 5’einde wel.
Omdat een neutrale mutatie in de genen minder vaak voorkomt dan die in psuedogenen terwijl er
geen echt verschil in effect is, is de codon usage bias opgesteld. Dit suggereert dat synonieme
mutaties niet helemal neutraal zijn, waarschijnlijk door een voorkeur voor het gebruik van bepaalde
codons t.o.v. anderen. Dit komt omdat er ander tRNA gebruikt wordt. De tRNA’s die het meest
voorkomen worden efficiënter getransleert en behoeven daarom de voorkeur.de tRNA is afhankelijk
van het codon, niet van het product.
De mutatiegrootte in mitochondria is veel hoger. Dit komt waarschijnlijk door meer vrije radicalen in
de mitochodria en door de kloon achtige overerving.
De moleculaire clock hypothese gebruikt de mutatie schaal (dus eigenlijk de substitutieschaal) om
een evolutionaire klok op te stellen aan de hand waarvan de soorten in tijd gerelteerd aan elkaar
opgesteld kunnen worden. Dit kan gebruikt worden om een boom van afstamming op te stellen,
fylogenetische boom. Dit is uiteindelijk gebruikt om bijvoorbeeld een onderscheid te maken tussen
eukaryoten, eubacteriën en archaebacteriën.
Multigenen families zijn families van genen die van eenzelfde voorvader afstammen. Globine is het
klassieke voorbeeld hiervan.
Zelfstudie 23 (H14 Russel):
Kwantitatieve eigenschappen kunnen op een schaal worden gemeten en zijn dus continu. Deze
eigenschappen hebben dus een range en verschillende genen produceren hetzelfde fenotype. Ze
worden ook wel de kwantitaieve trait loci genoemd die bijdagen aan het fenotype (QTL’s)
De mate waarin de variatie van een fenotype beïnvloed wordt door een genotype en de omgeving
kan in een formule worden weergegeven. De erfelijkheidsgraad is de maat waarin de genetische
factoren een rol spelen. Dit is dus een proportie. We maken hier onderscheid tussen de nauwe
erfelijkheidsgraad en de brede. De brede neemt ook allelinteracties in acht. Vaak echter kijken we
naar de nauwe omdat allelinteracties niet doorgegeven worden via gameten, de allelen zelf wel. Let
hier wel op! De variantie is de standaardafwijking (sigma) in het kwadraat!!!
Maternale effecten zijn de effecten door de moederlijke omgeving in de baarmoeder en daarna door
bijvoorbeeld moedermelk. De erfelijkheidsgraad geeft aan hoe goed je dieren kunt selecteren.
Zelfstudie 24 (Russel H14):
De respons op selectie van een volgende populatie wordt de selectierespons genoemd en kan
berekend worden. Deze hangt van twee zaken af, de erfelijkheidsgraad van een eigenschap en het
seletiedifferentiaal (S). Het selectiedifferntiaal is het verchil van de geselcteerde groep voor
voortplanting en de gehele populatie. R (respons) is de erfelijkheidsgraag maal het
selectiedifferentiaal. De selectierespons hangt af van de moederselectie en de vader selectie, dit is
de helft van de R voor vaders + de heft van de R voor moeders.
De makkelijkste manier om deze totale respons te zien is met de volgende formule:
H2 (erfelijkheidsgraad) * ((Sman) + (Svrouw))
R = ----------------------------------------------------------2
Wanneer twee fenotypen correleren het het fenotypishe correlatie. (blond haar/blauwe ogen).
Pleiotropie is echter het gevolg van genetische correlatie, evenals genetische linkage. Herinner:
pleiotropie is het effect van een gen op meerdere fenotypische kenmerken.