1.- GENETISCHE CODE
2.- NUCLEOTIDEN / BASEN / basic “triplets “
Genetische code
Beeldmateriaal http://pt.bestpicturesof.com/genetische%60code
Genetische code.
De combinatie van codon en aminozuur is specifiek en is vastgelegd in de genetische code. Drie mRNA-letters (een codon) coderen voor één aminozuur. Voor een
aantal aminozuren bestaan er meerdere codons (er zijn immers 64 mogelijkheden voor maar twintig aminozuren). Drie codons geven een Stop aan: het eiwit is af.
http://nl.wikipedia.org/wiki/Genetische_code
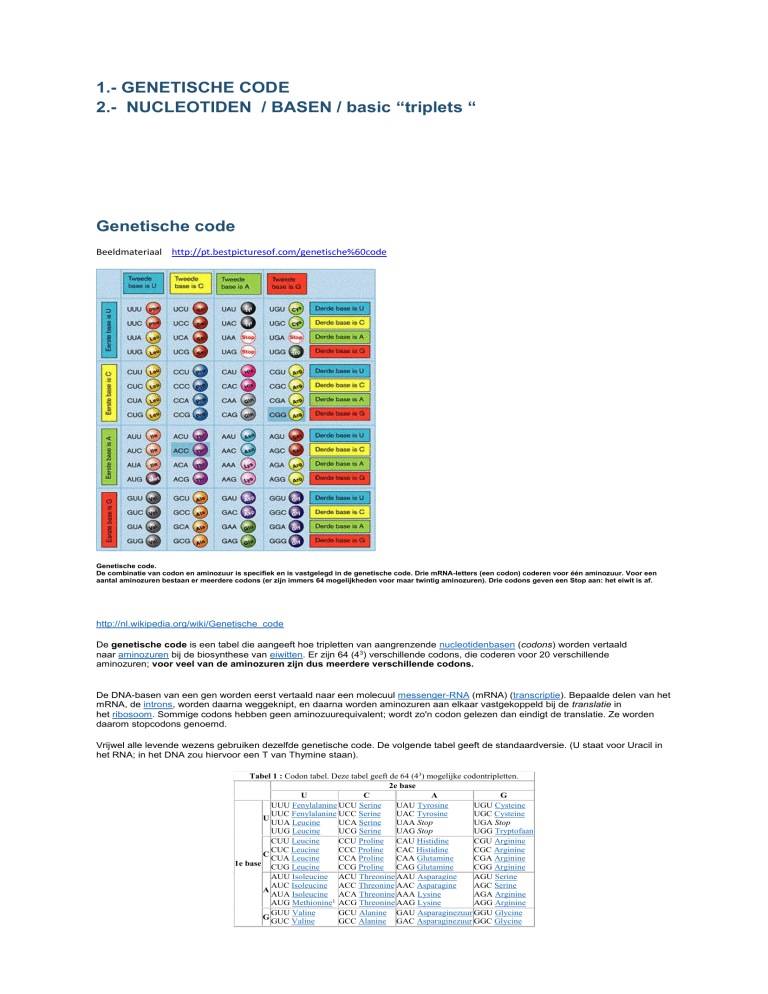
De genetische code is een tabel die aangeeft hoe tripletten van aangrenzende nucleotidenbasen (codons) worden vertaald
naar aminozuren bij de biosynthese van eiwitten. Er zijn 64 (43) verschillende codons, die coderen voor 20 verschillende
aminozuren; voor veel van de aminozuren zijn dus meerdere verschillende codons.
De DNA-basen van een gen worden eerst vertaald naar een molecuul messenger-RNA (mRNA) (transcriptie). Bepaalde delen van het
mRNA, de introns, worden daarna weggeknipt, en daarna worden aminozuren aan elkaar vastgekoppeld bij de translatie in
het ribosoom. Sommige codons hebben geen aminozuurequivalent; wordt zo'n codon gelezen dan eindigt de translatie. Ze worden
daarom stopcodons genoemd.
Vrijwel alle levende wezens gebruiken dezelfde genetische code. De volgende tabel geeft de standaardversie. (U staat voor Uracil in
het RNA; in het DNA zou hiervoor een T van Thymine staan).
Tabel 1 : Codon tabel. Deze tabel geeft de 64 (43) mogelijke codontripletten.
2e base
U
C
A
G
UUU Fenylalanine UCU Serine
UAU Tyrosine
UGU Cysteine
UUC Fenylalanine UCC Serine
UAC Tyrosine
UGC Cysteine
U
UUA Leucine
UCA Serine
UAA Stop
UGA Stop
UUG Leucine
UCG Serine
UAG Stop
UGG Tryptofaan
CUU Leucine
CCU Proline CAU Histidine
CGU Arginine
CUC Leucine
CCC Proline CAC Histidine
CGC Arginine
C
CUA Leucine
CCA Proline CAA Glutamine
CGA Arginine
1e base CUG Leucine
CCG Proline CAG Glutamine
CGG Arginine
AUU Isoleucine ACU Threonine AAU Asparagine
AGU Serine
AUC Isoleucine ACC Threonine AAC Asparagine
AGC Serine
A
AUA Isoleucine ACA Threonine AAA Lysine
AGA Arginine
AUG Methionine1 ACG Threonine AAG Lysine
AGG Arginine
GUU Valine
GCU Alanine GAU Asparaginezuur GGU Glycine
G
GUC Valine
GCC Alanine GAC Asparaginezuur GGC Glycine
GUA Valine
GUG Valine
GCA Alanine
GCG Alanine
GAA Glutaminezuur GGA Glycine
GAG Glutaminezuur GGG Glycine
1Het
AUG codon codeert voor methionine en dient tevens als startpunt; de eerste AUG in een mRNA is de plaats waar de translatie
begint.
Tabel 2 : Inverse codon tabel. Deze toont de 20 aminozuren en de codons waardoor ze kunnen worden
gecodeerd.
UUA, UUG, CUU, CUC, CUA, CUG
Ala GCU, GCC, GCA, GCG
Leu
CGU,
CGC,
CGA,
CGG,
AGA,
AGG
AAA, AAG
Arg
Lys
AUG
Asn AAU, AAC
Met
UUU, UUC
Asp GAU, GAC
Phe
CCU, CCC, CCA, CCG
Cys UGU, UGC
Pro
UCU, UCC, UCA, UCG, AGU, AGC
Gln CAA, CAG
Ser
ACU, ACC, ACA, ACG
Glu GAA, GAG
Thr
UGG
Gly GGU, GGC, GGA, GGG
Trp
UAU, UAC
His CAU, CAC
Tyr
AUU, AUC, AUA
GUU, GUC, GUA, GUG
Ile
Val
START AUG, GUG
STOP UAG, UGA, UAA
A=Adenine, C=Cytosine, T=Thymine, G=Guanine en U=Uracil
Externe links
Online DNA → Amino Acid Converter (http://www.geneseo.edu/~eshamb/php/dna.php) by bdesham
zie ook - ( J. Mol. Evol., 59:1 - 8 (2004) )
A new classification scheme of genetic code is based on a binary representation of :
purines (G,A) - 1
pyrimidines (C,U) - 0
The eight rows (23 = 8) and four columns are sufficient to place 20 amino acids, as well as the termination codons.
Code
6 hydrogen bonds
000
Pro CC (C/U) P
Ser UC (C/U) S
Leu CU (C/U) L
Phe UU (C/U) F
Proline
Serine
Leucine
Phenylalanine
Pro CC (A/G) P
Ser UC (A/G) S
Leu CU (A/G) L
Leu UU (A/G) L
Proline
Serine
Leucine
Leucine
Ala GC (C/U) A
Thr AC (C/U) T
Val GU (C/U) V
Ile AU (C/U) I
Alanine
Threonine
Valine
Isoleucine
001
100
101
010
011
Ala GC (A/G) A
010
011
<v: Thr AC (A/G) T Val GU (A/G) V
Ile/Met AU (A/G) I/M
Alanine
Threonine
Valine
Isoleucine / Methionine
Arg CG (C/U) R
Cys UG (C/U) C
His CA (C/U) H
Tyr UA (C/U) Y
Arginine
Cysteine
Histidine
Tyrosine
Arg CG (A/G) R
Arginine
101
5 hydrogen bonds 5 hydrogen bonds 4 hydrogen bonds
Ala GC (A/G) A
Stop/Trp UG (A/G) W Gln CA (A/G) Q
Tryptophan
Stop UA (A/G) *
Glutamine
<v: Thr AC (A/G) T Val GU (A/G) V
Ile/Met AU (A/G) I/M
Alanine
Threonine
Valine
Isoleucine / Methionine
Arg CG (C/U) R
Cys UG (C/U) C
His CA (C/U) H
Tyr UA (C/U) Y
Arginine
Cysteine
Histidine
Tyrosine
Arg CG (A/G) R
Arginine
Stop/Trp UG (A/G) W Gln CA (A/G) Q
Tryptophan
Glutamine
Stop UA (A/G) *
110
Gly GG (C/U) G
Glycine
111
Gly GG (A/G) G
Glycine
Ser AG (C/U) S Asp GA (C/U) D Asn AA (C/U) N
Serine
Aspartic acid
Asparagine
Arg AG (A/G) R Glu GA (A/G) E Lys AA (A/G) K
Arginine
Glutamic acid
Lysine
1. Each row contains exactly 4 different amino acids (including the termination codon).
In the standard code, exceptions are the second row with two leucines and in the fourth row the AU* start codon. Note that
here are also the deviations from the standard code. Interestingly, the yeast mitochondrial code shows no exception: each row
contains exactly four different entries in four different columns. In this spirit the yeast mitochondrial code is the most regular
one.
2. The mitochondrial code shows no exception: 32 positions contain exactly 32 entries.
3. There are 22 tRNA genes in the mammalian mitochondrial genomes: Table2.
"The mammalian mitochondrial genomes contain ONE gene for each tRNA, with the exceptions of tRNA Leucine and tRNA
Serine for which TWO genes are present." This is no exeption for our scheme, what can be seen in the mammalian
mitochondrial code via tRNA .
4. This corresponds to the known fact that transition mutations (e.g. purine A vs. purine G) occur more frequently than
transversion mutations (e.g. purine A vs. pyrimidine U).
Our scheme yields some support for the “adaptive genetic code” hypothesis (Freeland 2002) which states that the code has
evolved to minimize the deleterious effects of mutation and translation error (Haig and Hurst 1991, Freeland and Hurst 1998).
The purine-pyrimidine binary coding scheme, given in table, gives a much higher regularity than a binary coding according to
the base pairs (A,U – 1; G,C – 0).
5. In the first column the first two positions are G and C. These always pair with their anticodon base via 3 hydrogen bonds, i.e.
the first two bases together always guarantee 6 hydrogen bonds. For that reason Lagerkvist (1978) called them strong
codons. In the second and third column, the first two bases guarantee exactly 5 bonds (mixed codons) and in the fourth
column just 4 bonds (weak codons).
This pattern corresponds very well to the importance of the third base in the triplet codon: if the first bases are G and/or C (first
column), the third base is never important, and in the second and third column, the third base is important in exactly half of the
cases (if there is a purine in the second position – lower half of the table). In the fourth column the third base is always
necessary for the determination of the correct amino acid.
6. The deviations of non-standard genetic codes. As can be seen in table, nearly all deviations occur in codons with a purine at
the third position. The only exception is the yeast mitochondrial code where CU* does not code for Leu, but rather for Thr.
7. Two perfect symmetries in our scheme.
The first is the codon-anticodon symmetry: the thick horizontal line in Fig.2 marks the symmetry axis.
For instance, codon CCC (Pro, first column, first row) has the anticodon GGG (Gly, first column, last row).
The second is the point symmetry corresponding to Halitsky’s family – nonfamily symmetry operation (“E-M bifurcation”,
Halitsky 2003),
indicated by the point in the center of table. Halitsky observed that all the 32 “family codons” CC*, CU*, UC* GC*, GU*, AC*,
CG*, GG* can be mapped into the 32 “nonfamily codons” UU*, AU*, CA*, UG*, UA*, GA*, AG*, AA* by exchanging the two
amino bases A and C with one another, and the two keto bases U and G with one another. For instance, the family codon
GUA (Val) is mapped into the nonfamily codon UGC (Cys). Thus, this point symmetry is behind the family – nonfamily
symmetry in our scheme (shaded vs. unshaded regions).
8. In the fourth column all amino acids are ketogenic (leucine, lysine) or glucogenic and ketogenic (isoleucine, phenylalanine,
threonine, asparagine, methionine and tyrosine) .
The carbon skeletons of amino acids are generally conserved as carbohydrate, via gluconeogenesis, or as fatty acid via fatty
acid synthesis pathways. In this respect amino acids fall into three categories: glucogenic, ketogenic, or glucogenic and
ketogenic. Glucogenic amino acids are those that give rise to a net production of pyruvate or TCA cycle intermediates, such
as a-ketoglutarate or oxaloacetate, all of which are precursors to glucose via gluconeogenesis.
9. Correlation of codon strength and amino acid properties: Table 1.
10. Evolution of the genetic code: doublet code?
http://www.evolvingcode.net/index.php?page=Universal_Genetic_Code
against creationist misinformation
http://www.natcenscied.org/resources/articles/STRS.html
Historical adres
http://nobelprize.org/nobel_prizes/medicine/laureates/1962/crick-lecture.html
http://www.pandasthumb.org/archives/2004/08/meyer_v_univers.html
TTAGGG sequentie
Unieke individuele eigenschappen en de TTAGGG sequentie op de menselijke telomeren
Ieder individu reageert anders op geneesmiddelen, voeding en omgevingsstoffen, net zoals op micro-organismen.
In onze 3 miljard DNA-nucleotides zitten een 22.000 genen, die tot honderdduizenden eiwitproducten kunnen leiden.
In ieder van onze 100 biljoen cellen is de onderlinge verhouding van deze eiwitten ook weer anders; geen enkele cel is gelijk; net als individuen
nooit gelijk zijn ( zelfs eeneiige tweelingen niet ) .
Veranderingen op DNA-niveau kunnen leiden tot veranderingen op RNA- en eiwitniveau.
Denk hierbij niet alleen aan punt-mutaties, maar ook aan volledige deleties, alternatieve manieren om de exonen in het mRNA te koppelen bij het
weghalen( uitsnijden ) van de intronen, frame shifts,repeats en andere complexe zaken.
Alles wat fout kan gaan, is fout gegaan.
Het enige dat echt bewaard is gebleven is de TTAGGG sequentie die aan de staart van de chromosomen (telomeren) zit.
TTAGGG is een menselijke telomeer-sequentie van een basis -sequentie die bij alle (aardse )levende wezens aanwezig is (zie
tabel onder ) :
Telomeer <
Een telomeer bestaat uit enkelstrengs DNA en zit aan het uiteinde van een chromosoom. Telomeer betekent in het Grieks
eindstuk. Een telomeer komt niet voor bij prokaryotenmet een ringvormig chromosoom.
Oude cellen hebben chromosomen met een korter telomeer dan jonge cellen. Een uitzondering hierop werd echter in 2003 ontdekt bij het Vaal
stormvogeltje, waarbij de telomeren bij het ouder worden van de vogel juist in lengte toenemen.
Een telomeer is te vergelijken met het plastic uiteinde van een veter. Bij iedere deling rafelt het uiteinde een stukje uit en uiteindelijk kan de cel niet
meer delen en sterft. Menselijke cellen bereiken dit stadium na zo'n vijftig, zestig delingen.
Als de telomeerlengte behouden blijft, kan de cel zich blijven delen zonder dood te gaan. Onder de microscoop is een telomeer zichtbaar als een
oplichtend puntje.
T-loop
Een telomeer bestaat uit repeterende stukjes DNA dat de belangrijke genen beschermt tegen het korter worden van de chromosomen. Ook is het
telomeer belangrijk voor de stabiliteit van het chromosoom. Bij de mens bestaat het repeterende stukje DNA uit de sequentie TTAGGG en is het
telomeer tussen de 3 en 20 kilobasen lang. Tussen het telomeer en de rest van het chromosoom zit nog een stukje dat 100 tot 300 kilobasen lang is.
Hierdoor kan het telomeer een soort lasso, T-loop, vormen. De sequentie verschilt per organisme, maar bevat gewoonlijk veel GC
(Guanine en Cytosine) nucleotiden.
Telomerase
Tijdens de celdeling wordt het DNA via replicatie verdubbeld. Het enzym DNA-polymerase kan echter het eind van het chromosoom
niet repliceren, omdat het voortijdig van het DNA afvalt. Hierdoor worden bij elke celdeling de chromosomen korter. Het
enzym telomerase dat een reverse-transcriptase is, kan een telomeer weer langer maken en komt voor in de kiembaancellen die de
geslachtscellen maken, stamcellen, kankercellen en protozoa. Telomerase bestaat uit een enzymatisch deel en een RNA deel, de
complementaire sequentie.
Verantwoordelijk gen
Het gen dat verantwoordelijk is voor de vorming van telomerase is in 1998 door Geron Corporation in Californië gekloond en
ingebracht in cellen. Deze cellen hebben zich twee jaar lang gedeeld zonder enige vorm van veroudering. Ze lijken onsterfelijk te zijn
geworden.
DNA-herstelmechanisme
Elke cel beschikt over een DNA-herstelmechanisme dat beschadigd DNA repareert. Als het DNA te veel beschadigd is en niet meer
hersteld kan worden, stopt de cel met delen of gaat dood. Als het telomeer 4 kilobasen kort is geworden kan het geen lus meer maken
en ziet het DNA-herstelmechanisme het eind van het chromosoom als een gebroken chromosoom waardoor de cel stopt met delen of
dood gaat.
Klonen
Bij klonen van cellen vertrekt men van cellen die al kortere telomeren hebben. Het nieuw organisme vertrekt dus met kortere
telomeren.
Enkele bekende telomeren sequenties
Groep
Gewervelden
Organisme
Mens, muis, Xenopus
Draadvormende schimmels Neurospora crassa
Telomeer repeterend stukje DNA (repeat) (5' → 3' richting eind)
TTAGGG
TTAGGG
Slijmzwammen
Physarum, Didymium
Dictyostelium
TTAGGG
AG(1-8)
Kinetoplastiden protozoa
Trypanosoma, Crithidia
TTAGGG
Ciliophora (protozoa)
Tetrahymena, Glaucoma
TTGGGG
Paramecium
TTGGG(T/G)
Oxytricha, Stylonychia, Euplotes TTTTGGGG
Apicomplexan (protozoa)
Plasmodium
TTAGGG(T/C)
Hogere planten
Arabidopsis thaliana
TTTAGGG
Groene algen
Chlamydomonas
TTTTAGGG
Insecten
Bombyx mori
TTAGG
Rondwormen
Ascaris lumbricoides
TTAGGC
Celdelende gisten
Schizosaccharomyces pombe
TTAC(A)(C)G(1-8)
Saccharomyces cerevisiae
Knopvormende gisten
Candida glabrata
Candida albicans
Candida tropicalis
Candida maltosa
Candida guillermondii
Candida pseudotropicalis
Kluyveromyces lactis
TGTGGGTGTGGTG (van RNA 'mal' (template))
of G(2-3)(TG)(1-6)T (consensus)
GGGGTCTGGGTGCTG
GGTGTACGGATGTCTAACTTCTT
GGTGTA[C/A]GGATGTCACGATCATT
GGTGTACGGATGCAGACTCGCTT
GGTGTAC
GGTGTACGGATTTGATTAGTTATGT
GGTGTACGGATTTGATTAGGTATGT
zie ook : OUDE VADERS, JONGE MOEDERS ? http://evodisku.multiply.com/journal/item/539/oude_vaders_jonge_moeders_
Over de evolutie van de genetische code
Marleen op januari 2, 2012
aminozuren, basen, code, DNA, eiwitten, evolutie,genetische code,nucleotiden, RNA, tRNA, tRNA synthase, wobble positie
Eerst gaat het vaak over de abiogenesis, ofwel het ontstaan van de eerste macromoleculen (DNA, RNA en eiwitten) uit de
beschikbare moleculen in de ‘primordiale soep’. Daarbij springt vooral het werk van Ernesto Di Mauro (1, 2) in het oog, die liet zien
dat zich in water zonder katalyserende enzymen spontaan lange ketens van nucleotiden (ofwel basen) kunnen vormen. In zijn
experimenten ging het om heel eenvoudige ketens van maar één van de vier basen.
De genetische code: A, G, C en U zijn de vier basen van het RNA die de drieletterig code vormen voor de aminozuren (aangegeven met kleine
letters)
Nucleotiden ofwel basen zijn de letters van het DNA en het RNA. DNA kent iedereen als de moleculen waarin onze genen opgeslagen liggen. De
genen worden gelezen en zo wordt er in de celkern mRNA van gemaakt dat een bijna exacte kopie is van het DNA. Het DNA en het RNA bestaan uit
vier verschillende letters (A, G, C en T of U). Bij de productie van eiwitten, wordt het mRNA afgelezen waarbij de letters in groepjes van drie worden
herkend door het transferRNA ofwel het tRNA. Die groepjes van drie zijn de codons. Deze zijn complementair aan een tRNA dat een specifiek
aminozuur draagt. Aangezien we vier ‘letters’ hebben die zich in groepjes van drie kunnen combineren, bestaan er 4^3 = 64 verschillende codons.
De eiwitten, die uit ketens van aminozuren bestaan, zijn uit slechts 20 verschillende aminozuren opgebouwd. In de tabel zijn de codons met hun
corresponderende aminozuren te zien; dit is de genetische code. Het blijkt dat de code voor elk aminozuur voornamelijk bepaald wordt door de
eerste twee letters. De derde is vaak irrelevant. Deze derde positie wordt ook wel de Wobble positie genoemd en de code zelf is zogezegd
gedegenereerd. Het gangbare idee is dat de oorspronkelijke primordiale code slechts uit twee nucleotiden bestond en dat daar vervolgens een code
van drie basen uit ontstaan is.
Fig. 1 Translatie: Het tRNA hecht zich met het anti-codon aan het codon van het mRNA (groen). Het gedragen aminozuur wordt vastgehecht aan de
groeiende peptide.
Een belangrijke rol heeft het tRNA. Dit tRNA heeft een arm waarvan het uiteinde van de lus complementair is aan het codon van het mRNA. Het
tRNA, dat een specifiek aminozuur draagt, hecht zich met zijn anti-codon aan het mRNA zodra ‘zijn’ codon aan de beurt is om vertaald te worden (zie
fig. 1). Het aminozuur hecht zich aan de keten aminozuren en is daarmee deel van het groeiende eiwit. De code is gedegenereerd wat betekent dat
er verschillende tRNA’s zijn die hetzelfde aminozuur dragen.
De allerbelangrijkste speler is wel de synthase van het tRNA. Dit enzym is verantwoordelijk voor de juiste koppeling van een tRNA aan het
bijbehorende aminozuur (zie fig.2) . Het is de ware tweetalige protagonist die de brug legt tussen de vier lettercode van het DNA/RNA en de twintig
lettercode van de eiwitten. Hier is een filmpje te zien van hoe dat in zijn werk gaat. Een kant van het enzym leest het anti-codon en de andere kant
hecht het juiste aminozuur aan het andere uiteinde van het tRNA. Elk aminozuur heeft zijn eigen tRNA-synthase.
Fig. 2. tRNA synthase (geel), tRNA (rood)
Er bestaan twee groepen tRNA-synthase. Elke groep bestaat uit 10 enzymen. De twee groepen verschillen van elkaar voor wat betreft hun actieve
site, hun sequentie, en de plek waar het aminozuur vastgemaakt wordt. Op dezelfde manier kunnen de aminozuren in twee groepen verdeeld
worden. Dit heeft geleid tot het idee dat de twee groepen apart van elkaar geëvolueerd zijn of zelfs na elkaar. Het zou kunnen dat er ooit twee codes
naast elkaar bestonden en dat deze twee zich samengevoegd hebben. Het zou ook kunnen dat er in eerste instantie heel eenvoudige eiwitten
bestaan hebben van slechts 10 aminozuren (groep II) in plaats van 20. Studies van sterk geconserveerde sequenties, die dus erg oud zijn, tonen aan
dat deze voornamelijk uit groep II-aminozuren zijn opgebouwd. Dezeprimordiale verdubbeling van de genetische code laat zien dat de code
niet eenvoudigweg een ‘bevroren toeval’ is zoals Crick beweerde, maar wel degelijk een optimale code
is.
Uit: Apoorva Patel (2004); Woese et al. (2000); José et al. (2009)
WOBBLE POSITIE : De derde codonpositie is vaak niet relevant. waarom er dan nog 64 codons moeten bestaan ? Veel
overlap ?Redundantie ?
-"vaak " in de bovenstaande zin , moet genuanceerder zijn
: ....immers : 'De 3de
base(letter) ___De"wobble" positie ___ is wél relevant bij aminozuren die maar door 1,2 of 3 codons worden
gecodeerd en is niet relevant bij aminozuren die door 4 codons worden,
gecodeerd.'
-*De 64 codons zijn simpelweg het gevolg van het feit dat een codon
uit 3 basen bestaat. Daar volgen dus 4^3 = 64 (mogelijke combinaties ) codons uit. Deze zogenoemde "degenererende
"eigenschap ( met als resultaat een mogelijke "overbodige (=redundante )" overlap ") manifesteert zich op niveau van de aminozuren
waarvan er slechts 20 zijn voor 64 codons.
-In zekere zin is het toch wat
speciaal dat er tegenwoordig 64 codons in omloop zijn ....want er wordt serieus gespeculeerd over het feit dat de (oudste) code
ooit uit twee basen bestond i.p.v. drie. Je spreekt in die optie dan alleen niet van "degenereren " omdat de code van twee naar drie
basen zou gegaan zijn.
Enig idee waarom die code van 2 naar 3 ging terwijl er een
hoop wobbles zijn?
-Het waarom is lastig. Als je zoals Apoorva Patel ervan uitgaat dat er aan het begin van het leven twee codes bestonden met codons van twee
basen dan levert dat 2 x 4^2= 2 x 16 aminozuren.Meer dan genoeg voor de twintig die we kennen.
Dus een codon
van twee nucleotiden omdat het 'simpeler' was?
De derde nucleotide of base had absoluut geen rol. Maar hij zat er dus wel. Dit alles is gebaseerd op het feit dat de tRNA's van twee
klassen zijn. Elke klasse draagt 10 verschillende aminozuren.
Het anticodon van tRNA heeft drie nucleotiden/basen. Er wordt wel beweerd dat de kromming in de 'loop' van het anticodon ervoor zorgt dat
de derde base niet hoeft te matchen.
NOTES
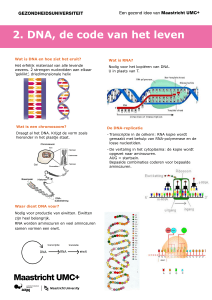
DE GENETISCHE CODE : In DNA vormen de nucleotide basen een eenvoudig vierletter alfabet dat kan coderen voor de 20
aminozuren die voorkomen in eiwitten.
De taal van de genetische code ligt bevat in codonen, woorden voor individuele aminozuren.
Elk codon is exact drie basen lang.
Een gen kan voorgesteld worden als een zin die volledig is samengesteld met deze drie letterwoorden. Op het eerste zicht kan dit
alfabet te beperkt lijken.
Maar als de vier letters in combinaties van drie worden gebruikt, zijn er 64 verschillende combinaties mogelijk.
Inderdaad, vele aminozuren kunnen beschreven worden met meer dan één codon.
Het basismechanisme van de genetische code is vergelijkbaar bij vele organismen.
Omwille van deze gelijkenis, is het soms mogelijk om een DNA segment van één bron te nemen en hetzelfde gedrag bij een andere te
voorspellen.
Een DNA segment kan bijvoorbeeld ingebracht worden in een gastheerorganisme, zoals een bacterie, waardoor de bacteriën het
menselijk eiwit zullen produceren.
Dit is de sleutel voor genetic engineering.
http://nl.wikipedia.org/wiki/Genetische_code
et een vier-letter code kunnen 64 verschillende combinaties van elk drie letters worden gemaakt, die de bouwstenen voor de eiwitten
coderen –
de aminozuren.
Omdat er maar 20 verschillende aminozuren in eiwitten voorkomen en er 64 combinaties mogelijk zijn, zijn er 44 combinaties over.
Drie daarvan worden gebruikt als stopsignalen (1a) die de moleculaire machientjes vertellen wanneer het eiwit klaar is, maar welke
functies zouden de
resterende 41 combinaties kunnen vervullen?
Het blijkt dat ze ook gewoon coderen voor één van de 20 aminozuren.
Sommige aminozuren worden namelijk door verschillende combinaties gecodeerd.(1b)
De aminozuren serine, arginine en leucine worden bijvoorbeeld op zes verschillende manieren gecodeerd.
Vijf andere aminozuren – alanine, glycine, proline, threonine en valine – worden door vier codes gespecificeerd.
De overige aminozuren worden door één, twee of drie codes gespecificeerd.
Vijf komt niet voor.
OPMERKINGEN :
Bron : blog van een creationist =
http://www.vkblog.nl/bericht/268733/Nogmaals_voor_dovemansoren...
( Lees vooral het commentaar van Peter Mudde )
Volgens intelligent Design aanhangers is deze hierboven (kort) beschreven code de " slimste " oplossing ...
Maar er zijn wel degelijk slimmere oplossingen te bedenken.
Je kunt een mutatiebestendige code bedenken, een extra controlesysteem ...(2)
Een code b.v. die bestaat uit een drie maal herhaald triplet..
Je hebt dan negen baseparen per aminozuur nodig, maar als je vervolgens in je systeem inbouwt dat als er van de drie tripletten er 1
afwijkt,
die weer hetzelfde gemaakt wordt als de twee andere tripletten, ben je bijna foutbestendig.
Je kunt ook eiwitten opbouwen uit minder dan 20 aminozuren zonder dat ze noemenswaard in werking achteruit gaan
De bewering
"Omdat er maar 20 verschillende aminozuren in eiwitten voorkomen .. tot... Elke andere andere verdeling is slechter."
is weinig steekhoudend.
Het klinkt mooi, maar waarom zou elke andere verdeling slechter zijn?
Het is ook maar hoe je het brengt..
Sommige aminozuren worden gecodeerd niet door drie, maar door slechts de eerste twee basenparen van een triplet..
Het derde paar hangt er voor spek en bonen bij..
-De derde positie ( =het derde basenpaar ) __door Peter Mudde terecht "Het derde paar hangt er ( soms ) voor spek en bonen bij..",
genoemd ___
is het zogenaamde "wobble" paar ... toch ?
(Las ik bij de leuvense geneticus prof . Cassiman )
NOTEN
(1)
a) naast de "stop-codons " is er slechts één "start " codon ( AUG-codon )
--> zie http://nl.wikipedia.org/wiki/Methionine
b) Het feit dat verschillende codons coderen voor dezelfde basisbouwstoffen ( pakweg 20 aminozuren )
"degenerancy" van de genetische code genoemd ...
Zie hier een aanklikbare link die over dit onderwerp gaat
arxiv.org/ftp/math/papers/0607...
(2)
alleen heb je dan geen bron van mutaties meer die het genetische "ontwerp" van een bestaand organisme in zijn afstammingslijnen
laat "adapteren"aan
het veranderend millieu en het daardoor vroeg of laat tot uitsterven doemt ..
= Het is dan ook niet nodig (of erg slim ) dat zulke systemen zich(altijd) vermenigvuldigen als ze dan toch gedoemd zijn tot uitsterven
DNA Structuur
Proteïnen worden gecodeerd door genen. Genen zijn op hun beurt samengesteld uit deoxyribonucleïnezuur of DNA.
Deze naam verwijst zowel naar de chemische samenstelling van de molecule als naar het feit dat ze voorkomt in de kern.
De kracht van de DNA molecule ligt in haar vermogen om te coderen voor alle genen die nodig zijn om de volledige diversiteit van het leven dat voorkomt op aarde,
te verzekeren.
De sleutel voor dit vermogen houdt verband met de fameuze dubbele helix die in 1952 werd ontwikkeld door James Watson en Francis Crick.
De dubbele helix verwijst naar de vorm van DNA die kan vergeleken worden met een spiraalvormige trap of een gedraaide ladder.
Als we de analogie met de ladder gebruiken, bestaan de buitenste staven uit suiker- en fosfaat moleculen, terwijl de sporten bestaan uit moleculen die “basen”
worden genoemd.
Een individuele unit die bestaat uit één suiker, één fosfaat en één base wordt een nucleotide genoemd. Op elke sport is een basepaar onderling verbonden door een
chemische verbinding.
DNA bevat enkel vier specifieke basen:
adenine (A), thymine (T), guanine (G) en cytosine (C).
De vier basen kunnen enkel op twee manieren met elkaar gepaard worden: A met T,
enG met C.
Als men de sequentie van de basen kent aan één zijde (streng) van de molecule kunnen de wetenschappers de sequentie aan de andere zijde bepalen.
Zoals eerst werd waargenomen eens de DNA structuur was bepaald, heeft DNA het inherente vermogen om gekopieerd te worden.
Omdat adenine steeds paart met thymine,
en guanine met cytosine, kan elke streng dienen als een template om identieke kopieën van de molecule te maken.
Wat toen nog niet duidelijk was, was hoe een molecule met beperkte diversiteit – slechts vier basen - informatie kan bevatten die vereist is om zeer diverse
moleculen zoals proteïnen aan te maken.
Biochemistry:
The similarity of DNA, blood proteins, and other organic molecules among organisms must be related to organisms that share a common ancestor.
DNA Molecule:
From: DNA Molecule - Two Views at http://www.accessexcellence.org/AB/GG/dna_molecule.html
The four nucleotide bases in DNA.
From: BIOL 1400 -- Lecture Outline 21 at http://www.accessexcellence.org/AB/GG/dna_molecule.html
The similarity of DNA among organisms is considered by many as the strongest line of evidence in favor of evolution.
Mar 9, '09
Verdraaid DNA
09-03-2009 Tomaso Agricola
De moleculaire structuur van DNA wordt veel gebruikt in (populair) wetenschappelijke illustraties op boeken, logo's en
posters om wetenschap te promoten. Op de 1 of andere manier heeft is er iets aan de structuur die esthetische lekker
overkomt.
In een ingezonden brief, vorige week in de Universiteits Krant (UK) van de Rijks Universiteit Groningen (RUG)
schrijft Maarten Linskens, Univeritair Hoofd Docent Biologie aan de RUG (aanleiding was een poster die de Universiteit
Groningen voor haar Lustrumactiviteiten gebruikt) over het fenomeen dat er op veel illustraties de verkeerde, namelijk
gespiegelde, structuur staat.
De echte natuurlijke structuur is de ‘rechtshandige' variant terwijl in illustraties soms de gespiegelde ‘linkshandige' variant
wordt gebruikt.
Ik heb regelmatig moeten vaststellen dat kunst het in sommige gevallen met de wetenschap niet erg nauw neemt. De
linkshandige variant is een niet-bestaande fictieve structuur, die helaas door veel ontwerpers wordt gebruikt, waarschijnlijk
om grafische redenen. Dit gebeurt in ongeveer 25 procent van de gevallen, soms op zeer prominente plaatsen, zoals de
omslag van het genoom-issue van het tijdschrift Science (23-10-1998)[zie plaatje hieronder, TA].
Ik had er nooit zo op gelet, maar aangezien ik zelf een DNA structuur in mijn banner had was wel meteen mijn aandacht getrokken. Het zou voor mijn status als
kennisinstituut heel slecht zijn wanneer mijn structuur niet klopt.
In de vergelijking met de omslag van Science draait de helix op de omslag de andere kant op dan die van mij. Vervolgens heb ik er ook mijn oude
studieboek Molecular Biology of the Cell maar eens op nageslagen en inderdaad draait daar het DNA de andere kant op dan op het plaatje op de omslag
vanScience en het draait dezelfde kant op als het DNA op wikipedia en mijn DNA op de banner.
Het zit dus wel snor. Maar blijf opletten en hoed u voor namaak!