Samenvatting
Moleculaire biologie: DNA Replication I: Basic Mechanism
and Enzymology
1
General Features of DNA replication
1.1 Semiconservative Replication
Er zijn drie hypotheses voor DNA replicatie. De eerste is het semi conservatieve model dat er van
uitgaat dat de twee ouderstrengen scheiden en dat elke streng dan dient als een template voor een
nieuw ontwikkelende streng. Dus elke dochter duplex heeft een ouderstreng en een nieuwe streng.
Een andere mogelijkheid is de conservatieve replicatie waar twee ouderlijke strengen bij elkaar
blijven en op een of andere manier een dochterhelix met twee nieuwe strengen produceren. Een
derde mogelijkheid is de dispersieve replicatie. Hierbij wordt het DNA gefragmenteerd zodat
nieuw en oud DNA samenzit in dezelfde streng na replicatie. Deze manier kan het probleem van
ontwinding van de twee DNA strengen vermijden.
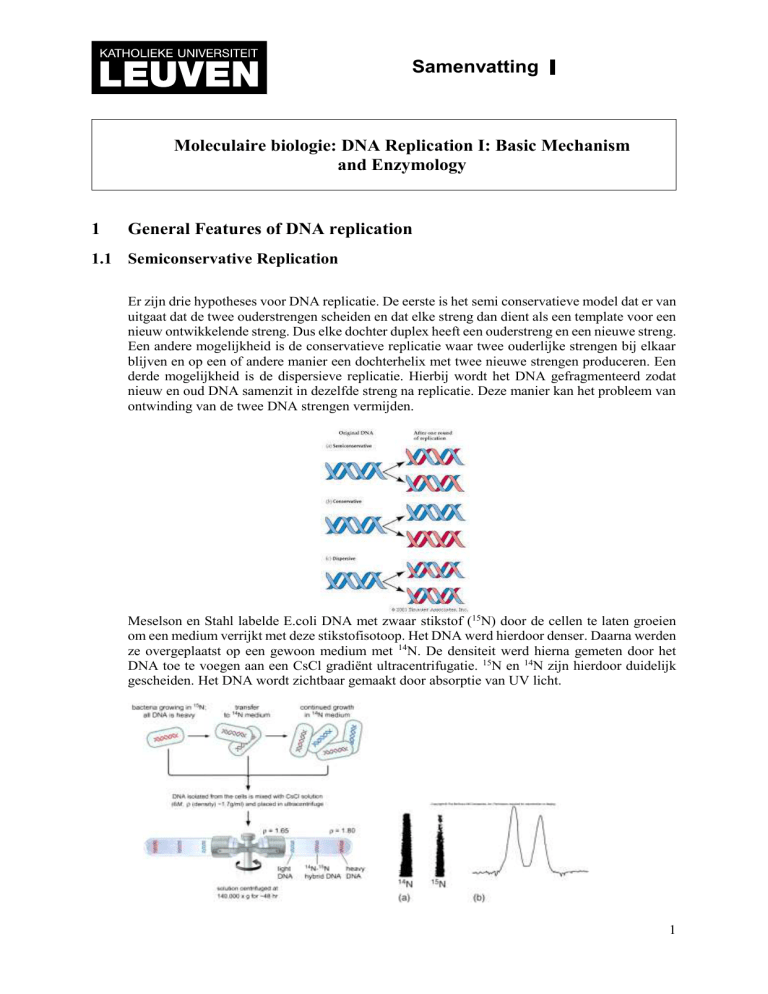
Meselson en Stahl labelde E.coli DNA met zwaar stikstof (15N) door de cellen te laten groeien
om een medium verrijkt met deze stikstofisotoop. Het DNA werd hierdoor denser. Daarna werden
ze overgeplaatst op een gewoon medium met 14N. De densiteit werd hierna gemeten door het
DNA toe te voegen aan een CsCl gradiënt ultracentrifugatie. 15N en 14N zijn hierdoor duidelijk
gescheiden. Het DNA wordt zichtbaar gemaakt door absorptie van UV licht.
1
De bekomen resultaten hangen af van het replicatiemodel.
LH
Een band van intermediaire densiteit is te zien tussen de twee andere banden (licht en zwaar) en
is alleen bij de 1ste generatie (1.0 en 1.1). Deze band komt overeen met een duplex van 15N en 14N.
Dit komt enkel overeen met het semi conservatieve model.
1.2 Semidiscontinous replication
Het replicatiemachine: DNA ontwindt en vormt zo een vork, twee nieuwe DNA strengen worden
doorlopend gesynthetiseerd in dezelfde richting als de beweging van de vork. Het machine moet
dus kunnen afschrijven in zowel de 5’ > 3’ richting als 3’ > 5’ richting doordat de twee strengen
antiparallel zijn. Het DNA polymerase kan enkel synthetiseren in de 5’ > 3’ richting door
nucleotiden toe te voegen aan het 3’-uiteinde van de groeiende streng. Okazaki concludeerde dat
het DNA polymerase theoretisch gezien een doorlopende streng (leading strand) maakt in de
5’>3’ richting, maar de andere streng (lagging strand) wordt discontinu aangemaakt.
De synthese van de lagging strand is in de omgekeerde richting tegenover de beweging van de
replicatie vork. Als de vork opent komt er een nieuwe regio van het DNA vrij om te repliceren,
maar de lagging strand groeit de andere kant uit, weg van de vork. De enige manier om de nieuw
DNA regio’s telkens te kunnen repliceren is door de DNA synthese telkens achter het stuk DNA
dat al gemaakt is geweest te laten herstarten. Deze start en herstart van de DNA synthese gebeurt
telkens opnieuw en de korte stukjes DNA worden daarna bij elkaar gezet tot een nieuwe
doorlopende streng.
2
Twee predicaties bij het semidiscontinu replicatiemodel dat experimenteel werd onderzocht door
Okazaki’s team:
1) Doordat de helft van het nieuwe gesynthetiseerde DNA eerst uit korte stukjes bestond is het
misschien mogelijk om deze te labelen en te vangen vooraleer ze aan elkaar worden geplakt
door enkel heel korte periodes (pulsen) van labelen met een radioactieve DNA precursor toe
te staan.
2) Als het enzym DNA ligase dat verantwoordelijk is voor het aan elkaar plakken wordt
geëlimineerd, dan kunnen deze korte stukjes ook gedetecteerd worden met lange pulsen van
DNA precursor.
Okazaki gebruikte hiervoor faagT4 DNA dat zeer simpel is en de beschikbaarheid T4ligase
mutanten. Om de eerste predictie te testen werden er korte pulsen (tot 2s) van 3H gelabelde
thymidine toegediend aan E.coli cellen dat T4DNA repliceren. Daarna meten ze de bekomen
groottes van de nieuw gevormde DNAs door ultracentrifugatie. Hierbij is te zien dat al na 2s wat
gelabelde DNA aanwezig was, in zeer kleine DNA stukjes, dat aan de bovenkant van de
centrifugatiebuis bleef. Bij toenemende pulsatietijd worden verdere pieken van gelabelde DNA
bereikt dichter bij de bodem van de buis. Dit is het resultaat van kleine gelabelde stukjes DNA
die vastgemaakt worden aan stukken DNA die al voor het labelen aan elkaar waren geplakt. Deze
stukken zijn pas te zien als het DNA ligase ze verbonden heeft met stukken gelabelde DNA. De
kleine stukjes DNA dat de initiële producten van de replicatie zijn, zijn de Okazaki fragmenten.
Deze Okazaki fragmenten accumuleren tot hoge levels als het DNAligase enzym niet werkt. Dit
werd ook uitgetest met een T4 mutant met een defectieve DNA ligase gen. Dit toont ook aan dat
deze fragmenten niet zomaar een artefact zijn van zeer korte labeltijden.
1.3 Priming of DNA Synthesis
Vooraleer DNA polymerase kan beginnen met afschrijven is er eerst een primer nodig (dit is niet
zo bij RNAsynthese!). Deze primer is een klein stukje RNA, polymerase kan dit vastnemen en
het kan verlengen door er nucleotiden aan het 3’-einde toe te voegen. Het mechanisme (figuur)
gaat als volgt: eerst opent het replicatievork, er worden dan korte RNA primers aangemaakt, DNA
polymerase voegt hieraan deoxyribonucleotiden toe en vormt DNA.
3
Het bewijs hiervoor werd gevonden in de replicatie van M13faag DNA in E.coli dat was
gëinhibeerd door het antibiotica rifampicin, dat normaal E.coli RNA polymerase inhibeert. M13
gebruikt dus E.coli RNA polymerase om zijn eigen RNA primers aan te maken. Dit is echter niet
een algemeen fenomeen, E.coli gebruikt namelijk niet zijn eigen RNA polymerasen voor priming
maar een speciaal enzym systeem. De beste verklaring is waarschijnlijk de ontdekking dat DNAse
de Okazaki fragmenten niet helemaal kan vernietigen. Er blijven kleine stukjes van 10-20nt lang
achter. Uit onderzoek bleek ook dat intacte primers ook 10-20nt lang zijn. Intacte primers zijn
moeilijk te detecteren in wild-type cellen door de aanwezigheid van nucleases (RNAseH). Primers
worden aangemaakt door primase.
1.4 Bidirectional replication
DNA replicatie begint met de creatie van een bubbel, een smalle regio waar de ouderstrengen zijn
gescheiden en een nieuwe streng wordt gesynthetiseerd. Als de bubbel uitbreidt begint het
replicatie DNA een theta vorm (Ɵ) aan te nemen.
De Ɵ-structuur bevat twee replicatievorken (X en Y). Is de replicatie nu unidirectioneel (1 vork
beweegt weg andere staat stil) of bidirectioneel (beide vorken bewegen in tegenoverstaande
richting)? Experiment: B.subtilis cellen groeien voor een kort tijd in de aanwezigheid van een
zwakke radioactive DNA precursor en daarna voor een korte tijd in een sterke radioactieve DNA
precursor. The pulsen warken kort genoeg zodat enkel de replicatiebubbels zichtbaar zijn. Hierbij
is te zien dat beide vorken de sterke radioactieve label hebben opgenomen wat dus wilt zeggen
dat deze werkten gedurende de pulsen. De DNA replicatie is dus bidirectioneel. De twee vorken
beginnen vanaf hetzelfde starpunt, the origin of replication of ori. En bewegen verder in
tegenovergestelde richting van de cirkel totdat ze elkaar tegenkomen langs de andere kant.
4
Bidirectionele DNA replicatie in eukaryoten:
Uitgevoerd bij Drosophila (fruitvlieg), eerst werd er gelabeld met hoog radioactief label en daarna
met laag radioactief label. Dit bracht een patroon op waarbij er paren zijn van strepen die in het
midden onderbroken zijn en spits lopen weg van het midden. Het midden is het ori en de strepen
zijn de twee replicatievorken. Wat dus wijst op een bidirectionele replicatie (a). Het patroon van
een unidirectionele replicatie (b) werd niet gevonden. Verder werd er nog gevonden dat dat de
corresponderende replicatie bij Triturus vulgaris op hetzelfde moment begonnen doordat ze een
constante grootte en vorm hebben. (c).
Niet alle DNAs repliceren bidirectioneel, de replicatie van de plasmide ColE1 in E.cli gebeurt
unidiretioneel met maar 1 replicatievork.
5
1.5 Rolling circle replication
Sommige circulaire DNAs repliceren via een mechanisme dat heet rolling circle replication.
E.coli heeft enkelstrengig circulaire DNA genomen zoals X174 dat dit mechanisme gebruikt. Een
dubbelstrengige replicatievorm (RFI) geeft aanleiding tot vele kopies van enkelstrenging
progreserende DNA. (σ model)
a) Een endonuclease creëert een nick in de positieve streng van de dubbelstrengige replicatieve
vorm.
b) Het vrije 3’-uiteinde (door de nick) dient als primer voor de positieve streng elongatie als het
andere uiteinde is verplaatst.
c) Verdere replicatie gebeurd als de positieve streng de dubbele lengte bereikt. De cirkel kan
dan beschouwd worden als rollen tegen de wijzers in.
d) Het stuk lengte van de positieve streng dat is verplaatst wordt afgeknipt door endonucleases
en wordt in een cirkelvorm gebracht.
e) De replicatie gaat door en produceert een nieuwe positieve streng door de negatieve streng
als template te gebruiken. Daarna herhaalt heel het proces zich zodat er een hoop positieve
enkelstrenige DNA kopies zijn gemaakt.
Deze methode wordt ook gebruikt voor het repliceren van dubbelstrengig DNA. Gedurende de
vroege fase van ʎ DNA replicatie volgt de faag de Ɵ mode om enkele circulaire kopies te maken.
Deze dienen verder als templates voor het rolling circle synthese voor lineair ʎ DNA dat wordt
verpakt. Dit vertoont ook de replicatievork structuur zoals in E.coli DNA replicatie. De streng
rond de cirkel is de leading strand en wordt doorlopend aangemaakt, de andere streng wordt
discontinu aangemaakt en is dus de laggin strand
Deze strengen reiken lengtes van verschillende genomen lang vooraleer ze worden verpakt.
6












